引用本文

刘彩霞, 焦如珍, 董玉红, 孙启武, 刘少文. 2015. 应用PLFA方法分析氮沉降对土壤微生物群落结构的影响[J]. 林业科学, 24(6): 155-162.

Liu Caixia, Jiao Ruzhen, Dong Yuhong, Sun Qiwu, Liu Shaowen. 2015. Effect of Nitrogen Deposition on Soil Microbial Community Structure Determined with the PLFA Method. Scientia Silvae Sinicae, 24(6): 155-162. DOI: 10.11707/j.1001-7488.20150619

应用PLFA方法分析氮沉降对土壤微生物群落结构的影响
刘彩霞,
焦如珍 
,
董玉红,
孙启武,
刘少文
中国林业科学研究院林业研究所 林木遗传育种国家重点实验室 国家林业局林木培育重点实验室 北京 100091
收稿日期:2014-10-08;修回日期:2014-12-06
基金项目:中央级公益性科研院所基本科研业务费专项资金项目( CAFYBB2012026)。
通讯作者:焦如珍
摘要:【目的】土壤微生物是土壤生态系统变化的敏感指标。本研究选择磷脂脂肪酸法(PLFA)分析微生物群落结构的变化,可以更准确地了解短期氮沉降对土壤生态系统的影响,从而预测氮沉降后土壤性质及植物生长的变化趋势,为氮饱和条件下人工林的可持续经营提供微生物参数和指标,对氮沉降的即时调控和实时治理具有指导意义。【方法】 2013年5月,在江西省分宜县山下林场约1 hm2的杉木幼龄林中建立30个1 m×1 m的样方,在30个样方中进行5种氮沉降量[N0(对照)、N1(20 kg·hm-2a-1)、N2(40 kg·hm-2a-1)、N3(60 kg·hm-2a-1)、N4(80 kg·hm-2a-1)]和2种氮形态(NH4+-N,I和NO3--N,II)的模拟沉降试验,沉降1年后用土钻进行土壤样品采集。磷脂脂肪酸提取方法为氢氧化钾-甲醇溶液甲酯化法,以十九烷酸为内标,采用Agilent 6850N测定,用Sherlock MIS4.5系统分析PLFA图谱,脂肪酸含量换算成每克干土中的含量(nmol)后进行分析。【结果】本研究共检测到PLFAs 72种,其中特征脂肪酸36种。分析特征脂肪酸种类和含量可知:各处理中土壤微生物群落均以原核微生物为主,不同氮处理样地中以磷脂脂肪酸总量表征的土壤微生物生物量范围20~44 nmol·g-1。沉降铵态氮时,土壤中PLFA总量、革兰氏阳性菌、革兰氏阴性菌脂肪酸含量均高于对照样地,细菌、真菌、放线菌和原生动物的脂肪酸含量变化趋势相同,均为随着氮沉降量的增加先升高再降低最后再升高,NH4+-N N4处理土壤微生物PLFAs的数量最多,NH4+-N N2处理土壤微生物PLFAs的丰度值和多样性值最高;沉降硝态氮时,土壤中PLFA总量、革兰氏阳性菌和革兰氏阴性菌PLFAs量随着硝态氮浓度的增加呈现出先增加后减少的趋势,在NO3--N N2处理达到最大值。细菌和放线菌的标记脂肪酸含量变化趋势相同。NO3--N N2处理微生物脂肪酸量最多,NO3--N N4浓度下微生物PLFAs多样性值最高。根据典型性相关分析,得出铵态氮对土壤中细菌和放线菌含量影响较为显著,土壤中硝态氮和含水量对细菌含量影响较为显著。【结论】当氮沉降量小于80 kg·hm-2a-1时,铵氮和硝态氮处理均促进了微生物的生长,但增长幅度不同。铵态氮的最高氮处理和硝态氮的中氮处理,更有利于土壤微生物总量的增长,铵态氮的中氮处理和硝态氮的最高氮处理,更有利于土壤微生物多样性的增加。铵态氮对土壤中细菌和放线菌含量影响较为显著,土壤中硝态氮和含水量对细菌含量影响较为显著。
关键词:
氮沉降 磷脂脂肪酸 土壤微生物 微生物多样性
Effect of Nitrogen Deposition on Soil Microbial Community Structure Determined with the PLFA Method
Liu Caixia,
Jiao Ruzhen
,
Dong Yuhong,
Sun Qiwu,
Liu Shaowen
State Key Laboratory of Tree Genetics and Breeding Key Laboratory of Tree Breeding and Cultivation of State Forestry Administration Research Institute of Forestry, CAF Beijing 100091
Abstract: [Objective] Soil microbial community is a sensitive indicator of changes in soil quality and soil ecosystem. The changes in soil microbe community structure in response to simulated nitrogen deposition were investigated by phospholipids fatty acids (PLFA) biomarkers. This study can be accurate to understand the effects of short-term nitrogen deposition on soil ecosystem, and hence predict the change of soil properties and plant growth. This study aims at providing microbial parameters and indicators for the sustainable management of artificial forest under nitrogen saturation condition, and would have guiding significance for the real-time control and governance of nitrogen deposition. [Method] In May 2013, we established 30 plots of 1 m × 1 m in a hectare of young Chinese fir forest, at the Shanxia forest farm in Fenyi, Jiangxi Province. Nitrogen loadings were designed at 5 levels such as N0, N1, N2, N3 and N4 at the doses of 0, 20, 40, 60 and 80 kg N·hm-2 a-1, respectively, with 2 nitrogen forms of NH4+-N (I) and NO3--N (II). After one year treatment, soil samples were collected with a soil drill. The fatty acid phospholipid was extracted with potassium hydroxide-methyl methanol solution, and assayed by Agilent 6850N with nineteen alkyl acid as internal standard. PLFA pattern was analyzed with the Sherlock MIS4.5 system, before the fatty acid content was converted into the number of nmol·g-1 dry soil. [Result] A total of 72 PLFAs were detected, and among them the characteristic fatty acids were 36 kinds. The analysis on type and content of the characteristic fatty acids indicated that prokaryotic microorganism was the predominant group in the plot soil treated with all nitrogen treatments. In different nitrogen treatment plots, the range of soil microbial biomass with characteristics of total PLFAs content was 20-44 nmol·g-1. In deposition of ammonium nitrogen, the amount of total PLFAs, Gram-positive bacterial PLFAs and Gram-negative bacterial PLFAs in the soil were higher compared with control group. The bacterial PLFAs, fungal PLFAs, actinomycete PLFAs and protozoa PLFAs had a same trend under the treatments of ammonium nitrogen. The NH4+-N N4 was the optimal concentration for the growth of microorganisms. In the NH4+-N N2 treatment, the PLFAs of soil microorganisms had the highest quantity and diversity. However, along with the increased rates of nitrate nitrogen deposition, the amount of total PLFAs, Gram-positive bacterial PLFAs and Gram-negative bacterial PLFAs showed a trend that they first increased and then decreased, with the peaks occurred at the NO3--N N2 treatment. Bacterial PLFAs had a same trend as actinomycete PLFAs. Moreover, the PLFAs in the NO3--N N4 treatment had the highest diversity, while the microbial fatty acids in NO3--N N2 treatment had the highest content. According to the Canonical Correlations, ammonium nitrogen in soil was positively correlated with the bacteria and actinomycetes, while nitrate nitrogen and soil moisture were most significantly correlated with bacterial. [Conclusion] When the nitrogen deposition amount was less than 80 kg N·hm-2 a-1, both ammonium nitrogen and nitrate nitrogen treatments could promote the growth of microorganisms, but the growth rate was different. The nitrogen treatments of the highest ammonium nitrogen concentration and the medium nitrate nitrogen were more conducive to the total soil microorganism growth, whereas treatments with the medium ammonium nitrogen and the highest nitrate nitrogen were more conducive to increase soil microbial diversity. Ammonium nitrogen in soil was correlated with the bacteria and actinomycetes, while the nitrate nitrogen and soil moisture is most significantly correlated with bacterial.
Key words:
nitrogen deposition phospholipid fatty acids soil microbe microbial diversity
近年来,随着工农业的发展和人类活动的变化,全球的氮沉降量不断升高(Elser et al., 2007; Fang et al., 2011),由此引发的诸多生态环境问题也引起了关注。氮沉降的增加会导致土壤酸化、营养物质失衡、氮素流失、生物多样性丧失以及森林蓄积量下降等一系列问题(MacDonald et al., 2002; Gilliam et al., 2006; Bobbink et al., 2010)。
微生物是土壤的重要组成部分,它不仅是土壤养分的重要来源,还对微环境十分敏感,当生态机制变化和环境改变时,微生物的群落结构也发生变化(张瑞娟等,2011)。所以研究微生物群落结构的变化对研究氮沉降所带来的土壤生态系统的变化具有指向性(Wallenstein,2004)。微生物群落结构的研究方法很多,包括Biolog法、磷脂脂肪酸(phospholipid fatty acid,PLFA)法、DGGE(denaturing gradient gel electrophoresis)法等。本研究选择PLFA法是因为它不需要分离和培养技术,适合微生物群落的动态追踪(齐鸿雁等,2003); 操作简便,结果较为精准、客观; 能够定量描述土壤环境中的微生物群体(黄懿梅,2008; 张秋芳等,2009)。
本研究通过土壤样品PLFA分析短期氮沉降对土壤微生物群落结构的影响,其变化趋势能更好地预测土壤性质及植物生长的变化趋势,为氮饱和条件下人工林的可持续经营提供微生物参数和指标,对氮沉降的即时调控和实时治理具有指导意义。
1 材料与方法
1.1 试验地概况
试验地位于江西省分宜县大岗山东北侧的山下林场。地处114°30′—114°45′ E,
27°30′—27°45′ N,属低山丘陵地貌,母岩以千枚岩为主,土壤为黄、红壤,地带性植被为常绿阔叶林,年均气温17.9 ℃,日最高温39.9 ℃,最低气温-8.3 ℃,年降水量1 593.7 mm,集中在4—7月,无霜期为268天(杨承栋等,1999)。
1.2 试验设计
2013年5月,在约1 hm2的杉木(Cunninghamia lanceolata)幼龄林中建立30个1 m×1 m的样方,每个样方间隔3 m的缓冲带,在30个样地中进行5种氮沉降量[N0(对照)、N1(20 kg·hm-2a-1)、N2(40 kg·hm-2a-1)、N3(60 kg·hm-2a-1)、N4(80 kg·hm-2a-1)]和2种氮形态(NH4+-N,I和NO3--N,II)的模拟沉降试验,每种氮处理设3个重复。根据微生物活性的季节性变化特征,试验中将年氮沉降量分成5等份,分别于5,6,7,9,11月中旬施入样地中。施氮时将每份氮肥溶解于1 L水中,用喷雾器在该样方均匀喷洒,对照只喷清水。
1.3 样品采集和测定
2014年5月,模拟氮沉降1年后进行土壤采集。取土深度为0~20 cm,不同土壤样品均通过3点采集,每个点取3个重复。取出土壤中的残留根系、石块及其他杂质,过2 mm筛后放入液氮中带回实验室,放置在-70 ℃的超低温冰箱内,并尽快完成PLFA分析。
土壤pH、含水量以及氮含量的测定参照文献(南京土壤研究所,1983)进行,表 1为测定结果。磷脂脂肪酸提取方法为氢氧化钾-甲醇溶液甲酯化法(Kourtev et al., 2002; Drenovsky et al., 2004),以十九烷酸为内标,采用Agilent 6850N 测定,用Sherlock MIS4.5系统分析PLFA图谱从而对脂肪酸的成分进行鉴定。
表 1 不同氮处理土壤中的氮相关指标
Tab.1 The properties of different nitrogen deposition soil

表 1 不同氮处理土壤中的氮相关指标
Tab.1
The properties of different nitrogen deposition soil
| 处理 Treatment | N沉降量Amount of nitrogen deposition/(kg·hm-2a-1) | pH | 含水量 Moisture(%) | 铵态氮Ammonium content/(mg·kg-1) | 硝态氮含量Nitrate content/(mg·kg-1 ) |
| NH4+ -N | N0 | 0 | 4.353±0.133 2 | 0.296 1±0.053 | 24.233±2.587 | 4.216 5±1.178 |
| N1 | 20 | 4.403±0.277 5 | 0.356 8±0.031 | 25.311±5.321 | 4.902 1±1.379 |
| NH4+-N N2 | 40 | 4.377±0.015 3 | 0.315 4±0.025 | 23.847±0.407 | 4.479 3±1.555 |
| N3 | 60 | 4.347±0.212 2 | 0.304 6±0.013 | 18.822±4.390 | 7.232 3±1.328 |
| N4 | 80 | 4.400±0.260 0 | 0.308 7±0.033 | 22.725±4.873 | 5.480 2±2.203 |
| NH3- -N | N0 | 0 | 4.360±0.108 2 | 0.297 3±0.017 | 27.861±6.674 | 3.982 9±0.589 |
| N1 | 20 | 4.410±0.182 5 | 0.337 5±0.034 | 24.989±5.127 | 6.776 5±1.112 |
| NO3--N N2 | 40 | 4.343±0.138 7 | 0.348 5±0.040 | 30.777±2.029 | 7.227 3±2.159 |
| N3 | 60 | 4.453±0.083 3 | 0.340 2±0.038 | 24.444±4.813 | 4.799 5±0.966 |
| N4 | 80 | 4.317±0.032 1 | 0.335 7±0.023 | 25.381±5.017 | 6.842 0±1.202 |
|
1.4 数据分析
PLFA作为通用生物标记物可以反映出总的土壤微生物生物量、革兰氏阳性菌和革兰氏阴性菌的含量。而总PLFA谱中某些特定脂肪酸作为特定生物标记,可以初步判定细菌、真菌、放线菌和原生动物的含量及其群落结构功能的多样性。表 2为部分特定生物标记的一些脂肪酸(Jain et al., 1997; Joergensen et al., 2005; Sakamoto et al., 2004)。测定结果中PLFAs含量的标定和计算参照刘波等(2010)方法,将脂肪酸换算成每克干土中的含量(nmol),计算公式如下:
表 2 表征微生物的PLFA①
Tab.2 PLFA characterizing microbes

表 2 表征微生物的PLFA①
Tab.2
PLFA characterizing microbes
| 微生物类型Microbial group | 磷脂脂肪酸标记Phospholipids fatty acid signatures |
| 细菌Bacteria | 含有以酯链与甘油相连的饱和或单不饱和脂肪酸(如15:0,i15:0,a15:0,16:0,i16:0,16:1ω5,16:1ω9,16:1ω7t,17:0,i17:0,a17:0,cy17:0,18:1ω5,18:1ω7,18:1ω7t,i19:0,a 19:0 和cy19:0 等)。Saturated or mono unsaturated fatty acid with a ester chain linked to glycerol(e.g. 15:0,i15:0,a15:0,16:0,i16:0,16:1ω5,16:1ω9,16:1ω7t,17:0,i17:0,a17:0,cy17:0,18:1ω5,18:1ω7,18:1ω7t,i19:0,a 19:0 and cy19:0 etc.). |
| 好氧细菌Aerobes | i15: 0,a15: 0,15: 0,i16: 0,16: 1ω9,16: 1ω7t,i17: 0,a17: 0,17: 0 |
| 厌氧细菌Anaerobes | cy17: 0,cy19: 0 |
| 革兰氏阳性菌Gram-positive bacteria | 含有多种支链脂肪酸(iso-, anteiso-)。Containing a variety of branched fatty acids(iso-, anteiso-)。 |
| 革兰氏阴性菌Gram-negative bacteria | 含有多种羟基脂肪酸(单烯脂肪酸、 环丙基脂肪酸等)。Containing a variety of hydroxy fatty acids (mono fatty acids, cyclic fatty acids etc.). |
| 真菌Fungi | 含有特有的磷脂脂肪酸( 如18:1ω9,18:2ω6,18:3ω6,18:3ω3)。Containing a unique phospholipid fatty acid (e.g. 18:1ω9,18:2ω6,18:3ω6,18:3ω3 etc. ). |
| 放线菌Actinomycetes | 10Me16:0,10Me17:0,10Me18:0 等(etc.) |
| 原生动物Protozoa | 20:3ω6,20:4ω6 |
| ①
i,a,cy 和Me 分别表示异、反异、环丙基和甲基分支脂肪酸;ω,c和t 分别表示脂肪端、顺式空间构造和反式空间构造。
i, a, cy and Me refer to iso, anteiso, cyclopropyl andmethyl branching fatty acids, respectively;ω, c and t refer to the aliphatic end, cis configuration and trans configuration, respectively.
下同The same below. |
|
|
$
\begin{array}{l}
N = \frac{{目标{\mathop{\rm Re}\nolimits} sponse}}{{19:0{\mathop{\rm Re}\nolimits} sponse}} \times 19:0浓度\\
\;\;\;\;\; \times \frac{{溶样体积}}{{样品干质量 \times FAME}}。
\end{array}
$
|
式中,N为脂肪酸含量(nmol·g-1DW),Response为生物标记的响应值,19:0为内标物c19: 0(ng·μL-1),FAME为脂肪酸甲酯的摩尔质量(g·mol-1),溶样体积单位μL,样品干质量单位g。
换算后的脂肪酸数据利用Excel进行数据初处理,Sigma Plot进行制图,SPSS19.0 软件进行单因素方差分析(显著水平为α=0.05)以及丰度(S)、均匀度(J)和多样性指数分析[Shannon-Wiener指数(H)和Simpson指数(D)]以及典型性相关分析。
2 结果与分析
2.1 各处理对土壤微生物总生物量的影响
土壤中磷脂脂肪酸的组成可以表示土壤微生物群落的生物量和结构(Bardgett et al., 1996)。使用直接从土壤中提取的磷酯类化合物的量可准确地表达为土壤微生物的生物量(于树等,2008)。本研究共检测到72种PLFA,其中可对应出微生物类型的特征PLFA为36种(表 3),C链长度为11~20,包括饱和、不饱和、甲基化分支和环化脂肪谱。
表 3 各处理下土壤微生物特征磷脂脂肪酸标记分析①
Tab.3 Analysis of PLFA biomarkers in soil of different treatments

表 3 各处理下土壤微生物特征磷脂脂肪酸标记分析①
Tab.3
Analysis of PLFA biomarkers in soil of different treatments
| nmol·g-1 |
| 磷脂脂肪酸名称Peak name |
微生物类型Microbial types |
NH
4
+-N |
NO
3
--N |
| N0 |
N1 |
N2 |
N3 |
N4 |
N0 |
N1 |
N2 |
N3 |
N4 |
| 12:00 |
Ba |
0.022 3 |
0.053 0 |
0.031 6 |
0.024 6 |
0.045 4 |
0.030 9 |
0.048 4 |
0.029 5 |
0.027 1 |
0.028 4 |
| 14:00 |
Ba |
0.232 1 |
0.385 1 |
0.306 5 |
0.251 9 |
0.429 7 |
0.335 3 |
0.396 2 |
0.430 6 |
0.387 6 |
0.344 9 |
| 15:1w6c |
Ba |
0.040 9 |
0.072 5 |
0.060 6 |
0.041 5 |
0.071 7 |
0.060 6 |
0.067 0 |
0.070 2 |
0.075 4 |
0.063 8 |
| 15:00 |
Ba |
0.209 4 |
0.325 6 |
0.293 9 |
0.235 2 |
0.379 7 |
0.299 8 |
0.337 1 |
0.417 2 |
0.393 0 |
0.326 1 |
| 19:0 iso |
Ba |
0.055 6 |
0.061 7 |
0.054 6 |
0.050 6 |
0.072 9 |
0.052 5 |
0.075 4 |
0.097 2 |
0.073 1 |
0.060 8 |
| 19:0 anteiso |
Ba |
— |
— |
— |
— |
— |
0.134 2 |
— |
— |
— |
— |
| 20:00 |
Ba |
0.091 9 |
0.133 0 |
0.131 5 |
0.097 6 |
0.240 6 |
0.141 9 |
0.230 6 |
0.297 2 |
0.167 7 |
0.157 3 |
| 17:0 iso 3OH |
Gn |
0.063 8 |
0.059 9 |
0.025 9 |
0.044 8 |
0.067 5 |
0.054 8 |
0.072 5 |
0.108 0 |
0.069 6 |
0.043 7 |
| 16:0 iso |
Gp |
1.303 1 |
1.872 8 |
1.790 4 |
1.454 0 |
2.141 7 |
1.804 3 |
1.826 1 |
2.634 0 |
2.233 6 |
1.968 8 |
| 15:0 iso |
Gp |
1.890 1 |
2.914 5 |
2.815 4 |
2.303 6 |
3.509 2 |
3.071 2 |
3.390 3 |
4.760 2 |
3.889 3 |
3.305 4 |
| 15:0 anteiso |
Gp |
0.828 1 |
1.196 6 |
1.071 3 |
0.908 8 |
1.259 7 |
1.155 6 |
1.301 7 |
1.706 1 |
1.443 8 |
1.294 9 |
| 12:0 3OH |
Gp |
— |
— |
— |
— |
0.012 7 |
0.003 6 |
0.002 9 |
0.008 8 |
0.004 7 |
— |
| 11:0 3OH |
Gp |
— |
0.020 9 |
— |
— |
— |
0.008 8 |
0.016 2 |
— |
— |
— |
| 15:0 3OH |
Ae/Gp |
— |
— |
— |
— |
— |
— |
— |
0.644 6 |
— |
0.504 1 |
| 14:0 iso |
Ae |
0.118 0 |
0.200 1 |
0.160 5 |
0.140 3 |
0.193 7 |
0.166 2 |
0.185 5 |
0.203 0 |
0.181 1 |
0.176 0 |
| 17:0 iso |
Ae/Gp |
1.054 2 |
1.208 4 |
1.189 0 |
1.089 5 |
1.292 9 |
1.147 1 |
1.352 8 |
1.585 9 |
1.500 5 |
1.262 5 |
| 18:0 iso |
Gp |
0.143 4 |
0.151 7 |
0.133 6 |
0.137 3 |
0.162 2 |
0.128 3 |
0.170 1 |
0.200 5 |
0.179 4 |
0.145 6 |
| 17:0 anteiso |
Gp |
0.510 7 |
0.642 1 |
0.595 8 |
0.522 4 |
0.677 4 |
0.627 9 |
0.672 9 |
0.932 2 |
0.797 7 |
0.689 4 |
| 16:0 anteiso |
Gp |
0.025 3 |
0.042 8 |
0.035 7 |
0.036 9 |
0.041 6 |
— |
0.047 8 |
0.055 9 |
0.058 2 |
0.039 0 |
| 19:0 cyclo w8c |
Gn |
1.956 6 |
3.090 7 |
2.821 3 |
2.449 5 |
3.750 4 |
3.313 2 |
3.170 3 |
5.654 5 |
4.601 2 |
3.446 3 |
| 17:1 w8c |
Gn |
0.104 4 |
0.148 7 |
0.149 8 |
0.123 2 |
0.189 9 |
0.139 0 |
0.168 9 |
0.213 4 |
0.214 2 |
0.160 9 |
| 17:0 cyclo |
Gn |
0.314 0 |
0.506 7 |
0.429 3 |
0.401 3 |
0.612 5 |
0.493 5 |
0.502 9 |
0.667 8 |
0.623 8 |
0.540 0 |
| 15:0 iso 3OH |
Gn |
— |
0.037 9 |
0.032 7 |
— |
0.048 2 |
0.038 9 |
0.041 0 |
0.055 6 |
0.039 3 |
0.038 7 |
| 13:0 iso |
F1 |
0.012 4 |
0.026 0 |
0.015 6 |
0.014 0 |
0.022 2 |
0.012 9 |
0.033 0 |
0.020 3 |
0.019 4 |
0.018 7 |
| 16:1 w5c |
Me/Gn |
0.393 0 |
0.650 2 |
0.492 6 |
0.405 0 |
0.703 7 |
0.632 9 |
0.708 8 |
0.928 4 |
0.796 5 |
0.639 4 |
| 14:1 w5c |
Ps |
0.027 0 |
0.046 9 |
0.036 8 |
0.044 4 |
0.035 7 |
0.039 5 |
0.041 8 |
0.041 4 |
0.033 4 |
0.033 5 |
| 16:00 |
Ps |
3.575 4 |
5.655 1 |
4.847 8 |
3.873 4 |
7.341 8 |
5.232 0 |
6.255 0 |
7.667 1 |
6.939 2 |
5.561 7 |
| 17:00 |
Ar |
0.149 5 |
0.251 9 |
0.210 0 |
0.161 2 |
0.299 9 |
0.215 5 |
0.263 7 |
0.277 4 |
0.300 2 |
0.251 1 |
| 18:1 w7c 11-methyl |
Ce |
0.166 4 |
0.252 4 |
0.199 1 |
0.191 0 |
0.300 7 |
0.243 4 |
0.284 9 |
0.433 7 |
0.371 7 |
0.265 2 |
| 18:00 |
Hy |
0.835 8 |
1.180 7 |
1.075 7 |
0.879 5 |
1.334 6 |
1.112 4 |
1.328 2 |
1.543 5 |
1.664 1 |
1.229 7 |
| 18:3 w6c (6,9,12) |
Fu |
0.061 7 |
0.082 6 |
0.074 1 |
0.079 4 |
0.096 2 |
0.073 2 |
0.097 1 |
0.127 3 |
0.096 4 |
0.081 2 |
| 18:1 w9c |
Fu |
1.448 1 |
2.178 8 |
2.178 9 |
1.596 3 |
2.718 4 |
2.075 0 |
2.664 9 |
3.141 3 |
3.896 4 |
2.362 9 |
| 17:0 10-methyl |
Ac |
0.218 6 |
0.329 4 |
0.326 9 |
0.261 1 |
0.402 5 |
0.306 2 |
0.329 9 |
0.488 9 |
0.410 7 |
0.350 9 |
| 18:0 10-methyl, TBSA |
Ac |
— |
1.183 8 |
1.121 3 |
— |
1.196 7 |
1.157 3 |
0.771 2 |
1.589 7 |
1.478 4 |
1.452 7 |
| 20:4 w6,9,12,15c |
Pr |
0.194 8 |
0.386 5 |
0.264 9 |
0.198 3 |
0.376 2 |
0.269 2 |
0.434 9 |
0.422 3 |
0.420 0 |
0.309 8 |
| 20:2 w6,9c |
Pr |
— |
— |
0.039 3 |
— |
— |
— |
— |
— |
0.065 7 |
— |
| ①Ba:细菌Bacteria;Ae耗氧细菌Aerobes;Gp:革兰氏阳性菌Gram-positwe bacteria;
Gn:革兰氏阴性菌Gram-negative bacteria;
Fl:黄杆菌属/革兰氏阴性菌:Flavbacterium balustinum/ Gram-negative bacterial;
Me:甲烷氧化菌:Methane-oxidizing bacteria;
Ps:假单胞杆菌:Pseudomonadaceae;
Ar:节杆菌属:Arthrobacter;
Ce:纤维素属:Cellulomonas;
Hy:嗜热解氢杆菌:Hydrogenobacter;
Fu:真菌Fungi;Ac:放线菌Actinomycetes;
Pr:原生动物Protoza. |
|
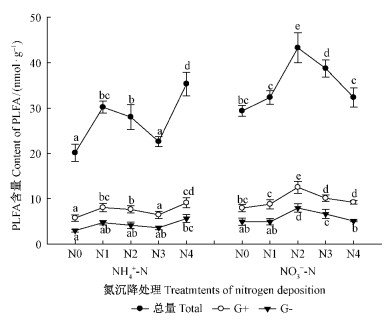
如图 1所示,不同氮处理样地中以磷脂脂肪酸总量表征的土壤微生物生物量范围为20~44
nmol·g-1。沉降铵态氮时,土壤中PLFA总量均高于对照样地,且随着氮沉降量的增加表现出先增加再减少最后增加的趋势,最终在NH4+-N N4处理达到最大值,高于对照处理75.45%。沉降硝态氮时,土壤中PLFA总量同样高于对照样地,且随着硝态氮沉降量的增加呈现出先增加后减少的趋势,在NO3--N N2处理达到最大值,高于对照处理47.37%。不同氮沉降浓度下革兰氏阴性菌和革兰氏阳性菌均与PLFA总量变化趋势相同,但变化幅度略小。
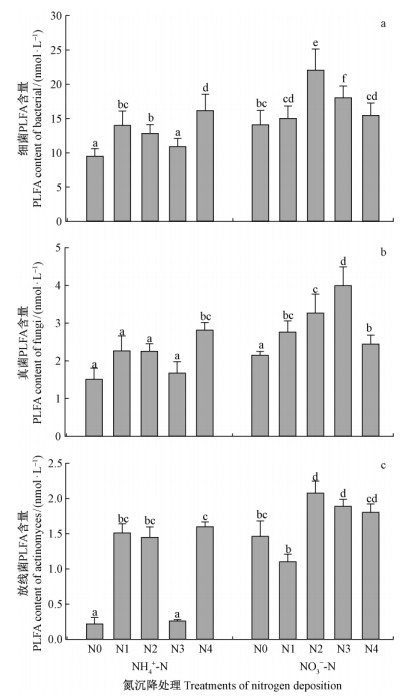
2.2 各处理对土壤细菌、真菌和放线菌
式中,N为脂肪酸含量(nmol·g-1DW),Response为生物标记的响应值,19:0为内标物c19: 0(ng·μL-1),FAME为脂肪酸甲酯的摩尔质量(g·mol-1),溶样体积单位μL,样品干质量单位g。含量的影响由图 2a可见,不同沉降氮浓度间土壤细菌含量存在一定差异。其中,各处理均高于对照样地。沉降铵态氮时,其变化趋势为先升高再降低最后再升高,NH4+-N N4处理细菌含量为最大值,高于对照处理69.84%;NH4+-N N3处理为最小值,高于对照处理14.60%。沉降硝态氮时,其变化趋势先升高再降低,最大值出现在NO3--N N2处理,高于对照处理56.29%;最小值出现在NO3--N N4处理,高于对照处理9.61%。比较2种氮形态整体的增长幅度,可得出铵态氮含量变化更利于细菌量的增长,并且土壤微生物细菌的含量变化与微生物总含量变化大致相同,这也证明了土壤微生物是以细菌为主体的群落结构。
图 2b表明:不同沉降氮沉降量间土壤真菌含量存在一定差异。沉降铵态氮时,其变化趋势与细菌相同,NH4+-N N4处理达到最大值,高于对照86.43%。沉降硝态氮时,其变化趋势也同于细菌,但是NO3--N N3处理达到最大值,即真菌比细菌更易于生活在高硝态氮的土壤中。
图 2c为不同氮处理土壤中放线菌含量的变化。沉降铵态氮时,其变化趋势与真菌相同,均是NH4+-N N3处理与对照数量较为相近,NH4+-N N4处理为最大值,且其变化幅度较大。沉降硝态氮时,NO3--N N1处理的放线菌生物量低于对照24.76%,NO3--N N2处理达到最大值,高于对照42.04%。在本研究中,NH4+-N N4和NO-3-N N2处理最利于放线菌生长。
2.3 不同处理对土壤微生物群落结构的影响
通过对标记性的特征脂肪酸进行分析,可以得出细菌、真菌、放线菌和原生动物的脂肪酸含量存在一定差异,各处理间的多样性指数也各不相同(表 4)。NH4+-N N2处理,土壤微生物PLFA多样性指数最高。磷脂脂肪酸丰富度为70,均匀度为0.960 5;Simpson和Shannon指数分别为3.500 5和0.823 9。所以铵态氮N2处理更有利于微生物的生长,微生物多样性较高,而NH4+-N N3处理的多样性指数最低。沉降硝态氮时,NO3--N N4处理指数值最大:丰富度72,均匀度0.967 1;Simpson和Shannon指数分别为3.682 4和0.861 0,即在NO3--N N4处理更利于微生物的生长,微生物多样性指数高,其他硝态氮处理的多样性指数差异较小。
表 4 不同氮沉降处理下土壤微生物PLFA多样性指数
Tab.4 Diversity indices of PLFA in soil of
different nitrogen deposition

表 4 不同氮沉降处理下土壤微生物PLFA多样性指数
Tab.4
Diversity indices of PLFA in soil of
different nitrogen deposition
| 处理类型Treatments | S | J | H | D |
| NH4+-N | N0 | 48 | 3.137 4 | 0.810 4 | 0.942 0 |
| N1 | 50 | 3.163 7 | 0.808 7 | 0.941 7 |
| NH4+-N N2 | 70 | 3.500 5 | 0.823 9 | 0.960 5 |
| N3 | 48 | 3.130 2 | 0.808 6 | 0.941 5 |
| N4 | 52 | 3.279 3 | 0.829 9 | 0.934 4 |
| NH3--N | N0 | 51 | 3.140 2 | 0.798 7 | 0.940 9 |
| N1 | 51 | 3.145 0 | 0.799 9 | 0.939 4 |
| NO3--N N2 | 52 | 3.149 7 | 0.797 1 | 0.938 2 |
| N3 | 53 | 3.145 9 | 0.792 4 | 0.939 6 |
| N4 | 72 | 3.682 4 | 0.861 0 | 0.967 1 |
|
为了进一步研究环境因子对微生物群落结构的影响,使用SPSS进行典型性相关分析。U代表环境变量的标准化变量,V代表群落结构生物量的标准化变量。如表 5所示,将U与V进行Bartlett的χ2检验,得出5组相关系数。由Sig.值判断得知,在α=0.05时,前2个典型相关系数是极显著的,2组变量的相关系数均为1。
表 5 典型变量相关系数和显著性检验
Tab.5 Canonical correlation coefficient and
significant test

表 5 典型变量相关系数和显著性检验
Tab.5
Canonical correlation coefficient and
significant test
| 组Group | Wilk’s | Chi-Sq | DF | Sig. | r |
| 1 | 0.000 | 0.000 | 30.000 | 0.000 | 1.000 |
| 2 | 0.000 | 112.277 | 20.000 | 0.000 | 1.000 |
| 3 | 0.084 | 7.442 | 12.000 | 0.827 | 0.895 |
| 4 | 0.422 | 2.587 | 6.000 | 0.859 | 0.697 |
| 5 | 0.820 | 0.596 | 2.000 | 0.742 | 0.424 |
|
由表 6可知环境变量的典型变量。U1=0.425×N沉降量+1.030×pH值-0.446×含水量-0.006×铵态氮含量-0.202×硝态氮含量,U1的主要影响因子为pH值和含水量。U2=0.129×N沉降量+0.222×pH值-0.158×含水量+1.027×铵态氮含量-0.559×硝态氮含量,U2的主要因子为铵态氮含量。表 7为微生物群落结构因子的典型变量,V1=7.014×微生物总量-14.440×细菌含量+0.001×真菌含量+0.759×放线菌含量,V1的主要代表因子为细菌;V2=0.659×微生物总量+9.435×细菌含量-0.175×真菌含量-1.774×放线菌含量,即V2的主要因子为细菌。
表 6 典型变量U与原始标准化变量X的相关系数
Tab.6 Correlation coefficients between canonical
variables U and standardized variables X

表 6 典型变量U与原始标准化变量X的相关系数
Tab.6
Correlation coefficients between canonical
variables U and standardized variables X
| 因子Factor | U1 | U2 | U3 | U4 | U5 |
| N沉降量Amount of nitrogen deposition | 0.425 | 0.129 | 0.742 | 0.626 | -0.020 |
| pH | 1.030 | 0.222 | -0.346 | -0.039 | -0.138 |
| 含水量Moisture | -0.446 | -0.158 | 0.601 | -1.169 | 0.215 |
| 铵态氮含量Ammonium content | -0.006 | 1.027 | -0.149 | 0.135 | 0.198 |
| 硝态氮含量Nitrate content | -0.202 | -0.559 | 0.194 | -0.650 | 1.039 |
|
表 7 典型变量V与原始标准化变量Y的相关系数
Tab.7 Correlation coefficients between canonical
variables V and standardized variables Y

表 7 典型变量V与原始标准化变量Y的相关系数
Tab.7
Correlation coefficients between canonical
variables V and standardized variables Y
| 类型Type | V1 | V2 | V3 | V4 | V5 |
| 微生物总量Total microorganism | 7.014 | 0.659 | 14.512 | 15.193 | 9.586 |
| 细菌含量Bacteria content | -14.440 | 9.435 | -55.460 | -59.189 | -5.503 |
| 真菌含量Fungi content | 0.001 | -0.175 | -3.020 | -3.836 | -2.291 |
| 放线菌含量Actinomycetes content | -0.759 | -1.774 | -0.558 | -1.716 | -1.062 |
|
根据U和V的典型相关关系分析可知,土壤pH、土壤含水量及土壤中铵态氮含量对微生物群落中细菌含量影响较显著。N沉降对土壤微生物群落中细菌的影响最大,放线菌次之,真菌最小。
3 结论
研究表明,在各处理中多不饱和脂肪酸相对含量较低,单不饱和脂肪酸和支链脂肪酸为优势脂肪酸类群,即各处理土壤中微生物群落结构中原核微生物为主要类群,细菌为其群落结构中的优势菌群,真核微生物所占比例较小。
沉降铵态氮时,土壤中PLFA总量、革兰氏阳性菌、革兰氏阴性菌脂肪酸含量均高于对照样地。细菌、真菌、放线菌和原生动物的脂肪酸含量变化趋势相同。NH4+-N N4处理土壤微生物PLFAs的数量最多,NH4+-N N2处理的土壤微生物PLFAs丰度值和多样性值最高。沉降硝态氮时土壤中PLFA总量、革兰氏阳性菌和革兰氏阴性菌PLFAs量随着硝态氮沉降量的增加呈现出先增加后减少的趋势,在NO3--N N2处理达到最大值。细菌和放线菌的标记脂肪酸含量变化趋势相同。NO3--N N2处理的微生物脂肪酸量最多,NO3--N4处理的微生物PLFAs多样性值最高。
根据典型性相关分析可知,铵态氮对土壤中细菌和放线菌含量影响较为显著,土壤中硝态氮和含水量对细菌含量影响较为显著。
4 讨论
本研究中氮沉降增加了土壤中营养物质的含量,为细菌生长提供了丰富的营养来源,因此一定氮含量的氮沉降
处理可促进细菌的生长。随着氮沉降量的增加,土壤中营养物质的有效性发生变化,微生物对底物的利用模式也发生变化,导致中高氮处理时细菌含量减少。氮沉降影响真菌含量变化可能是由于氮沉降促使土壤有机质含量增加(Van Diepen et al., 2010),导致土壤pH值下降(Turner et al., 2009),土壤中真菌分泌物含量也增加,最终造成真菌生物量的增加或其群落结构发生改变。本研究发现低氮水平促进了真菌含量的增长,但降低了真菌菌群的多样性,而高氮水平抑制了真菌含量的增长,真菌菌群多样性降低,从而降低了真菌的微生物生物量,这些均与前人的研究结果一致(Wallenstein et al., 2004)。
本研究中,沉降高浓度铵态氮时各微生物类型的PLFA均有升高趋势,所以仍需升高浓度进行持续的沉降试验来确定其变化趋势。PLFA 法在一定程度上克服了传统培养方法的不全面性,但因其脂肪酸类型及含量会随生长条件和环境污染情况变化,所以它们还未形成严格的专一性,可能会引起错误的群落指征(陈振翔等,2005)。所以PLFA含量换算成微生物生物量时有关环境因子的转换系数仍有待于进一步研究。
参考文献(References)
| [1] |
陈振翔,于鑫,夏明芳,等.2005.磷脂脂肪酸分析方法在微生物生态学中的应用. 生态学杂志,24(7): 828-832.
(Chen Z X,Yu X,Xia M F,et al. 2005.Application of phospholipid fatty acid (PLFA) analysis in microbial ecology.Chinese Journal of Ecology,24(7): 828- 832[in Chinese]).( 1) 1)
|
| [2] |
黄懿梅. 2008.黄土丘陵区植被自然恢复过程中土壤微生物指标的演变. 杨凌:西北农林科技大学博士学位论文,79-92.
(Huang Y M.2008.Soil microbial indices response to vegetation natural rehabilitation on the loess hilly-gully area.Yangling: PhD thesis of Northwest A&F University,79-92[in Chinese]).( 1) 1)
|
| [3] |
刘波,胡桂萍,郑雪芳,等. 2010.利用磷脂脂肪酸(PLFAs)生物标记法分析水稻根际土壤微生物多样性.中国水稻科学,24(3): 278-288.
(Liu B, Hu G P, Zheng X F, et al.2010.Analysis on microbial diversity in the rhizosphere of rice by phospholipid fatty acids biomarkers.Chin J Rice Sci, 24 (3) : 278-288[in Chinese]).( 1) 1)
|
| [4] |
齐鸿雁,薛凯,张洪勋. 2003.磷脂脂肪酸谱图分析方法及其在微生物生态学领域的应用. 生态学报,23(8):1576- 1582.
(Qi H Y, Xue K, Zhang H X.2003.Phospholipid fatty acid analysis and its applications in microbial ecology.Acta Ecologica Sinica,23(8): 1576- 1582[in Chinese]).( 1) 1)
|
| [5] |
薛璟花,莫江明,李炯,等. 2007.土壤微生物数量对模拟氮沉降增加的早期响应. 广西植物,27(2): 174-179.
(Xue J H, Mo J M, Li J, et al. 2007.The short term response of soil microorganism number to simulated nitrogen deposition.Guihaia, 27(2): 174 - 179[in Chinese]).
|
| [6] |
杨承栋,焦如珍,盛炜彤,等.1999. 江西省大岗山湿地松林土壤性质的变化. 林业科学研究,12(4): 392-397.
(Yang C D, Jiao R Z, Sheng W T, et al. 1999.Change of soil properties under slash pine in Dagangshan, Jiangxi Province.Forest Research,12(4): 392-397[in Chinese]).( 1) 1)
|
| [7] |
于树,汪景宽,李双异,等. 2008.应用PLFA方法分析长期不同施肥处理对玉米地土壤微生物群落结构的影响. 生态学报,28(9):4221- 4227.
(Yu S, Wang J K, Li S Y,et al.2008.Effect of long-term fertilization on soil microbial community structure in corn field with the method of PLFA.Acta Ecologica Sinica,28(9): 4221-4227[in Chinese]).( 1) 1)
|
| [8] |
张秋芳,刘波,林营志,等. 2009.土壤微生物群落磷脂脂肪酸PLFA生物标记多样性. 生态学报,29(8):4127- 4137.
(Zhang Q F, Liu B, Lin Y Z,et al.2009.The diversity of phospholipid fatty acid (PLFA) biomarker for the microbial community in soil.Acta Ecologica Sinica,29(28): 4127-4137[in Chinese]).( 1) 1)
|
| [9] |
张瑞娟,李华,林勤保,等. 2011.土壤微生物群落表征中磷脂脂肪酸(PLFA)方法研究进展.山西农业科学, 39(9):1020-1024.
(Zhang R J,Li H,Lin Q B,et al.2011.Research progress of PLFA method in the soil microbial community.Journal of Shanxi Agricultural Sciences,39(9): 1020-1024[in Chinese]).( 1) 1)
|
| [10] |
Bardgett R D, Hobbs P J, Frostegård Å. 1996. Changes in soil fungal bacterial ratios following reductions in the intensity of management of up land grassland. Biology and Fertility of Soils, 22(3): 261- 264.( 1) 1)
|
| [11] |
Bobbink R, Hicks K, Galloway J, et al. 2010. Global assessment of nitrogen deposition effects on terrestrial plant diversity: a synthesis.Ecological Applications, 20(1): 30-59.( 1) 1)
|
| [12] |
Drenovsky R E, Elliott G N, Graham K J, et al. 2004. Comparison of phospholipid fatty acid (PLFA) and total soil fatty acid methyl esters(TSFAME) for characterizing soil microbial communities.Soil Biology and Biochemistry, 36(11): 1793- 1800.( 1) 1)
|
| [13] |
Elser J J, Bracken M E S, Cleland E E, et al. 2007. Global analysis of nitrogen and phosphorus limitation of primary producers in reshwater, marine and terrestrial ecosystems. Ecology Letters, 10(12): 1135-1142.( 1) 1)
|
| [14] |
Fang Y T, Yoh M, Koba K, et al. 2011. Nitrogen deposition and forest nitrogen cycling along an urban-rural transect in southern China. Global Change Biology, 17(2): 872-885.( 1) 1)
|
| [15] |
Frey S D, Knorr M, Parrent J L, et al. 2004. Chronic nitrogen enrichment affects the structure and function of the soil microbial community in temperate hardwood and pine forests. Forest Ecol Manag, 196(1): 159-171.
|
| [16] |
Gilliam F S. 2006. Response of the herbaceous layer of forest ecosystems to excess nitrogen deposition. Journal of Ecology, 94(6): 1176-1191.( 1) 1)
|
| [17] |
Jain D K, Stroes-Gascoyne S, Providenti M, et al. 1997. Characterization of microbial communities in deep groundwater from granitic rock. Can J Microbial, 43(3): 272-283.( 1) 1)
|
| [18] |
Joergensen R G, Potthoff M. 2005. Microbial react ion in activity, biomass,and community structure after longterm continuous mixing of a grassland soil. Soil Biol Biochem, 37(7) : 1249-1258.( 1) 1)
|
| [19] |
Kourtev P S,Ehrenfeld J G,Haiggelom M. 2002. Exotic plant species alter the microbial community structure and function in the soil. Ecology,83(11): 3152-3166.( 1) 1)
|
| [20] |
MacDonald J A, Dise N B, Matzner E, et al. 2002. Nitrogen input together with ecosystem nitrogen enrichment predict nitrate leaching from European forests. Global Change Biology, 8(10): 1028-1033.( 1) 1)
|
| [21] |
Sakamoto K, Iijima T, Higuchi R. 2004. Use of specific phospholipid fatty acids for identifying and quantifying the external hyphae of the arbuscular mycorrhizal fungus Gigaspora rosea. Soil Biol Biochem, 36(11) : 1827-1834.( 1) 1)
|
| [22] |
Turner M M,Henry H A L, 2009. Interactive effects of warming and increased nitrogen deposition on 15N tracer retention in a temperate old field: seasonal trends.Global Change Biology, 15(12) : 2885-2893.( 1) 1)
|
| [23] |
Van Diepen L,Lilleskov E,Pregitzer K,et al.2010. Simulated nitrogen deposition causes a decline of intra- and extraradical abundance of arbuscular mycorrhizal fungi and changes in microbial community structure in northern hardwood forests. Ecosystems, 13(5) : 683-695.( 1) 1)
|
| [24] |
Wallenstein M D. 2004. Effects of increased nitrogen deposition on forest soil nitrogen cycling and microbial community structure. PhD thesis of Duke University, 47-55.( 2) 2)
|
1
本文献在全文中的定位:
...
污染情况变化,所以它们还未形成严格的专一性,可能会引起错误的群落指征(
陈振翔等,2005)。所以PLFA含量换算成微生物生物量时有关环境因子的转换系数仍有待于进
...
1
本文献在全文中的定位:
...
测定结果中PLFAs含量的标定和计算参照
刘波等(2010)方法,将脂肪酸换算成每克干土中的含量(nmol),计算公式如下:
...
1
本文献在全文中的定位:
...
研究选择PLFA法是因为它不需要分离和培养技术,适合微生物群落的动态追踪(
齐鸿雁等,2003); 操作简便,结果较为精准、客观; 能够定量描述土壤环境中的微生物群体
...
1
本文献在全文中的定位:
...
最高温39.9 ℃,最低气温-8.3 ℃,年降水量1 593.7 mm,集中在4—7月,无霜期为268天(
杨承栋等,1999)。
...
1
本文献在全文中的定位:
...
直接从土壤中提取的磷酯类化合物的量可准确地表达为土壤微生物的生物量(
于树等,2008)。本研究共检测到72种PLFA,其中可对应出微生物类型的特征PLFA为36种
...
1
本文献在全文中的定位:
...
环境十分敏感,当生态机制变化和环境改变时,微生物的群落结构也发生变化(
张瑞娟等,2011)。所以研究微生物群落结构的变化对研究氮沉降所带来的土壤生态系统的变
...
2
本文献在全文中的定位:
...
生物群落结构的变化对研究氮沉降所带来的土壤生态系统的变化具有指向性(
Wallenstein,2004)。微生物群落结构的研究方法很多,包括Biolog法、磷脂脂肪
...
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 1)
1)
 2)
2)
 2015, Vol. 24
2015, Vol. 24

