文章信息
- 尹立伟, 池玉杰
- Yin Liwei, Chi Yujie
- 猴头菌MnP1基因全长cDNA克隆及生物信息学分析
- Cloning and Bioinformatics Analysis of MnP 1 cDNA Gene from Hericium erinaceum
- 林业科学, 2015, 51(5): 68-77
- Scientia Silvae Sinicae, 2015, 51(5): 68-77.
- DOI: 10.11707/j.1001-7488.20150508
-
文章历史
- 收稿日期:2014-09-06
- 修回日期:2014-11-13
-
作者相关文章
2. 东北林业大学林学院 哈尔滨 150040
2. School of Forestry, Northeast Forestry University Harbin 150040
木质素降解酶在分解木质素的过程中,能够产生一系列胞外氧化酶参与对木素大分子聚合物的最初裂解反应,其中锰过氧化物酶(manganese peroxidases/MnPs)、漆酶(laccases)和木质素过氧化物酶(lignin peroxidases/LiPs)构成了木质素主要的降解酶系统(Morgenstern et al., 2008)。这3种酶集中分布在白腐菌中,木质素降解酶具有生物降解、纸浆漂白、染料脱色(Muhammad et al., 2013)及污水处理等功能,在日常生活中广泛开发和应用在化工和造纸等工业生产中。木质素的降解主要依靠真菌中分泌的木质素降解酶来完成,其中的锰过氧化物酶(MnPs;EC1.11.1.13)是一种依赖H2O2的亚铁血红素糖蛋白酶,其分子由10条长的蛋白质单链和1条短的单链构成,含有一系列Fe(s)-卟啉环(IX)血红素辅基的同功酶,能够氧化分解芳香环多聚体、断裂酚和非酚的木质素内部单元结构的Cα- Cβ键和醚键(Pribnow et al., 1989;Orth et al., 1994;Phil et al., 2007)。GenBank中有近2 000多条与MnPs相关的基因序列,多数来自真菌中的高等担子菌,其中包括担子菌亚门层菌纲非褶菌目中的多孔菌科(Polyporaceae)、猴头菌科(Hericiaceae)、灵芝菌科(Ganodermataceae)、革菌科(Thelephoraceae)、刺革菌科(Hymenochaetaceae)、韧革菌科(Stereaceae)、伏革菌科(Corticiaceae)、粉孢革菌科(Coniophoraceae)、齿菌科(Hydnaceae)、珊瑚菌科(Clavariaceae)和裂褶菌科(Schizophyllaceae),还有银耳目(Tremellales)、木耳目(Auriculariales)、花耳目(Dacrymycetales)中的大部分种,伞菌目中的口蘑科(Tricholomataceae)以及子囊菌亚门核菌纲球壳菌目中的团炭角菌科(Xylariaceae)等(池玉杰等,2007)。国外已开展对MnPs基因的酶活特性、基因结构(朱刚等,2013;Nagai et al., 2007)、转录表达(Yeo et al., 2007;Janusza et al., 2012;Hirofumi et al., 2013)、生物学功能与作用机制(Kum et al., 2011)等研究,已日益受到木质素研究者的关注。
猴头菌(Hericium erinaceum)属于木材白腐菌,多选择栎属(Quercus)树木作为寄主,寄生于木材腐朽部分,以分解木材中的木质素而获得碳源、氮源、矿质等营养,现广泛进行人工栽培。猴头菌的营养功效、多糖提取物、抗病机制在医疗及工业领域上有重要的经济价值。目前对白腐菌产生MnPs基因的功能分析和开发利用的研究并不多,对猴头菌MnPs基因的克隆及序列分析尚无报道。本研究以猴头菌为试验材料,克隆MnPs基因全长cDNA序列(命名为He-mnp1),对He-mnp1基因及蛋白序列进行分析,为今后研究猴头菌MnPs基因功能、转录调控和构建优良工程菌株提供参考。
1 材料与方法 1.1 菌种猴头菌菌株CB1(H.erinaceum CB1)源于长白山,分离后保藏于东北林业大学森林保护学科森林病虫病理实验室。此菌株CB1经分子生物学ITS鉴定(GenBank登录号为GU584100)。
1.2 试剂大肠杆菌(Escherichia coli)Top10感受态细胞(TIANGEN);pMD 20 T Simple Vector(TaKaRa);DNAquick-快捷型植物基因组DNA(TIANGEN);E.Z.N.A真菌RNA提取试剂盒(OMEGA);2步法RT-PCR试剂盒(TaKaRa);E.Z.N.A凝胶回收试剂盒(OMEGA);质粒DNA小提试剂盒(OMEGA)。
1.3 引物的设计根据GenBank中已报道的10条白腐菌锰过氧化物酶同源基因,包括变色栓菌(Trametes versicolor)KN9522(cmp2)AY677129;变色栓菌KN9522(cmp3)AY677130;偏肿革裥菌(Lenzites gibbosa)CB-1(mnp2)JQ388597;平菇侧耳(Pleurotus ostreatus)(mnp)U21878;平菇侧耳(mnp3)AB011546;弯孢拟蜡孔菌(Ceriporiopsis subvermispora)(mnp1)AF013257;C.subvermispora (mnp4)AY217670;黄孢原毛平革菌(Phanerochaete chrysosporium)(MnP-1)M60672;黄孢原毛平革菌(mnp2)L29039;原毛平革菌属的P.sordida (mnp3)AB078606。利用软件ClustalX 2.0进行相似序列比对,查找到核苷酸同源序列的保守区,根据保守区序列设计上游引物为5′-SYNATGTNATH TYCTCCNA-3′,下游引物为5′-NANRCNGCGRAA NCNGCT -3′,进行简并PCR的扩增。
1.4 猴头菌的总RNA和DNA的提取收集猴头菌新鲜菌丝体,提取猴头菌总RNA(OMEGA真菌提取试剂盒)和猴头菌基因组DNA(TIANGEN),检验总RNA和dsDNA的质量(完整性),其浓度可用变性琼脂糖凝胶电泳和核酸检测仪测量分析A260/A280及A260/A230的比值,纯度为1.8~2.0。具体操作参照试剂盒说明书进行。
1.5 猴头菌MnP的cDNA基因克隆根据MnP基因的简并引物,以提取的猴头菌DNA为模板,PCR扩增到He-mnp1的DNA片段。MnP基因cDNA片段利用2步法RT-PCR试剂盒(TaKaRa),PrimeScriptTM RTase酶和Oligo dT引物(TaKaRa独自设计的dT区域),以反转录反应液为模板扩增体系如下: 10×PCR Buffer 5 μL,dNTP(2.5 mmol·L-1)8 μL,简并引物(10 μmol·L-1)各1 μL,rTaq DNA聚合酶(5 U·μL)0.5 μL,模板1 μL,用ddH2O补足至50 μL。扩增参数: 94 ℃ 3 min,94 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,30个循环,72 ℃ 10 min。猴头菌MnP基因的5′和3′末端cDNA序列,采用RACE克隆技术首先设计5′特异性引物GSP: 5′-CGACGCAATTGGTTTCTCTCAG-3′,使用SMARTerTM RACE cDNA试剂盒快速扩增(Clontech,USA);然后设计3′端外侧特异性引物GSP: 5′-TTCCACGACGCAATTGGTTTC-3′和3′端内侧特异性引物NGSP: 5′- CAGCATTCTTTCTC GCTTCACT-3′,使用3′-Full RACE试剂盒(TaKaRa)扩增出3′末端cDNA序列。利用ContigExpress拼接软件,重新设计MnP的cDNA基因的上游引物: 5′-ATGGCGTTCAAAACTTTCGCC-3′;下游引物:5′- TTAAGACGGGGGGACGGGG-3′(下划线分别为起始密码子和终止密码子),最终PCR获得He-mnp1基因的cDNA全长序列。
1.6 猴头菌MnP的生物信息学分析克隆获得猴头菌He-mnp1基因的全长cDNA序列,通过NCBI数据库进行BLAST同源搜索,ORF Finder查找该基因的完整开放阅读框(open reading frame,ORF),分析起始密码子ATG和终止密码子TAA的正确位置。利用Expasy数据库和BioEdit软件预测He-mnp1蛋白质的理化特性及氨基酸的组成,同时进行亲/疏水性及跨膜区的分析。采用SignalP 4.1软件进行蛋白信号肽的预测,并利用Clustal W和MEGA 5.1软件对He-mnp1蛋白序列同源性比对和构建白腐菌MnPs系统发育树。利用Conserved Domain Database(CDD)蛋白保守结构域的预测,查看He-mnp1血红素、基质及锰、钙等结合位点。采用PredictProtein软件和SWISS-MODEL软件进行He-mnp1蛋白二级结构的预测和同源三维建模(Canales et al., 1998)。
2 结果与分析 2.1 猴头菌He-mnp1 DNA与cDNA基因片段的扩增同时以提取的猴头菌总DNA和总RNA为模板,进行PCR扩增产物的验证,经1.0%的琼脂糖凝胶电泳图谱,见图 1中CK为空对照,1-2泳道表明猴头菌He-mnp1 DNA产物在600 bp左右有单一亮带,经过回收PCR产物测序得到He-mnp1基因片段573 bp;3-4泳道为He-mnp1 cDNA产物在400 bp左右处有单一亮带,经过RT-PCR产物测序得到He-mnp1cDNA基因片段412 bp。同时将猴头菌He-mnp1 DNA和cDNA基因片段进行BLAST比对,He-mnp1 2条目的基因与其他白腐菌的MnPs基因序列最大相似度为72%,确定猴头菌He-mnp1为一新的MnP基因。He-mnp1基因的DNA序列比cDNA序列长161 bp,表明DNA水平上的He-mnp1含有一段内含子序列。
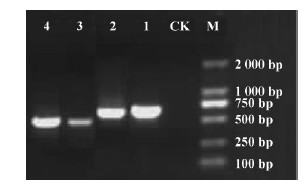 |
图 1 猴头菌He-mnp1 DNA与cDNA基因片段 Fig. 1 He-mnp1 DNA and cDNA gene fragment from H.erinaceum M: DNA标记2 000 bp DL 2 000 Marker; CK:空对照 Control; 1-2: He-mnp1 DNA基因片段 He-mnp1 DNA gene fragment; 3-4: He-mnp1cDNA基因片段 He-mnp1 cDNA gene fragment. |
利用RACE试剂盒扩增He-mnp1 3′端和5′端cDNA目的片段,首先以反转录的cDNA为模板,3′端进行嵌套PCR,外侧引物进行PCR验证,经1.0%的琼脂糖凝胶电泳检测,结果未扩增出目的条带,见图 2中1-2泳道,可能是基因自身丰度较低,需要以第1次PCR产物为模板,利用内侧引物进行第2次PCR反应,见图 2中3-4泳道,回收产物经测序得到3′基因片段410 bp,测序结果显示目的基因序列之后有一段明显的poly A序列;5′端采用SMART RACE技术,利用特异引物进行RT-PCR扩增得到723 bp目的条带,回收并验证结果见图 2中5-6泳道。分别将序列进行BLAST比对,与其他白腐菌的MnPs相似度各达74%和62%。利用ContigExpress拼接软件,将3′端和5′端及cDNA序列对接,查找各特异性引物,最终PCR获得He-mnp1的cDNA全长序列1 080 bp,见图 2中第7泳道;同时以猴头菌总DNA为模板,PCR获得He-mnp1的DNA全长序列1 647 bp作为目的基因对照,见图 2中第8泳道。在NCBI中ORF Finder推导He-mnp1基因含有1条1 080 bp的完整开放阅读框,GenBank注册新基因登录号为HM116841,起始密码子ATG,终止密码子TAA,5′UTR有68个核苷酸,3′UTR有131个核苷酸,共编码蛋白359 aa。
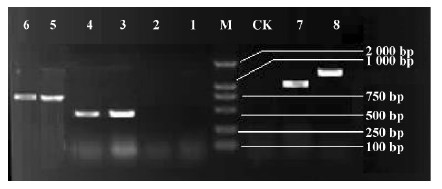 |
图 2 He-mnp1 基因全长cDNA序列 Fig. 2 Full length cDNA products of He-mnp1 1-4: He-mnp1 3′端片段He-mnp1 3′terminal fragment; 5-6: He-mnp15′端片段5′ terminal fragment; 7-8: He-mnp1的cDNA和DNA全长序列Full length cDNA and DNA products of He-mnp1. |
一般生物的蛋白质由20种基本氨基酸组合而成。使用BioEdit软件分析了猴头菌He-mnp1的359 aa,其氨基酸组成为:丙氨酸(Ala)11.98%、半胱氨酸(Cys)2.23%、天冬氨酸(Asp)7.24%、谷氨酸(Glu)6.13%、苯丙氨酸(Phe)6.96%、甘氨酸(Gly)7.52%、组氨酸(His)1.95%、异亮氨酸(Ile)6.41%、赖氨酸(Lys)1.95%、亮氨酸(Leu)6.69%、蛋氨酸(Met)1.67%、天冬酰胺(Asn)3.9%、脯氨酸(Pro)7.52%、谷氨酰胺(Gln)3.9%、精氨酸(Arg)3.61%、丝氨酸(Ser)5.85%、苏氨酸(Thr)8.64%、缬氨酸(Val)5.29%、色氨酸(Trp)0.56%和酪氨酸(Tyr)0,其中丙氨酸(Ala)含量最高,酪氨酸(Tyr)无。
2.3.2 猴头菌He-mnp1蛋白质的的理化特性利用Expasy数据库中protparam tool和Compute pI/Mw tool预测He-mnp1的分子质量约为38.18 kDa,等电点(pI)为4.35,其中含有48个带负电荷的氨基酸残基(ASP+Glu)和20个带正电荷的氨基酸残基(Arg+Lys)。N-末端含硫类的蛋氨酸M(MET)序列,在酵母和大肠杆菌中的半衰期分别大于10和20 h,不稳定指数达52.97,脂肪族氨基酸指数较高,达到78.38,这是由于其中含有较多的Gly(7.5%)、Ala(12%)、Asp(7.2%)、Phe(7.0%)、Pro(7.5%)和Thr(8.6%)。
2.3.3 猴头菌He-mnp1信号肽的预测与蛋白序列同源性比对采用SignalP 4.1 Server系统对猴头菌He-mnp1氨基酸序列的信号肽进行了预测,使用Nature Methods方法预测最可能的信号肽切割位点位于18-19 aa之间: TNA-AI,He-mnp1前18个氨基酸残基为信号肽序列(MAFKTFAAFIA LWGVTNA),He-mnp1成熟蛋白N末端序列(VACPDGVNT ATNA)。使用Clustal W在线软件对5种不同白腐真菌猴头菌、Heterobasidion irregulare、变色栓菌、地中海嗜蓝孢孔菌(Fomitiporia mediterranea)、平菇侧耳的MnPs同工酶基因进行同源蛋白多序列比对,比对结果显示了成熟蛋白(用虚线框隔开)、保守氨基酸残基及半胱氨酸等信息,图中名称均为缩写(例如:变色栓菌的MnP基因缩写为Tv- mnp),结果见图 3。
 |
图 3 He-mnp1与同源蛋白的多重比对 Fig. 3 Multiple alignment of homologous protein and He-mnp1 *:保守的氨基酸残基The conserved residues; □:保守的Mn2+结合结构域 The conserved Mn2+ binding domain; ↓:使Mn2+氧化的锰结合位点 The Mn-binding site enabling oxidation of Mn2+ ; ◆ :参与芳香族底物的氧化残基Residues that are involved in oxidation of aromatic substrates; ▲:保守半胱氨酸The conserved cysteine residues; ◇ :催化作用中必不可少的2个组氨酸(H) The two histidines(H)that are essential for catalysis. |
在NCBI数据库中进行BLAST序列比对,将克隆的He-mnp1全长cDNA序列翻译成含有359 aa,tblastn结果未搜索到相似度为100%的基因序列,确定猴头菌He-mnp1为新基因序列,GenBank注册新基因登录号为HM116841。在190株菌种中选择10株白腐菌与猴头菌He-mnp1进行同源性分析,采用MEGA 5.1软件Neighbor-Joining法并构建He-mnp1蛋白系统进化树。结果表明黄孢原毛平革菌编码蛋白mnp1,mnp2,mnp3(AAA33744,AAA33745,AAB39652),弯孢拟蜡孔菌编码序列mnp1和mnp2(AAC05222和AAB92247),地中海嗜蓝孢孔菌mnp1和mnp2(ADK60889和ADK60890),包括在第1组群(Group I),它们都是由5个二硫键的10半胱氨酸组成的MnPs。而猴头菌CB1编码蛋白He-mnp1(ADK26471)(图 4中左箭头所示),平菇侧耳mnp3(BAA33449),冬生多孔菌(Polyporus brumalis)mnp1(HQ910417)和变色栓菌KN9522编码cmp2序列(AAT90349),包括在第2组群(GroupⅡ),它们都是由4个二硫键的8半胱氨酸组成的MnPs。另外,选择2株变色栓菌木质素过氧化物酶基因编码序列LiP1和LiP2(CAA53334,CAA53333)作为外源群组,包括在第3组群(Group Ⅲ)。系统发育分析表明第1组群、第2组群和第3组群之间有较为清晰的界限。猴头菌He-mnp1与平菇侧耳、冬生多孔菌、变色栓菌的MnPs基因遗传距离较近、同源性最高,而与黄孢原毛平革菌、弯孢拟蜡孔菌和地中海嗜蓝孢孔菌MnPs基因遗传距离较远,从系统进化树中也可看出同一物种的MnPs基因同工酶之间在遗传距离上会较近(图 4)。
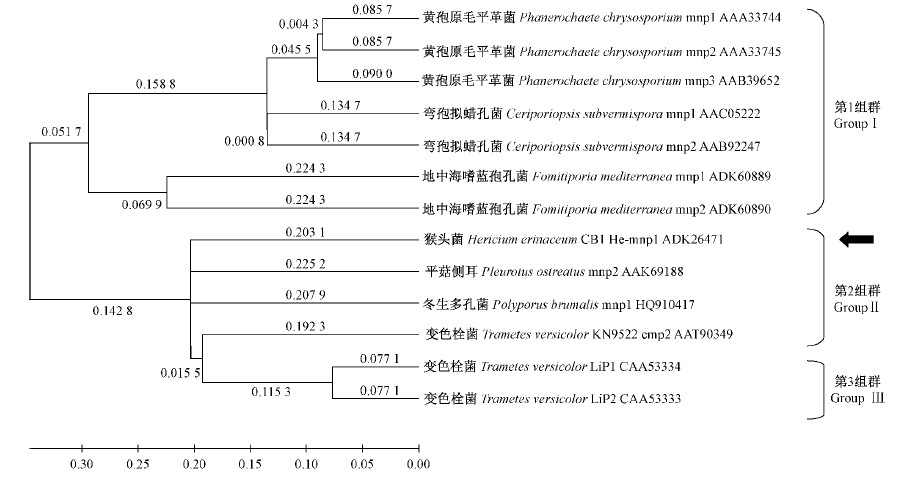 |
图 4 He-mnp1 的蛋白系统发育树分析 Fig. 4 Protein phylogenetic tree analysis of He-mnp1 |
通过ExPASy蛋白预测专家系统中Protscale软件采用Kyte & Doolittle方法分析猴头菌He-mnp1蛋白序列的亲/疏水性及蛋白跨膜区,结果显示He-mnp1的蛋白有明显的亲水区,仅存在81-105、121-141间有2处疏水区域,见图 5(a)。与利用TMpred在线工具分析He-mnp1蛋白跨膜区,疏水性分析结果一致,见图 5(b)跨膜蛋白的拓扑结构模型的预测参数TM-helix螺旋长度都在17~33之间,结果He-mnp1由内向外存在3处跨膜核心区,内侧在1-21(21)→1671+、85-109(25)→749++、125-145(21)→551+,以749++为强烈的跨膜取向。由外向内存在4处跨膜核心区,1-21(21)→1421、24-44(21)→157++、85-103(19)→455,130-152(23)→462,N末端外存在最优的跨膜拓扑结构模型,2处强烈的跨膜螺旋位点分别为1-21(21)→1421和85-109(25)→ 749。蛋白的开始区域有较强的疏水性,可能与分泌蛋白的信号肽性质相关,此蛋白平均亲水指数为-0.042,是一个亲水蛋白。
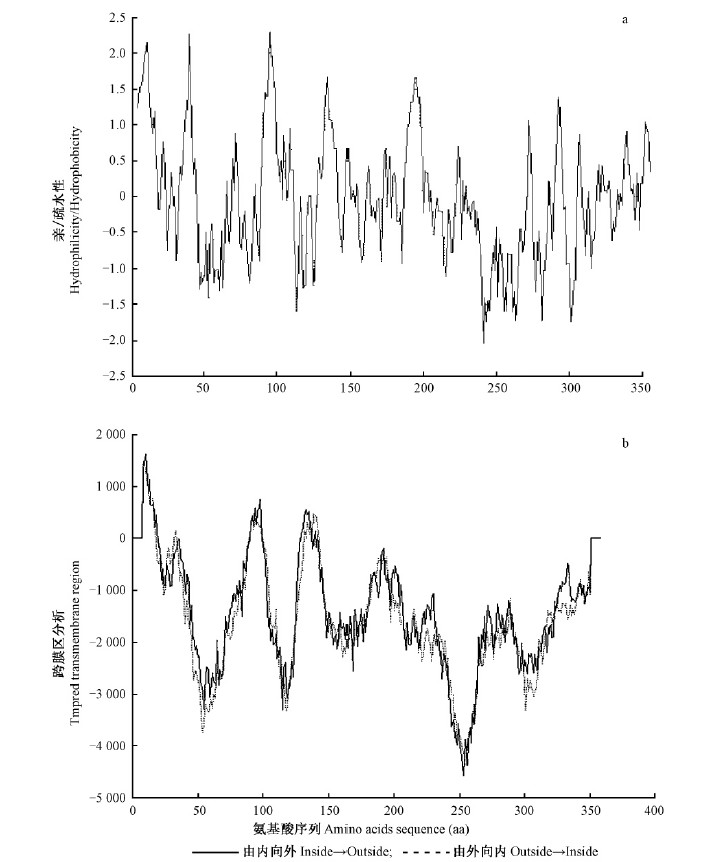 |
图 5 He-mnp1 亲/疏水性(a)和跨膜区(b) Fig. 5 Hydrophilicity /Hydrophobicity (a) and transmembrane region (b) of He-mnp1 |
首先根据NCBI数据库的Conserved Domain Database(CDD)预测到He-mnp1基因存在一个保守结构域,并对其酶的作用物或抑制剂的结合位点、血红素结合位点、基质结合位点、锰结合位点、钙结合位点做了预测(Ruiz-Dueйas et al., 2009)。保守结构域分析显示He-mnp1为特定群体-真菌木质素降解酶,属Class Ⅱ类的真菌血红素过氧化物酶蛋白家族(Morgenstern et al., 2010)。此家族成员共享一个血红素辅基和催化多元氧化反应,以过氧化氢为电子受体。Class Ⅱ类过氧化物酶是真菌糖蛋白,与木质素氧化分解相关,含有多处血红素结合位点、3个基质结合位点、 3个Mn2+结合位点和2处保守的Ca2+结合域(图 6)(Sundaramoorthy et al., 1994;2005;Blodig et al., 2001)。
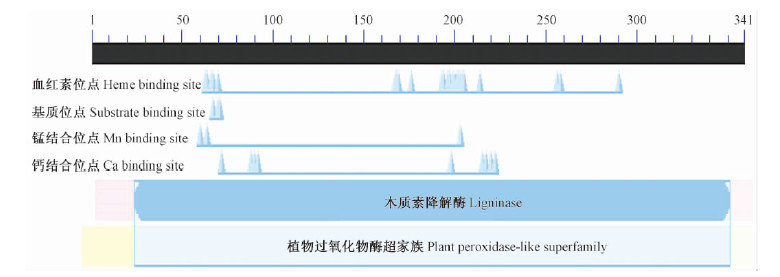 |
图 6 He-mnp1蛋白保守结构域 Fig. 6 Conserved domain of He-mnp1 |
使用PREDATOR在线软件预测He-mnp1蛋白二级结构中α-螺旋(Alpha helix,Hh)的氨基酸有110个,所占比例为30.99%;β-折叠(Extended str and ,Ee)氨基酸有12个,占3.38%;无规则卷曲(R and om coil,Cc)的氨基酸有233个,占65.63%。由图 7可见,He-mnp1蛋白以α-螺旋(蓝色)和无规则卷曲(紫色)为主,β-折叠(红色)所占比率最低,验证了He-mnp1蛋白二级结构属于稳定蛋白。
 |
图 7 He-mnp1蛋白二级结构预测 Fig. 7 The secondary structure prediction of He-mnp1 |
使用SWISS-MODEL的Cn3D软件预测工具构建猴头菌He-mnp1蛋白三维结构模型,结构模型有不同的结构形式,其中有螺纹模型、线性模型、球棍模型及空间结构模型,见图 8为He-mnp1蛋白三维螺纹模型。将He-mnp1成熟蛋白序列通过VAST Search比对后,将结果输入Cn3D软件进行功能位点模拟,结果显示从He-mnp1蛋白三维结构模型中得到1个Fe血红素,2个Ca2+、1个Mn2+的结合位点及组氨酸残基等。
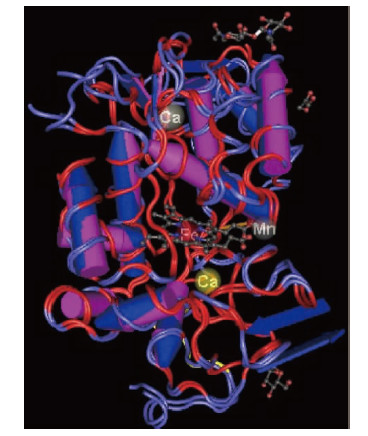 |
图 8 He-mnp1三维结构模型的构建 Fig. 8 The construction of the three-dimensional structure model of He-mnp1 |
我国林区、贮木及建材腐朽或材质降低,牵涉到胞外大分子氧化降解和胞内小分子分解代谢(严东辉等,2003;戴玉成等,2008;曾祥谓等,2008),在真菌分泌降解木质素的非特异性胞外氧化还原酶中,MnPs基因起着至关重要的作用。其白腐菌的分子特征和转录调控已在逐步开展研究(Manubens et al., 2003;Johansson et al., 2002;Ryu et al., 2014),发现一些白腐菌[黄孢原毛平革菌、弯孢拟蜡孔菌、地中海嗜蓝孢孔菌、射脉菌(Phlebia radiata)等]存在多个MnPs同工酶基因,它们受不同的诱导条件,如氮源、温度、pH值(Kazumichi et al., 2013)、吐温80、热稳定性、血红素、静置与振荡、 Mn2+浓度(Mancilla et al., 2010;Sangcheol et al., 2004)等影响,而猴头菌He-MnPs同工酶基因仍需进一步研究。本文所克隆的猴头菌He-mnp1基因全长cDNA序列登录号(HM116841.3),在2010年提交序列之后又经过2次更新,He-mnp1全长cDNA序列共1 279 bp,含有1条1 080 bp的完整开放阅读框,DNA测序获得1 647 bp,与He-mnp1的cDNA全长序列相对比,具有10条内含子,内含子平均长度为51~62 bp;11条外显子,平均长度为13~208 bp,并翻译成359 aa。
通过Signal软件预测信号肽,发现He-mnp1蛋白质多肽前体包含1个18 aa的信号肽及1个5 aa的中间前导短肽,这样He-mnp1成熟蛋白编码序列为336 aa;利用Clustal W软件进行同源MnPs蛋白多重比对,结果表明白腐菌MnPs序列之间存在一些相似性及保守区域,具体分析成熟蛋白与芳香结合位点于143-146之间(I-P-E-P),在催化作用中必不可少的2个组氨酸分别为H 48远端组氨酸和H 174近端组氨酸,精氨酸残基位于R 44,构成4个二硫键的8半胱氨酸残基分别位于C 26: C 38,C 39: C 58,C 143: C 271和C 307: C 336。从He-mnp1氨基酸组成结构来看,白腐菌MnPs基因的催化机制、晶体结构和功能位点与相关的氨基酸残基都是相对保守的。并构建了白腐菌MnPs蛋白系统进化树,He-mnp1与平菇侧耳mnp2、冬生多孔菌mnp1和变色栓菌cmp2的亲缘关系最为接近。通常白腐菌同一物种的MnPs基因同工酶会聚类在一起,除非同一物种中含有4个或5个二硫键的组成不同,MnPs基因就会分配在不同的组群(Groups)。Hildén等(2005)对射脉菌的Pr-MnP2和Pr-MnP3基因进行了系统发育和结构分析,研究结果显示,Pr-MnP2含有10个半胱氨酸,而Pr-MnP3含有8个半胱氨酸,它们在进化树的位置就会出现不同(Hildén et al., 2005;Hakala et al., 2006)。白腐菌中这种二硫键频繁出现在胞外蛋白中,把不同的肽链连接在一起形成稳定的蛋白质三维结构,使蛋白质分子不易被降解,可提高锰过氧化酶的活力和热稳定性。另外选择2株变色栓菌木质素过氧化物酶基因LiP1和LiP2作为外源群组,2条LiPs基因在遗传相似系数上极为接近,虽与MnPs基因同属木质素降解酶基因,但在遗传距离上变色栓菌LiPs基因与第1、第2组群有较为清晰的界限。
根据CDD数据库预测He-mnp1蛋白存在一个保守结构域,该成熟蛋白保守结构域与同源蛋白多序列比对分析发现,结构域中有多处血红素结合位点;基质结合位点分别位于R 44,F 47,H 48;构成Mn2+结合有2处保守结构域(37-40和174-187),其中He-mnp1蛋白配体的Mn2+结合位点分别位于E 37,E 41,D 174;2处保守的Ca2+结合域,其中4个与近侧末端的Ca连接氨基酸结合位点分别位于(D49,G 65,D 67,S 69)和5个远侧Ca结合位点分别位于(T 175,D 192,T 194,I 197,D 199)。以上说明,He-mnp1的蛋白结构域中有多处结合位点,包括血红素、Mn2+、Ca2+和基质的存在,推测猴头菌可能存在多条MnPs同工酶基因,且参与猴头菌He-mnps基因的转录水平,调控着锰过氧化物酶表达量的高低。同源蛋白多序列的比对结果显著地提高了He-mnp1蛋白二级结构预测的精确度,此蛋白二级结构中存在α-螺旋、β-折叠和一些无规则卷曲,He-mnp1属于稳定蛋白。与此同时,通过SWISS-MODEL同源建模法构建了He-mnp1成熟蛋白三维结构螺纹模型,结果该功能蛋白所结合的主要位点区域,显示有1个Fe血红素、2个Ca2+、1个Mn2+离子的结合,一些组氨酸残基以及肽链向外突出的缠绕等。该He-mnp1蛋白三维结构与解析的MnPs蛋白空间特征基本吻合,这些生物信息为He-mnp1蛋白的功能预测及认识其MnPs基因的转录表达提供初步参考。
通过Protscale软件分析猴头菌He-mnp1蛋白序列的亲/疏水性及蛋白跨膜区,结果显示He-mnp1是一个亲水蛋白,平均亲水指数为-0.042,He-mnp1由内向外存在3处跨膜核心区,跨膜片段含有多处的疏水残基,但He-mnp1拓扑结构分析结果还需要进一步验证。通过分析其理化特性、功能位点、亲/疏水性及蛋白跨膜区、蛋白结构等,利用多种生物信息学软件进行猴头菌He-mnp1蛋白的预测,可知He-mnp1蛋白具有白腐真菌MnPs基因典型的保守结构域和同源性,表明所克隆的猴头菌He-mnp1是高等真菌MnPs基因家族的新成员,属Class Ⅱ类的真菌血红素过氧化物酶蛋白家族,可为猴头菌筛选MnPs基因工程菌株提供基础信息,但对基因功能的改造有待深入研究。目前本研究小组已针对大肠杆菌、毕赤酵母(Pichia pastoris)和构巢曲霉(Aspergillus nidulans)等生物系统开展MnPs基因转录与异源表达研究,并将深入研究主要的功能蛋白。
| [1] |
池玉杰, 伊洪伟. 2007. 木材白腐菌分解木质素的酶系统-锰过氧化物酶、漆酶和木质素过氧化物酶催化分解木质素的机制. 菌物学报, 26 (1): 153-160. (Chi Y J, Yi H W.2007. Lignin degradation mechanisms of ligninolytic enzyme system,manganese peroxidase, laccase and lignin peroxidase, produced by wood white rot fungi. Mycosystema, 26(1): 153-160[in Chinese]).(  1) 1)
|
| [2] |
戴玉成, 徐梅卿, 杨 忠, 等. 2008. 中国储木及建筑木材腐朽菌(Ⅰ).林业科学研究, 21 (1): 49-54. (Dai Y C, Xu M Q, Yang Z, et al. 2008. Wood-decaying fungi on timber or wooden constructions in China(I). Forest Research, 21 (1): 49-54[in Chinese]).(  1) 1)
|
| [3] |
严东辉, 姚一建. 2003. 菌物在森林生态系统中的功能和作用研究进展.植物生态学报, 27 (2): 143-150. (Yan D H, Yao Y J. 2003.Recent advance of research on fungi in forest ecosystem. Acta Phytoecologica Sinica, 27 (2): 143-150[in Chinese]).(  1) 1)
|
| [4] |
曾祥谓, 崔宝凯, 徐梅卿, 等. 2008. 中国储木及建筑木材腐朽菌(Ⅱ). 林业科学研究, 21 (6): 783-791. (Zeng X W, Cui B K, Xu M Q, et al. 2008. Wood-decaying fungi on timber or wooden constructions in China(Ⅱ). Forest Research, 21(6): 783-791[in Chinese]).(  1) 1)
|
| [5] |
朱 刚, 吴 林, 陈明杰, 等. 2013. 草菇锰过氧化物酶编码基因生物信息学分析及其转录水平和酶活性的测定.菌物学报, 32 (5): 919-927.[in Chinese])(Zhu G, Wu L, Chen M J, et al. 2013. Bioinformatic, gene expression and enzyme activity analyses of manganese peroxidases in Volvariella volvacea. Mycosystema, 32 (5): 919-927.[in Chinese])( 1) 1)
|
| [6] |
Blodig W, Smith A T, Doyle W A, et al. 2001.Crystal structures of pristine and oxidatively processed lignin peroxidase expressed in Escherichia coli and of the W171F variant that eliminates the redox active tryptophan 171. Implications for the reaction mechanism. J Mol Biol, 305 (4): 851-861.( 1) 1)
|
| [7] |
Canales M, Lobos S, Vicuйa R. 1998. Molecular modeling of manganese peroxidase from the lignin-degrading fungus Ceriporiopsis subvermispora and structural comparison with other peroxidases. Elec J Biotech, 1 (2): 96-102.( 1) 1)
|
| [8] |
Hakala T, Hilden K, Maijala P, et al. 2006. Differential regulation of manganese peroxidases and characterization of two variable MnP encoding genes in the white rot fungus Physisporinus rivulosus. Appl Microbiol Biotechnol, 73 (4): 839-849.( 1) 1)
|
| [9] |
Hildén K, Martinez A T,Hatakka A, et al. 2005. The two manganese peroxidases Pr-MnP2 and Pr-MnP3 of Phlebia radiata, a lignin-degrading basidiomycete, are phylogenetically and structurally divergent. Fungal Genetics and Biology, 42 (5): 403-419. ( 1) 1)
|
| [10] |
Hirofumi H, Kenta M, Tomohiro S, et al. 2013. Improvement of manganese peroxidase production by the hyper lignin-degrading fungus Phanerochaete sordida YK-624 by recombinant expression of the 5-aminolevulinic acid synthase gene.Current Microbiology, 67: 708-711. ( 1) 1)
|
| [11] |
Janusza G, Katarzyna K H, Pawlik A, et al. 2012. Fungal laccase, manganese peroxidase and lignin peroxidase: Gene expression and regulation. Enzyme and Microbial Technology, 52 (1): 1-12.( 1) 1)
|
| [12] |
Johansson T, Nymann P, Cullen D. 2002. Differential regulation of mnp2, a new manganese peroxidase encoding gene from the ligninolytic fungus Trametes versicolor PRL572. Appl Environ Microbiol, 68 (4): 2077-2080.( 1) 1)
|
| [13] |
Kazumichi F, Mari U, Chie H, et al. 2013. Environmental control of lignin peroxidase, manganese peroxidase, and laccase activities in forest floor layers in humid Asia.Soil Biology and Biochemistry, 57: 109-115.( 1) 1)
|
| [14] |
Kum H, Lee S, Ryu S, et al. 2011. Degradation of endocrine disrupting chemicals by genetic transformants with two lignin degrading enzymes in Phlebia tremellosa. J Microbiol, 49 (5): 824-827.( 1) 1)
|
| [15] |
Mancilla R A,Canessa P, Manubens A, et al. 2010. Effect of manganese on the secretion of manganese-peroxidase by the basidiomycete Ceriporiopsis subvermispora. Fungal Genetics and Biology, 47 (7): 656-661. ( 1) 1)
|
| [16] |
Manubens A, Avila M, Canessa P, et al. 2003. Differential regulation of genes encoding manganese peroxidase (MnP) in the basidiomycete Ceriporiopsis subvermispora. Curr Genet, 43 (6): 433-438.( 1) 1)
|
| [17] |
Morgenstern I, Klopman S, Hibbett D S. 2008. Molecular evolution and diversity of lignin degrading heme peroxidases in the agaricomycetes. J Mol Evol, 66 (3): 243-257.( 1) 1)
|
| [18] |
Morgenstern I, Robertson D L, Hibbett D S. 2010. Characterization of three mnp genes of Fomitiporia mediterrane and report of additional class Ⅱ Peroxidases in the order hymenochaetales. Applied and Environmental Microbiology, 76 (19): 6431-6440.( 1) 1)
|
| [19] |
Muhammad A,Bazgha A,Hafiz M N I. 2013. Novel catalytic and effluent decolorization functionalities of sol-gel immobilized Pleurotus ostreatus IBL-02 manganese peroxidase produced from bio-processing of wheat straw. Chinese Journal of Catalysis, 34 (9): 1756-1761.( 1) 1)
|
| [20] |
Nagai M, Sakamoto Y, Nakade K, et al. 2007. Isolation and characterization of the gene encoding a manganese peroxidase from Lentinula edodes. Mycoscience, 48 (2): 125-130. ( 1) 1)
|
| [21] |
Orth A, Rzhetskaya M, Cullen D, et al. 1994. Characterization of a cDNA encoding a manganese peroxidase from Phanerochaete chrysosporium: genomic organization of lignin and manganese peroxidase genes. Gene, 148 (1): 161-165.( 1) 1)
|
| [22] |
Phil K, Dan C. 2007. Extracellular oxidative systems of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Fungal Genetics and Biology, 44 (2): 77-87.( 1) 1)
|
| [23] |
Pribnow D, Mayfield M B, Nipper V J, et al. 1989. Characterization of a cDNA encoding a manganese peroxidase, from the lignin-degrading basidiomycete Phanerochaete chrysosporium. J Biol Chem, 264 (9): 5036-5040.( 1) 1)
|
| [24] |
Ruiz-Dueйas F J, Morales M, Garcia E, et al. 2009. Substrate oxidation sites in versatile peroxidase and other basidiomycete peroxidases. J Exp Bot, 60 (2): 441-452.( 1) 1)
|
| [25] |
Ryu S H, Kim B, Kim M, et al. 2014. Molecular characterization of manganese peroxidases from white-rot fungus Polyporus brumalis. Bioprocess Biosyst Eng, 37 (3): 393-400. ( 1) 1)
|
| [26] |
Sangcheol J, Kyunghoon K, Hyoung C T, et al. 2004. Characterization of Mn-peroxidase isoenzymes from the white-rot fungus Trametes versicolor in Korea. Journal of Huazhong Agricultural University, 23 (1): 146-149.( 1) 1)
|
| [27] |
Sundaramoorthy M, Kishi K, Gold M H, et al. 1994.The crystal structure of manganese peroxidase from Phanerochaete chrysosporium at 2.06-A resolution. J Biol Chem , 269 (52): 32759 -32767.( 1) 1)
|
| [28] |
Sundaramoorthy M, Youngs H L, Gold M H, et al. 2005. High-resolution crystal structure of manganese peroxidase: substrate and inhibitor complexes. Biochemistry, 44 (17): 6463 -64670.( 1) 1)
|
| [29] |
Yeo S, Park N, Song H G, et al. 2007. Generation of a transformant showing higher manganese peroxidase (MnP) activity by overexpression of MnP gene in Trametes versicolor.J Microbiol, 45 (3): 213-218.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51

