文章信息
- 罗群凤, 胥猛, 冯源恒, 徐嘉娟, 李火根
- Luo Qunfeng, Xu Meng, Feng Yuanheng, Xu Jiajuan, Li Huogen
- 北美鹅掌楸LtCHS基因的克隆及生物信息学与组织表达特征分析
- Cloning and Bioinformatics of Chalcone Synthase Gene (CHS) in Liriodendron tulipifera and Characterization of Its Tissue Expression
- 林业科学, 2015, 51(5): 37-45
- Scientia Silvae Sinicae, 2015, 51(5): 37-45.
- DOI: 10.11707/j.1001-7488.20150505
-
文章历史
- 收稿日期:2014-07-10
- 修回日期:2014-08-12
-
作者相关文章
2. 广西壮族自治区林业科学研究院 南宁 530002
2. Guangxi Institute of Forestry Science Nanning 530002
查尔酮合成酶(chalcone synthase,CHS)是黄酮类化合物生物合成途径中的第1个关键酶(Huang et al.,2004),该酶催化4-香豆酸-CoA与丙二酰-CoA反应,合成柚皮素查尔酮(何水林等,2004)。查尔酮为类黄酮类物质提供了基本的碳架结构,为类黄酮、黄酮醇、黄烷酮、花青素糖苷及其他次生代谢物质的合成提供了保障(蒋明等,2007; Martin,1993)。查尔酮合成酶基因在类黄酮类物质合成中扮演着重要的角色,参与色素合成、防御反应、植物育性等诸多生理生化过程,在植物的生长发育中起着至关重要的作用。
CHS基因广泛存在于被子植物中,具有相同的氨基酸活性残基,在生物学功能和进化上均较为保守。CHS和CHS类基因可能来自共同的祖先,在物种分化和新类群产生过程中发生了不同程度的重复和变异(Durbin et al., 1995; 2000; Austin et al., 2003)。目前CHS基因已在数百种植物中被分离克隆出来,如银杏(Ginkgo biloba)(李琳玲等,2010)、大豆(Glycine max)(单丽伟等,2012)、葡萄(Vitis vinifera)(周军等,2010)、百合(Lilium brownii)(安利清等,2011)、玉米(Zea mays)(Franken et al., 1991)、莲(Nelumbo nucifera)(王曼玲等,2006)等,通常作为研究花色调控的候选基因。
自然界中,木兰科(Magnoliaceae)鹅掌楸属(Liriodendron)现仅存2种,即分布于我国长江流域和越南北部的鹅掌楸(L. chinense),以及天然分布于美国东部和加拿大南部的北美鹅掌楸(L. tulipifera)(Harlow et al.,1994)。而杂交鹅掌楸(L. tulipifera × L. chinense)则为北美鹅掌楸与鹅掌楸的人工杂交种。鹅掌楸属树种的花形似郁金香,因而又称之为郁金香树(tulip tree),其花大、花色艳丽,观赏性佳,且种间差异明显(图 1),是研究木本植物花色形成与调控机制的较理想材料。但迄今为止,有关鹅掌楸属树种花色调控基因研究尚为空白。由于查尔酮合成酶(CHS)是植物花色素合成中的一个关键酶,其表达受UV照射(Kreuzaler et al.,1983)、真菌侵染(Lamb et al.,1989)等条件诱导,且在花中特异性表达(Johzuka-Hisatomi et al.,1999),其表达量的改变可能导致植物花色的变异(Elomaa et al.,1993),对花色形成具有重要影响; CHS基因又是研究科以下系统发育的理想材料,种间进化差异较大。因此CHS基因用于研究鹅掌楸属花色差异形成机制有较强的可行性。本研究以北美鹅掌楸的花芽为材料,采用同源克隆和RACE技术克隆查尔酮合成酶(CHS)基因,并对其进行生物信息学及组织表达分析,以期为鹅掌楸属树种花色的形成和调控机制研究提供参考。
 |
图 1 鹅掌楸属树种的花色 Fig. 1 The flower colors in Liriodendron A: 鹅掌楸 L. chinense B: 北美鹅掌楸 L. tulipifera C: 杂交鹅掌楸 L. tulipifera × L. chinense |
2 012年春季,在江苏句容南京林业大学下蜀实习林场的鹅掌楸属种源试验林内采集1株北美鹅掌楸(引自美国路易斯安那州,编号: LYS)的花芽用于基因克隆,同时,采集另1株北美鹅掌楸(种源不详,编号: BM),以及鹅掌楸(引自福建武夷山,编号: WYS)、杂交鹅掌楸(编号: F1)各1株,取其花萼、花瓣、雄蕊和叶片4个组织作为基因表达分析的试验材料,液氮速冻后置于-80 ℃冰箱中保存备用。
1.2 基因组DNA和RNA的提取及cDNA第1链的合成采用德国Qiagen公司的DNeasy Plant Mini Kit提取DNA,采用北京TIANGEN公司的RNAplant plus Reagent和德国Qiagen公司的RNeasy Plant Mini Kit提取RNA,并对其分别进行DNA和RNA的消化。取适量样品用1%琼脂糖凝胶电泳检测其完整性,并用紫外分光光度计检测其浓度与纯度。
以总RNA为模板,并以Oligo(dT)18 Primer为引物,参照Thermo Scientific公司的RevertAid First Str and cDNA Synthesis Kit合成第1链cDNA,存于-20 ℃备用。
1.3 RACE扩增3′RACE和5′RACE按照TaKaRa公司试剂盒说明书进行。根据NCBI数据库中查找到的北美鹅掌楸CHS基因(LtCHS)的部分序列设计基因特异引物3GSP1(5′-GACATGCCTGGTGCTGATTA-3′),3GSP2(5′-GCGTGTTCTTGTTGTGTGCT-3′),5GSP1(5′-CAACACACCATATCATACGCAC-3′),5GSP2(5′-CTTCCCTTCCTCTACAGATTTCTT -3′)和5GSP3(5′-GTGGACAACGAACCTTAACTCT-3′),5GSP4(5′-AATCCAGCCGTTCTCCAGTC-3′)。3′端分别以3GSP1、3GSP2进行第1轮和第2轮嵌套式PCR扩增。5′端分别以5GSP1、5GSP2和5GSP3、5GSP4进行第1轮和第2轮扩增,得到2个5′RACE产物。将PCR产物经2%琼脂糖凝胶电泳检测,切下目的条带用胶回收试剂盒(北京TIANGEN)进行回收,产物与pMD19-T Vector(TaKaRa)进行连接,转化大肠杆菌(Escherichia coli)TOP10感受态细胞,最后将筛选到的阳性克隆菌液送上海英骏生物技术有限公司测序。
1.4 全长cDNA序列和基因组DNA序列的扩增采用BioXM软件对LtCHS基因的3′RACE和5′RACE的测序结果进行比对拼接,通过NCBI预测分析全长编码序列,设计全长引物CHS1-F(5′-ATGATGTACCAACAAGGTTG-3′),CHS1-R(5′-AACACACCATATCATACGCAC-3′)和CHS2-F(5′- CCCTTTGAGAGAGAACTATTAC-3′),CHS2-R(5′-TGGACAACGAACCTTAACTCT-3′)分别扩增北美鹅掌楸(LYS)的LtCHS1和LtCHS2 cDNA和gDNA全长序列,最后将目的片段进行克隆测序。
1.5 生物信息学分析利用ORF Finder软件对序列进行分析,利用ProtParam软件计算蛋白质的分子质量和理论等电点。采用InterProScan(http://www.ebi.ac.uk/InterProScan/index.html)在线软件工具进行结构域和功能位点预测,运用ProtScale程序(http://web.expasy.org/protscale/)绘制蛋白质的亲疏水性系列谱; 利用欧洲生物学实验室提供的蛋白质序列和结构预测服务网站PredictProtein(http://www.predictprotein.org/)预测蛋白质的二级结构; Swiss-Model Workspace(http://swissmodel.expasy.org/workspace/)预测蛋白质三级结构(Guex et al.,1997; Schwede et al.,2003; Arnold et al.,2006; Kiefer et al.,2009)。
1.6 CHS基因的组织表达分析分别提取北美鹅掌楸(BM)、鹅掌楸(WYS)及杂交鹅掌楸(F1)的花萼、花瓣、雄蕊和叶片4个组织的总RNA,以反转录得到的第1链cDNA为模板, CHS3-F(5′-GGCTCATCTCAACGAACATC-3′),CHS3-R(5′-AACACACCATATCATACGCAC-3′)和CHS4-F(5′-CACCGCACAAACCATACTAC-3′),CHS4-R(5′-TGGACAACGAACCTTAACTCT-3′)为引物进行RT-PCR扩增,检测2个CHS基因在不同种及不同组织中的表达情况。内参基因上下游引物分别为ACT-F(5′-TCGAGCAGGAGCTAGAGACA-3′),ACT-R(5′-AAGAGATGGCTGGAAGAGGA-3′)。反应体系为20 μL,包括10×PCR Buffer(Mg2+ Free)2.0 μL,MgCl2(25 mmol·L-1)1.5 μL,dNTP Mixture(2.5 mmol·L-1)1.3 μL,引物各1.0 μL,模板cDNA 1.0 μL,rTaq 1.0 μL,ddH2O 11.2 μL。反应程序为: 94 ℃ 3 min; 94 ℃ 30 s,58 ℃ 30 s,72 ℃ 1 min,28个循环; 最后延伸10 min。
2 结果与分析 2.1 LtCHS基因全长序列的获得与分析通过3′RACE和5′RACE 的2轮PCR扩增获得2个CHS基因,在BioXM上进行比对拼接,获得基因全长,命名为LtCHS1、LtCHS2。根据拼接得到的序列设计特异性引物,将PCR扩增获得的目的条带送上海英骏生物技术有限公司进行测序,测序结果和拼接结果完全吻合。
LtCHS1基因的cDNA全长875 bp,其中包括1个13 bp的5′-UTR,1个702 bp的ORF,1个160 bp的3′-UTR。该基因在14-16位为起始密码子ATG,下游有同框终止密码子TAA和polyA尾。推导该序列编码233个氨基酸,蛋白质分子量为 71.88 kDa,等电点为5.09。用ORF Finder软件对序列进行分析,LtCHS1基因的ORF结构如图 2所示。
 |
图 2 LtCHS1全长cDNA序列及推导氨基酸序列 Fig. 2 The full-length of cDNA and cDNA-derived amino acid sequences of LtCHS1 |
LtCHS2基因的cDNA全长1 457 bp,其中包括1个96 bp的5′-UTR,1个1 185 bp的ORF,1个176 bp的3′-UTR。该基因在97-99位为起始密码子ATG,下游有同框终止密码子TAA和polyA尾。该基因编码394个氨基酸,蛋白质分子量为120.0 kDa,等电点为 4.97。用ORF Finder软件对序列进行分析,LtCHS2基因的ORF结果如图 3所示。
 |
图 3 LtCHS2全长cDNA序列及推导氨基酸序列 Fig. 3 The full-length of cDNA and cDNA-derived amino acid sequences of LtCHS2 |
将北美鹅掌楸(LYS)的基因组序列PCR扩增产物在2%琼脂糖凝胶上进行电泳检测,将目的片段切下,转化大肠杆菌后送上海英骏公司测序,得到LtCHS1和LtCHS2基因的基因组序列。利用Gene Structure Display Server(GSDS)(http://gsds.cbi.pku.edu.cn/)将它们的gDNA与cDNA进行比较(郭安源等,2007)后发现: LtCHS1基因只含有1个外显子,没有内含子; 而LtCHS2基因gDNA序列长2 842 bp,含有2个外显子(外显子Ⅰ185 bp,外显子Ⅱ1 000 bp),含1个内含子,长1 559 bp。LtCHS2基因比LtCHS1基因的gDNA序列长很多。2个基因的外显子/内含子结构见图 4。
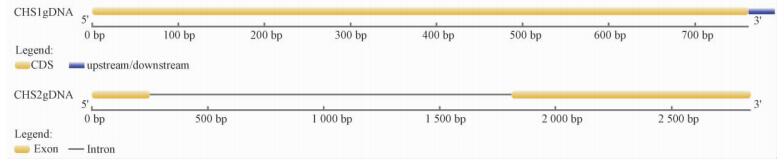 |
图 4 北美鹅掌楸CHS基因外显子/内含子结构 Fig. 4 Exon / intron structure diagram for CHS gene in L. tulipifera |
根据InterProScan在线软件分析CHS基因编码产物的结构域和功能位点,发现LtCHS2具有典型的CHS蛋白保守结构域: N-末端结构域(从Ile6到Pro230)、C-末端结构域(从Gln239到Pro389)、聚合酶Ⅲ结构域(从Met1到Thr391); 而LtCHS1只含有N-末端结构域(从Met1到Pro71)、C-末端结构域(从Gln80到Pro230),没有聚合酶Ⅲ结构域。
将LtCHS1、LtCHS2蛋白质氨基酸序列提交到ProtScale网站(http://web.expasy.org/protscale/)进行亲疏水性分析,其蛋白质亲疏水性序列谱见图 5。从图中可以看出: 这2个蛋白质同时具有疏水性和亲水性区域。LtCHS1蛋白质的疏水性氨基酸残基明显多于亲水性氨基酸残基,属于疏水性蛋白质,疏水指数为-2.244~2.689,疏水区域最高值(Score=2.689)出现在第184个氨基酸残基,亲水区域的最高值(Score=-2.244)出现在第196个氨基酸残基。而LtCHS2蛋白质的亲疏水性氨基酸残基分布较为均衡,疏水指数为-2.578~2.422,疏水区域的最高值(Score=2.422)出现在第343个氨基酸残基,亲水区域的最高值(Score=-2.578)出现在第355个氨基酸残基。
 |
图 5 北美鹅掌楸CHS基因编码蛋白质的亲疏水性序列谱 Fig. 5 Hydropathy profile of protein coded by CHS gene in L. tulipifera |
LtCHS1基因所编码蛋白质由36.05%的H(α-helix)、17.17%的E(β-sheet)和46.78%的L(coil)组成,其中有49.79%的氨基酸残基表面裸露16%以上。LtCHS1蛋白质有跨膜结构(图 6)。
 |
图 6 北美鹅掌楸CHS蛋白质的PHD跨膜结构预测结果 Fig. 6 Predicted helical transmembrane region of CHS of L. tulipifera |
LtCHS2基因所编码蛋白质由39.34%的H(α-helix)、11.93%的E(β-sheet)和48.73%的L(coil)组成,其中有44.16%的氨基酸残基表面裸露16%以上。LtCHS2蛋白质也有跨膜结构(图 6)。
2.4 LtCHS基因编码蛋白质三级结构预测蛋白质三级结构预测结果(图 7)显示: LtCHS1基因编码蛋白质以Swiss-Model Workspace上编号为1i88B的模型作为模板,CHS1蛋白质与模板有较高相似性的是1-231位氨基酸,相似性为78.45%。LtCHS2基因编码蛋白质以Swiss-Model Workspace上编号为1bq6A的模型作为模板,CHS2蛋白质与模板有较高相似性的是4-390位氨基酸,相似性达83.25%。
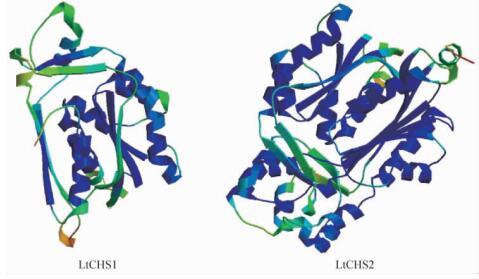 |
图 7 北美鹅掌楸CHS基因所编码蛋白质的三级结构模型 Fig. 7 Predicted tertiary structure model of protein coded by CHS in L. tulipifera |
通过半定量RT-PCR的方法对鹅掌楸属不同种及不同组织的表达情况进行分析,结果(图 8)显示: LtCHS1、LtCHS2基因在北美鹅掌楸(BM)、杂交鹅掌楸(F1)、鹅掌楸(WYS)各个组织中的表达存在差异。LtCHS1基因在各个样本、组织中表达量均不高,在北美鹅掌楸的花萼和花瓣中有少量表达,杂交鹅掌楸只有花萼中有表达,而在鹅掌楸的4个组织中表达均很微弱,几乎无法检测。LtCHS2基因在鹅掌楸、杂交鹅掌楸的各个组织中都有表达,表达量明显增加,且在花萼和花瓣的表达量都明显高于雄蕊和叶片,而在北美鹅掌楸4个组织中几乎无法检测到表达。
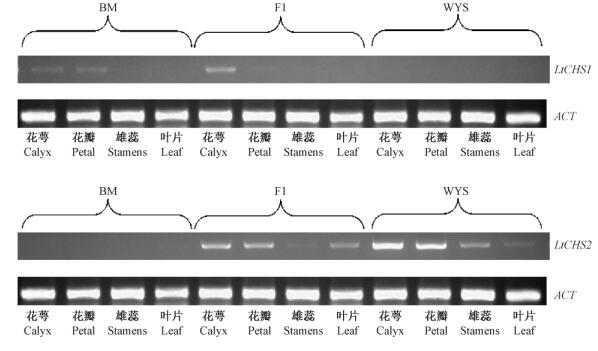 |
图 8 鹅掌楸属CHS基因半定量RT-PCR检测 Fig. 8 Detection of CHS gene in Liriodendron based on semi-quantitative RT-PCR |
自1983年获得第1个CHS基因序列(Reimold et al.,1983)以来,己从苔藓、蕨类、裸子植物和被子植物中克隆了约700个CHS基因及其相关基因的序列,并对其功能进行了研究和分类。CHS基因是一个多基因家族,其基因结构非常保守(Lanz et al.,1991),多数CHS基因包含2个外显子和1个内含子,并且内含子的位置在所有己知序列中相似。本研究采用同源克隆策略和RACE技术克隆得到2个北美鹅掌楸CHS基因全长序列。LtCHS1基因仅有1个外显子,无内含子; LtCHS2基因与大多数CHS相似,包含2个外显子和1个内含子。将2个LtCHS基因序列进行比对后发现,北美鹅掌楸LtCHS基因序列与其他物种的CHS序列相似度较高。因此可以认为,LtCHS基因属于CHS基因家族成员。至于鹅掌楸属中是否还存在其他CHS基因家族成员,还有待进一步的研究。
LtCHS基因在各组织中的半定量RT-PCR结果显示,2个基因的表达不仅在同一树种不同组织间存在差异,而且在种间也存在表达差异。在所涉及的4个组织中,LtCHS1基因只在北美鹅掌楸和杂交鹅掌楸中表达,而LtCHS2基因则在杂交鹅掌楸和鹅掌楸中表达。这可能是由于LtCHS1与LtCHS2基因虽属于同一基因家族但执行不同功能,其具体的功能差异还有待进一步的分析。
杂交鹅掌楸与鹅掌楸、北美鹅掌楸的花色存在明显差异,如图 1所示,杂交鹅掌楸的花色兼具双亲特征。查尔酮合成酶(CHS)在植物的花青素的合成、防UV照射和外源基因的表达等过程中起着重要的作用(Koes et al.,1989)。本研究发现,杂交鹅掌楸同样兼具双亲各自独有的CHS基因,表明 CHS基因可能与鹅掌楸花色形成有关。结合LtCHS基因组织差异表达结果与鹅掌楸种间花色特征,可初步推测,LtCHS1基因与LtCHS2基因可能分别调控不同类型花青素的合成,LtCHS1基因合成的花青素颜色偏红,而LtCHS2基因合成的花青素颜色偏绿。当然,该推论尚需进一步的基因功能验证。
此外,由于CHS基因非常保守,被视为研究物种进化关系的适合工具(Ursula et al.,1987; Soltis et al.,1998)。Durbin等(2000)指出,CHS非常适合用于基因家族起源和基因复制的研究。而Soltis等(1998)则大胆地推测CHS可能适合于科以下居群间的进化研究。因此,LtCHS基因也有可能作为鹅掌楸属种间进化研究的候选基因之一。
| [1] |
安利清, 杨 凯, 张 克, 等. 2011. 百合查尔酮合成酶基因的克隆与分析. 西北植物学报, 31 (3): 492-498. (An L Q, Yang K, Zhang K, et al. 2011. Cloning and analysis of chalcone synthase gene of Lily. Acta Botanica Boreali-Occidentalia Sinica, 31 (3): 492-498[in Chinese]).(  1) 1)
|
| [2] |
郭安源, 朱其慧, 陈 新, 等. 2007. GSDS:基因结构显示系统.遗传, 29(8): 1023-1026. (Guo A Y, Zhu Q H, Chen X, et al. 2007. GSDS: a gene structure display server. Hereditas, 29(8):1023-1026[in Chinese]).(  1) 1)
|
| [3] |
何水林, 郑金贵, 林 明, 等. 2004. 植物芪类次生代谢物的功能、合成调控及基因工程研究进展. 农业生物技术学报,12 (1): 102-108. (He S L, Zheng J G, Lin M, et al. 2004. Advances of biological function, regulatory mechanism of biosynthesis and genetic engineering of stilbenes in plant. Journal of Agricultural Biotechnology, 12 (1): 102-108[in Chinese]).(  1) 1)
|
| [4] |
蒋明, 曹家树. 2007. 查尔酮合成酶基因.细胞生物学杂志, 29(4): 525-529. (Jiang M, Cao J S. 2007. Chalcone synthase gene. Chinese Journal of Cell Biology, 29(4): 525-529[in Chinese]).(  1) 1)
|
| [5] |
李琳玲, 程 华, 程水源, 等. 2010. 银杏查尔酮合成酶基因启动子(GbCHSP)调控元件及功能分析. 园艺学报, 37 (12): 1919-1928. (Li L L, Chen H, Cheng S Y, et al. 2010. Regulatory elements and functional analysis of chalcone synthase gene promoter from Ginkgo biloba L. Acta Horticulturae Sinica, 37 (12): 1919-1928[in Chinese]).(  1) 1)
|
| [6] |
单丽伟, 汪 勇, 王美玲, 等. 2012. 大豆类黄酮生物合成关键酶CHS基因的克隆及表达分析. 西北植物学报, 32 (11): 2164-2168. (Shan L W, Wang Y, Wang M L, et al. 2012. Cloning and expression analysis of CHS genes involved in the biosynthesis of flavonoids in Soybean. Acta Botanica Boreali-Occidentalia Sinica, 32 (11): 2164-2168[in Chinese]).(  1) 1)
|
| [7] |
王曼玲, 董 臣, 朱虹琳, 等. 2006. 莲查尔酮合酶基因的克隆及序列分析. 分子植物育种, 4 (3): 30-35. (Wang M L, Dong C, Zhu H L, et al. 2006. Molecular cloning and analysis of chalcone synthase gene in Nelumbo nucifera (Lotus). Molecular Plant Breeding, 4 (3): 30-35[in Chinese]).(  1) 1)
|
| [8] |
周军, 陶建敏, 彭日荷, 等. 2010. 巨峰葡萄查尔酮合成酶基因4的克隆及表达特性的RT-PCR分析. 南京农业大学学报, 33(2):39-44. (Zhou J, Tao J M, Peng R H, et al. 2010. Cloning and expression analysis of CHS 4 of Kyoho grape by RT-PCR. Journal of Nanjing Agricultural University, 33(2):39-44[in Chinese]).(  1) 1)
|
| [9] |
Arnold K, Bordoli L, Kopp J, et al. 2006. The SWISS-MODEL Workspace: a web-based environment for protein structure homology modelling. Bioinformatics, 22(2):195-201.( 1) 1)
|
| [10] |
Austin M B, Noel J P. 2003.The chalcone synthase superfamily of type III polyketide synthases. Natural Product Reports, 20(1): 79-110.( 1) 1)
|
| [11] |
Durbin M L, Learn G H, Huttley G A, et al. 1995. Evolution of the chalcone synthase gene family in the genus Ipomoea. Proceedings of the National Academy of Sciences of the United States of America, 92(8): 3338-3342.( 1) 1)
|
| [12] |
Durbin M L, McCaig B, Clegg M T. 2000. Molecular evolution of the chalcone synthase multigene family in the morning glory genome. Plant Mol Biol, 42(1): 79-92.( 2) 2)
|
| [13] |
Elomaa P, Honkanen J, Puska R, et al. 1993. Agrobacterium-mediated transfer of antisense chalcone synthase cDNA to gerbery hybrida inhibits flower pigmentation. Biotechnology, 11(4): 508-511.( 1) 1)
|
| [14] |
Franken P, Niesbach K U, Weydemann U, et al. 1991. The duplicated chalcone synthase genes C 2 and Whp (white pollen) of Zea mays are independently regulated; evidence for translational control of Whp expression by the anthocyanin intensifying gene in. EMBO J, 10(9):2605-2612.( 1) 1)
|
| [15] |
Guex N, Peitsch M C.1997. Swiss-Model and the Swiss-PdbViewer: An environment for comparative protein modelling. Electrophoresis, 18: 2714-2723.( 1) 1)
|
| [16] |
Harlow W M, Harrar E S. 1994. Textbook of dendrology. New York, the USA: McGrau-Hill Book Company, 397-400.( 1) 1)
|
| [17] |
Huang J X, Qu L J, Gu H Y. 2004. A preliminary study on the origin and evolution of chalcone synthase (CHS) gene in angiosperms. Acta Botanic Sinica, 46 (1): 10-19.( 1) 1)
|
| [18] |
Johzuka-Hisatomi Y, Hoshino A, Mori A, et al. 1999. Characterization of the chalcone synthase genes expressed in flowers of the common and Japanese morning glories. Genes Genet Syst, 74(4): 141-147.( 1) 1)
|
| [19] |
Kiefer F, Arnold K, Künzli M, et al. 2009. The SWISS-MODEL repository and associated resources. Nucleic Acids Research, 37: D387-D392.( 1) 1)
|
| [20] |
Koes R E, Spelt C E, van den Elzen P J, et al. 1989. Cloning and molecular characterizaton of chalcone synthase multigene family of Petunia hybrida. Gene, 81(2):245-257.( 1) 1)
|
| [21] |
Kreuzaler F, Ragg H, Fautz E, et al. 1983. UV-induction of chalcone synthase mRNA in cell suspension culture of Petroselinum hortense. Proc Nat Acad Sci USA, 80(9): 2591-2593.( 1) 1)
|
| [22] |
Lamb C J, Lawton M A, Dron M, et al. 1989. Signal and construction mechanisms for activation of plant defenses against microbial attack. Cell, 56(2): 215-224.( 1) 1)
|
| [23] |
Lanz T, Tropf S, Marner F J, et al. 1991. The role of cysteines in polyketide synthase site directed mutagenesis of resveratrol and chalcone synthase, two enzymes in different plant-specific pathways. J Biol Chem, 266: 9971-9976.( 1) 1)
|
| [24] |
Martin C R. 1993. Structure, function, and regulation of the chalcone synthase (CHS). Interation Review of Cytology, 147: 233-284.( 1) 1)
|
| [25] |
Reimold U, Kroeger M, Kreuzaler F, et al. 1983.Coding and 3' non-coding nucleotide sequence of chalcone synthase messenger RNA and assignment of amino acid sequence of the enzyme. EMBOJ, 2(10): 1801-1805.( 1) 1)
|
| [26] |
Schwede T, Kopp J, Guex N, et al. 2003.SWISS-MODEL: an automated protein homology-modeling server. Nucleic Acids Research, 31: 3381-3385.( 1) 1)
|
| [27] |
Soltis D E, Soltis P S. 1998. Choosing an approach and an appropriate gene for phylogenetic analysis//Sotis D E. Molecular systematics of plants II. DNA sequencing. Alphen aan den Rijn, Holland: Kluwer Academic Publishers, 1-42.( 1) 1)
|
| [28] |
Ursula N K, Barzen E B. 1987. Chalcone synthase gene in plants: a tool to study evolutionary relationships. J Mol Evol, 26(3): 213-225.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51
