文章信息
- 胡慧, 周明兵, 杨萍, 汤定钦
- Hu Hui, Zhou Mingbing, Yang Ping, Tang Dingqin
- 毛竹微型颠倒重复序列转座子PhTourist1的克隆与分析
- Cloning and Analysis of Miniature Inverted Repeat Transposable Elements PhTourist 1 from Phyllostachys edulis
- 林业科学, 2015, 51(5): 127-134
- Scientia Silvae Sinicae, 2015, 51(5): 127-134.
- DOI: 10.11707/j.1001-7488.20150515
-
文章历史
- 收稿日期:2014-09-01
- 修回日期:2015-01-19
-
作者相关文章
转座子(transposable elements,TEs)是广泛存在于真核生物中的一类可以移动的DNA片段(Feschotte et al.,2002),最早由McClintock在玉米(Zea mays)中发现(McClintock,1951)。转座子根据转座机制可分为Ⅰ、Ⅱ2类,类型Ⅰ转座子又叫反转录转座子(retro-transposons),以RNA为中间媒介,遵循“复制-粘贴”的转座方式;类型Ⅱ转座子又叫DNA转座子(DNA transposons),通过DNA中间体进行转座,遵循“剪切-粘贴”的转座方式(Shirasawa et al.,2012)。根据转座自主性又可把DNA转座子分为自主转座子(autonomous elements)、非自主转座子(non-autonomous elements)和微型颠倒重复序列(miniature inverted repeat transposable elements,MITEs)3类。自主转座子自身可编码转座酶等产物,催化自身发生转座;非自主转座子是自主转座子发生部分缺失的产物,自身不具备编码转座酶的功能,在相应自主转座子提供转座酶的情况下可以发生转座(Shirasawa et al.,2012)。
MITEs是一类类似非自主转座子但比非自主转座子短的转座子,长度为100~800 bp,不编码转座酶,有短末端反向重复序列(short terminal inverted repeat,sTIR)和靶位点重复序列(target site duplication,TSD),高A/T含量,多拷贝(1 000~15 000),易形成二级结构,偏好插在基因内部或基因附近(Kidwell et al.,1997)。根据其TIR和TSD的不同分为不同家族,在植物中存在最多的是Stowaway-like MITE(2 bp TSD,TA)和Tourist-like MITE(3 bp TSD,TWA)两大家族。Stowaway-like MITE与Tc 1 /mariner 超家族转座子在TIR和TSD上的同源性很高,Stowaway-like MITE在Tc 1 /mariner超家族自主转座子编码的转座酶催化下可以发生转座(Feschotte et al.,2000);Tourist-like MITE与PIF/Harbinger超家族在TIR和TSD上的同源性很高,Tourist-like MITE在PIF/Harbinger超家族自主转座子编码的转座酶催化下可以发生转座(Yang et al.,2007)。
第1个被证明有活性的MITE是mPing,mPing来源于水稻(Oryza sativa)基因组(Jiang et al.,2003;Kikuchi et al.,2003;Nakazaki et al.,2003),属于Tourist-like MITE,其长度为430 bp,有15 bp的TIR(GGCCAGTCACAATGG)、3 bp的TSD(TTA或TAA)。mPing偏好插在基因附近或基因内部(Naito et al.,2009),在水稻和拟南芥(Arabidopsis thaliana)中的插入偏好序列为YTMTWAKAR(Yang et al.,2007;Naito et al.,2006)。mPing在基因组中的跳跃还具有2个特性: 1)很少留下转座足迹,例如在酵母基因组转座时,99%的切除为精确切除(Hancock et al.,2010),在拟南芥基因组转座时,82%的切除为精确切除(Yang et al.,2007);2)偏好插入基因附近且不会插在自己原来所处位置附近(Hancock et al.,2011)。这些特性使mPing在用作基因标签方面优于之前常见的Ac/Ds系统和Tn 1 ,已成功应用于大豆(Glycine max)、水稻基因组功能基因的鉴定与分析(Hancock et al.,2011)。
毛竹(Phyllostachys edulis)为散生竹,是我国经济价值最高的竹种,占全国竹林面积的2/3以上(江泽慧,2002),且变异丰富(钟浩等,2010)。毛竹为典型的二倍体,其基因组草图由国际竹藤中心、中国林业科学研究院林业研究所等机构学者于2013年2月公布(Peng et al.,2013)。
本研究根据已知的mPing的特点在毛竹基因组中寻找与mPing同源的Tourist-like MITE,系统分析了该类型Tourist-like MITE的结构特点及在基因组中的插入特性,研究它们在毛竹生长发育过程中的转座情况,及对宿主基因表达丰度的影响。
1 材料与方法 1.1 材料与试剂所用的24株毛竹实生苗由广西灵川同一株开花毛竹的种子繁育,于温室中培养,依次编号为1,2,3,…,24。取生长3个月的新鲜叶片用作试验。
所用克隆受体菌为大肠杆菌(Escherichia coli)DH5α,为本实验室保存。 DNeasy Plant Mini Kit和RNeasy Mini Kit购自QIAGEN,克隆载体pMD18-T,Ex Taq Polymerase,Prime ScriptTM RT Master Mix和SYBR® Premix Ex Taq Ⅱ(Tli RNaseH Plus)均购自Takara。
1.2 试验方法1)毛竹基因组中mPing-like MITE的鉴定和结构特征分析 根据水稻mPing的TSD和TIR保守序列(TA/TggccagtcacaatggA)(Jiang et al.,2003),利用transpo软件(Santiago et al.,2002),在TIR序列上允许错配碱基为2个,检索目标序列长度设置为100~800 bp,其他为默认参数,在毛竹基因组中检索与mPing在TIR,TSD序列上同源性高的MITE。
利用MEGA5.1中MUSCLE程序中的neighbor joining算法分析得到的mPing-like MITE序列之间的同源性,将其中碱基完全一致的序列分为一类,共17类,每类选出1条共17条和mPing一起用DNAStar软件进行同源比对,同时得到mPing-like MITE的一致序列。
根据得到的mPing-like MITE在毛竹scafold中的位置提取这些mPing-like MITE的两侧侧翼序列,利用MEGA5.1中MUSCLE程序中的neighbor joining算法分析其TSD同源性及插入偏好性,用WebLogo 3-Create在线软件(http://weblogo.threeplusone.com/create.cgi)作图。
2)mPing-like MITE的插入时间分析 在1)中获得了mPing-like MITE的一致序列,用Clustal W对每个序列和一致序列进行比对,然后利用Kimura2参数模型计算每个成员与一致序列之间的核苷酸替换情况(k),依据公式T=k/(2r)计算每个mPing-like MITE的插入时间,式中,r为核苷酸每年每个位点的替换率(1.56×10-8,Han et al.,2010)。
3)mPing-like MITE转座活性分析 根据mPing-like MITE的侧翼序列分别设计引物,以验证这些mPing-like MITE在基因组中的位置(引物信息见表 1),反应体系如下: 0.2 μL Ex Taq Polymerase(5 U ·μL-1),0.8 μL primer-5(10 μmol ·L-1),0.8 μL primer-3(10 μmol ·L-1),2 μL 10×Ex Taq Buffer,1.6 μL dNTP mix(2.5 mmol ·L-1),100 ng DNA,加无菌水补齐20 μL。反应条件为: 预变性94 ℃ 5 min;变性94 ℃ 30 s,退火50 ℃ 30 s(根据引物选择合适的退火温度),延伸72 ℃ 30 s,35个循环;72 ℃ 2 min,4 ℃ 10 min。
|
|
PCR产物琼脂糖电泳,割胶回收,连接到pMD18-T载体上送测序。
4)mPing-like MITE下游基因表达丰度分析 选取基因组中同一个位点2条染色体上皆未发生缺失的6,11,15号植株和2条染色体上皆发生缺失的8,13,20号植株的叶片为材料,分别提取其RNA,反转录成cDNA,通过荧光定量PCR(real-time fluorescence quantitative PCR,RTFQ PCR)分析其下游基因的表达丰度(反应体系及程序依据说明书),引物信息见表 1。
2 结果与分析 2.1 mPing-like MITE的鉴定通过transpo得到30个mPing-like MITEs,同源比对发现其相似度高达99%,可以确定它们来自于同一个祖先,命名为PhTourist1,每一条分别命名为PhTourist1-#(#:1-30)。
将序列完全一致的PhTourist1分为一类,共17类,每一类选出1条,17条序列与mPing同源比对的结果显示PhTourist1拥有共同的序列为GGCCAGTCTCAATG 的14 bp的TIR,比mPing的TIR(GGCCAGTCACAATGG)少1 bp,且内部有1个碱基的不同,PhTourist1为T,mPing为A。17条序列与mPing相比还存在3处明显的片段缺失,其余位置的比对发现PhTourist 1 与mPing在5′端的同源性高于3′端,平均相似度在68%~69%之间(图 1)。
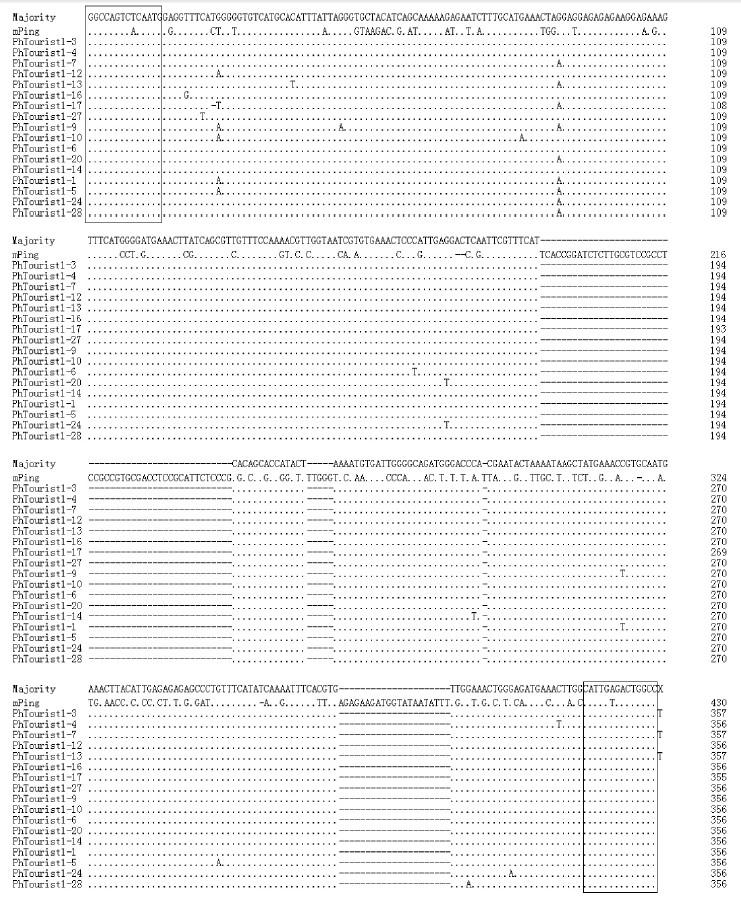 |
图 1 PhTourist1与mPing序列同源比对结果 Fig.1 Sequence homologous alignment result between PhTourist1 and mPing 黑色框标记的为TIR。TIR is boxed in black. |
经MEGA5.1分析得到的结果表明PhTourist1的TSD为TWA,9 bp的插入偏好序列比较明显,为(C/T)T(C/A)T(T/A)A(G/T)A(A/C)(图 2)。
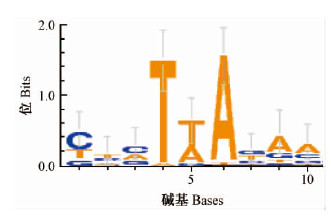 |
图 2 PhTourist1插入偏好序列 Fig.2 Preference insertion sequence of PhTourist1 |
插入时间的计算结果(表 2)显示PhTourist1的插入时间比较集中,14个在19万年前插入,10个在10万年前插入,2个插入发生在将近26万年前,4个在最近发生插入。
|
|
已有报道表明,mPing倾向于插入基因附近(Natio et al.,2009)。分析30个 PhTourist1在毛竹基因组scafold上与基因的距离,除有6个PhTourist1所在的毛竹基因组scafold太短,没法获得附近基因的信息外,剩余的24个PhTourist1中17% PhTourist1位于距离基因上下游2 kb范围内,21%位于距离基因上下游4 kb范围内,8%位于基因内含子中(表 3)。
|
|
针对24株毛竹实生苗中的30个PhTourist1的位点进行PCR验证并测序,得到的结果显示各位点的PhTourist1及其侧翼序列同已报导的毛竹基因组序列一致。但30个位点中有29个位点在24株毛竹实生苗基因组中皆表现为插入,无多态性。只有PhTourist1-3呈现插入多态性:24株实生苗中12株表现为 PhTourist1-3在1对同源染色体上都缺失,所占比例为50%;6株表现为1条染色体上PhTourist1-3保留,其同源染色体上等位位点处PhTourist1-3缺失,所占比例为25%;剩余6株表现为PhTourist1-3在1对同源染色体都保留,所占比例为25%(图 3)。分析PhTourist 1-3 的转座足迹,发现PhTourist 1-3 的切除接近精确切除,除自身完全切除外还缺失1 bp TSD(图 4)。
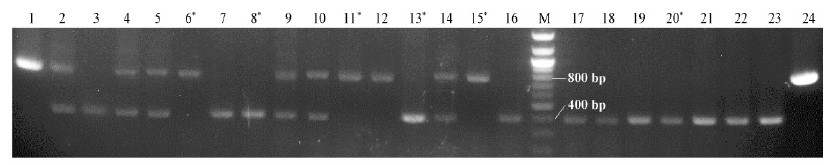 |
图 3 24株毛竹半同胞实生苗中PhTourist1-3 PCR验证结果
Fig.3 PCR result of PhTourist 1-3 in genomes of 24 Phyllostachys edulis half-sib seedlings
PCR产物长度少于400 bp的表示PhTourist 1-3 发生转座,长度在800 bp左右的表示PhTourist 1-3 留在该位置,“*”标识的为做RTFQ PCR的植株。
The PCR products less than 400 bp reveal the transposition of PhTourist 1-3 . The PCR products about 800 bp reveal that PhTourist 1-3 still existed at this genomic location. The seedlings whose cDNA used in RTFQ PCR are marked by the asterisk. |
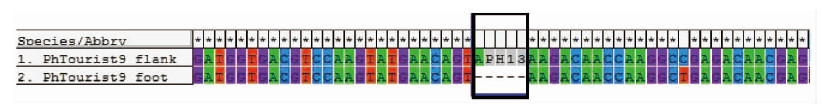 |
图 4 PhTourist1-3转座足迹 Fig.4 Transposition footprint of PhTourist1-3 黑色框标出的为PhTourist1-3的位置。Black box marked the location of PhTourist 1-3. |
PhTourist1-3插入位点位于PH01000402G0860基因上游1 585 bp处,针对PhTourist1-3插入位点多态性,分析了PhTourist1-3存在与否对下游基因表达丰度的影响。RTFQ PCR的结果显示,PhTourist1-3缺失之后下游基因表达量相对缺失前平均增加了8.04倍(图 5)。
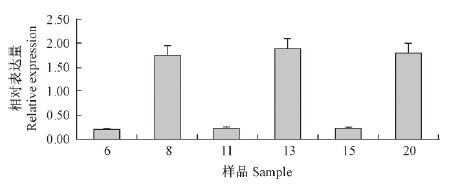 |
图 5 PhTourist1-3下游基因RTFQ PCR结果 Fig.5 Result of RTFQ PCR of the gene downstream PhTourist1-3 |
本研究利用transpo软件共寻找到30个mPing-like MITEs,同源比对之后确定它们源自于共同祖先,统一命名为PhTourist1,对其TIR、TSD、插入位点序列偏好性、插入时间和分布进行了系统分析。
PhTourist1的TIR与mPing的TIR(GGCCAGTC ACAATGG)基本一致,为GGCCAGTCACAATG,TIR的G/C含量为57.1%;9 bp的偏好序列为YTMTWAKAR;发生转座的PhTourist1-3位点上,转座子除自身完整切除,还缺失1 bp TSD。这些都表明PhTourist1 具备了Tourist-like MITE的典型特点。
验证30个PhTourist1在来源于同一母本的24株毛竹实生苗基因组中的插入位点,发现插入多态性很低,只有PhTourist1-3存在插入多态性,在75%实生苗中PhTourist1-3发生了转座,从而造成了该位点在24株实生苗基因组中的插入多态性。已有报道表明,mPing在外源压力作用下会发生转座,如辐射、高静水压、细胞培养和近期驯化(Jiang et al.,2003;Nakazaki et al.,2003;Naito et al.,2006;Lin et al.,2006);而本试验中的24株实生苗是于相同的适宜条件下培养的,其生长过程中受到的外源压力具有一致性,因此PhTourist1-3的转座可能不是由于外源压力作用引起的。有报道显示mPing的转座发生在胚胎发育阶段,从球状体时期到子叶期转座事件逐渐增多,但是到苗期转座事件变得很少(Hancock et al.,2011);考虑到每株实生苗的胚胎发育都是独立的,PhTourist1-3可能在种子萌发过程中发生了转座,从而导致PhTourist1-3的插入多态性。本试验中24株实生苗源自同一母本,母本基因型一致,但因为花药来源不能确定而无法确定其父本基因型;有文章报道种间杂交会促使mPing发生转座(Yasuda et al.,2012),分析PhTourist1-3的转座也可能是由于父本来源不同引起。
RTFQ PCR还表明PhTourist1-3的缺失对其下游基因PH01000402G0860表达量有显著影响,PH01000402G0860的表达量在PhTourist1-3缺失后比缺失前增加了8.04倍。水稻中关于mPing的插入影响试验显示,mPing插在基因附近或内部会造成该基因转录增多或者对该基因没有影响(Naito et al.,2009),但mPing在基因上游启动子区域的插入会影响顺式作用元件和反式作用元件间的相互作用(Yasuda et al.,2013)。本试验中PhTourist1-3的转座使下游基因表达量增加,可能原因是PhTourist1-3的存在使其附近的作用元件作用方式发生了变化从而抑制了PH01000402G0860的表达,PhTourist1-3转座离开后,这种抑制效果消失了,从而PH01000402G0860的表达量明显增加。PhTourist1-3转座对下游基因表达的调控机制有待进一步研究。
本研究鉴定到一类与mPing高度同源的转座子PhTourist1,其在毛竹基因组中可以独立发生转座,并对宿主基因表达产生了显著的影响。这些结果表明,作为毛竹基因组重要组成部分的转座子可以通过自身转座参与宿主基因组多态性的形成,调节宿主基因表达,进而参与毛竹生长发育的调控。
| [1] |
江泽慧.2002.世界竹藤.沈阳:辽宁科学技术出版社,3-4. (Jiang Z H.2002. Bamboo and rattan in the world. Shenyang: Liaoning Science and Technology Publishing House, 3-4.[in Chinese])(  1) 1)
|
| [2] |
钟浩, 周明兵, 白友煌, 等. 2010.毛竹Stowaway-like MITEs转座子的分离与分析. 林业科学, 46(4):37-42. (Zhong H, Zhou M B, Bai Y H, et al. 2010. Isolation and analysis of Stowaway-like MITEs from DNA libraries enriched in Phyllostachys edulis. Scientia Silvae Sinicae, 46(4):37-42[in Chinese]).(  1) 1)
|
| [3] |
Feschotte C, Mouches C. 2000. Evidence that a family of miniature inverted-repeat transposable elements (MITEs) from the Arabidopsis thaliana genome has arisen from a pogo-like DNA transposon. Mol Biol Evol, 17(5):730-737.( 1) 1)
|
| [4] |
Feschotte C, Jiang N, Wessler S R. 2002. Plant transposable elements: where genetics meets genomics. Nat Rev Genet, 3(5):329-341.( 1) 1)
|
| [5] |
Han M J, Shen Y H, Gao Y H, et al. 2010. Burst expansion, distribution and diversification of MITEs in the silkworm genome. BMC Genomics, 11:520.( 1) 1)
|
| [6] |
Hancock C N, Zhang F, Wessler S R. 2010. Transposition of the Tourist-MITE mPing in yeast: An assay that retains key features of catalysis by the Class 2 PIF/Harbinger super-family. Mobile DNA, 1(1): 5.( 1) 1)
|
| [7] |
Hancock C N, Zhang F, Floyd K, et al. 2011. The rice (Oryza sativa) MITE mPing is an effective insertional mutagen in soybean (Glycine max). Plant Physiology Preview, 10(1104):111.( 3) 3)
|
| [8] |
Jiang N, Bao Z R, Zhang X Y, et al. 2003. An active DNA transposon family in rice. Nature, 421(6919): 163-167.( 3) 3)
|
| [9] |
Kidwell M G, Lisch D. 1997. Transposable elements as sources of variation in animals and plants. Proceedings of the National Academy of Sciences of the United States of America, 94(15):7704-7711.( 1) 1)
|
| [10] |
Kikuchi K, Terauchi K, Wada M, et al. 2003. The plant MITE mPing is mobilized in anther culture. Nature, 421(6919):167-170.( 1) 1)
|
| [11] |
Lin X, Long L, Shan X, et al. 2006. In plant a mobilization of mPing and its putative autonomous element Pong in rice by hydrostatic pressurization. Journal of Experimental Botany, 57(10):2313-2323.( 1) 1)
|
| [12] |
McClintock B. 1951. Chromosome organization and gene expression. Cold Spring Harbor Symp, 16:13.( 1) 1)
|
| [13] |
Naito K, Cho E, Yang G J, et al. 2006. Dramatic amplification of a rice transposable element during recent domestication. Proceedings of the National Academy of Sciences of the United States of America, 103(47):17620-17625.( 2) 2)
|
| [14] |
Naito K, Zhang F, Tsukiyama T, et al. 2009. Unexpected consequences of a sudden and massive transposon amplification on rice gene expression. Nature, 461(7267):1130-1134.( 3) 3)
|
| [15] |
Nakazaki T, Okumoto Y, Horibata A, et al. 2003. Mobilization of a transposon in the rice genome. Nature, 421(6919): 170-172.( 2) 2)
|
| [16] |
Peng Z H, Lu Y, Li L B, et al. 2013. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla). Nature Genetics, 45(4):456-461.( 1) 1)
|
| [17] |
Santiago N, Herraiz C, Goñi J R, et al. 2002. Genome-wide analysis of the emigrant family of MITEs of Arabidopsis thaliana. Journal of Molecular Biology and Evolution, 19(12):2285-2293.( 1) 1)
|
| [18] |
Shirasawa K, Hirakawa H, Tabata S, et al. 2012. Characterization of active miniature inverted-repeat transposable elements in the peanut genome. Theor Appl Genet, 124(8):1429-1438.( 2) 2)
|
| [19] |
Yang G J, Zhang F, Hancock C N, et al. 2007.Transposition of the rice miniature inverted repeat transposable element mPing in Arabidopsis thaliana. Proceedings of the National Academy of Sciences of the United States of America, 104(26):10962-10967.( 3) 3)
|
| [20] |
Yasuda K, Tsukiyama T, Karki S, et al. 2012. Mobilization of the active transposon mPing in interspecific hybrid rice between Oryza sativa and O. glaberrima. Euphytica, 192(1):17-24.( 1) 1)
|
| [21] |
Yasuda K, Ito M, Sugita T, et al. 2013. Utilization of transposable element mPing as a novel genetic tool for modification of the stress response in rice. Mol Breeding, 32(3):505-516.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51

