文章信息
- 贾会霞, 姬慧娟, 胡建军, 卢孟柱
- Jia Huixia, Ji Huijuan, Hu Jianjun, Lu Mengzhu
- 杨树新品种的SSR指纹图谱构建和倍性检测
- Fingerprints of SSR Markers and Ploidy Detection for New Populus Varieties
- 林业科学, 2015, 51(2): 69-79
- Scientia Silvae Sinicae, 2015, 51(2): 69-79.
- DOI: 10.11707/j.1001-7488.20150209
-
文章历史
- 收稿日期:2014-02-26
- 修回日期:2014-04-22
-
作者相关文章
2. 南京林业大学 林木遗传与生物技术省部共建教育部重点实验室 南京 210037
2. Key Laboratory of Forest Genetics & Biotechnology of Ministry of Education Nanjing Forestry University Nanjing 210037
杨树(Populus)物种丰富,栽培和利用历史悠久,是重要的绿化和防护树种,也是重要的能源、纸浆、胶合板等用材树种(胡建军等,2010)。近几十年来,我国的杨树育种工作发展迅速,培育出大量速生优良新品种;但是,由于杨树的生物学特性易受生长发育周期的限制和外界环境的干扰,且新品种遗传基础狭窄,品种之间形态学差异越来越小,给杨树新品种的鉴定和知识产权保护增加了困难。所以,如何对杨树新品种进行准确、快速的区分已成为当务之急。此外,在杨树杂交育种过程中,通常采用套袋隔离授粉,子代系谱关系比较清楚,但不排除杂交繁育过程中由于授粉组合多、育种工作量大使操作人员出现失误造成花粉污染或者材料混淆的可能,在申请和鉴定新品种时,建议提供新品种的亲本信息,并鉴定其系谱关系,以便更准确地鉴别和保护新品种。
随着分子生物学快速发展,DNA指纹图谱作为一种遗传种质分析的新方法,可以直接反映植物遗传物质在DNA分子水平上的差异,具有高效、准确、经济、便捷、不受季节和环境影响等优点(王忠华,2006; Guichoux et al., 2011),可有效解决杨树品种鉴定中的困难。构建DNA指纹图谱的分子标记方法有多种,其中简单重复序列(simple sequence repeat,SSR)标记具有多态性高、重复性好、共显性、多等位点基因变异等优点,为杨树新品种鉴定、系谱分析和遗传多样性分析等提供了有利的工具(Powell et al., 1996; 黄秦军等,2002; Guichoux et al., 2011)。Dayanandan等(1998)利用基因组文库鉴定分离了美洲山杨(P. tremuloides)的SSR位点,其中4个SSR位点在36个美洲山杨中共检测到29个等位基因,该研究是SSR标记在杨树中的首次利用。2006年,毛果杨(P. trichocarpa)全基因组测序工作顺利完成,大量的杨树SSR引物信息公布于国际杨树基因组委员会的官方网站上,解决了SSR标记的关键技术。随后,SSR标记在杨树的品种鉴定、杂交子代鉴定、指纹图谱构建和遗传多样性分析等方面得到了广泛应用(王辉等,2008; 卫尊征等,2008; 张亚东等,2009; 刘春英等,2013);冯锦霞等(2011)利用SSR标记鉴定杨树转基因与非转基因无性系的SSR位点差异,证实SSR标记也可用于鉴别基因工程手段获得的品种差异。TP-M13-SSR(simple sequence repeat with tailed primer M13)毛细管电泳自动检测法是基于自动荧光测序技术的SSR检测体系(Schuelke,2000),该技术只需1条荧光标记的M13通用引物,极大降低了检测成本,同时,相比于常规的聚丙烯酰胺凝胶电泳检测技术,毛细管电泳自动检测具有操作简便、高通量、高分辨率和自动统计数据等优点。
流式细胞仪(flow cytometry,FCM)是20世纪70年代发展起来的一种集光电测量技术、激光技术、计算机技术和流体力学理论为一体的新型细胞检测技术,可以快速测定细胞中的DNA含量并进行倍性分析(Loureiro et al., 2005; Doležel et al., 2007),近年来,在中华猕猴桃(Actinidia chinensis)(朱道圩等,2007)、杜仲(Eucommia ulmoides)(张海凤等,2008)、柑橘(Citrus)(Aleza et al., 2010)、咖啡(Coffea arabica)(Clarindo et al., 2012)等多种植物中得到广泛的应用。
本研究采用TP-M13-SSR毛细管电泳自动检测法对24份杨树种质进行指纹图谱构建和遗传多样性分析,并利用FCM进行倍性检测,为杨树新品种鉴定和知识产权保护提供科学的理论依据,同时为杨树的育种工作奠定坚实的基础。
1 材料与方法 1.1 材料供试的24份杨树种质(表 1)于2012年7月采自中国林业科学研究院苗圃,每份种质均随机选取2个单株,取幼嫩的叶片放入自封袋中,用冰盒带回实验室,于-80 ℃冰箱中备用。
|
|
采用CTAB法(Allen et al.,2006)提取幼嫩叶片中的DNA,0.8%琼脂糖凝胶电泳检测DNA的完整性,紫外分光光度计测定样品DNA的浓度和纯度,将浓度稀释至50 ng·μL-1,置于-20 ℃冰箱中备用。
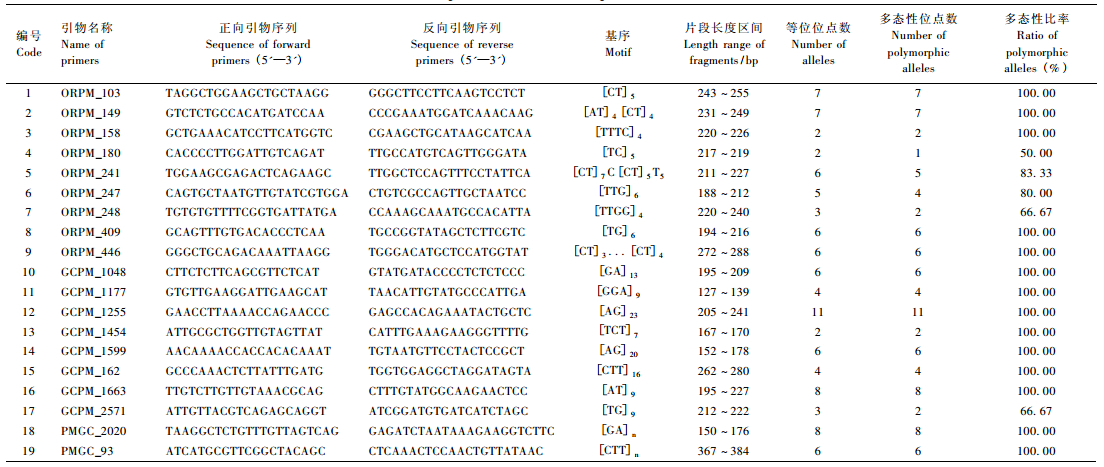
1.3 SSR引物的筛选从国际杨树基因组委员会官方网站(http://web.ornl.gov/sci/ipgc/ssr_resources.htm)的杨树SSR数据库中选取200对SSR引物,对遗传背景差异大的4份杨树材料进行引物筛选,选出19对扩增条带清晰、具有多态性且重复性好的引物对24份供试材料进行SSR-PCR扩增。
1.4 PCR扩增和GeXP毛细管电泳检测采用TP-M13-SSR毛细管电泳荧光检测法进行PCR扩增和产物检测。设计3条引物[分别为5′端带有Cy5荧光标记的M13引物(5′-TGTAAAACGACGGCCAGT-3′)、5′端接有M13序列的SSR正向引物(F+M13引物)和SSR反向引物(R引物)]进行PCR扩增。PCR反应体系:10×PCR buffer 2 μL,dNTP 1.6 μL,MgCl2 1.2 μL,rTaq 0.2 μL,F+M13引物0.4 μL,R引物和M13引物各1.6 μL,水11.4 μL,共20 μL。PCR反应程序为:94 ℃ 5 min→(94 ℃ 30 s,57 ℃ 45 s,72 ℃ 45 s),30个循环→(94 ℃ 30 s,53 ℃ 45 s,72 ℃ 45 s),8个循环→72 ℃ 10 min→4 ℃保存。采用GenomeLabTM GeXP遗传分析系统检测带有荧光标记的SSR扩增产物,检测依照贾会霞等(2013)中的毛细管电泳法进行。
1.5 倍性测试利用FCM对24份杨树种质进行倍性测试。取供试种质刚展开的嫩叶用刀片在滴有裂解液的培养皿中切碎,用300目尼龙网过滤,将滤液放入离心机,1 500 r·min-1离心5 min,倒掉上清液,加入200 μL碘化丙啶(propidium iodide,PI)染液,置于4 ℃冰箱染色30 min,利用美国BD公司的流式细胞仪FACSCalibur进行倍性鉴定。
1.6 数据分析本研究进行了2次生物学重复和2次技术重复,以确保试验的准确性和可靠性。GeXP系统中的Fragments分析软件可以自动处理收集的原始数据,根据分子量内标得出片段大小。按Nei等(1979)方法计算品种间的相似系数(genetic similarity,GS)和遗传距离(genetic distance,GD):GS=2Nij/(Ni+Nj),GD=1-GS;式中: Ni和Nj为i和j品种的谱带数,Nij为i和j品种共有的谱带数。利用NTSYS-pc 2.10e软件对各供试材料采用UPGMA(unweighted pair group method arithmetic averages)法进行聚类分析。
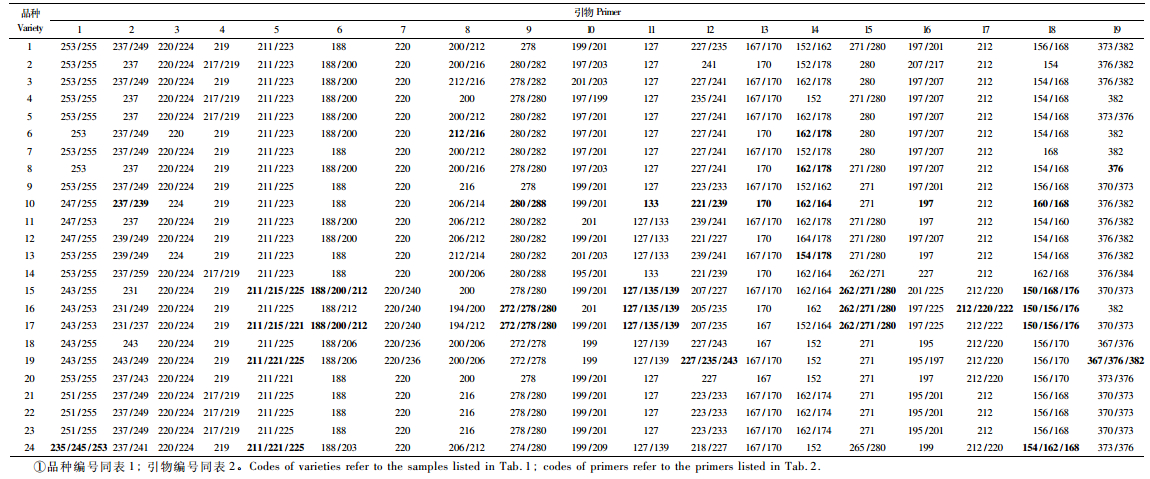
2 结果与分析 2.1 SSR引物的多态性分析和指纹图谱构建利用筛选出的19对SSR引物对24份杨树种质的基因组DNA进行扩增,共检测到102个片段,其中多态性片段97个,占95.10%,每个位点的等位基因数为2~11个,平均每对引物为5.37个。其中引物ORPM_158,ORPM_180和GCPM_1454的等位基因数最少(2个),引物GCPM_1255的等位基因数最多(11个)。19对SSR引物的扩增信息见表 2,构建的指纹图谱见表 3,图 1是引物ORPM_247在部分(10份)杨树种质中的扩增图谱。中林2025杨是以I-69杨为母本、以意大利罗马农林研究中心提供的美洲黑杨为父本,经过杂交选育得到的优良无性系;中红杨是中林2025杨的芽变品种;全红杨是在中红杨的基础上进一步选育而来。19对SSR引物均检测不到中林2025杨、中红杨和全红杨的条带差异。除了这3个品种外,3对高效的引物ORPM_103,ORPM_180和GCPM_1255可以将21份杨树种质完全区分开。
|
|
|
|
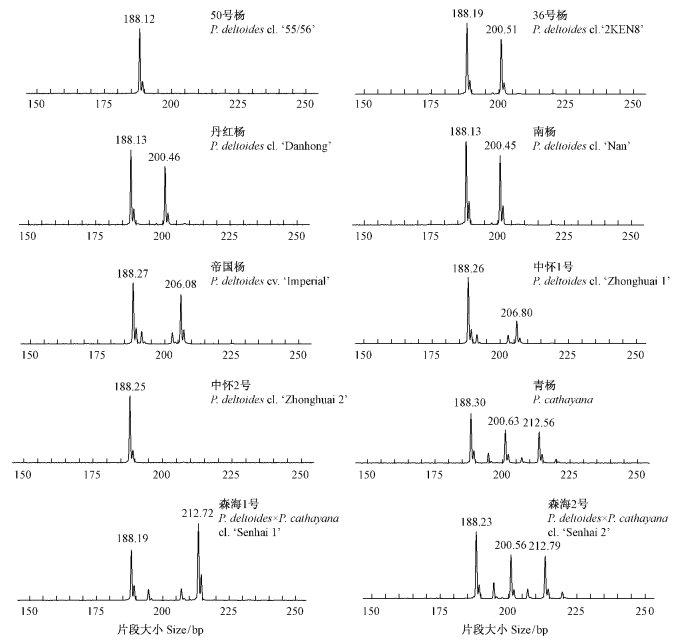 |
图 1 引物ORPM_247在10份杨树种质中的扩增图谱 Fig. 1 Polymorphic fingerprints detected by ORPM_247 for 10 Populus germplasms |
以从意大利引进的美洲黑杨无性系50号杨为母本、36号杨为父本杂交,从子代中选育出了3个优良无性系,分别为丹红杨、南杨和239;其中丹红杨和南杨速生杨树新品种,适合速生丰产林、农田防护林和四旁绿化。以丹红杨为母本、南杨为父本杂交,从子代中选育出3个优良子代,分别为1-116、中成1号和中成2号。对这8份杨树种质进行SSR分析鉴定,19对引物中有15对能够鉴别这8份种质的差异条带(表 3)。其中引物ORPM_409,GCPM_1599和PMGC_93在杂交子代中出现不符合亲本信息的条带,以引物ORPM_409扩增的条带为例,丹红杨和南杨分别为212 bp/216 bp和200 bp,1-116为212 bp/216 bp,未出现父本南杨200 bp的条带。
2.2.2 帝国杨、创新杨、丹红杨以及其杂种子代的SSR鉴定以I-69杨和I-63杨杂交得到的南抗杨1B 34-135为母本、帝国杨为父本杂交,从子代中选育出了优良子代创新杨;以丹红杨为母本、创新杨为父本杂交,选育出3个优良子代,分别为中豫1号、中成3号和中成4号。对帝国杨、创新杨、丹红杨、中豫1号、中成3号和中成4号进行SSR分析鉴定,19对引物中有18对能够鉴别这6份种质的差异条带(表 3)。引物ORPM_149,ORPM_446,GCPM_1177,GCPM_1255,GCPM_1454,GCPM_1599,GCPM_1663和PMGC_2020在检测创新杨时,创新杨出现与帝国杨不相符的条带。引物GCPM_1599在检测中成4号时,154 bp的条带在亲本中均未出现。
2.2.3 50号杨、帝国杨以及其杂种子代的SSR鉴定以50号杨为母本、帝国杨为父本杂交,从子代中选育出了2个优良无性系,中怀1号和中怀2号;对这4份杨树种质进行SSR鉴定分析,19对引物中有16对能够鉴别这4份种质的差异条带(表 3)。经检测,所有的条带信息均符合系谱关系。其中引物ORPM_241,GCPM_1255和PMGC_93对中怀1号进行检测时,均扩增出3条差异条带,且这些条带均有父本帝国杨杂合的等位基因位点;推测在培育中怀1号时,50号杨的卵细胞与帝国杨未经减数分裂的二倍体花粉受精,得到了三倍体植株。
2.2.4 50号杨、青杨以及其杂种子代的SSR鉴定以50号杨为母本、青杨为父本杂交,从子代中选育出了2个优良子代,森海1号和森海2号;对这4份杨树种质进行SSR分析鉴定,19对引物中有17对能够鉴别这4份种质的差异条带(表 3)。检测青杨时,引物ORPM_241,ORPM_247,GCPM_1177,GCPM_162和PMGC_2020扩增出3条带;检测森海1号时,ORPM_446,GCPM_1177,GCPM_162,GCPM_2571和PMGC_2020扩增出3条差异条带,检测森海2号时,引物ORPM_241,ORPM_247,ORPM_446,GCPM_1177,GCPM_1414,GCPM_162和PMGC_2020扩增出3条差异条带;推测青杨、森海1号和森海2号是三倍体,本研究中的青杨种质并非是森海1号和森海2号的真正父本,所以子代的条带与其存在较大的差异。
2.3 聚类分析将19对SSR引物扩增出的条带转换为0、1矩阵,用NTSYS-pc 2.10e软件进行遗传多样性分析,得到聚类分析图(图 2),相似系数在0.50~1.00之间。聚类结果显示,在相似系数为0.56时,24份杨树种质可分为5大类。
 |
图 2 基于SSR结果的24份杨树种质的聚类 Fig. 2 Dendrogram of 24 Populus germplasms based on SSR data |
第1类包括50号杨、中怀1号、中怀2号、I-69、帝国杨、中林2025杨、中红杨和全红杨。中怀1号和中怀2号是50号杨和帝国杨的杂交后代,中怀1号的遗传信息偏向父本,中怀2号的遗传信息偏向母本。中林2025杨的母本是I-69,2个品种相似系数较高,为0.86;未测到中林2025杨、中红杨和全红杨的条带差异。
第2类包括青杨、森海1号和森海2号。森海1号和森海2号是50号杨和青杨的杂交子代,遗传信息均偏向于青杨。
第3类包括36号杨、丹红杨、南杨、239、1-116、中成1号、中成2号、中成3号、中成4号和中豫1号。50号和36号杨杂交得到丹红杨、南杨和239,丹红杨和南杨杂交得到了1-116、中成1号和中成2号,聚类结果显示这些品种在相似系数为0.73时聚为一类,与其杂交系谱关系相符合。中成3号、中成4号和中豫1号的杂交亲本为丹红杨和创新杨,其遗传信息偏向母本。
第4类包括北抗杨和创新杨。北抗杨是南抗杨1A 34-17(I-69×I-63)与D175(引自美国的美洲黑杨)的杂交子代,创新杨是南抗杨1B 34-135(I-69×I-63)和帝国杨的杂交子代,2个品种都包含南抗杨的遗传信息,在相似系数为0.74时聚在一起。
第5类中只有中林46杨。中林46杨是以美洲黑杨为母本、钻天杨和俄罗斯杨的混合花粉为父本得到的杂交子代,与其他23份杨树种质亲缘关系较远。
2.4 倍性测试结果利用流式细胞仪FACSCalibur对24份杨树种质进行倍性测试,图 3是其中12份杨树的倍性测试结果。从图 3可以看出,当所测种质的G1期和G2期DNA相对含量分别位于50和100附近时,为二倍体;G1期和G2期DNA相对含量分别位于75和150附近时,为三倍体。研究结果表明,中怀1号、森海1号、森海2号、青杨、中林46均为三倍体,其他19份杨树种质均为二倍体,与SSR鉴定推测的结果一致。本研究说明SSR标记检测可以很好地反映植物的倍性情况。
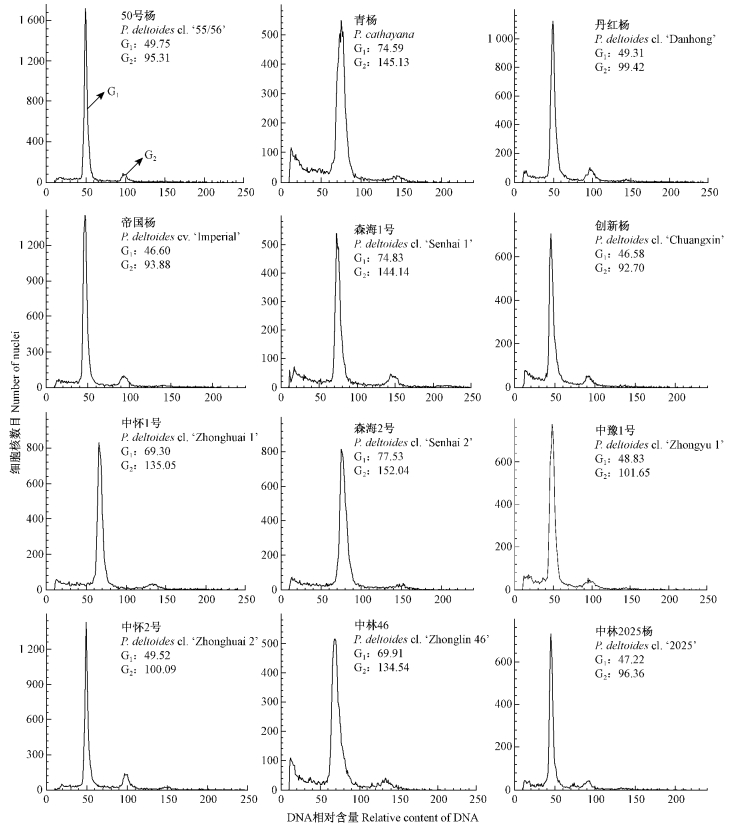 |
图 3 12份杨树种质的倍性测试 Fig. 3 Ploidy pattern of 12 Populus germplasms |
目前由于许多杨树新品种的遗传基础狭窄以及杂交组合亲本间差异较小,培育出的新品种之间形态学相似,所以很难从表型差异方面进行准确区分,DNA指纹图谱被认为是快速鉴别新品种的重要技术手段。本研究采用TP-M13-SSR技术利用3对高效引物ORPM_103,ORPM_180和GCPM_1255可将其中的21份杨树种质完全区分开,为新品种申请和育种者权利保护等提供了科学准确的理论依据。中林2025杨、中红杨和全红杨3份种质相对于其他21份种质,有特异的共同指纹条带,但是它们三者之间并没有差异条带,可能是由于中红杨和全红杨是中林2025杨的基础上选育来的新品种,在基因组水平差异较小或者没有差异。本研究得到的指纹图谱与其系谱关系基本一致。但是,在个别种质中出现了与亲本不相符的条带,推测原因如下:一是杂交子代SSR标记的个别基序(motif)重复数目发生变化,如1-116仅有1个不相符的位点;二是在杂交过程中,亲本种质混淆,如创新杨出现许多与帝国杨不相符的位点,推测其父本不是帝国杨。
与已报道的研究结果不同,冯锦霞等(2011)对50号杨、青杨及其杂交子代森海1号和森海2号进行SSR分析鉴定时,未检测到3个等位基因差异位点,例如引物GCPM_1177在青杨、森海1号和森海2号中只检测到127 bp和135 bp 2个位点;而本研究中多对引物检测到青杨、森海1号和森海2号有3个不同等位基因位点,例如引物GCPM_1177在青杨、森海1号和森海2号中均检测到3个等位位点,分别为127,135,139 bp。在梁海永等(2005)和王辉等(2008)的文献中,利用SSR标记未检测到中林46有3个等位基因差异位点,本研究中引物ORPM_103,ORPM_241和PMGC_2020均检测到3个不同等位基因位点。
在进行SSR检测时,除了发现青杨、森海1号、森海2号和中林46出现3个不同等位基因位点外,还发现中怀1号也出现3个不同位点,其余19份杨树种质只出现1个位点或2个位点。利用FCM对24份杨树种质进行倍性测试,发现扩增出3个位点的5个杨树种质均为三倍体,与SSR标记检测结果一致。Hu等(2012)对中林46进行染色体观察计数,发现其为三倍体,与本研究结果相符。Yagi等(2009)利用SSR标记和FCM对香石竹(Dianthus caryophyllus)进行倍性检测分析,2种方法得到了一致结果。本研究同样证实了SSR标记检测能较好地反映植物的倍性情况,可以作为检测植物倍性的重要依据。
在读取SSR检测结果的电泳图谱时,经常会发现有一些标记中出现大于或小于真实等位基因条带,称之为“影子带”。“影子带”大多数发生于双核苷酸重复序列中,由于DNA复制过程中双核苷酸重复序列发生滑动错配导致生成各种不同长度的片段(Hauge et al., 1993)。“影子带”会严重干扰对真实等位基因的识别,在进行PCR扩增时,可以通过touchdown-PCR的方法来减少“影子带”的产生,但该方法不能真正地避免该问题(Testolin et al., 2000)。因此,在进行SSR指纹图谱构建时,应挑选稳定性高和特异性好的引物进行扩增,同时应利用系谱材料对“峰”与“等位基因”进行一一对应确证,避免错误识别等位基因。
本研究利用TP-M13-SSR技术对24份杨树种质进行了指纹图谱构建、遗传多样性分析和倍性测试,证实了用SSR标记可以检测亲本与子代之间的系谱关系,并很好地反映植物的倍性,为杨树新品种鉴定和知识产权保护提供科学的理论依据,同时给杨树育种工作奠定坚实的基础。但是本研究未对24份杨树种质进行染色体压片镜检,在今后的工作中将采用此方法进一步确定染色体倍数并进行核型分析。
| [1] |
冯锦霞, 张川红, 郑勇奇, 等. 2011. 利用荧光SSR标记鉴别杨树品种. 林业科学, 47(6): 167-174. (Feng J X, Zhang C H, Zheng Y Q, et al. 2011. Identification of poplar varieties by SSR markers using capillary electrophoresis with fluorescence detection. Scientia Silvae Sinicae, 47(6): 167-174[in Chinese]).(  2) 2)
|
| [2] |
胡建军, 杨敏生, 卢孟柱. 2010. 我国抗虫转基因杨树生态安全性研究进展. 生物多样性, 18(4): 336-345. (Hu J J, Yang M S, Lu M Z. 2010. Advances in biosafety studies on transgenic insect-resistant poplars in China. Biodiversity Science, 18(4): 336-345[in Chinese]).(  1) 1)
|
| [3] |
黄秦军, 苏晓华, 张香华. 2002. SSR分子标记与林木遗传育种. 世界林业研究, 15(3): 14-21. (Huang Q J, Su X H, Zhang X H, et al. 2002. Microsatellite marker and its application in tree genetics and breeding. World Forestry Research, 15(3): 14-21[in Chinese]).(  1) 1)
|
| [4] |
贾会霞, 吴立栓, 胡建军, 等. 2013. 柳树种质资源遗传多样性和亲缘关系的CE-AFLP分析. 林业科学, 49(6):37-44. (Jia H X, Wu L S, Hu J J, et al. 2013. Genetic diversity and genetic relationship of Salix germplasms revealed by CE-AFLP analysis. Scientia Silvae Sinicae, 49(6):37-44[in Chinese]).(  1) 1)
|
| [5] |
梁海永, 刘彩霞, 刘兴菊, 等. 2005. 杨树品种的SSR分析及鉴定. 河北农业大学学报, 28(4): 27-31. (Liang H Y, Liu C X, Liu X J, et al. 2005. Simple sequence repeat (SSR) analysis and identify of different cultivars in Populus L. Journal of Agricultural University of Hebei, 28(4): 27-31[in Chinese]).(  1) 1)
|
| [6] |
刘春英, 樊军锋, 高建社, 等. 2013. 杨树新杂种的SSR分析及鉴定. 西北林学院学报, 28(2): 70-73. (Liu C Y, Fan J F, Gao J S, et al. 2013. Simple sequence repeat analysis and identification of new Populus hybrid. Journal of Northwest Forestry University, 28(2): 70-73[in Chinese]).(  1) 1)
|
| [7] |
王 辉, 杨敏生, 朱建峰. 2008. 利用SSR对杨属部分种及杂种的分析鉴定. 东北林业大学学报, 36(12): 4-6. (Wang H, Yang M S, Zhu J F, et al. 2008. SSR analysis of some species and hybrids in Populus. Journal of Northeast Forestry University, 36(12): 4-6.[in Chinese]) (  2) 2)
|
| [8] |
王忠华. 2006. DNA指纹图谱技术及其在作物品种资源中的应用. 分子植物育种, 4(3): 425-430. (Wang Z H. 2006. DNA fingerprinting technology and its application in crop germplasm resources. Molecular Plant Breeding, 4(3): 425-430[in Chinese]).(  1) 1)
|
| [9] |
卫尊征, 张金凤, 张德强, 等. 2008. 白、青杨派间杂交幼胚培养及杂种子代的分子鉴定. 北京林业大学学报, 30(5): 73-77. (Wei Z Z, Zhang J F, Zhang D Q, et al. 2008. Immature ovule culture of inter-sectional hybrids between (Populus tomentosa × P.bolleana) and P. simonii and molecular identification of the hybrid filial generation. Jourmal of Beijing Forestry University, 30(5): 73-77[in Chinese]).(  1) 1)
|
| [10] |
张海凤, 郭宝林, 张成合, 等. 2008. 杜仲四倍体的诱导与鉴定. 园艺学报, 35(7): 1047-1052. (Zhang H F, Guo B L, Zhang C H, et al. 2008. Induction and identification of tetraploids in Eucommia ulmoides Oliv. Acta Horticulturae Sinica, 35(7): 1047-1052[in Chinese]).(  1) 1)
|
| [11] |
张亚东, 胡兴宜, 宋丛文. 2009. 利用新型分子标记EST-SSR鉴定湖北省内的主栽黑杨品种. 分子植物育种, 7(1): 105-109. (Zhang Y D, Hu X Y, Song C W. 2009. Identification of Populus varieties from Hubei Province by EST-SSR marker. Molecular Plant Breeding, 7(1): 105-109[in Chinese]).(  1) 1)
|
| [12] |
朱道圩, 杨 宵, 郅玉宝, 等. 2007. 用流式细胞仪鉴定中华猕猴桃的多倍体. 植物生理学通讯, 43(5): 905-908. (Zhu D Y, Yang X, Zhi Y B, et al. 2007. Identification of polyploidy in Actinidia chinensis Planch. by flow cytometer. Plant Physiology Communications, 43(5): 905-908[in Chinese]).(  1) 1)
|
| [13] |
Aleza P, Juárez J, Cuenca J, et al. 2010. Recovery of citrus triploid hybrids by embryo rescue and flow cytometry from 2x×2x sexual hybridisation and its application to extensive breeding programs. Plant Cell Reports, 29(9): 1023-1034.( 1) 1)
|
| [14] |
Allen G, Flores V M, Krasynanski S, et al. 2006. A modified protocol for rapid DNA isolation from plant tissues using cetyltrime thylammonium bromide. Nature Protocols, 1(5): 2320-2325.( 1) 1)
|
| [15] |
Clarindo W R, Carvalho C R, Mendonça M A C. 2012. Ploidy instability in long-term in vitro cultures of Coffea arabica L. monitored by flow cytometry. Plant Growth Regulation, 68(3): 533-538.( 1) 1)
|
| [16] |
Dayanandan S, Rajora O, Bawa K. 1998. Isolation and characterization of microsatellites in trembling aspen (Populus tremuloides). Theoretical and Applied Genetics, 96(6/7): 950-956.( 1) 1)
|
| [17] |
Doležel J, Greilhuber J, Suda J. 2007. Estimation of nuclear DNA content in plants using flow cytometry. Nature Protocols, 2(9): 2233-2244.( 1) 1)
|
| [18] |
Guichoux E, Lagache L, Wagner S, et al. 2011. Current trends in microsatellite genotyping. Molecular Ecology Resources, 11(4): 591-611.( 2) 2)
|
| [19] |
Hauge X, Litt M. 1993. A study of the origin of 'shadow bands' seen when typing dinucleotide repeat polymorphisms by the PCR. Human Molecular Genetics, 2(4): 411-415.( 1) 1)
|
| [20] |
Hu B, Wang B, Wang C, et al. 2012. Microarray analysis of gene expression in triploid black poplar. Silvae Genetica, 61(4/5): 148-157.( 1) 1)
|
| [21] |
Loureiro J, Pinto G, Lopes T, et al. 2005. Assessment of ploidy stability of the somatic embryogenesis process in Quercus suber L. using flow cytometry. Planta, 221(6): 815-822.( 1) 1)
|
| [22] |
Nei M, Li W H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proceedings of the National Academy of Sciences, 76(10): 5269-5273.( 1) 1)
|
| [23] |
Powell W, Machray G C, Provan J. 1996. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1(7): 215-222.( 1) 1)
|
| [24] |
Schuelke M. 2000. An economic method for the fluorescent labeling of PCR fragments. Nature Biotechnology, 18(2): 233-234.( 1) 1)
|
| [25] |
Testolin R, Marrazzo T, Cipriani G, et al. 2000. Microsatellite DNA in peach (Prunus persica L. Batsch) and its use in fingerprinting and testing the genetic origin of cultivars. Genome, 43(3): 512-520.( 1) 1)
|
| [26] |
Yagi M, Kimura T, Yamamoto T, et al. 2009. Estimation of ploidy levels and breeding backgrounds in pot carnation cultivars using flow cytometry and SSR markers. Journal of the Japanese Society for Horticultural Science, 78(3): 335-343.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51