文章信息
- 袁红雨, 马宁, 杨会敏, 庞秋芬, 谢素霞, 程琳
- Yuan Hongyu, Ma Ning, Yang Huimin, Pang Qiufen, Xie Suxia, Cheng Lin
- 茶树Ⅱ型核糖体失活蛋白基因CsRIP1和CsRIP2的克隆与表达分析
- Cloning and Expression of Type Ⅱ Ribosome Inactivating Protein Genes CsRIP 1 and CsRIP 2 from Camellia sinensis
- 林业科学, 2015, 51(2): 147-153
- Scientia Silvae Sinicae, 2015, 51(2): 147-153.
- DOI: 10.11707/j.1001-7488.20150218
-
文章历史
- 收稿日期:2014-04-21
- 修回日期:2014-07-19
-
作者相关文章
2. 红河学院生物科学与技术学院 蒙自 661100
2. College of Life Science and Technology, Honghe University Mengzi 661100
植物核糖体失活蛋白(ribosome inactivating proteins,RIPs)是一种rRNA N-糖苷酶,作用于真核生物核糖体大亚基28S rRNA上的一个高度保守的茎环结构(ricin/sarcin),切除其上的一个特定的腺嘌呤残基,使延伸因子EF-2不再与核糖体结合,从而抑制蛋白质的生物合成(Lapadula et al.,2012)。除了rRNA N-糖苷酶活性外,RIPs还表现出一系列其他酶学活性,例如超氧化物歧化酶和核酸酶活性。一些RIPs的N-糖苷酶活性还能将腺嘌呤从rRNA的其他位点上释放出来,或者作用于病毒的RNA和基因组DNA释放腺嘌呤(Peumans et al.,2001)。RIPs可分为2种主要类型:Ⅰ型RIPs为单链结构,具有rRNA N-糖苷酶活性;Ⅱ型RIPs为一异二聚体,由1条相当于Ⅰ型RIP的A链和1条具有凝集素性质的B链通过1个二硫键连接而成,这2条多肽链均来自一个前体蛋白。
尽管目前对RIPs确切的生理活性尚未形成一致的看法,但是越来越多的研究表明,RIPs在植物应答各种环境胁迫中发挥重要作用,是植物防御体系的组成部分。RIP基因的表达受多种环境胁迫的诱导,其中非生物胁迫包括干旱、聚乙二醇、盐胁迫、H2O2、热激和渗透压等,生物胁迫包括伤害、病毒、真菌和昆虫等(Kawade et al.,2009)。在体外,几乎所有被检测过的RIPs都具有广谱抗病毒活性,转基因植物研究又为RIPs的抗病毒活性提供了新的证据(Chen et al.,2002)。RIPs还对昆虫表现出一定的毒性,Bertholdo-Vargas等(2009)发现2种鳞翅目(Lepidoptera)昆虫摄食添加5种Ⅰ型核糖体失活蛋白的叶片后,生长速度和存活率显著降低,细胞的DNA损伤增加;表达西洋接骨木(Sambucus nigra)Ⅱ型核糖体失活蛋白SNA-I′的转基因烟草(Nicotiana tabacum cv. Samsun NN)对烟蚜(Myzus nicotianae)和甜菜夜蛾(Spodoptera exigua)产生抗性(Shahidi-Noghabi et al.,2009);辛纳毒蛋白对棉铃虫(Helicoverpa armigera)和淡色库蚊(Culex pipiens pallens)的幼虫具有毒性(Zhou et al.,2000)。
Yang等(2011)在研究茶树(Camellia sinensis)对刺吸式口器昆虫的应答反应时,从茶树‘凫早2号’(C. sinensis ‘Fuzao 2’)叶片中分离出一种受假眼小绿叶蝉(Empoasca vitis)取食诱导的Ⅱ型核糖体失活蛋白基因的cDNA片段,命名为Cs-Ev2(GenBank登录号:GH618807)。了解茶树Ⅱ型核糖体失活蛋白基因在植物抗虫中的作用,对林木抗虫基因工程具有重要意义。本研究以假眼小绿叶蝉取食的茶树幼叶SMART cDNA 文库为模板,采用RACE技术克隆了该基因的全长cDNA(CsRIP1),并利用PCR技术分离出另一个在茶树种子中表达的Ⅱ型RIP基因(CsRIP2)。同时还分析了这2个茶树Ⅱ型RIP基因在假眼小绿叶蝉和机械伤害处理不同时间的表达情况。试验结果将为进一步分析CsRIP1和CsRIP2的亚功能化以及其在茶树抗虫中的作用创造条件。
1 材料与方法 1.1 材料与处理以在营养钵生长2年的茶树栽培品种‘凫早2号’的幼苗为试验材料进行假眼小绿叶蝉和机械伤害处理。处理前将茶树幼苗置于温度25~28 ℃、光照强度160 μmol·m-2s-1、光照时间12 h·d-1的条件下生长7 天。
假眼小绿叶蝉处理:每一茶树幼苗接种50头左右的假眼小绿叶蝉成虫,用通风良好的塑料网罩将茶树幼苗罩上,接虫3,6,12,24,48 h后剪取充分伸展的茶树叶片。机械伤害:用剪刀将茶树叶片的边缘剪去,分别在处理3,6,12,24,48 h后剪取叶片。每种处理3次重复,每次取6株,以培养在相同条件下未进行处理的茶树幼苗作为对照。对照和各种处理的叶片采集后迅速用液氮处理,并置于-80 ℃冰箱中保存。
1.2 CsRIP1 基因全长cDNA的克隆根据Cs-Ev2 cDNA片段的核苷酸序列设计1对特异性引物CsRIP-F1(5′-ATACAGTTGTGGCCGTGTGA-3′)和CsRIP-R1(5′-TGACTCTGCGCTGAGGACTA-3′)用于5′RACE和3′ RACE。以5′-ready RACE cDNA文库为模板(谢素霞等,2013),利用5′ 通用引物和CsRIP-R1扩增该基因cDNA的5′ 末端,反应程序为:96 ℃ 2 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,30个循环。以3′-ready RACE cDNA文库为模板,利用3′通用引物和CsRIP-F1扩增该基因cDNA的3′ 末端,反应程序为:96 ℃ 2 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 2 min,30个循环。
拼接5′RACE产物和3′ RACE产物,得到CsRIP1的全长cDNA,并利用NCBI网站预测其ORF,根据5′和3′非编码区序列设计引物CsRIP-F2(5′-CTGCTACTGAAACGAAATATGAAAG-3′)和CsRIP-R2(5′-TAGGGAATGACAAACAACCAAGTAGGC-3′),扩增该基因完整的ORF,反应程序为:96 ℃ 2 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 3 min,30个循环。
1.3 CsRIP2基因cDNA的克隆利用RNeasy plant mini kit(Qiagen,德国)从茶树子叶中提取总RNA,用DNase I(Promega,美国)处理总RNA以去除其中残余的DNA。用First Str and cDNA Synthesis试剂盒(Fermentas)合成第1链cDNA,以第1链cDNA为模板,利用特异引物CsRIP-F2和CsRIP-R2扩增CsRIP2基因的cDNA序列。将PCR产物凝胶电泳后,回收目的片段,将目的片段与pMD19-T 载体(大连宝生物工程有限公司)连接并转化DH5α,重组质粒鉴定后测序。
1.4 CsRIP基因组DNA的克隆按照Plant DNA Isolation Reagent 试剂盒(大连宝生物工程有限公司)说明书,以茶树叶片为材料提取茶树的基因组DNA。以基因组DNA为模板,利用特异引物CsRIP-F2和CsRIP-R2扩增CsRIP基因的gDNA序列。
1.5 Real-time qRT-PCR分析CsRIP基因的表达分别选取茶树的幼根、茎、叶片和子叶,用Trizol Reagent(Invitrogen)提取总RNA,通过琼脂糖凝胶电泳和紫外分光光度计检测RNA的完整性和浓度。用First Str and cDNA Synthesis试剂盒(Fermentas)合成第1链cDNA,用于检测CsRIP1和CsRIP2在不同组织中的表达水平。选取不同处理的茶树幼苗完全伸展的叶片,分别提取总RNA,以检测CsRIP基因的表达水平。
根据2个CsRIP基因3′ UTR的差异性区域,分别设计CsRIP1基因特异性的实时荧光定量RT-PCR分析引物CsRIP1-F(5′-CCAATCCAATCCTCC AGCAA-3′)和CsRIP1-R(5′-GGGCCTTCTTTGG ATTCAGCG-3′),以及CsRIP2基因特异性的实时荧光定量RT-PCR引物CsRIP2-F(5′-CCAATCC AATCCTCCAGCAA-3′)和CsRIP2-R(5′-ACAAACGA CGATTGAGCGCA-3′)。采用茶树的α-tubulin基因(上游引物:5′-CCACTCATTCCCTCCTTGAA-3′;下游引物:5′-ATGGCTCCATCAAACCTCAG-3′)作为内参对不同样品cDNA的模板量进行校准。
按照TaKaRa公司SYBR Premix Ex TaqTM(Tli RNaseH Plus)试剂盒说明书进行实时荧光定量PCR反应,反应体系为:cDNA 模板2 μL,2×SYBR Premix Ex TaqTM10 μL,上、下游引物(10 μmol · L-1)各0.4 μL,50×ROX Reference Dye 0.4 μL,ddH2O 6.8 μL。每个样品设3 次重复。PCR反应在ABI PRISM 7300 Real-Time PCR仪(Applied Biosystems)上进行,程序为:95 ℃ 1 min;95 ℃ 15 s,58 ℃ 15 s,72 ℃ 30 s,40个循环;设定程序,使扩增结束后PCR 仪自动运行绘制熔解曲线(95 ℃ 15 s,60 ℃ 15 s,95 ℃ 15 s)。数据分析按2-ΔΔCT方法(Livak et al.,2001)进行。在组织表达分析中,用表达量最低的组织作为对照,在胁迫处理的表达分析中,以未处理的材料作为对照,计算试验组目的基因的表达相对于对照组的变化倍数。
2 结果与分析 2.1 CsRIP基因克隆与序列分析以SMART cDNA为模板,5′ RACE和3′ RACE扩增后,1%的琼脂糖凝胶电泳检测,分别得到1条特异的条带。克隆测序后发现,5′RACE产物长1 330 bp(图 1A),其3′端序列与Cs-Ev2的5′端序列完全重叠;3′ RACE产物长936 bp(图 1B),其5′端与Cs-Ev2的3′端完全重叠;拼接后得到2 032 bp的全长cDNA,命名为CsRIP1(GenBank登录号:FJ648831)。该序列包含5′ 非翻译区73 bp、3′ 非翻译区224 bp、阅读框1 713 bp,编码570个氨基酸残基,其预测分子质量为63.55 kDa,等电点为4.75。根据CsRIP1基因的5′ UTR和3′ UTR分别设计引物CsRIP-F2和CsRIP-R2,以上述SMART cDNA为模板扩增得到1 893 bp长的cDNA序列,含有一个与拼接序列相同的阅读框(图 1C)。
 |
图 1 CsRIP1基因的cDNA扩增
Fig. 1 The amplification of CsRIP1 cDNA with PCR method
M:DNA Marker; A:CsRIP1的5′ RACE产物;B:CsRIP1的
3′RACE产物;C:CsRIP-F2和CsRIP-R2的扩增结果。 M: DNA Marker; A: Product of 5′ RACE; B: Product of 3′RACE; C: Product of amplification with CsRIP-F2 and CsRIP-R2. |
植物的Ⅱ型RIPs常常在植物的种子中积累,本研究以茶树子叶(~210 DAF)的mRNA为模板,采用CsRIP-F2和CsRIP-R2进行RT-PCR扩增。对扩增产物克隆测序后,发现了1个新的茶树Ⅱ型核糖体失活蛋白基因,命名为CsRIP2(GenBank登录号:GU951535)。CsRIP2与CsRIP1有98%的序列一致性,也含有一个1 713 bp的ORF,编码产物与CsRIP1具有96%的相似性。与CsRIP1相比,已克隆的CsRIP2的3′ UTR缺失了一段15 bp的序列。
利用CsRIP-F2和CsRIP-R2,以来自叶片的基因组DNA为模板进行PCR扩增,克隆CsRIP1和CsRIP2的基因组序列。测序后发现,CsRIP1和CsRIP2基因的gDNA都不含内含子。
2.2 CsRIP基因编码产物的结构域分析和同源性比较通过Signal 4.1软件分析发现,CsRIP含有1个由22个氨基酸残基组成的信号肽,其切割位点在第22位的缬氨酸和第23位的半胱氨酸之间。SMART在线预测软件分析表明,CsRIPs含有1个RIP结构域(氨基酸残基36—289)和2个ricin B-like(RBL)结构域(氨基酸残基316—442,446—569)。RIP结构域相当于Ⅱ型RIP的A链,而B链由2个RBL结构域组成。
BLASTP搜索GenBank数据库,发现CsRIPs与樟(Cinnamomum camphora)的辛纳毒蛋白Ⅲ前体、蓖麻(Ricinus communis)毒素前体、相思子(Abrus precatorius)毒素-a、美丽相思子(Abrus pulchellus)的prepropulchellin Ⅳ具有较高的序列相似性(61%~54%)。序列比对表明相思子毒素-a和蓖麻毒素A链的N-糖苷酶活性中心裂隙中所有的氨基酸残基均出现在CsRIPs的相应位置上(图 2)。
 |
图 2 不同物种Ⅱ型核糖体失活蛋白的氨基酸序列比对 Fig. 2 Amino acid multiple alignment of type Ⅱ RIPs from different species 黑色表示在所有6种蛋白质氨基酸序列中都相同的氨基酸残基;灰色表示在5种蛋白质氨基酸序列中都相同的氨基酸残基;“*”表示构成A链N-糖苷酶活性位点的氨基酸残基; “▲”和“:”分别表示B链中形成碳水化合物结合位点的氨基酸残基;“+”表示形成链间和链内二硫键的半胱氨酸残基;“QxW”表示QxW基序。CsRIP1:茶树核糖体失活蛋白1(ACV60361);CsRIP2:茶树核糖体失活蛋白2(ADF45510);ricin:蓖麻毒素前体(XP_002534649);abrin-a:相思子毒素-a(AAA32624);Lectin Ⅰ:白果槲寄生凝集素Ⅰ(AAR25545);cinna Ⅲ:樟辛纳毒蛋白Ⅲ前体(AAK82460)。 Amino acids identical in all six proteins are shaded black, while amino acid residues that are conserved in at least five of the six sequences are shaded grey. The amino acids that form the RNA N-glycosidase active site in A-chain are indicated with (*), whereas amino acids that form two separate carbohydrate-binding sites in B-chain are marked by (▲) and (:). Ten cysteines that form one interchain and four intrachain disulfide bonds in six RIPs are marked by (+). QxW motifs in B chain are indicated with (QxW). CsRIP1: Camellia sinensis ribosome inactivating protein 1 (ACV60361); CsRIP2: C. sinensis ribosome inactivating protein 2 (ADF45510); ricin: Ricinus communis preproricin (XP_002534649); abrin-a: Abrus precatorius abrin-a (AAA32624);Lectin Ⅰ: Viscum album lectin Ⅰ(AAR25545); cinna Ⅲ: Cinnamomum camphora cinnamomin Ⅲ precursor (AAK82460). |
白果槲寄生(Viscum album)的凝集素I、蓖麻毒素和相思子毒素-a的B-链含有2个同源的RBL结构域,每个RBL结构域具有3个谷氨酰胺-任意氨基酸-色氨酸(QxW)重复基序。序列比对显示,CsRIPs的每个RBL结构域在对应位置上均含有3个保守的QxW基序。蓖麻毒素的2个碳水化合物结合位点各由5个保守的氨基酸残基组成,这2组氨基酸残基在CsRIPs的B链中也高度保守(图 2)。与其他Ⅱ型RIPs一样,CsRIPs的A链靠近C末端的位置上有1个半胱氨酸残基,B链上有9个保守的半胱氨酸残基。
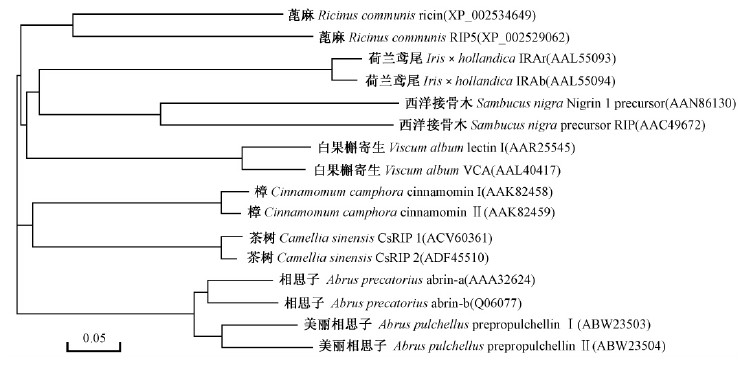 |
图 3 基于不同物种Ⅱ型RIP氨基酸序列构建的系统进化树 Fig. 3 Phylogenetic tree derived from the alignment of the amino acid sequences of type Ⅱ RIPs from different plant species |
核糖体失活蛋白以多基因家族的形式存在于植物的基因组中。分别从8种植物中选取序列差别最大的2种Ⅱ型RIPs,同茶树的2种Ⅱ型RIPs一起,采用DNAMAN软件中的邻接法(neighbor-joining)构建进化树。结果表明,同一物种的RIPs首先聚类合并,茶树的2种RIPs与樟的RIPs聚为一支,蓖麻、荷兰鸢尾(Iris × holl and ica)、西洋接骨木和白果槲寄生的RIPs聚为一支,美丽相思子和相思子的RIPs聚为一支(图 3)。
2.4 CsRIP基因在不同组织中的表达分析利用Real-time qRT-PCR技术检测CsRIP1和CsRIP2在茶树4种不同组织中的表达模式,结果(图 4)显示,CsRIP1和CsRIP2在幼根、茎、叶和子叶中均有表达。CsRIP1在叶中的表达水平最高,约为子叶的5.5倍、幼根的4.4倍、茎的2.4倍;CsRIP2在子叶中的表达水平最高,约为叶的6倍、幼根的3倍、茎的1.6倍。
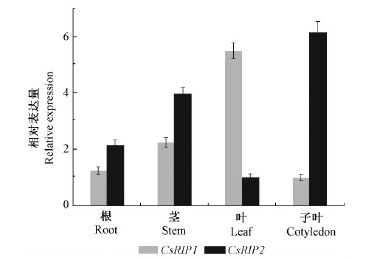 |
图 4 CsRIP基因组织特异性表达分析 Fig. 4 Expression of two CsRIP genes in different tissues of C. sinensis |
荧光定量RT-PCR分析表明,2个CsRIP基因的表达均受假眼小绿叶蝉取食的强烈诱导,并且具有类似的表达谱。从图 5可知,假眼小绿叶蝉取食3 h后,CsRIP1和CsRIP2表达开始升高,并且取食时间越长表达量越高,取食48 h后,CsRIP1和CsRIP2转录本水平大约分别是对照的120和100倍(图 5)。机械伤害处理后,CsRIP1表达升高,处理后6 h达到最大值,随后逐渐下降,处理后48 h再度增强(图 6);CsRIP2在机械伤害处理后6 h,表达水平显著升高,12 h达到最高水平,约为对照的9.5倍,以后逐渐下降,处理后48 h,降至对照水平的1/2。
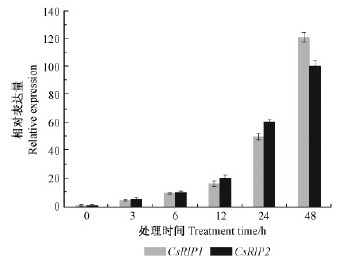 |
图 5 假眼小绿叶蝉处理对CsRIP基因表达的影响 Fig. 5 Expression of two CsRIP genes in response to E. vitis feeding |
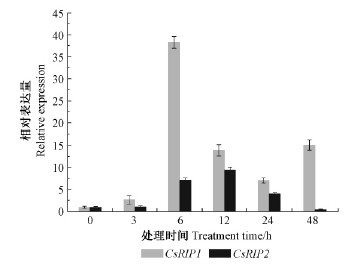 |
图 6 机械伤害处理对CsRIP基因表达的影响 Fig. 6 Expression of two CsRIP genes in response to mechanical damage |
植物体常常编码一组不同的RIPs,例如对水稻(Oryza sativa)和蓖麻的基因组进行扫描,发现水稻的基因组含有31个Ⅰ型RIP基因(Jiang et al.,2008),蓖麻的基因组含有7个Ⅱ型RIP基因(Leshin et al.,2010),RIP基因家族不同成员的表达常常表现出组织特异性和发育阶段的特异性,并且应答不同的环境胁迫。本研究从茶树中分离出2个Ⅱ型核糖体失活蛋白基因,这2个RIP基因均含有1个1 713 bp的读码框,编码570个氨基酸残基。CsRIP1的5′ UTR和3′ UTR的长度分别是73 bp和224 bp,与CsRIP1基因的3′ UTR相比,除了1个位置上的碱基不同之外,CsRIP2基因已克隆的3′ UTR缺失了一段15 bp的核苷酸序列。CsRIP1和CsRIP2编码产物具有98%的一致性,结构域分析表明,CsRIPs的编码产物由信号肽、A链、链接肽和B链组成。Ⅱ型核糖体失活蛋白前体在成熟的过程中,信号肽和链接肽被切除,A链和B链通过1个二硫键连接起来。
Ⅱ型RIPs的A链具有N-糖苷酶活性,氨基酸序列的多重比对结果表明,其活性中心位点保守的氨基酸残基均出现在CsRIPs A链的相应位置上。另外,低水平的赖氨酸残基也是A链发挥其酶学活性的关键因素,蓖麻毒蛋白的A链含有2个赖氨酸残基,而CsRIPs的A链仅带有1个赖氨酸残基。低水平的赖氨酸残基可能有助于A链躲避细胞质中蛋白酶体的降解(Di et al.,2005),作用于rRNA。
Ⅱ型RIPs的B链能够结合细胞表面寡糖链上的半乳糖残基,介导毒蛋白的内吞过程。通过对蓖麻毒蛋白和abrin-a的晶体结构分析发现,Ⅱ型RIPs的B链含有2个重复的结构域,而每个结构域又进一步分成3个大约由40个氨基酸残基组成的亚结构域。因此,Ⅱ型RIPs的B链可能起源于一个编码40个氨基酸残基的基因,该原初基因首先发生3次重复,形成RBL结构域的编码序列,然后再发生1次重复,形成2个RBL结构域。由于每个亚结构域中存在1个QxW基序,因此RBL结构域又称为(QxW)3结构域(Hazes,1996)。相同的结构模式和保守的氨基酸残基也存在于CsRIPs的B链上。
尽管2个CsRIP基因在已检测的4种组织中均有表达,但仍表现出一定的组织特异性。CsRIP1在叶片中的表达水平最高,而CsRIP2在发育的子叶中高度表达。在本试验中,假眼小绿叶蝉取食快速诱导CsRIPs基因的表达,并且随着取食时间的延长,CsRIPs基因的表达量逐渐增加。机械伤害显著诱导CsRIPs基因的表达,但是在伤信号的诱导下,CsRIP1和CsRIP2的表达模式存在较大差别。结果表明CsRIPs基因可能参与茶树的防卫反应,且已经发生亚功能化。系统进化树分析显示,一种植物的Ⅱ型RIPs首先聚类,说明Ⅱ型核糖体失活蛋白基因在植物体中发生扩增是一个近期事件。序列分析显示,CsRIPs与樟的cinnamomin Ⅲ的一致性最高,约为63%。cinnamomin被认为是樟种子的贮藏蛋白(Liu et al.,2002),因此可以设想昆虫取食的选择压力导致CsRIPs演变成一种防卫蛋白,并在进化的过程中获得了在叶片中表达且受昆虫取食诱导的特性。
| [1] |
谢素霞, 程琳, 曾威, 等. 2013. 茶树HCT基因的克隆及表达. 东北林业大学学报, 41(6):19-22. (Xie S X, Cheng L, Zeng W, et al. 2013. Molecular cloning and expression of HCT gene from Camellia sinensis. Journal of Northeast Forestry University, 41(6): 19-22[in Chinese]).(  1) 1)
|
| [2] |
Bertholdo-Vargas L R, Martins J N, Bordin D, et al. 2009. Type 1 ribosome-inactivating proteins-entomotoxic, oxidative and genotoxic action on Anticarsia gemmatalis (Hübner) and Spodoptera frugiperda (J.E. Smith) (Lepidoptera: Noctuidae). J Insect Physiol, 55(1): 51-58.( 1) 1)
|
| [3] |
Chen Y, Peumans W J, Van Damme E J. 2002. The Sambucus nigra type-2 ribosome-inactivating protein SNA-I' exhibits in planta antiviral activity in transgenic tobacco. FEBS Lett, 516(1/3): 27-30.( 1) 1)
|
| [4] |
Di C A, Frigerio L, Lord J M, et al. 2005. Endoplasmic reticulum-associated degradation of ricin A chain has unique and plant-specific features. Plant Physiol, 137(1): 287- 296.( 1) 1)
|
| [5] |
Hazes B. 1996. The (QxW)3 domain: a flexible lectin scaffold. Protein Sci, 5(8): 1490-1501.( 1) 1)
|
| [6] |
Jiang S Y, Ramamoorthy R, Bhalla R, et al. 2008. Genome-wide survey of the RIP domain family in Oryza sativa and their expression profiles under various abiotic and biotic stresses. Plant Mol Biol, 67(6):603-614.( 1) 1)
|
| [7] |
Kawade K, Masuda K. 2009. Transcriptional control of two ribosome-inactivating protein genes expressed in spinach (Spinacia oleracea) embryos. Plant Physiol Biochem, 47(5):327-334.( 1) 1)
|
| [8] |
Lapadula W J, Sanchez-Puerta M V, Ayub M J. 2012. Convergent evolution led ribosome inactivating proteins to interact with ribosomal stalk. Toxicon, 59(3):427-432.( 1) 1)
|
| [9] |
Leshin J, Danielsen M, Credle J J, et al. 2010. Characterization of ricin toxin family members from Ricinus communis. Toxicon, 55(2/3):658-661.( 1) 1)
|
| [10] |
Liu R S, Wei G Q, Yang Q, et al. 2002. Cinnamomin, a type II ribosome-inactivating protein, is a storage protein in the seed of the camphor tree (Cinnamomum camphora). Biochem J, 362(3): 659-663.( 1) 1)
|
| [11] |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 25(4):402-408.( 1) 1)
|
| [12] |
Peumans W J, Hao Q, Van Damme E J. 2001. Ribosome-inactivating proteins from plants: more than RNA N-glycosidases. FASEB J, 15(9):1493-1506.( 1) 1)
|
| [13] |
Shahidi-Noghabi S, Van Damme E J, Smagghe G. 2009. Expression of Sambucus nigra agglutinin (SNA-I') from elderberry bark in transgenic tobacco plants results in enhanced resistance to different insect species. Transgenic Res, 18(2):249-259.( 1) 1)
|
| [14] |
Yang H M, Xie S X, Wang L, et al. 2011. Identification of up-regulated genes in tea leaves under mild infestation of green leafhopper. Scientia Horticulturae, 130(2): 476-481.( 1) 1)
|
| [15] |
Zhou X, Li X D, Yuan J Z, et al. 2000. Toxicity of cinnamomin - a new type II ribosome-inactivating protein to bollworm and mosquito. Insect Biochem Mol Biol, 30(3): 259-264.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51
