文章信息
- 王雁红, 俞琦, 杨佳, 赵鹏, 李忠虎, 赵桂仿
- Wang Yanhong, Yu Qi, Yang Jia, Zhao Peng, Li Zhonghu, Zhao Guifang
- 基于核微卫星的短柄枹栎居群遗传多样性和遗传结构
- Genetic Diversity and Population Structure of Quercus serrata var.brevipetiolata Revealed by nSSR Markers
- 林业科学, 2015, 51(12): 121-131
- Scientia Silvae Sinicae, 2015, 51(12): 121-131.
- DOI: 10.11707/j.1001-7488.20151215
-
文章历史
- 收稿日期:2014-11-06
- 修回日期:2015-01-14
-
作者相关文章
遗传多样性和遗传分化是了解物种(树种)在其进化历史中如何响应气候变化的基础(Hedrick,2004)。近年来,随着分子生物学技术的快速发展,SSR分子标记已被普遍地应用到物种的遗传多样性和群体遗传结构研究中(Tautz,1989; Chen et al.,2003; Bertini et al.,2006; Chen et al.,2006; Guo et al.,2007;徐斌等,2013)。SSR(simple sequence repeats)即简单序列重复,亦称微卫星DNA(microsatellite DNA),此标记具有共显性、重复性好以及多态性丰富等优点,并且其侧翼部分在相近种间具有高度保守性和转移通用性,被广泛应用于物种的遗传多样性和保护生物学等领域(陈怀琼等,2009;刘牧,2012)。
短柄枹栎(Quercus serrata var. brevipetiolata)是壳斗科(Fagaceae)栎属(Quercus)中的一种多年生落叶乔木,是我国热带和南亚热带地区的重要组成树种之一(徐永椿等,1998;曹倩等,2014)。其木材坚硬,可供建筑、车辆等用材;种子富含淀粉,可用于酿酒和制作饮料;树皮可供提取栲胶,叶子可饲养柞蚕,是一种很重要的经济树种(Aldrich et al.,2011)。短柄枹栎的自然地理分布区从我国东部、南部地区的江苏、广东、广西一直向西延伸到秦岭山脉和青藏高原以东地区,日本和朝鲜等国也有少量分布(徐永椿等,1998)。短柄枹栎多分布于海拔200~2 000 m的山地或沟谷林中,是我国南方重要的森林建群树种和优势树种,在维持森林生态系统中起着重要作用。前期的采样调查表明短柄枹栎现今主要分布在我国的南部山区。这一地区在第四纪冰期扮演了重要的物种避难所的角色,对物种遗传多样性的保持和维护提供了重要的作用(Liu et al.,2012)。然而,对于短柄枹栎以及我国广泛分布的多种栎属植物的分子遗传学研究则很少,现有的对不同区域分布的栎类的遗传多样性研究也呈现差异的结论。例如,在我国栎属植物研究中,刘牧(2012)使用9个核SSR分子标记研究了蒙古栎(Quercus mongolica)和辽东栎(Quercus wutaishanica)的群体遗传多样性,表明2个近缘种都具有较高水平的遗传多样性;秦英英等(2012)利用11个核SSR位点对8个辽东栎自然群体的遗传多样性进行研究,揭示该物种具有较高的遗传多态性(I =1.75,He=0.75,Na=10.27);曹倩等(2014)采用7对SSR引物对短柄枹栎天然林中的4个次生林居群的遗传多样性和遗传结构进行了研究,也发现该物种具有较高的遗传多样性水平(He=0.88,Na=14.42),但4个次生林居群不能代表该物种的整体遗传信息。在国外栎属植物的研究中,基于核SSR标记的欧洲栓皮栎(Quercus suber)的研究发现此物种杂合度为0.65,具有高水平的遗传变异(Hornero et al.,2001);Ortego等(2012)对北美洲的Engelmann oak (Quercus engelmannii)的研究发现不同的SSR位点显现出差异的遗传多样性,认为该物种的遗传多样性和遗传结构与其分布区域的气候变化密切相关。
基于以上分析,本研究选用8对具有稳定多态性的核SSR分子标记对采集到的24个短柄枹栎居群进行研究,研究群体覆盖短柄枹栎现今的分布范围,以期揭示现今该物种的遗传多样性和遗传结构,探讨短柄枹栎的遗传特性与其进化历史以及生活环境的联系,为短柄枹栎的利用和保护提供科学依据。
1 材料与方法 1.1 材料来源2010—2013年期间,对短柄枹栎的自然分布区进行全面的野外调查和采样,采集到24个短柄枹栎的居群共计398个个体(表 1),样本个体之间至少间隔50 m。采集新鲜、健康的叶片使用变色硅胶干燥保存。凭证标本保存于西北大学生命科学学院标本馆。
|
|
从每份样品中取30 mg干燥叶片利用植物基因组DNA提取试剂盒(天根生化科技有限公司)提取总基因组DNA。DNA经浓度检测后保存在-4 ℃备用。
2)引物筛选从前人已开发的其他栎属SSR引物(Dow et al.,1995; Steinkellner et al.,1997; Kampfer et al.,1998; Sullivan et al.,2013)中筛选出8对多态性较高、条带清晰且稳定的引物(表 2)进行扩增,引物在部分居群中的扩增情况如图 1所示。
|
|
 |
图 1 8对引物在部分样品中的扩增结果 Fig. 1 The part of amplification products of all primers (a)L1p; (b)L1/2; (c)L13; (d)L3d; (e)L3a; (f)L110; (g)L2p; (h)L11. M: Marker. |
3)PCR扩增 PCR程序在PTC-200(MJ Research,USA)热循环仪上进行; 10 μL PCR反应体系如下: 2×Taq PCR Mix 5 μL(含Taq DNA聚合酶、dNTPs、MgCl2、反应缓冲剂等),10 μmol·L-1正、反向引物各0.2 μL,20 ng·μL-1模板DNA 1.0 μL,3.6 μL ddH2O。PCR反应程序如下: 94 ℃预变性5 min; 94 ℃加热变性30 s,退火30 s,72 ℃延伸30 s,34个循环;最后72 ℃延伸7 min,扩增产物在4 ℃保存。
4)电泳检测 PCR扩增产物经1%琼脂糖凝胶电泳检测合格后,再使用10%聚丙烯酰胺凝胶进行电泳检测,电泳结果采用银染法进行条带显色。
1.3 数据统计与SSR分析使用Quantity One软件读取和统计扩增产物的片段长度,将结果录入Excel表格中。采用POPGENE 1.32(Yeh et al.,1999)检测分子标记的Hardy-Weinberg平衡、连锁不平衡(95%置信区间)以及中性检验(1 000次随机运算)。同时利用Micro-checker软件(Van Oosterhout et al.,2004)对哑等位基因进行检测(置信区间设置为95%)。
利用GenALEx 6.5(Peakall et al.,2006)软件计算各居群的遗传多样性,如平均等位基因数(Na)、平均有效等位基因数(Ne)、居群及微卫星位点的期望杂合度(He)和观测杂合度(Ho)、Shannon信息指数(I)、多态位点百分率(PPL),并且估算基因流(Nm)。采用IBD(Isolation by Distance)分析居群的遗传距离和地理距离之间的相关性及其显著性水平(Bohonak,2002)。为了衡量群体的遗传分化程度,在GenALEx 6.5中统计Fis,Fit,FST等F统计量指数(Nei,1973)。
为了估计居群之间、居群内部以及群组之间的遗传差异,使用Arlequin v3.1(Excoffier et al.,2005)软件对物种水平和STRUCTURE分组水平下的遗传变异分别进行分子变异(AMOVA)分析。
为反映居群之间的遗传差异和相关性,使用STRUCTURE v 2.3软件(Pritchard et al.,2000)计算短柄枹栎居群的遗传结构。软件设置为: burn-in,105; MCMC,106。并进行10次随机运算(K值设置为1~10)。采用2个不同分组判别标准来确定合适的分组: 1)取值最大且前后变化平缓的lnP(D)值; 2)ΔK统计法(Evanno et al.,2005),当ΔK值最大时的K为最理想的分组值。同时利用Alleles In Space软件中的景观遗传插值分析(L and scape Shape Interpolation analysis)(Miller,2005)对短柄枹栎群体间的遗传距离进行空间遗传模式构建,距离权重值设置为a=1,分析不同群体之间的遗传差异。其中X轴和Y轴分别对应居群地理分布坐标,Z轴对应居群间的相对遗传距离,单位网格设置为50×50以保证覆盖短柄枹栎整个地理分布范围。
2 结果与分析 2.1 Hardy-Weinberg平衡、连锁不平衡和中性检验对8个微卫星位点进行的Hardy-Weinberg平衡检验显示,除L13位点符合Hardy-Weinberg平衡以外,其余的7个位点均偏离Hardy-Weinberg平衡。同时,在这些偏离Hardy-Weinberg平衡的位点上也检测到可能的哑等位基因存在(表 3)。多位点分析检验表明在7个位点中,24个居群均在2~5个位点上偏离Hardy-Weinberg平衡(除江苏句容居群JR和湖南长沙居群CS)。在L13位点上,只有湖北恩施居群(ES)和陕西山阳居群(SY)偏离Hardy-Weinberg平衡。哑等位基因的存在导致观测杂合子减少、纯合子增加,进而引起Hardy-Weinberg平衡的偏离。
|
|
连锁不平衡检验显示8个微卫星位点在95%的置信水平上均不存在连锁。24个群体内的连锁不平衡情况显示,陕西安康(AK)、湖北十堰(JX)、陕西平利(PL)和安徽霍山群体(HS)呈现出极显著的不平衡状况(P<0.01),陕西商南(SN)和陕西佛坪群体(FP)呈现出显著不平衡(P<0.05)。中性检验显示2个位点偏离中性进化(L1/2和L3a),而大部分位点遵循中性进化(表 3)。
2.2 短柄枹栎居群的遗传多样性和遗传分化24个短柄枹栎自然居群的Nei’s遗传多样性指数(He)为0.21~0.46,平均0.43,其中遗传多样性最高的居群是浙江绍兴(SX),最低的是安徽休宁(XN);平均等位基因数(Na)为3.76,最高的是湖北恩施(ES)(5.38),最低的是湖南长沙居群(CS)(2.13);平均的Shannon’s信息指数(I)为0.66,其中浙江绍兴居群(SX)最高(0.95),安徽休宁(XN)最低(0.41)。多态位点百分率(PPL)平均为82.81%(表 4)。除河南内乡(NX),江西庐山(LS)和句容(JR),安徽宣城(XC)、霍山(HS)和休宁(XN)群体的近交系数(Fis)是负值外,其余18个居群均大于0,表明多个居群存在杂合子不足的情况,群体内部存在一定程度的近交(表 4)。
|
|
24个短柄枹栎居群中有10个居群含有私有等位基因,分别为重庆巫山(WS)、陕西安康(AK)、湖北十堰(JX)、湖南新宁(HL)、陕西商南(SN)、江西庐山(LS)、安徽霍山(HS)、江西南昌(NC)、陕西佛坪(FP)以及江苏句容(JR)居群。安徽霍山居群(HS)在L110位点含有2个私有等位基因,其余居群只含有1个私有等位基因。私有等位基因频率最高是江苏句容居群(JR),为0.143,最低是安徽霍山(HS)、江西庐山(LS)以及陕西商南(SN)居群,为0.025。
F统计结果显示短柄枹栎不同基因位点间的遗传分化系数(FST)存在差异,其中分化最高的位点是L11(FST =0.49),最低的是L1/2(FST=0.04),物种的平均基因流(Nm)为1.88(表 3)。AMOVA分析结果显示物种水平的遗传变异主要存在于居群内部(78%),群体间的遗传分化相对较小(表 5)。
|
|
IBD(isolation by distance)结果显示短柄枹栎居群之间的遗传距离和地理距离没有显著的线性关系(R2=0.011,P=0.07)(图 2)。24个短柄枹栎居群中,河南内乡居群(NX)和安徽宣城居群(XC)之间存在最大的遗传距离,其Nei’s pairwise遗传距离为0.421;而湖北恩施(ES)和浙江杭州居群(HZ)之间有较大的遗传相似性,其遗传距离为0.020。
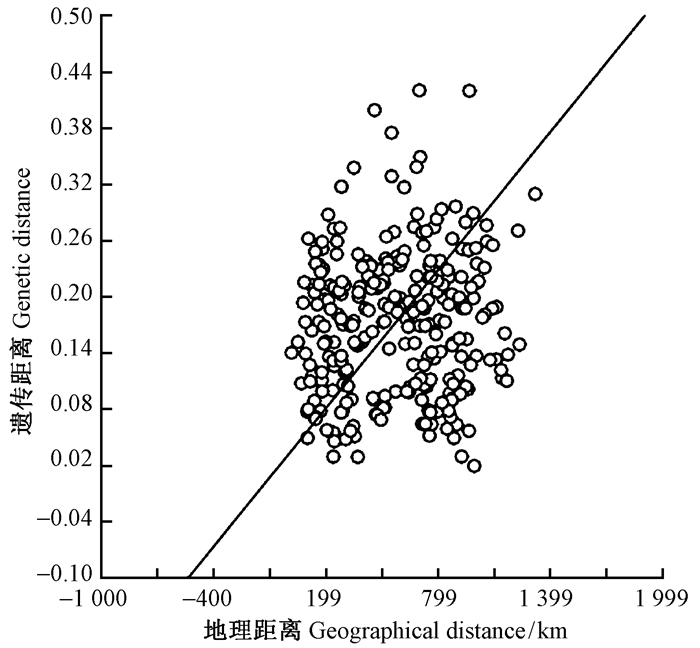 |
图 2 短柄枹栎遗传距离和地理距离的相关性检验(Isolation by Distance) Fig. 2 IBD (Isolation by Distance) test of Q. serrata var.brevipetiolata between the genetic distance and geographical distance |
基于贝叶斯的聚类分析(STRUCTURE)将短柄枹栎的24个居群划分为2组(K=2时,平均的lnP(D)=-6 430.54; ΔK最大值=364.97)(图 3b),居群间均存在一定程度的遗传混杂(基因交流)现象。结合STRUCTURE分组的居群遗传结构分布(图 3a)可看出,虽然居群间存在遗传混杂,但东部和西部群组之间仍存在较为明显的分化。
 |
图 3 基于8对nSSR的短柄枹栎的遗传结构及地理分布 Fig. 3 Genetic structure and geographical distribution of Q. serrata var. brevipetiolata populations based on 8 nSSRs (a)基于STRUCTURE分组的居群地理分布; (b) 基于STRUCTURE的24个短柄枹栎居群的遗传结构(K=2)。(a) Geographical distribution of the genetic groups based on the STRUCTURE analysis; (b) Genetic structure of the 24 Q. serrata var. brevipetiolata populations using STRUCTURE analysis (K=2). |
根据STRUCTURE的分组结论,F统计分析显示西部和东部群组内部的平均基因流(Nm)分别是2.31和2.66,均高于物种整体水平的基因流(1.88)。西部和东部群组的遗传分化系数(FST)分别是0.17和0.18,均低于物种水平的遗传分化(FST=0.22)。分组水平的AMOVA分析也显示东部居群和西部居群之间的遗传差异较小(FST=0.25,P<0.001)(表 5)。
居群间的景观遗传插值分析与STRUCTURE的遗传分组结果一致,显示短柄枹栎的东部居群和西部居群之间存在较高的峰值。分布于西部的居群之间也存在多个峰值,而东部居群之间则显现出较低的遗传插值。位于东部边缘地带的群体之间存在较高的峰值(图 4)。
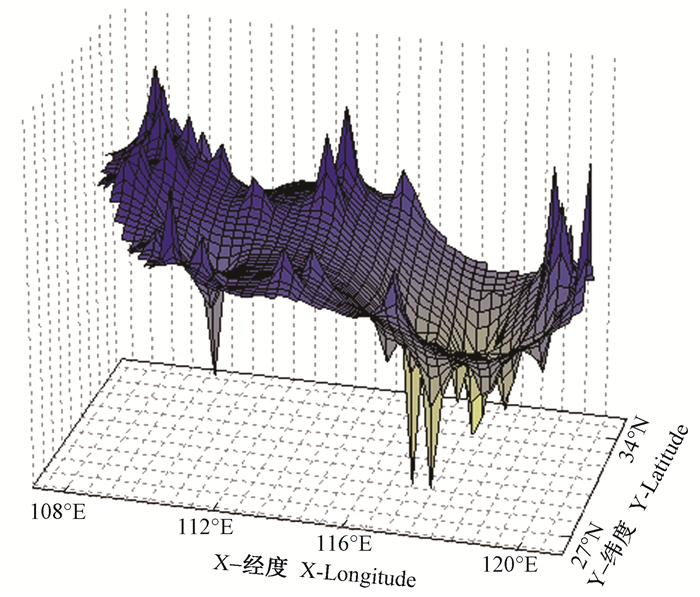 |
图 4 短柄枹栎群体景观遗传插值分析 Fig. 4 Landscape shape interpolation analysis of Q. serrata var. brevipetiolata populations |
遗传多样性是物种长期进化过程中的历史产物,物种遗传多样性水平的高低决定了物种的进化潜力或抵御不良环境因素的能力(陈灵芝,1993)。遗传多样性越高的物种,就越可能更好地适应环境或者更容易存活;相反,遗传多样性越低的物种,则可能更容易受到恶劣环境的影响甚至灭绝(杨梅等,2014)。本研究基于8对nSSR引物对24个短柄枹栎自然居群的遗传多样性进行研究,得到平均的遗传多样性为: He=0.43, I =0.66,PPL=82.81%,显示丰富的遗传多样性(表 4)。目前对多种栎属植物的遗传多样性的研究发现不同地区的栎属物种具有不同的遗传多样性水平。例如,De Greef等(1998)使用RAPD分子标记对无梗栎(Quercus petraea)的种内遗传变异进行了研究,发现其种内存在丰富的遗传变异。Degen等(1999)利用SSR标记研究了德国北部2个夏栎(Quercus robur)居群的遗传结构,发现2个居群均具有较高水平的遗传多样性(Ho=0.898,Na=19.75)。 López-Aljorna等(2007)利用SSR标记研究了西班牙4个区域的欧洲栓皮栎的遗传变异,揭示了该物种较高水平的遗传变异(He=0.65, Ho=0.72,Na=9.33,Ne=4.16)。与国外丰富的遗传多样性研究相比,我国关于栎属植物遗传背景的研究相对较少。李文英(2003)使用等位酶技术以及AFLP技术对我国境内的8个蒙古栎自然群体的遗传多样性水平进行了研究,结果显示该物种的遗传多样性偏低。徐小林等(2004)使用16对SSR引物标记对5个栓皮栎(Quercus variabilis)居群的遗传多样性进行分析,结果显示较高水平的遗传多样性(He=0.81,Ho=0.42,Na=8.44,Ne=5.95,I =1.83)。曹倩等(2014)采用7对SSR引物对短柄枹栎天然林中的4个次生林居群的遗传多样性、遗传结构进行了研究,发现平均的He为0.88,居群之间的遗传杂合度相对较高。然而以上对栓皮栎、短柄枹栎等的研究只采集了物种分布区内较少的居群,居群之间也存在较远的地理距离隔离,并不能完全反映物种水平下的遗传多样性和遗传结构。
尽管本文对24个短柄枹栎居群的遗传多样性研究显示丰富的遗传多样性(表 4),但仍然低于国内外对部分栎属植物的遗传多样性的研究结论。可能的原因如下:首先,短柄枹栎具有较大的有效种群数量以及慢的进化速率,加之居群之间存在一定水平的基因流(Nm=1.88),一定程度上减弱了群体间的遗传分化。其次,短柄枹栎现今的分布区主要集中在中国南部、东部区域,这一区域被认为在第四纪时期未受到大的冰盖覆盖影响,但短柄枹栎居群在冰期时下降的温度环境下仍然可能发生了居群范围的退缩,祖先的有效群体缩小,可能伴随有瓶颈效应的发生,引起祖先基因池中部分等位基因的消失,降低了物种的遗传多样性。第三,对于8个SSR位点的哑基因检测显示大部分位点(除L13外)可能存在哑等位基因,这也是导致等位基因减少的原因,不能完全真实地反映物种的遗传多样性(文亚峰等,2013)。另外,逐渐缩小的群体其遗传多样性也会相应降低(Bruschi et al.,2003),因此近期人为活动的影响(如砍伐、作物替代等)也会影响短柄枹栎的遗传多样性。本研究的调查显示短柄枹栎大部分居群主要由次生林组成(如湖南长沙CS,江苏句容JR,河南内乡NX等居群),人为砍伐影响较大,个体主要以散生方式存在,很少形成纯林,居群很大程度上丢失了祖先的遗传多样性。再者,对于风媒传粉的植物,距离的增加会降低花粉的浓度和传播效率,导致产生的种子数量下降(Silander,1978; Antonovics et al.,1980; House,1993; Ghazoul et al.,1998),距离增加后,孤立的个体容易产生近交种子(Murawski et al.,1991;1992)。本研究结果也表明多数位点存在近交(除了L110外)(表 3),物种缺乏足够的杂合子来维持高的遗传多样性。最后,24个居群平均约17个样本水平也可能会降低短柄枹栎的遗传多样性结论,后期对物种进行密集采样和增加个体数量,以及结合不同类型的分子标记(如叶绿体基因、核基因)能够更准确地反映短柄枹栎的遗传现状。
3.2 短柄枹栎的遗传结构和遗传分化基于贝叶斯的聚类分析(STRUCTURE)(图 3b)和景观遗传插值分析(图 4)将短柄枹栎居群分为2组,地理上大致分布在东、西2个区域(图 3a)。不仅东部区域和西部区域之间存在较为明显的差异,西部区域内居群间也表现出明显的遗传分化。造成这种分化的可能原因是:首先,我国东、西部自然地理环境的差异。西部地区往往多山,而短柄枹栎主要在山脉地区分布,这些复杂的山脉、河流流向对基因流造成阻碍,一定程度上限制了居群间的基因交流,引起居群的分化;东部山脉则海拔较低,地势较为平缓,基因交流障碍小,居群之间的差异也相对较小;东、西部之间较大的遗传差异则源于中部地区平缓的地势所产生的地理隔离(如湖北中东部区域没有短柄枹栎分布,东、西部群体产生的花粉在长距离传播过程中可能失去效力)(Lagache et al.,2013)。其次,生境片段化影响。生境片断化不仅影响生态系统的种类组成、数量结构、生态过程以及非生物因素(Saunders et al.,1991),同时也会对物种的遗传结构产生较大的影响(Templeton et al.,1990;Young et al.,1996)。作者的野外调查研究发现短柄枹栎现今的生活环境差异很大,有些居群在其分布区内形成优势种(如安徽霍山HS、陕西安康AK居群),有些居群则自然生境退化或遭到严重的人为破坏,导致居群分布呈现片段化(如湖南长沙CS、江苏句容JR、河南内乡NX等居群),后者必然会限制群体间和群体内部的基因流,引起遗传分化。因此,短柄枹栎自然居群的遗传结构可能受到了其生活史特征、地理和环境因素、生境片段化以及人类活动的共同作用。
居群分化是由于居群空间上的隔离,突变后环境因子的差异造成选择差异,随机的遗传漂变以及基因流的隔离导致的居群遗传结构的空间异质性(Jacquemyn et al.,2004)。Kremer等(1993)分析得出栎属种内群体间的遗传分化很小,分化系数在0.01~0.17之间,平均值为0.06。本研究中的AMOVA分析(表 5)和F统计(表 3)结果与栎属植物的遗传分化结论一致,表明遗传变异主要存在于居群内部,群体间的遗传差异较小(FST=0.22,P <0.001)。然而短柄枹栎的遗传分化明显高于栎属植物遗传分化的平均值(FST=0.06),也比栓皮栎(FST=0.05)和辽东栎(FST=0.04)的遗传分化值都高,这可能与短柄枹栎主要的东、西2个群组的地理分布有关。中国中部平原地区缺少短柄枹栎的分布,弱化了东、西群体的居群之间的基因交流(西部居群Nm为2.31,东部居群Nm为2.66,总体Nm为1.88)(图 3),加剧了东、西分布群组的遗传分化。相比之下,栓皮栎则具有更广泛的分布范围,而辽东栎则主要分布在我国北部山地,分布相对连续。
4 结 论本研究使用8对nSSR标记对短柄枹栎24个居群的遗传多样性、遗传分化以及整体水平的遗传结构的研究表明,该物种具有丰富的遗传多样性和对应东、西地理分布的遗传分化。作为我国南方森林的主要建群种之一,短柄枹栎的平均遗传多样性(Ho=0.28,I =0.66)低于其他栎属植物的遗传多样性,而群体之间已呈现一定程度的遗传分化(FST=0.22)。前期的采样调查也发现部分居群受人为影响严重退化,结实率很低,具有很低的遗传多样性(如安徽休宁XN,湖南长沙CS居群)。这些居群在遗传保护中需要给予相应的重视。通过统计分析也发现24个居群中有10个居群存在私有等位基因,这些私有等位基因的出现可能是短柄枹栎适应局部环境变化的结果,对维持居群的遗传结构发挥重要作用,也为短柄枹栎的遗传多样性的保持和遗传保护提供了一定的依据。
| [1] |
曹倩,刘义飞,黄宏文. 2014.人工砍伐后短柄枹栎的居群遗传重建研究.热带亚热带植物学报,22(1):68-76. (Cao Q, Liu Y F, Huang H W. 2014. Restoration of genetic diversity of Quercus glandulifera var. brevipetiolata population after artificial logging. Journal of Tropical and Subtropical Botany, 22(1):68-76[in Chinese]).(  3) 3)
|
| [2] |
陈怀琼,隋春,魏建和. 2009.植物SSR引物开发策略简述.分子植物育种,7(4):845-851. (Chen H Q, Sui C, Wei J H. 2009. Summary of strategies for developing SSR primer. Molecular Plant Breeding, 7(4):845-851[in Chinese]).(  1) 1)
|
| [3] |
陈灵芝. 1993.中国的生物多样性——现状及其保护对策.北京:科学出版社. (Chen L Z. 1993. China's biodiversity:Current strategy and its conservation strategy. Beijing:Science Press.[in Chinese])(  1) 1)
|
| [4] |
李文英. 2003.蒙古栎天然群体遗传多样性研宄.北京:北京林业大学博士学位论文. (Li W Y. 2003. Study on genetic diversity of natural populations in Quercus mongolica. Beijing:PhD thesis of Beijing Forestry University[in Chinese]).(  1) 1)
|
| [5] |
刘牧. 2012.蒙古栎和辽东栎的遗传进化关系研究.哈尔滨:东北林业大学硕士学位论文. (Liu M. 2012. The research on genetic evolution relationship of Quercus mongolica and Quercus wutaishanica. Harbin:MS thesis of Northeast Forestry University[in Chinese]).(  2) 2)
|
| [6] |
秦英英,韩海荣,康蜂蜂,等. 2012.基于SSR标记的山西省辽东栎自然居群遗传多样性分析.北京林业大学学报,34(2):61-66. (Qin Y Y, Han H R, Kang F F, et al. 2012. Genetic diversity in natural populations of Quercus liaotungensis in Shanxi Province based on nuclear SSR markers. Journal of Beijing Forestry University, 34(2):61-66[in Chinese]).(  1) 1)
|
| [7] |
文亚峰,韩文军,谢伟东,等. 2013.微卫星标记中的无效等位基因.生物多样性, 21(1):117-126. (Wen Y F, Han W J, Xie W D, et al. 2013. Null alleles in microsatellite markers. Biodiversity Science, 21(1):117-126[in Chinese]).(  1) 1)
|
| [8] |
徐斌,张方秋,潘文,等. 2013.我国红锥天然群体的遗传多样性和遗传结构.林业科学, 49(10):162-166. (Xu B, Zhang F Q, Pan W, et al. 2013. Genetic diversity and genetic structure in natural populations of Castanopsis hystrix from its main distribution in China. Scientia Silvae Sinicae, 49(10):162-166[in Chinese]).(  1) 1)
|
| [9] |
徐小林,徐立安,黄敏仁,等. 2004.栓皮栎天然群体SSR遗传多样性研究.遗传,26(5):683-688. (Xu X L, Xu L A, Huang M R, et al. 2004. Genetic diversity of microsatellites (SSRs) of natural populations of Quercus variabilis. Hereditas, 26(5):683-688[in Chinese]).(  1) 1)
|
| [10] |
徐永椿,任宪威. 1998.中国植物志.北京:科学出版社,235-236. (Xu Y C, Ren X W. 1998. Flora of China. Beijing:Science Press, 235-236.)(  2) 2)
|
| [11] |
杨梅,张敏,师守国,等. 2014.武当木兰种群遗传结构的ISSR分析.林业科学,50(1):76-81. (Yang M, Zhang M, Shi S G, et al. 2014. Analysis of genetic structure of Magnolia sprengeri populations based on ISSR markers. Scientia Silvae Sinicae, 50(1):76-81[in Chinese]).(  1) 1)
|
| [12] |
Aldrich P R, Cavender-Bares J. 2011. Quercus//Kole C. Wild crop relatives:Genomic and breeding resources. Berlin Heidelberg:Springer, 89-129.( 1) 1)
|
| [13] |
Antonovics J, Levin D A. 1980. The ecological and genetic consequences of density-dependent regulation in plants. Annual Review of Ecology and Systematics, 11:411-452.( 1) 1)
|
| [14] |
Bertini C H C, Schuster I, Sediyama T, et al. 2006. Characterization and genetic diversity analysis of cotton cultivars using microsatellites. Genetics and Molecular Biology, 29(2):321-329.( 1) 1)
|
| [15] |
Bohonak A J. 2002. IBD (Isolation By Distance):a program for analyses of isolation by distance. Journal of Heredity, 93(2):153-154.( 1) 1)
|
| [16] |
Bruschi P, Vendramin G G, Bussotti F, et al. 2003. Morphological and molecular diversity among Italian populations of Quercus petraea (Fagaceae). Annals of Botany, 91(6):707-716.( 1) 1)
|
| [17] |
Chen G, Du X M. 2006. Genetic diversity of source germplasm of upland cotton in China as determined by SSR marker analysis. Acta Genetica Sinica, 33(8):733-745.( 1) 1)
|
| [18] |
Chen X M, He Z H, Shi J R, et al. 2003. Genetic diversity of high quality winter wheat varieties (lines) based on SSR markers. Acta Agronomica Sinica, 29(1):13-19.( 1) 1)
|
| [19] |
De Greef B, Triest L, De Cuyper B, et al. 1998. Assessment of intraspecific variation in half-sibs of Quercus petraea (Matt.) Liebl. 'plus' trees. Heredity, 81(3):284-290.( 1) 1)
|
| [20] |
Degen B, Streiff R, Ziegenhagen B. 1999. Comparative study of genetic variation and differentiation of two pedunculate oak (Quercus robur) stands using microsatellite and allozyme loci. Heredity, 83(5):597-603.( 1) 1)
|
| [21] |
Dow B D, Ashley M V, Howe H F. 1995. Characterization of highly variable (GA/CT) n microsatellites in the bur oak, Quercus macrocarpa. Theoretical and Applied Genetics, 91(1):137-141.( 2) 2)
|
| [22] |
Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:A simulation study. Molecular Ecology, 14(8):2611-2620.( 1) 1)
|
| [23] |
Excoffier L, Laval G, Schneider S. 2005. Arlequin (version 3.0):An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, 1(1):47-50.( 1) 1)
|
| [24] |
Ghazoul J, Liston K A, Boyle T J B. 1998. Disturbance-induced density-dependent seed set in Shorea siamensis (Dipterocarpaceae), a tropical forest tree. Journal of Ecology, 86(3):462-473.( 1) 1)
|
| [25] |
Guo W, Cai C, Wang C, et al. 2007. A microsatellite-based, gene-rich linkage map reveals genome structure, function and evolution in Gossypium.Genetics, 176(1):527-541.( 1) 1)
|
| [26] |
Hedrick P W. 2004. Recent developments in conservation genetics. Forest Ecology and Management, 197(1):3-19.( 1) 1)
|
| [27] |
Hornero J, Gallego F J, Martinez I, et al. 2001. Testing the conservation of Quercus spp. microsatellites in the cork oak, Q. suber L.Silvae Genetica, 50(3/4):162-166.( 1) 1)
|
| [28] |
House S M. 1993. Pollination success in a population of dioecious rain forest trees. Oecologia, 96(4):555-561.( 1) 1)
|
| [29] |
Jacquemyn H, Honnay O, Galbusera P, et al. 2004. Genetic structure of the forest herb Primula elatior in a changing landscape.Molecular Ecology, 13(1):211-219.( 1) 1)
|
| [30] |
Kampfer S, Lexer C, Glössl J, et al. 1998. Characterization of (GA)n microsatellite loci from Quercus robur. Hereditas, 129(2):183-186.( 2) 2)
|
| [31] |
Kremer A, Petit R J. 1993. Genetic diversity in natural populations of oak species. Annals of Forest Science, 50(suppl.1):186s-202s.( 1) 1)
|
| [32] |
Lagache L, Klein E K, Guichoux E, et al. 2013. Fine-scale environmental control of hybridization in oaks. Molecular Ecology, 22(2):423-436.( 1) 1)
|
| [33] |
Liu J Q, Sun Y S, Ge X J, et al. 2012. Phylogeographic studies of plants in China:Advances in the past and directions in the future. Journal of Systmatics and Evolution, 50(4), 267-275.( 1) 1)
|
| [34] |
López-Aljorna A, Bueno M Á, Aguinagalde I, et al. 2007. Fingerprinting and genetic variability in cork oak (Quercus suber) elite trees using ISSR and SSR markers. Annals of Forest Science, 64(7):773-779.( 1) 1)
|
| [35] |
Miller M P. 2005. Alleles In Space (AIS):Computer software for the joint analysis of interindividual spatial and genetic information.Journal of Heredity, 96(6):722-724.( 1) 1)
|
| [36] |
Murawski D A, Hamrick J L. 1991. The effect of the density of flowering individuals on the mating systems of nine tropical tree species. Heredity, 67(2):167-174.( 1) 1)
|
| [37] |
Murawski D A, Hamrick J L. 1992. The mating system of Cavanillesia platanifolia under extremes of flowering-tree density:A test of predictions. Biotropica, 24:99-101.( 1) 1)
|
| [38] |
Nei M. 1973. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences, 70(12):3321-3323.( 1) 1)
|
| [39] |
Ortego J, Riordan E C, Gugger P F, et al. 2012. Influence of environmental heterogeneity on genetic diversity and structure in an endemic southern Californian oak. Molecular Ecology, 21(13):3210-3223.( 1) 1)
|
| [40] |
Peakall R O D, Smouse P E. 2006. GENALEX 6:Genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 6(1):288-295.( 1) 1)
|
| [41] |
Pritchard J K, Stephens M, Donnelly P. 2000. Inference of population structure using multilocus genotype data. Genetics, 155(2):945-959.( 1) 1)
|
| [42] |
Saunders D A, Hobbs R J, Margules C R. 1991. Biological consequences of ecosystem fragmentation:A review. Conservation Biology, 5(1):18-32.( 1) 1)
|
| [43] |
Silander Jr J A. 1978. Density-dependent control of reproductive success in Cassia biflora. Biotropica, 10:292-296.( 1) 1)
|
| [44] |
Steinkellner H, Fluch S, Turetschek E, et al. 1997. Identification and characterization of (GA/CT)n microsatellite loci from Quercus petraea. Plant Molecular Biology, 33(6):1093-1096.( 3) 3)
|
| [45] |
Sullivan A R, Lind J F, McCleary T S, et al. 2013. Development and characterization of genomic and gene-based microsatellite markers in North American red oak species. Plant Molecular Biology Reporter, 31(1):231-239.( 5) 5)
|
| [46] |
Tautz D. 1989. Hypervariabflity of simple sequences as general source for polymorphic DNA markers. Nucleic Acids Research, 17(16):6463-6467.( 1) 1)
|
| [47] |
Templeton A R, Shaw K, Routman E, et al. 1990. The genetic consequences of habitat fragmentation. Annals of the Missouri Botanical Garden, 77(1):13-27.( 1) 1)
|
| [48] |
Van Oosterhout C, Hutchinson W F, Wills D P M, et al. 2004. MICRO-CHECKER:Software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4(3):535-538.( 1) 1)
|
| [49] |
Yeh F C, Yang R C, Boyle T. 1999. POPGENE version 1.32, Microsoft window-based freeware for population genetic analysis. Edmonton, Canada:University of Alberta.( 1) 1)
|
| [50] |
Young A, Boyle T, Brown T. 1996. The population genetic consequences of habitat fragmentation for plants. Trends in Ecology & Evolution, 11(10):413-418.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51

