文章信息
- 文亚峰, 韩文军, 周宏, 徐刚标
- Wen Yafeng, Han Wenjun, Zhou Hong, Xu Gangbiao
- 杉木转录组SSR挖掘及EST-SSR标记规模化开发
- SSR Mining and Development of EST-SSR Markers for Cunninghamia lanceolata Based on Transcriptome Sequences
- 林业科学, 2015, 51(11): 40-49
- Scientia Silvae Sinicae, 2015, 51(11): 40-49.
- DOI: 10.11707/j.1001-7488.20151106
-
文章历史
- 收稿日期:2015-06-01
- 修回日期:2015-07-23
-
作者相关文章
2. 中南林业科技大学林学院 长沙 410004;
3. 广东省韶关市林业局 韶关 512000
2. College of Forestry, Central South University of Forestry and Technology Changsha 410004;
3. Shaoguan Forestry Administration, Guangdong Province Shaoguan 512000
微卫星也称SSR(simple sequence repeat,简单重复序列),是以1~6个核苷酸碱基(bp)为重复单元(motif)组成的简单串联重复序列(short t and em repeat)。与同类分子标记技术相比,微卫星标记具有多态性高、呈共显性遗传、重复性好、实验操作容易等特点(Jarne et al., 1996),在人类医学、动物、植物及微生物等学科领域得到了广泛应用。然而,微卫星标记的特异性一定程度上限制了其引物的通用性(Glenn et al., 2005)。对基因序列未知的物种而言,微卫星标记的开发较为困难。传统微卫星标记开发以基因文库构建法(包括SSR富集文库)为主(Hamilton et al., 1999; Zane et al., 2002),其实验过程繁杂、费时费力、效率较低。微卫星标记还可以利用公共基因数据库(NCBI,EMBL,DDBJ)中的共享序列来设计开发,但对于非模式生物或新物种来说,有限的基因序列资源依然是微卫星标记开发的瓶颈。2005年以来,第二代高通量测序技术的发展为规模化遗传变异检测(O’Neill et al., 2013)和标记位点开发(Davey et al., 2011; Yu et al., 2011; Pandey et al., 2013)带来了新机遇。但利用高通量测序数据开发SSR标记,目前仍面临2个方面的挑战: 一是海量测序数据对生物信息学方法和技术的挑战,二是规模化SSR挖掘对标记位点开发的挑战。如何利用高通量测序数据高效、快速地开发微卫星标记位点,是当前分子遗传学领域研究的热点之一。
杉木(Cunninghamia lanceolata)是我国南方重要的用材林树种,具有生长快、产量高、材质好、用途广等特点。但杉木分子遗传学研究基础薄弱,严重滞后于杨树、桉树、松树等其他用材林树种。目前杉木分子研究所用的标记仍以RAPD(尤勇等,1998)、ISSR(齐明,2008)和AFLP(Chung et al., 2004)等显性标记为主。近年来,杉木微卫星标记开发有所报道(张圣等,2013; 徐阳等,2014),其原始序列来源于公共数据库中有限的EST或基因组数据,因而开发得到的SSR位点多态性均不理想。为解决杉木SSR标记数量不足、多态性位点质量差等问题,本文以杉木转录组测序数据为基础,从中批量挖掘SSR,利用四色荧光标记通用引物多重PCR(multiplex-PCR)技术,规模化开发杉木EST-SSR标记。新方法不仅能大幅度提高SSR开发效率,而且能够显著降低开发试验成本,目前已得到了一定数量的高质量EST-SSR多态性位点。SSR标记规模化挖掘与开发新技术的应用,将极大地促进杉木分子遗传学相关研究,对其他非模式生物或新物种SSR标记开发也具有重要的参考作用。
1 材料与方法杉木转录组序列数据(Accession: SRX151872)从NCBI(National Center of Biotechnology Information)的SRA(Sequence Read Archive)数据库下载(http://www.ncbi. nlm.nih.gov/sra/SRX151872)。该转录组以Illumina Genome Analyzer Ⅱ为平台测序获得,20 μg RNA测序样本由杉木种子、叶、茎和根部组织所提取的RNA等量混合而成。
1.1 序列的预处理利用商业软件CLC遗传工作平台5.0(http:// www.clcbio.com)提供的de novo assembly程序对预处理后的转录组数据进行拼接。序列质量控制参数设置为: Removal of low quality sequence,limit=0.05; Removal of ambigious nucleotides,maximal 2 nucleotides allowed。序列拼接完成后,剔除其中100 bp以下的短序列,将序列数据转化为fasta文件待用。
1.3 SSR批量挖掘与引物设计利用CMiB软件包(Ueno et al., 2012)进行规模化SSR挖掘、批量引物设计及电子PCR。CMiB有效组合了CD-HIT-EST(Li et al., 2006)、MISA(Thiel et al., 2003)、Primer3(Rozen et al., 2000)、ipcress(Slater et al., 2005)和BlastCLUST(Altschul et al., 1990)等程序,其操作流程如图 1所示: 首先用MISA软件对拼接得到的contigs进行SSR检测,得到含有设定参数(二、三、四、五、六核苷酸重复单元,其重复数分别须大于6,5,4,3,3)的SSR序列文件; CD-HIT-EST程序对含有SSR的EST序列进行聚类并排序(按序列长度从大到小),剔除其中相似或相同的冗余序列; Primer3用于批量引物设计; ipcress程序根据所设计的引物进行电子PCR,得到特定位点可能的PCR产物序列; 最后,BlastCLUST程序会对电子PCR产物进行聚类排序,筛选其中最短的序列作为该位点的最优扩增结果。
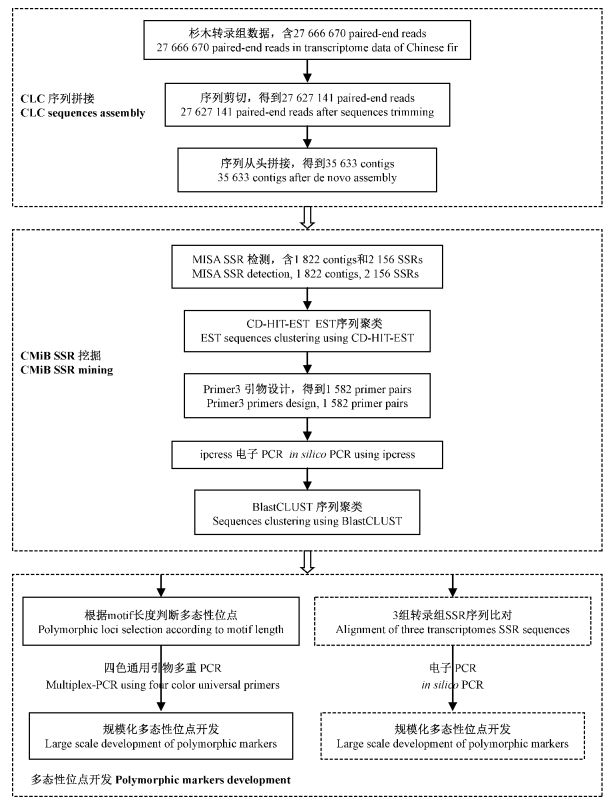 |
图 1 利用杉木转录组序列开发EST-SSR标记技术流程 Fig. 1 Flow chart for EST-SSR markers development from transcriptome sequences of Cunninghamia lanceolata |
转录组拼接序列(contigs)经CMiB软件包处理后,最终得到的是所有SSR位点的详细信息,包括检测序列中所含的SSR数量、SSR重复单元频率与分布、SSR位点引物对及电子PCR产物长度等。
1.4 四色荧光标记通用引物多重PCR检测技术从挖掘得到的SSR序列中选择motif重复数大于9的35个位点合成引物,正向引物5′端添加通用序列接头(A,B,C或D,其序列分别为: Tail A 5′-GCCTCCCTCGCGCCA-3′; Tail B 5′-GCCTTGCC AGCCCGC-3′; Tail C 5′-CAGGACCAGGCTACCGTG-3′; Tail D 5′-CGGAGAGCCGAGAGGTG-3′),利用四色荧光标记通用引物多重PCR技术(Blacket et al., 2012)对候选位点的多态性进行规模化检测。多重PCR扩增体系中含有3个引物,包括添加了通用序列接头的正向引物(F)、反向引物(R)以及有荧光标记的通用引物Tail A(FAM)、Tail B(HEX)、Tail C(NED)或Tail D(PET)。同一PCR反应中,正向引物(F)须与相应的荧光标记通用引物一致。四色荧光标记通用引物多重PCR基因分型的步骤如下:
首先,选用2个杉木样本材料检测候选位点能否成功扩增,单个位点PCR扩增按QIAGEN® Multiplex PCR 试剂盒方法进行,PCR反应体系(10.0 μL)中含2 × Multiplex PCR master混合液5.0 μL,10 × 引物混合液1.0 μL和5~10 ng DNA模板1.0 μL。10 × 引物混合液由1.0 μmol·L-1的荧光标记通用引物(Tail A,Tail B,Tail C或Tail D)、2.0 μmol·L-1正向引物(F)和2.0 μmol·L-1反向引物(R)组成。PCR反应条件为: 95 ℃预变性15 min; 94 ℃变性30 s,60 ℃退火90 s,72 ℃延伸60 s,共35个循环; 60 ℃延伸30 min。扩增产物利用2%的琼脂糖凝胶电泳检测其是否能成功扩增(有无扩增条带)。
其次,利用多重PCR技术对成功扩增位点的多态性进行检测。16个杉木优树单株(不同家系或无性系)用于评估其多态性,其中12株(Y6,Y18,J5,J80,Ht14,Ht16,Jh10,Jh16,Y26,J18,2-110,2-1116)来源于湖南省攸县杉木种子园(27°18′ N,113°47′ E),4株(Lc6,Lc12,Lc18,Lc418)来源于广东省乐昌市龙山杉木种子园(25°12′ N,113°28′ E)。多重PCR反应体系(10.0 μL)中含2× Multiplex PCR master 混合液5.0 μL,10×引物混合液1.0 μL,5~10 ng DNA模板1.0~2.0 μL(根据引物数量进行调整)。10 ×引物混合液由5~6个候选位点引物混合而成,其中含0.5~2.0 μmol·L-1的荧光标记通用引物(引物浓度根据不同颜色荧光强度或基因片段峰值大小进行调整)、2.0 μmol·L-1正向引物(F)和2.0 μmol·L-1反向引物(R),PCR反应条件与第1轮相同。多重PCR产物稀释5~10倍后,在ABI3100 测序仪上基因分型(Liz 600为内标),GeneScan收集基因分型结果,基因片段分析用Genotyper3.7软件。
最后,利用GenAlEx 6.3软件(Peakall et al., 2006)分析不同位点遗传多样性参数,包括每个位点的等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)、近交系数(FIS)等。Ms-tool软件用于位点多态性信息含量分析,Genepop v4.0.10(http://genepop. curtin.edu.au/index.html)用于Hardy-Weinberg和Linkage Disequilibrium检验,无效等位基因检测用Micro-check 2.2.3软件,Blast2GO软件对含有SSR的EST序列进行基因功能注释。
1.5 多态性位点的有效性评估利用GenAlEx 6.3软件对16个杉木优树(不同家系或无性系)进行主成分分析(PCA),确定其遗传关系。并根据不同优树的来源和已知遗传信息,对多态性位点的有效性进行评估。
2 结果与分析 2.1 转录组序列拼接结果杉木转录组序列经CLC剪切(去除低质量和污染序列)后有27 627 141 paired-end reads用于后续拼接。de novo assembly序列拼接结果见表 1,共得到35 633个contigs,其中最小拼接长度155 bp,最大23 794 bp,总长度31.5 Mb,平均长度884 bp,N75,N50,N25长度分别达到了705,1 396,2 218 bp。
|
|
35 633个contigs中挖掘得到2 156个SSR位点,分布于1 822个contigs中,其中256个contigs中包含1个以上SSR位点,复合型SSR数量为118个,SSR平均分布密度为68.4个·Mb-1。杉木SSR不同重复类型数量与分布比例见表 2,其中三核苷酸SSR重复类型数量最大,有900个,占SSR总数的41.7%; 其次为六核苷酸重复单元类型和五核苷酸重复类型,分别占SSR位点总数的29.8%和12.7%; 二核苷酸重复类型较少,占总数的11.1%。在三核苷酸重复类型的位点中,AAG重复单元分布频率最高,共有239个,占该重复类型位点数的26.6%。最高比例的二核苷酸重复单元是AG和AT,各有109个,分别占45.6%(图 2)。
|
|
 |
图 2 杉木二、三核苷酸重复单元SSR分布频率及重复数 Fig. 2 Frequency distribution of di- and tri-SSRs by motif and repeats number of C. lanceolata |
试验发现,多重PCR扩增及基因分型过程中,不同引物的荧光强度(基因片段峰值)存在较大差异。可以通过调整荧光标记通用引物的浓度,使不同颜色引物的荧光强度达到较为一致的水平,以便于基因型检测和分析。Tail C(NED)引物荧光强度最强,其浓度以0.5 μmol·L-1为宜,Tail B(HEX)引物浓度以1.0 μmol·L-1为宜,Tail A(FAM)引物浓度以1.5 μmol·L-1为宜,而Tail D(PET)引物的荧光强度普遍较弱,浓度以2.0 μmol·L-1较为合适。如果不同颜色引物的荧光强度均较低,可将DNA模板的量增加到2.0 μL,以满足多个引物对DNA量的较大需求。
35个候选标记位点中,29个成功扩增位点(表 3)根据其正向引物F端接头所对应的通用引物荧光标记颜色(FAM,HEX,NED或PET),以及各位点扩增片段分子量大小,可分为5个引物组合(每组各含5~6个位点),即需要5次PCR和基因分型可完成全部29个位点的多态性检测。与早期所使用的通用引物(M13等)基因分型方法相比较,本试验所建立的杉木四色荧光标记通用引物基因分型方法结果清晰、稳定可靠(图 3),不但试验成本仅为原来的10%~15%,而且,结合多重PCR扩增技术,使试验效率提高了5~6倍。
|
|
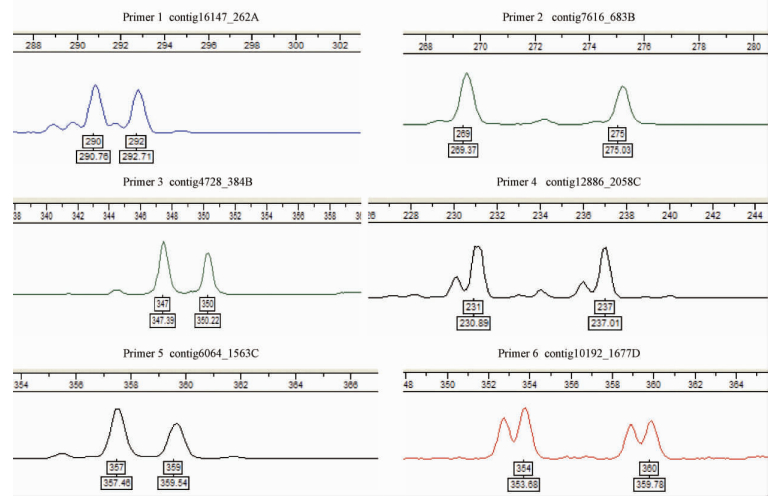 |
图 3 同一引物组合中6个EST-SSR位点多重PCR基因分型结果 Fig. 3 GeneScan result of 6 EST-SSR loci in a same primer combination by multiplex PCR |
2 156个SSR位点中有1 582个能成功设计引物,占SSR位点总数的73.4%。从二、三核苷酸重复单元类型中选择SSR重复数大于9的位点35个合成引物,从中筛选多态性位点。16个杉木样本基因分型结果表明,所检测的35个EST-SSR位点中,5个位点没有扩增成功(无扩增条带),1个位点有杂带(条带数量大于2),29个位点产生1~2条带(表 3),其中28个位点具有多态性(Wen et al., 2013),多态性位点比率达到80%。多态性位点的等位基因数为3~12不等,位点多态信息含量(PIC)变动幅度为0.210~0.881,平均为0.573,大于0.5,说明所开发的EST-SSR位点具有很高的多态性。
2.5 多态性位点的有效性PCA分析结果(图 4)显示,16个杉木优良单株可被清楚地分开,其中来源于乐昌市龙山杉木种子园的4株优树(Lc6,Lc12,Lc18和Lc418)位于二维散点图的右上端,彼此间显示出较近的亲缘关系。来源于湖南攸县杉木种子园的12株优树,在图中分布较为松散,部分个体间有较远的亲缘关系,这是因为这些优树来源于地理距离较远的不同种源。PCA分析结果表明,28个EST-SSR多态性位点具有很强的鉴别杉木不同地理种源,甚至同一种源不同单株的能力。
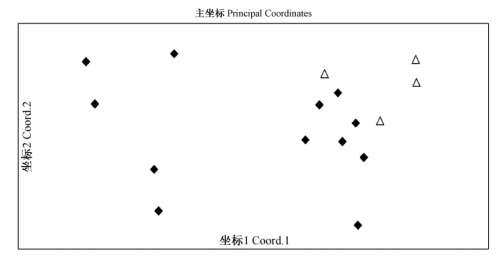 |
图 4 来源于2个种子园16个杉木优树的PCA分析结果
Fig. 4 PCA result of 16 individuals from two seed orchards of C. lanceolata
△ 优树来源于乐昌市龙山杉木种子园 Plus trees collected from Lechang seed orchard of Chinese fir ◆ 优树来源于湖南攸县杉木种子园 Plus trees collected from Youxian seed orchard of Chinese fir |
本文利用公共序列数据库(NCBI)中的杉木转录组开发EST-SSR标记位点,得到以下主要结论: 1)与传统SSR标记开发技术相比较,建立的杉木EST-SSR高效开发流程和方法有效结合了海量序列数据挖掘与通用引物多重PCR技术,大幅度提高了SSR标记开发效率,显著降低了试验成本。2)杉木转录组SSR平均分布密度为68.4个·Mb-1,其中,三核苷酸和六核苷酸是主要的SSR重复类型,占SSR位点总数的71.5%。开发得到的28个EST-SSR位点多态性高,分辨率强,能有效用于杉木遗传多样性保护研究。
4 讨论 4.1 杉木转录组SSR和EST-SSR位点特征杉木转录组SSR平均分布密度为68.4个·Mb-1,明显低于日本柳杉(Cryptomeria japonica)(Ueno et al., 2012),但高于火炬松(Pinus taeda)、白云杉(Picea glauca)、恩格曼云杉(Picea engelmannii)和北美云杉(Picea sitchensis)(Bérubé et al., 2007)。SSR重复类型(motif)中,三核苷酸重复类型数量最大,有900个,占SSR总数的41.7%; 其次为六核苷酸重复类型,占SSR位点总数的29.8%。这可能与基因密码子有关,生物体信使RNA分子上的3个碱基决定1个氨基酸,EST序列位于基因的编码区,直接与功能基因表达相关,因此转录组序列中三核苷酸、六核苷酸SSR重复类型的比例也相应较大。
与之前杉木SSR位点的开发研究相比(张圣等,2013; 徐阳等,2014),本研究以杉木转录组序列为基础,海量序列为高质量多态性位点的选择提供了充分的数据保证。所合成的35个引物中,多态性位点比例达到了80%。根据作者的经验,SSR位点重复单元的重复数如果小于6,则很难产生多态性。本次开发得到的28个EST-SSR多态性位点,SSR重复单元重复数均超过9,有18个位点的多态性信息含量(PIC)大于0.5,是高多态性位点。16个优良单株的检测表明,28个位点的等位基因数3~12不等,平均为5.71。这些EST-SSR位点已用于杉木遗传多样性保护研究。
4.2 利用第二代测序技术开发SSR位点的共性问题第二代高通量测序技术解决了传统微卫星标记开发的瓶颈问题,开启了大规模微卫星标记开发的新时代(程晓凤等,2011; Castoe et al., 2012; Zalapa et al., 2012)。在当前高通量测序成本依然较高、研究经费有限的情况下,利用公共基因序列数据库(NCBI,EMBL和DDBJ等)中的共享第二代测序数据不失为明智选择。目前,基于第二代测序技术开发微卫星位点依然存在诸多共性问题,值得进一步探讨。
4.2.1 生物信息学软件的比较序列拼接是测序数据处理的前提与基础,对于高通量测序产生的海量短序列,拼接与组装显得尤为重要。目前有多种序列拼接软件可供选择使用,但还没有一个中立的机构能够对这些软件的拼接质量和性能进行系统、公正的评估。Bräutigam等(2011)对目前常用的6种拼接软件(SOAP,Velvet,MIRA,CAP3,TGICL,CLC)的性能进行了模拟评价,评价参数包括拼接后的contigs数量、未拼接的reads百分率、N25、N50长度等。模拟试验结果表明CAP3,TGICL,CLC拼接质量稳定可靠,适合用于非模式物种序列从头拼接(de novo assembling),而且TGICL和CLC在处理错误(变异)序列方面性能更胜一筹。因此,本文选用CLC软件对杉木转录组序列进行拼接,共得到35 633个contigs,平均长度为884 bp。Qiu等(2013)利用SOAP软件也对该转录组进行过拼接,得到59 669个unigenes,平均长度为497 bp。从2个软件的拼接结果来看,CLC的拼接质量明显好于SOAP。
规模化SSR挖掘软件除早期的SSRIT,Sputnik,TROLL和MISA外,近年来,海量序列数据促生了包括CMiB(Ueno et al., 2012),ESMP(Sarmah et al., 2012),SSRLocator(da Maia et al., 2008)以及QDD(Meglécz et al., 2010)等大型软件的出现,这类软件整合了SSR检测、引物设计、电子PCR等多个程序,实现了SSR标记开发的高效化和自动化,可选择这类软件来提高SSR的开发效率。
4.2.2 提高多态性位点选择效率的方法第二代高通量测序技术显著提高了SSR开发效率和通量。但如何从数以万计的SSR中鉴别、筛选多态性位点,是面临的又一挑战。短序列拼接错误、电子PCR错误等都有可能使筛选位点无PCR产物或片段大小与预期不一致。因此,通过序列拼接得到的SSR位点必须经过PCR试验验证,确认其真实性后才能用于相关研究。可以根据研究材料(新物种或重测序物种)、研究目的(遗传多样性或遗传图谱构建)、所需标记位点数量的不同,灵活运用重复单元(motif)长度判断法和电子(in silico)预测法,借助多重PCR扩增技术进一步提高多态性位点选择效率。
较多的试验研究表明,微卫星位点的多态性与SSR长度(重复序列的长度)存在正相关(Smulders et al., 1997; He et al., 2003;Ueno et al., 2012),完美型SSR位点的多态性高于复合型(Buschiazzo et al., 2006)。可以根据重复单元长度判断法,选择具有较长SSR的完美型位点来设计引物以提高多态性检测效率。本研究中所选择的35个候选位点的重复单元重复数均大于9,检测发现其中28个具有多态性,多态性位点比例达到了80%,远高于其他相关研究结果。
电子预测法(in silico)是近年发展起来的大规模多态性位点选择方法(Tang et al., 2008; Hoffman et al., 2011; Victoria et al., 2011; Duran et al., 2013)。其原理是在序列拼接的基础上,借助序列比对软件对contigs中的SSR变异进行检测,以contigs中的等位基因数来判断(预测)特定位点的多态性。利用该方法的前提是原始序列中至少应包含2个或2个以上个体(样本)的序列数据。今后还可以通过多组杉木转录组数据的比较(SRX151872,SRX139598和SRX320120),进一步判断所获得的1 582个EST-SSR位点的多态性(图 1右下虚线部分)。
四色荧光标记通用引物多重PCR技术能将不同颜色标记(FAM,NED,HEX或PET)的通用引物和不同位点的SSR引物置于同一PCR反应中进行扩增,利用毛细管电泳技术一次获得多个位点的多态性信息,具有高效、高产率、低成本等优点(Blacket et al., 2012)。针对大规模多态性位点选择,可以利用该技术提高多态性位点选择效率。本试验中,利用建立的杉木多重PCR扩增技术,将5~6对不同引物置于同一PCR反应中进行扩增,基因分型结果清晰、稳定,显著提高了多态性位点开发效率,降低了试验成本。
当前,第三代测序技术呼之欲出,更高精度、更长读长、更低成本的单分子测序技术将使全基因组遗传变异分析变成现实。目前尚不能预测基于局部位点检测的分子标记技术的变化和发展前途,但可以肯定的是,第三代测序技术能有效解决大规模微卫星标记开发中的难题,“一条染色体,一个contig”的序列拼接目标会使SSR标记开发、功能基因定位更为便捷。同样,高精度的多样本平行测序会使大规模多态性位点电子检测成为可能,真正实现多态性位点选择的规模化和高效化。
| [1] |
程晓凤,黄福江,刘明典,等. 2011. 454测序技术开发微卫星标记的研究进展.生物技术通报, (8):82-90. (Cheng X F, Huang F J, Liu M D, et al. 2011. Development of microsatellite markers using 454 pyrosequencing. Biotechnology Bulletin, (8):82-90[in Chinese]).(  1) 1)
|
| [2] |
齐明. 2008.杉木远交亲本群体遗传多样性研究.植物研究, 28(3):299-303. (Qi M. 2008. Genetic diversity of wide cross population of Cunninghamia lanceolata and Platycladus orientalis. Bulletin of Botanical Research, 28(3):299-303[in Chinese]).(  1) 1)
|
| [3] |
徐阳,陈金慧,李亚,等. 2014.杉木EST-SSR与基因组SSR引物开发.南京林业大学学报, 38(1):9-14. (Xu Y, Chen J H, Li Y, et al. 2014. Development of EST-SSR and genomic-SSR in Chinese fir. Journal of Nanjing Forestry University, 38(1):9-14[in Chinese]).(  2) 2)
|
| [4] |
尤勇,洪菊生.1998. RAPD标记在杉木种源遗传变异上的应用.林业科学, 34(4):33-38. (You Y, Hong J S. 1998. Application of RAPD marker of genetic variation of Chinese fir provenances. Scientia Silvae Sinicae, 34(4):33-38[in Chinese]).(  1) 1)
|
| [5] |
张圣,黄华宏,林二培,等. 2013.杉木与台湾杉EST-SSR标记的开发与应用.林业科学, 49(10):173-180. (Zhang S, Huang H H, Lin E P, et al. 2013. Development and application of EST-SSR markers for Cunninghamia lanceolata and Taiwania cryptomerioides. Scientia Silvae Sinicae, 49(10):173-180[in Chinese]).(  2) 2)
|
| [6] |
Altschul S F, Gish W, Miller W, et al. 1990. Basic local alignment search tool. Journal of Molecular Biology, 215(3):403-410.( 1) 1)
|
| [7] |
Bérubé Y, Zhuang J, Rungis D, et al. 2007. Characterization of EST-SSRs in loblolly pine and spruce. Tree Genetics and Genomes, 3(3):251-259.( 1) 1)
|
| [8] |
Blacket M J, Robin C, Good R T, et al. 2012. Universal primers for fluorescent labelling of PCR fragments-an efficient and cost-effective approach to genotyping by fluorescence. Molecular Ecology Resources, 12(3):456-463.( 2) 2)
|
| [9] |
Bräutigam A, Mullick T, Schliesky S, et al. 2011. Critical assessment of assembly strategies for non-model species mRNA-Seq data and application of next-generation sequencing to the comparison of C3 and C4 species. Journal of Experimental Botany, 62(9):3093-3102.( 1) 1)
|
| [10] |
Buschiazzo E, Gemmell N J. 2006. The rise, fall and renaissance of microsatellites in eukaryotic genomes. BioEssays, 28(10):1040-1050.( 1) 1)
|
| [11] |
Castoe T A, Poole A W, de Koning A P J, et al. 2012. Rapid microsatellite identification from Illumina paired-end genomic sequencing in two birds and a snake. PLoS ONE, 7(2):e30953.( 1) 1)
|
| [12] |
Chung J D, Lin T P, Tan Y C, et al. 2004. Genetic diversity and biogeography of Cunninghamia konishii (Cupressaceae), an island species in Taiwan:a comparison with Cunninghamia lanceolata, a mainland species in China. Molecular Phylogenetics and Evolution, 33(3):792-801.( 1) 1)
|
| [13] |
da Maia L C, Palmieri D A, de Souza V Q, et al. 2008. SSRLocator:tool for simple sequence repeat discovery integrated with primer design and PCR simulation. International Journal of Plant Genomics, doi:10.1155/2008/412696.( 1) 1)
|
| [14] |
Davey J W, Hohenlohe P A, Etter P D, et al. 2011. Genome-wide genetic marker discovery and genotyping using next-generation sequencing. Nature Reviews Genetics, 12(7):499-510.( 1) 1)
|
| [15] |
Duran C, Singhania R, Raman H, et al. 2013. Predicting polymorphic EST-SSRs in silico. Molecular Ecology Resources, 13(3):538-545.( 1) 1)
|
| [16] |
Glenn T C, Schable N A. 2005. Isolating microsatellite DNA loci. Methods in Enzymology, 395:202-222.( 1) 1)
|
| [17] |
Hamilton M B, Pincus E L, Di-Fiore A, et al. 1999. Universal linker and ligation procedures for construction of genomic DNA libraries enriched for microsatellites. BioTechniques, 27(3):500-507.( 1) 1)
|
| [18] |
He C, Poysa V, Yu K. 2003. Development and characterization of simple sequence repeat (SSR) markers and their use in determining relationships among Lycopersicon esculentum cultivars. Theoretical and Applied Genetics, 106(2):363-373.( 1) 1)
|
| [19] |
Hoffman J I, Nichols H J. 2011. A novel approach for mining polymorphic microsatellite markers in silico. PLoS ONE, 6(8):e23283.( 1) 1)
|
| [20] |
Jarne P, Lagoda P J L. 1996. Microsatellites, from molecules to populations and back. Trends in Ecology and Evolution, 11(10):424-429.( 1) 1)
|
| [21] |
Li W, Godzik A. 2006. Cd-hit:a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics, 22:1658-1659.( 1) 1)
|
| [22] |
Meglécz E, Costedoat C, Dubut V, et al. 2010. QDD:a user-friendly program to select microsatellite markers and design primers from large sequencing projects. Bioinformatics, 26(3):403-404.( 1) 1)
|
| [23] |
O'Neill E M, Schwartz R, Bullock C T, et al. 2013. Parallel tagged amplicon sequencing reveals major lineages and phylogenetic structure in the North American tiger salamander (Ambystoma tigrinum) species complex. Molecular Ecology, 22(1):111-129.( 1) 1)
|
| [24] |
Pandey G, Misra G, Kumari K, et al. 2013. Genome-wide development and use of microsatellite markers for large-scale genotyping applications in foxtail millet[Setaria italica (L.)]. DNA Research, 20(2):197-207.( 1) 1)
|
| [25] |
Peakall R, Smouse P E. 2006. GENALEX 6:genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 6(1):288-295.( 1) 1)
|
| [26] |
Qiu Z B, Wan L C, Chen T, et al. 2013. The regulation of cambial activity in Chinese fir (Cunninghamia lanceolata) involves extensive transcriptome remodeling. New Phytologist, 199(3):708-719.( 1) 1)
|
| [27] |
Rozen S, Skaletsky H J. 2000. Primer3 on the WWW for general users and for biologist programmers. Bioinformatics Methods and Protocols, 132:365-386.( 1) 1)
|
| [28] |
Sarmah R, Sahu J, Dehury B, et al. 2012. ESMP:a high-throughput computational pipeline for mining SSR markers from ESTs. Bioinformation, 8(4):206-208.( 1) 1)
|
| [29] |
Slater G S, Birney E. 2005. Automated generation of heuristics for biological sequence comparison. BMC Bioinformatics, 6(4):31.( 1) 1)
|
| [30] |
Smulders M J M, Bredemeijer G, Rus-Kortekaas W, et al. 1997. Use of short microsatellites from database sequences to generate polymorphisms among Lycopersicon esculentum cultivars and accessions of other Lycopersicon species. Theoretical and Applied Genetics, 94(2):264-272.( 1) 1)
|
| [31] |
Tang J F, Baldwin S J, Jacobs J M E, et al. 2008. Large-scale identification of polymorphic microsatellites using an in silico approach. BMC Bioinformatics, 9:374.( 1) 1)
|
| [32] |
Thiel T, Michalek W, Varshney R, et al. 2003. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L). Theoretical and Applied Genetics, 106(3):411-422.( 1) 1)
|
| [33] |
Ueno S, Moroguchi Y, Uchiyama K, et al. 2012. A second generation framework for the analysis of microsatellites in expressed sequence tags and the development of EST-SSR markers for a conifer, Cryptomeria japonica. BMC Genomics, 13:136.( 4) 4)
|
| [34] |
Victoria F C, da Maia L C, de Oliveira A C. 2011. In silico comparative analysis of SSR markers in plants. BMC Plant Biology, 11:15.( 1) 1)
|
| [35] |
Wen Y, Ueno S, Han W, et al. 2013. Development and characterization of 28 polymorphic EST-SSR markers for Cunninghamia lanceolata (Taxodiaceae) based on tranxcriptome sequences. Silave Gentica, 62(3):137-141.( 1) 1)
|
| [36] |
Yu J N, Won C, Jun J, et al. 2011. Fast and cost-effective mining of microsatellite markers using NGS technology:an example of a Korean water deer Hydropotes inermis argyropus. PLoS ONE, 6:e26933.( 1) 1)
|
| [37] |
Zalapa J E, Cuevas H, Zhu H Y, et al. 2012. Using next-generation sequencing approaches to isolate simple sequence repeat (SSR) loci in the plant sciences. American Journal of Botany, 99(2):193-208.( 1) 1)
|
| [38] |
Zane L, Bargelloni L, Patarnello T. 2002. Strategies for microsatellite isolation:A review. Molecular Ecology, 11(1):1-16.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51
