文章信息
- 占明明, 杨毅, 程子彰, 苏光灿, 胡伟, 陈华萍, 黄乾明
- Zhan Mingming, Yang Yi, Cheng Zizhang, Su Guangcan, Hu Wei, Chen Huaping, Huang Qianming
- 基于SRAP标记的油橄榄品种遗传多样性分析
- Genetic Diversity of Olive Varieties Based on SRAP Markers
- 林业科学, 2015, 51(1): 157-164
- Scientia Silvae Sinicae, 2015, 51(1): 157-164.
- DOI: 10.11707/j.1001-7488.20150119
-
文章历史
- 收稿日期:2014-04-01
- 修回日期:2014-10-22
-
作者相关文章
2. 四川农业大学生命科学学院 雅安 625014;
3. 四川省凉山州中泽新技术开发有限责任公司 西昌 615000
2. College of Life Sciences, Sichuan Agricultural University Ya'an 625014;
3. Sichuan Liangshan New Technology Development Co., Ltd. Xichang 615000
油橄榄(Olea europaea)为木犀科(Oleaceae)木犀榄属(Olea)常绿乔木,早期在环地中海和小亚细亚地区栽培,因其橄榄油中高含量的单不饱和脂肪酸以及酚类、维生素等天然营养成分而被誉为“地中海的液体黄金”,目前油橄榄已成为地中海地区重要农业资源之一(Bandelj et al.,2004)。近几十年来在美国、澳大利亚、南非等国家已有大量引种栽培,20世纪60年代我国首次从阿尔巴尼亚引种油橄榄并成功栽培(Breton et al.,2006)。
油橄榄经过长时间的进化与变异,其遗传资源相当丰富,在形态学和农艺学方面都具有丰富的遗传多样性,包括大量的栽培型和野生型(Cantini et al.,1999)。据Rugini等(1992)统计全世界共有2 600个油橄榄栽培品种,仅在意大利就有至少300个品种。我国从20世纪60年代起大量引种油橄榄,目前已经取得很大进展。随着人们对油橄榄价值认识度的提高,橄榄油的需求量必将与日俱增,如何提高我国橄榄油产量与质量,这就对油橄榄遗传多样性研究和种质鉴定提出了更高的要求。
目前国外利用分子标记对油橄榄进行遗传多样性分析、遗传连锁图谱构建和基因定位研究(De la Rosa et al.,2003;Wu et al.,2004)已相当成熟,RAPD(Besnard et al.,2001;Guerin et al.,2002;Martins-Lopes et al.,2008)、AFLP(Angiolillo et al.,2006;Montemurro et al.,2008)、SSR(Cipriani et al.,2002;Dastkar et al.,2013;Erre et al.,2010)、ISSR(ElSaied et al.,2012;Gomes et al.,2009;Souza et al.,2012)、SRAP(Işket al.,2011)等DNA分子标记技术在国外已被广泛应用于油橄榄遗传育种研究,而国内利用分子标记研究油橄榄遗传多样性的工作还处于起步阶段,仅出现少量、单一的RAPD、SSR和ISSR研究报道。马万里等(2006)和邱源等(2008)分别将RAPD技术用于油橄榄品种研究,李金花等(2012)利用荧光SSR标记对油橄榄品种进行了遗传多样性分析,陈海云等(2013)和宁德鲁等(2013)分别利用ISSR技术对油橄榄品种做了遗传多样性和聚类分析,而运用SRAP技术对油橄榄进行遗传多样性和分子鉴定的研究国内尚未见报道。
SRAP(sequence-related amplified polymorphism)是一种基于PCR技术的新型标记系统,与AFLP和RAPD标记相比,具有操作简单、重复性好和多态性高等优势(Li et al.,2001)。本研究目的在于利用SRAP标记技术对采自四川西昌市的32个油橄榄品种进行遗传多样性分析,为油橄榄品种鉴定和遗传育种提供理论依据。
1 材料与方法 1.1 试验材料试验材料采自四川西昌市中泽公司油橄榄品种园,每个品种选取1个单株。共32个品种(表 1),其中5个为我国选育的品种(表 2)。
|
|
|
|
1)基因组DNA的提取采用改良的CTAB法从油橄榄幼嫩叶片中提取总DNA,在0.8%琼脂糖凝胶上检测所提样品DNA的质量。根据测定的OD260值计算出DNA浓度,将所提基因组DNA稀释至50 ng·μL-1工作浓度,-20 ℃保存备用。
2)SRAP分析采用Li等(2001)和Budak等(2004)所提出的引物(表 3),在此基础上组成208对引物,最终筛选出25对扩增产物多态性高、易于统计分析的引物组合(表 4)。PCR反应总体系为20 μL,包括2×Taq PCR Master Mix 10 μL,上、下游引物(0.2 μmol·L-1)各1 μL,DNA(50 ng·μL-1)1 μL,ddH2O 7 μL。扩增程序为:94 ℃预变性5 min;94 ℃变性40 s、35 ℃退火35 s、72 ℃延伸1 min,5个循环;94 ℃变性40 s、50 ℃退火35 s、72 ℃延伸1 min,35个循环;72 ℃延伸8 min,4 ℃保存。用8%的非变性聚丙酰胺凝胶电泳对扩增产物进行分离、检测,电泳缓冲液为1×TBE,120 V电压下电泳1.5 h。电泳结束,用EB(1 ng·mL-1)对凝胶进行20 min染色处理,染色结束后利用DocTM EZ System(Bio-Rad)凝胶成像系统对其拍照、保存。
|
|
|
|
3)数据处理与分析根据电泳条带的有或无,以0,1二元型数据记录不同标记的电泳谱带结果,即在相同位置有条带记为“1”,无条带记为“0”。利用NTsys-pc 2.10e软件(Rohlf,2000)中的UPGMA算法对32个油橄榄品种构建树状图和主坐标分析(PCA)图。用Cervus Version 3.0.3软件计算多态性信息含量(PIC)、期望杂合度(He)和一致性概率(PI)。
2 结果与分析 2.1 SRAP标记多态性利用25对筛选出的SRAP引物组合(表 4)对32个油橄榄基因组DNA进行扩增,共得到293条多态性谱带,平均每对引物扩增出11.72条多态性条带。其中引物对M5E5扩增出的多态性条带数量最多(图 1),为37条;引物对M3E11,M9E16,M12E9扩增出的多态性条带数量最少,为6条。所有引物对扩增条带大小为25~2 000 bp,大部分条带分布在50~1 000 bp。
 |
图 1 引物组合M5E5扩增结果 Fig. 1 Amplified result of primer M5E5 M: pBR322 DNA/Msp I. |
根据计算(表 4),所有引物扩增条带平均多态性比率为90.75%,其中引物对M1E14,M2E10,M2E15,M3E14,M4E11,M4E12,M5E5,M5E9,M5E16,M11E8扩增的多态性条带比率均为100.0%,M3E3扩增的多态性条带比率最低,为64.30%。PIC分析结果表明25对SRAP引物具有较高的扩增DNA多态性的能力(PIC> 0.7),而对应的随机个体一致性概率PI低于0.057,同样说明所有引物均为高信息量标记。期望杂合度在0.804~0.958之间,较高的平均期望杂合度(0.896)表明本试验选用的油橄榄品种之间存在着高度的遗传变异。
2.2 UPGMA聚类分析32个油橄榄品种的部分形态学特征见表 5。从UPGMA聚类图(图 2)中可以看出,32个品种间相似性系数为0.59~0.89,平均值为0.74。所有品种可以分为3大类。第1大类有6个引种品种,包括‘美佳伦’、 ‘北庆米扎’、‘奥托卡’、‘尼肖特’、‘美兰开和’‘圆果卡林’。第2大类有21个品种,主要来源于意大利和阿尔巴尼亚。在遗传相似性系数0.747处,又可将第2大类分为4个亚类:第1亚类包括3个品种,其中‘阿斯’与‘中山24’聚在一起,2个品种都具有果实质量(>6.13 g)与含油率高(>18%)的特点(表 5),且‘中山24’又是南京中山植物园通过‘阿斯’实生选优而来的,故二者存在亲缘关系;第2亚类含有9个品种,其中‘希腊3号’与‘配多灵’之间的遗传相似性系数(0.89)最高,且果实都比较轻(1.5~2.0 g),同时引种品种‘佛奥’与其改良品种‘科新佛奥’也聚在一类;第3亚类含有6个引种品种,其中‘贝拉特’、‘未知米扎’与‘米扎’的另外一个类型‘哥朗米扎’聚为一类,这3个品种含油率都很高且都来自阿尔巴尼亚;第4亚类包括1个选育品种‘张林’和2个引种品种‘科拉蒂’和‘小苹果’。第3大类包括1个引种品种‘莱星’和一个国内选育品种‘鄂植’。3个独立的分支分别为‘豆果’、‘皮瓜尔’和‘卡拉马达’,均为引种品种。整体来看,国内选育品种分布于不同的类群内,大部分是从引种品种种子繁殖产生的实生群体中选育并驯化的。而来源于相同国家的引种品种并没有完全聚为一类,即油橄榄品种不是完全按照地理起源的标准来划分的,而是根据品种间遗传学与形态学数据结合来决定的。
|
|
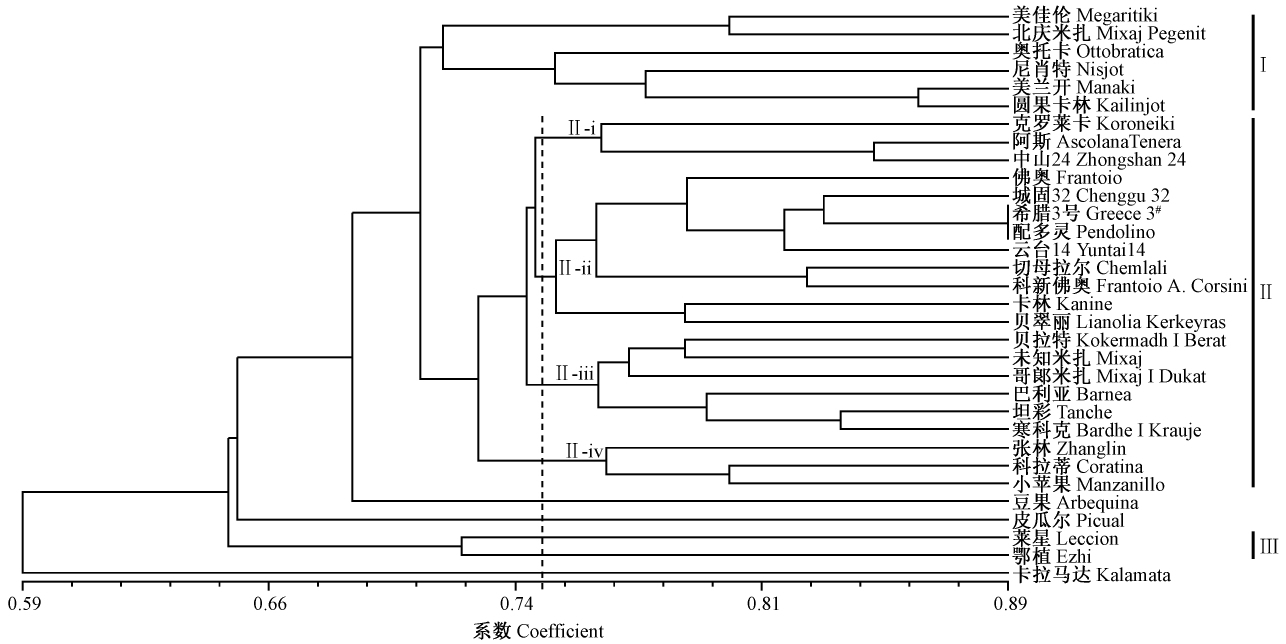 |
图 2 利用相似性系数和非加权平均法对32个油橄榄品种的SRAP聚类分析 Fig. 2 SRAP cluster analysis of 32 olive cultivars with similarity coefficient and unweighted pair group method with arithmetic (UPGMA) |
根据主坐标分析(principal coordinate analysis,PCA)将所有油橄榄品种聚类为3个群体(图 3),3个坐标的累计贡献率为20.4%。UPGMA分析中的第Ⅱ类的所有品种都聚在Group Ⅰ内,‘美佳伦’与‘北庆米扎’遗传相似性(0.80)较高而被单独聚在Group Ⅲ内,‘卡拉马达’是单独处于整个族群边缘的1个品种。PCA分析结果与UPGMA分析结果基本一致。
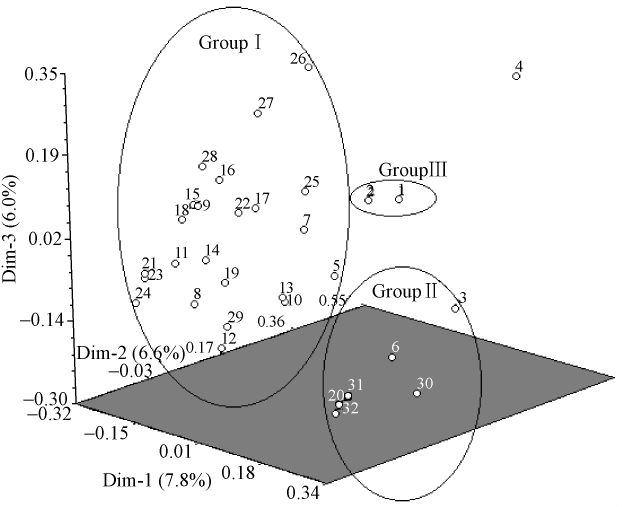 |
图 3 32个油橄榄品种的主坐标分析 Fig. 3 Principal coordinate analysis of 32 olive cultivars 图中数字与表1中品种编号对应。 Dim-1, Dim-2, Dim-3为每个维度贡献率。 The digits correspond to the cultivar code in Tab. 1. Dim-1, Dim-2 and Dim-3 is the cumulative contribution rate of each dimension. |
近些年来,国外常有运用SRAP技术来研究油橄榄遗传多样性和种质鉴定等方面的报道。Neslihan等(2011)运用SSR和SRAP标记对来自土耳其的66个油橄榄品种进行遗传多样性分析,选出的13对SRAP引物共扩增出103个多态性条带,平均PIC为0.24。Aydin等(2012)联合SRAP和RAPD技术对土耳其的24个油橄榄品种进行遗传多样性研究,其中利用18对SRAP引物扩增出了69个多态性条带,平均多态性比率为84%。本试验筛选出的SRAP引物扩增得到293个多态性条带,多态性比率为90.75%,多态性信息含量(PIC > 0.8)和一致性概率(PI < 0.057)证明本试验筛选的SRAP引物鉴别能力更强,同时期望杂合度(He> 0.804)也说明所采取的油橄榄品种之间遗传资源丰富、变异度较高(Do Val et al.,2012)。
UPGMA分析和主坐标分析结果表现出一致性,油橄榄品种的分类并未与其地理起源保持完全的一致性,而是与油橄榄形态学和遗传学等特点具有密切的相关性。这一结果与李金花等(2012)、陈海云等(2013)的研究结果相似。这是由于油橄榄种植历史悠久,长时间的进化与变异导致其遗传资源相当丰富,加之油橄榄品种在国际与地区间频繁交流以及大量自交、杂交新品种的选育,这些因素导致地理起源对油橄榄遗传多样性的影响逐渐降低。
实生选优育种是油橄榄品种改良的重要途径之一,近几十年来,我国利用实生选优育种己取得了很大进展。本研究采取的‘城固32’、‘中山24’、‘云台14’、‘鄂植’都是来自实生选优。我国油橄榄实生选优大都以引种品种为基础,因此国内选育品种分布于不同的类群中,且与一些引种品种保持较高的遗传相似性(徐纬英,2001)。油橄榄花为异株授粉,在长期的栽培与生产过程中将会产生一些基因的变异。我国从南斯拉夫和阿尔巴尼亚引入‘配多灵’在西昌等处做授粉树,效果良好。调查发现,‘配多灵’广泛种植于西昌油橄榄园中,与‘希腊3号’具有高度相似的形态学特征,因此‘希腊3号’可能为‘配多灵’的杂交后代。‘北庆米扎’与‘哥朗米扎’是‘米扎’品种的2个类型,聚类分析中‘未知米扎’与‘哥朗米扎’聚为一类。可见,我国在油橄榄引种与栽培过程中存在管理不善、品种相关背景信息不明确等问题。
丰富的遗传多样性可能是受到外界环境、自交和杂交等因素相互影响的结果(Martins-Lopes et al.,2007)。李金花等(2012)在利用SSR标记分析中国油橄榄遗传多样性时也发现我国油橄榄品种内存在着高度的遗传变异,可能是由于引种过程中品种混淆和交流频繁造成的。据邓明全等(2011)统计,中国已从国外引进大量油橄榄品种,被大量栽培的品种有‘豆果’、‘米扎’、‘佛奥’、‘莱星’、‘贝拉特’、‘皮瓜尔’、‘皮肖利’等,然而大部分品种还处于命名混乱、遗传背景不清楚的情况,需要做大量的品种鉴定工作。研究发现所采取的油橄榄品种存在着丰富的遗传多样性,这对油橄榄品种的鉴定和种质开发提供了有价值的理论指导。
| [1] |
陈海云, 宁德鲁, 李勇杰, 等. 2013. 59个油橄榄种质的 ISSR 分子鉴定. 东北林业大学学报, 41(3):13-17. (Chen H Y, Ning D L, Li Y J, et al. 2013. Molecular indentification among 59 cultivars based on ISSR markers. Journal of Northeast Forestry University, 41(3):13-17[in Chinese]).(  2) 2)
|
| [2] |
邓明全, 俞宁. 2011. 油橄榄引种栽培技术. 北京:中国农业出版社. (Deng M Q, Yu N. 2011. Olive introduction & cultivation techniques. Beijing: China Agriculture Press[in Chinese]).(  1) 1)
|
| [3] |
李金花, 俞宁. 2012. 利用荧光SSR标记分析中国油橄榄品种遗传多样性. 林业科学, 48(6):47-55. (Li J H, Yu N. 2012. Genetic diversity of olive cultivars in China based on fluorescent SSR markers. Scientia Silvae Sinicae, 48(6):47-55[in Chinese]).(  3) 3)
|
| [4] |
马万里, Collins G. 2006. 运用RAPD技术鉴定澳大利亚油橄榄品种的研究. 内蒙古师范大学学报, 35(3):337-339. (Ma W L, Collins G. 2006. Identification to cultivars in Australian olives by RAPD-PCR. Journal of Inner Mongolia Normal University: Natural Science Edition, 35(3):337-339[in Chinese]).(  1) 1)
|
| [5] |
宁德鲁, 陈少瑜, 陈海云, 等. 2013. 48个油橄榄品种的遗传多样性及聚类分析. 农业科学与技术,14(9):1215-1219. (Ning D L, Chen S Y, Chen H Y, et al. 2013. Genetic diversity and clustering analysis of 48 cultivars of Olea europaea L. Agricultural Science &Technology, 14(9):1215-1219[in Chinese]).(  1) 1)
|
| [6] |
邱源, 韩华柏, 李俊强, 等. 2008. 23个油橄榄品种的RAPD分析. 林业科学, 44(1):85-89. (Qiu Y, Han H B, Li J Q, et al. 2008. RAPD analysis of 23 olive (Olea europaea) cultivars. Scientia Silvae Sinicae, 44(1):85-89[in Chinese]).(  1) 1)
|
| [7] |
徐纬英. 2001. 中国油橄榄种质资源与利用. 长春:长春出版社. (Xu W Y. 2001. China olive germplasm resources and utilization. Changchun: Changchun Publishing House[in Chinese]).(  1) 1)
|
| [8] |
Angiolillo A, Reale S, Pilla F, et al. 2006. Molecular analysis of olive cultivars in the Molise region of Italy. Genetic Resources and Crop Evolution, 53(2):289-295.( 1) 1)
|
| [9] |
Aydin U, Ubeyit S, Cengiz T. 2012. Molecular marker based analysis of genetic diversity and relationships in some turkish and foreign olive cultivars and accessions. SABRAO Journal of Breed Genetics, 44(1): 177-188.( 1) 1)
|
| [10] |
Bandelj D,Jakše J,Javornik B. 2004. Assessment of genetic variability of olive varieties by microsatellite and AFLP markers. Euphytica, 136(1):93-102.( 1) 1)
|
| [11] |
Besnard G, Breton C, Baradat P, et al. 2001. Cultivar identification in olive based on RAPD markers. Journal of the American Society for Horticultural Science, 126(6):668-675.( 1) 1)
|
| [12] |
Breton C, Medail F, Pinatel C, et al. 2006. From olive tree to oleaster: origin and domestication of Olea europaea L. in the Mediterranean basin. Cahiers Agricultures, 15(4):329-336.( 1) 1)
|
| [13] |
Budak H, Shearman R, Parmaksiz I, et al. 2004. Molecular characterization of Buffalograss germplasm using sequence-related amplified polymorphism markers. Theoretical and Applied Genetics, 108(2):328-334.( 1) 1)
|
| [14] |
Cantini C,Cimato A,Sani G. 1999. Morphological evaluation of olive germplasm present in Tuscany region. Euphytica, 109(3):173-181.( 1) 1)
|
| [15] |
Cipriani G, Marrazzo M, Marconi R, et al. 2002. Microsatellite markers isolated in olive (Olea europaea L.) are suitable for individual fingerprinting and reveal polymorphism within ancient cultivars. Theoretical and Applied Genetics, 104(2/3):223-228.( 1) 1)
|
| [16] |
Dastkar E, Soleimani A, Jafary H, et al. 2013. Genetic and morphological variation in Iranian olive (Olea europaea L.) germplasm. Crop Breeding Journal, 3(2):99-106.( 1) 1)
|
| [17] |
De la Rosa R, Angiolillo A, Guerrero C, et al. 2003. A first linkage map of olive (Olea europaea L.) cultivars using RAPD, AFLP, RFLP and SSR markers. Theoretical and Applied Genetics, 106(7):1273-1282.( 1) 1)
|
| [18] |
Do Val A, Ferreira J, Vieira Neto J, et al. 2012. Genetic diversity of Brazilian and introduced olive germplasms based on microsatellite markers. Genetics and Molecular Research, 11(1):556-571.( 1) 1)
|
| [19] |
El Saied S H, Hegazi A A, Tawfik A A, et al. 2012. Molecular characterization of local and imported olive cultivars grown in Egypt using ISSR technique. Journal of Horticultural Science, 4(2):148-154.( 1) 1)
|
| [20] |
Erre P, Chessa I, Muñoz-Diez C, et al. 2010. Genetic diversity and relationships between wild and cultivated olives (Olea europaea L.) in Sardinia as assessed by SSR markers. Genetic Resources and Crop Evolution, 57(1):41-54.( 1) 1)
|
| [21] |
Gomes S, Martins-Lopes P, Lopes J, et al. 2009. Assessing genetic diversity in Olea europaea L. using ISSR and SSR markers. Plant Molecular Biology Reporter, 27(3):365-373.( 1) 1)
|
| [22] |
Guerin J R, Sweeney S M, Collins G G, et al. 2002. The development of a genetic database to identify olive cultivars. Journal of the American Society for Horticultural Science, 127(6):977-983.( 1) 1)
|
| [23] |
Işk N, Do Dğanlar S, Frary A. 2011. Genetic diversity of Turkish olive varieties assessed by simple sequence repeat and sequence-related amplified polymorphism markers. Crop Science, 51(4):1646-1654.( 1) 1)
|
| [24] |
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics, 103(2/3):455-461.( 2) 2)
|
| [25] |
Martins-Lopes P, Gomes S, Santos E, et al. 2008. DNA markers for Portuguese olive oil fingerprinting. Journal of Agricultural and Food Chemistry, 56(24):11786-11791.( 1) 1)
|
| [26] |
Martins-Lopes P, Lima-Brito J, Gomes S, et al. 2007. RAPD and ISSR molecular markers in Olea europaea L.: Genetic variability and molecular cultivar identification. Genetic Resources and Crop Evolution, 54(1):117-128.( 1) 1)
|
| [27] |
Montemurro C, Pasqualone A, Simeone R, et al. 2008. AFLP molecular markers to identify virgin olive oils from single Italian cultivars. European Food Research and Technology, 226(6):1439-1444.( 1) 1)
|
| [28] |
Neslihan Işlk, Sami Do Dğanlar, Anne Frary. 2011. Genetic diversity of Turkey olive varieties assessed by simple sequence repeat and sequence-related amplified polymorphism markers. Crop Science, 51(4):1646-1654.( 1) 1)
|
| [29] |
Rohlf F. 2000. Multivariate Analysis System: Version 2.10 e. Applied Biostatistics Inc, New York.( 1) 1)
|
| [30] |
Rugini E, Lavee S. 1992. Olive//Hammerschlag F A, Litz R E. Biotechnology of perennial fruit crops. Biotechnology in Agriculture, vol. 8. CAB International, Wallingford, 371-382.( 1) 1)
|
| [31] |
Souza R, Ferreira J L, Braga F T, et al. 2012. Outcrossing rate in olive assessed by microsatellite and inter simple sequence repeat (ISSR) markers. African Journal of Biotechnol, 11(53):11580-11584.( 1) 1)
|
| [32] |
Wu S B, Collins G, Sedgley M. 2004. A molecular linkage map of olive (Olea europaea L.) based on RAPD, microsatellite, and SCAR markers. Genome, 47(1):26-35.( 1) 1)
|
 2015, Vol. 51
2015, Vol. 51

