文章信息
- 王金星, 张利军, 廖资亿, 张运根, 邱乾栋, 孙鹏, 孙宇涵, 胡瑞阳, 卢楠, 李云
- Wang Jinxing, Zhang Lijun, Liao Ziyi, Zhang Yungen, Qiu Qiandong, Sun Peng, Sun Yuhan, Hu Ruiyang, Lu Nan, Li Yun
- 刺槐实时定量PCR分析中内参基因的选择
- The Selection of Reference Genes for Real-Time Quantitative PCR Normalization in Black Locust(Robinia pseudoacacia)
- 林业科学, 2014, 50(9): 167-172
- Scientia Silvae Sinicae, 2014, 50(9): 167-172.
- DOI: 10.11707/j.1001-7488.20140923
-
文章历史
- 收稿日期:2013-12-04
- 修回日期:2014-03-28
-
作者相关文章
2. 北京风沙源育苗中心 北京 102115;
3. 福建省三明市国有林场管理处 三明 365000;
4. 福建省宁化国有林场 宁化 365413;
5. 中国林业科学研究院经济林研究开发中心 郑州 450003
2. Beijing Sandstorm Source Nursery Center Beijing 102115;
3. Administrative Department of State-Owned Forest Farm of Sanming City in Fujian Province Sanming 365000;
4. The State-Owned Forest Farm of Ninghua County in Fujian Province Ninghua 365413;
5. Non-Timber Forestry Research and Development Center, Chinese Academy of Forestry Zhengzhou 450003
实时荧光定量PCR是由美国Applied Biosystems公司于1996年在传统的PCR技术基础上发展起来的一种新的核酸定量技术(Mackay,2004),具有定量准确、灵敏度高、特异性强、重复性好及高通量等优点(Huggett et al.,2005),已成为医学、农学、林学、微生物学等众多领域中进行基因表达和转录组分析的常用技术之一(Postollec et al.,2011;袁亚男等,2008)。然而,在其分析过程中,RNA的产量和质量、反转录效率的差异以及其他因素都可能会对目的基因的表达分析结果的准确性产生影响(Paolacci et al.,2009),因此,需要合适的内参基因进行校正和标准化,从而减少试验结果的误差(Nolan et al.,2006)。理想的内参基因应该在不同的器官、植物发育的不同阶段及不同的试验条件下表达均保持一致,常用的内参基因是构成细胞骨架的主要组分或是参与生物体基本代谢过程的一些基因(如ACT,TUB,UBQ等基因),这些基因基本上可以在植物不同器官中稳定地表达(Gutierrez et al.,2008)。近来的研究表明,没有任何一种内参基因的表达是始终稳定的,在不同的植物、同一种植物不同器官及不同生长发育阶段、生物及非生物胁迫下都会引起内参基因的特异性表达,因此每种特定条件下都有特定的稳定表达的内参基因(Vandesompele et al.,2002;Bustin et al.,2009),如不加筛选盲目地以一种内参基因作为所有试验条件下的内参,很难得到准确的定量结果。如Vandesompele等(2002)证实一种内参基因在10%的样品中可导致6.4倍的表达误差。因此,在qPCR试验中,根据特定的试验材料选择合适的内参基因极其重要(王彦杰等,2012)
刺槐(Robinia pseudoacacia )原生于北美洲,后被广泛引种到欧洲、亚洲等地。刺槐生长速度快、材质坚硬、抗腐能力强,是重要的速生用材树种(习洋等,2012)。其叶片含有大量粗蛋白,可作饲料,根部有根瘤,可通过生物固氮增加土壤肥力;刺槐的花属于蝶形花,开花时香气四溢,并且可供食用(Keeler,1900)。此外,刺槐还是一种重要的蜜源植物(孙鹏等,2012)。综上所述,刺槐具有很好的生态和经济价值。但目前刺槐的分子生物学研究基础比较薄弱,内参基因的稳定性验证还未见报道,该方面的空缺极大地限制了刺槐分子生物学的研究。
本研究以4株不同遗传背景的刺槐单株上的叶片以及同一植株刺槐的不同器官(叶、花、根、茎、豆荚)为材料,通过分析这些材料的刺槐荧光定量数据,对6个候选内参基因(ACT,Helicase,18S rRNA,SAMDC,GAPDH,EF1-α)在不同单株以及不同器官的稳定性进行鉴定。通过使用GeNorm和Bestkeeper 2个内参基因稳定性分析软件,在一系列不同功能的候选内参基因中筛选出最为稳定的内参基因用于目标基因的校正和标准化,从而达到准确定量的试验目的,为刺槐的分子生物学研究打下基础。
1 材料与方法 1.1 试验材料于2013年5月份在北京市延庆县米家堡林场的刺槐成年实生林中,随机选择相距50 m以上的4株单株(以确保单株的遗传背景不同,防止根萌植株)。采集这4株单株上的叶片用以比较不同株系间内参基因的稳定性,同时采集其中1单株的根、茎、叶、花、豆荚器官,用以比较不同器官间内参基因的稳定性。样品采集后液氮速冻,-80 ℃贮藏备用。
1.2 RNA提取及cDNA第1链合成利用Aidlab公司的植物总RNA提取试剂盒提取各个样品的总RNA,步骤按照说明书进行,用Nanodrop ND-1000测定RNA浓度,RNA的完整性通过1.2%的琼脂糖凝胶电泳检测。反转录按照Aidlab反转录cDNA第1链试剂盒的操作方法,以各个样品中1 000 ng总RNA为模板将各个样品反转录合成cDNA第1链,获得的产物直接用于PCR或-20 ℃储藏备用。
1.3 内参基因的选择及荧光定量PCR引物的设计选取刺槐肌动蛋白基因(actin,ACT)、18S核糖体RNA( 18S rRNA)、腺苷甲硫氨酸脱羧酶基因(SAMDC)、甘油醛-3-磷酸脱氢酶基因(GAPDH)、解旋酶基因(Helicase)、转录延伸因子基因(EF1-α),所有基因片段均为本实验室自行克隆,登录号分别为KJ587742,KJ587743,KJ587744,KJ587745,KJ587746,KJ587747。利用Primer Premier5.0软件,根据荧光定量PCR引物的设计原则,分别设计ACT,GAPDH,18S rRNA,SAMDC,GAPDH,Helicase 6个内参基因的PCR引物。引物序列见表 1,引物由睿博兴科生物技术有限公司合成。
|
|
6种内参基因的PCR扩增在ABI PCR扩增仪上进行,以各个样品的cDNA为模板进行PCR反应。反应体系为cDNA模板2 μL,正反引物1 μL,MIX12.5 μL,加ddH2O补齐至25 μL。反应条件: 95 ℃预变性3 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,共35个循环;72 ℃延伸5 min。最后用1.5%的琼脂糖凝胶电泳检测扩增引物的特异性。
1.5 实时定量PCR用TAKARA公司的SYBR Green I荧光定量PCR试剂和ABI公司的7500Fast荧光定量PCR仪进行RT-PCR试验。反应体系20 μL: 2×SYBR Premix Ex Taq 10 μL;ROX 0.5 μL;ddH2O 7.5 μL;cDNA template 1 μL;正反引物各0.5 μL。采用两步法进行PCR扩增,扩增条件如下: 95 ℃预变性30 s;95 ℃变性3 s,60℃退火30 s,共40个循环;之后进行55~95 ℃溶解曲线分析。每个样品3次生物学重复。
1.6 数据处理和分析用GeNorm和NormFinder软件,根据6个常用内参基因在刺槐不同器官中的相对表达量(Q)对各个内参基因的表达稳定性进行统计学分析,从而筛选出表达相对稳定的内参基因。根据公式Q=E△Ct,式中: E为基因扩增效率,E一般默认为2即扩增效率为100%;△Ct=Ctmin-Ct样品,Ct样品为每个样品的Ct值,Ctmin为不同样品中内参基因的最小Ct值。
2 结果与分析 2.1 RNA样品的质量检测所提取刺槐根、茎、叶、花、豆荚样品的RNA经Nanodrop ND-1000紫光分光光度计检测浓度和纯度,所测样品的A260/A280比值均为2.0左右,且浓度均较高。琼脂糖凝胶电泳(图 1)显示,刺槐每个器官RNA电泳图谱清晰,28S rRNA的亮度约为18S rRNA的2倍。表明所提取的RNA质量好,可用于后续的分子生物学试验。
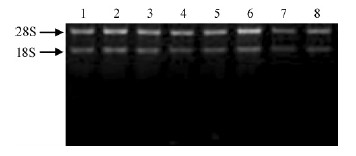 |
图 1 刺槐各试验样品总RNA 电泳图谱
Fig. 1 Agarose gel analysis of total RNA extracted from different samples of Robinia pseudoacacia
1-4: 单株1-4; 5: 茎; 6: 根; 7: 花; 8: 豆荚。 1-4: Individual tree 1-4; 5: Stem; 6: Root; 7: Flower; 8: Pod. |
以刺槐单株1的叶的cdna为模板,通过普通pcr扩增出与各内参基因预期大小一致的基因片段,且没有见明显的引物二聚体及非特异条带(图 2),表明各内参基因引物均能特异地扩增出相应的目的片段,可用于后续的qpcr试验。
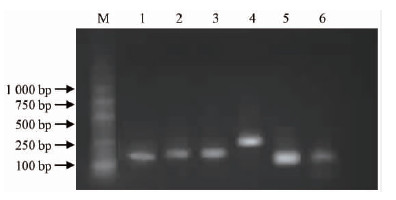 |
图 2 6种内参基因的PCR扩增产物 Fig. 2 PCR products of six reference genes M: DNA marker; 1: ACT; 2: EF1-α; 3: Helicase; 4: SAMDC; 5: 18S rRNA; 6: GAPDH |
以刺槐不同器官的cDNA为模板,进行荧光定量PCR分析。结果(图 3)表明,6个内参基因的引物在各个材料的溶解曲线只有明显的单一峰,反映出具有较高的专一性,且每个待测样品的扩增曲线重复性也很好,说明试验结果的可靠性很高。
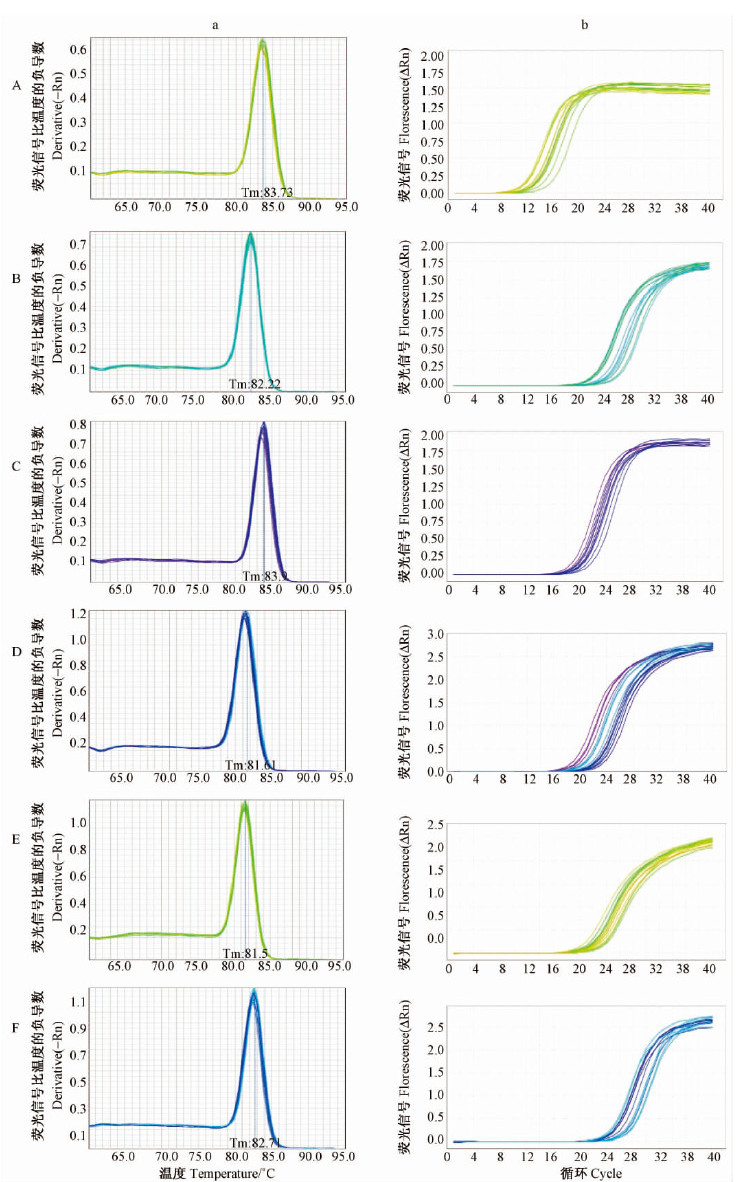 |
图 3 刺槐不同单株和器官中6个内参基因的real-time PCR溶解曲线(a)和扩增曲线(b) Fig. 3 The real-time PCR amplification curve(b) and melting curves(a) of 6 reference genes in different individuals and tissues of Robinia pseudoacacia A.18S rRNA; B. Helicase; C. SAMDC; D. GAPDH; E. ACT; F. EF1-α |
为筛选合适的内参基因,用GeNorm软件对6个内参基因在不同单株、不同器官中的表达稳定性进行统计,GeNorm软件通过计算内参基因的表达稳定性的M值来分析内参基因的表达稳定性,M值越大则稳定性越差,M值越小则稳定性越高。结果如图 4所示。当以不同单株为试验材料时,各内参基因的表达稳定性各异,ACT和18S rRNA的M值(0.67)最小,表明二者表达最为稳定,其次是GAPDH和Helicase,表达最不稳定的是SAMDC和EF1-α。当以同一单株不同器官为试验材料时,ACT与GAPDH的M值(0.89)最小,表示二者表达最为稳定,其次是18S rRNA和Helicase,表达最不稳定的是SAMDC和EF1-α。
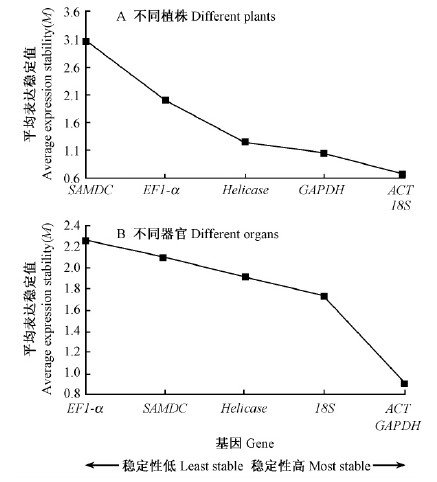 |
图 4 用GeNorm软件分析不同植株和不同器官中6种内参基因的表达稳定值 Fig. 4 Average expression stability value of six reference genes in different plant and organs calculated by GeNorm |
NormFinder软件经运算后产生表达稳定值,表达稳定值最小的基因作为最稳定的基因。结果(表 2)显示,在不同单株、不同器官间ACT表达最为稳定,为最优的内参基因,其次分别为GAPDH和18S rRNA。其结果与GeNorm程序分析结果基本相符,这在一定程度上验证了结果的可靠性。
|
|
实时定量PCR技术实现了从定性到定量的飞跃,已成为分子生物学中研究基因表达量的常用技术之一,但是使用稳定的内参基因是定量结果可靠的先决条件(Chang et al.,2012)。不同物种间内参基因表达存在差异,在某种植物中表达稳定性高的内参基因在另一种植物中不一定会有较好的表达稳定性,因此根据具体的试验样品筛选适当的内参基因对目的基因的表达进行校正是RT-PCR分析中至关重要的一步。随着生物信息学和基因组学的发展,已有很多物种的内参基因的稳定性得到了评价与鉴定,大豆(Glycine max)(Jian et al.,2008;Hu et al.,2009)、水稻(Oryza sativa)(Jain,2009)、小麦(Triticum aestivum)(Long et al.,2010)、拟南芥(Arabidopsis thaliana)(Remans et al.,2008)等植物都鉴定出了在各自不同发育阶段、不同试验条件下表达最为稳定的内参基因。
可用于评价内参基因稳定性的软件有多种,例如qBasePlus软件(Hellemans et al.,2007)、REST软件(Pfaffl et al.,2002)、BestKeeper软件(Pfaffl et al.,2004)等(赵文静等,2010)。GeNorm程序是通过计算内参基因的表达稳定性的M值来分析内参基因的表达稳定性,M值越大则稳定性越差,M值越小则稳定性越高,且GeNorm软件可以同时筛选一对内参基因;NomFinder软件的运行原理与GeNorm软件的原理类似,经运算后产生表达稳定值,然后根据表达稳定值的大小排序,最后将表达稳定值最小的基因作为最稳定的基因(Giulietti,2001;魏永赞等,2012)。
经2种软件计算得出,在不同植株、不同器官间ACT,GAPDH,18S rRNA相对比较稳定,而SAMDC,EF1-α,Helicase基因则相对不稳定。Iskandar(2004)分析了甘蔗(Saccharum sinense)不同品种、不同器官中4个常见的内参基因(ACT,25S rRNA,GAPDH,TUB)的表达稳定性,结果表明,GAPDH在不同器官中表达稳定性最好。Mascia等(2010)分析了8个内参基因(ACT,CYP,EF1-α,GAPDH,TUB,UBI,18S rRNA,UBQ)在番茄(Lycopersicon esculentum)不同病毒侵染下的表达稳定性,结果表明GAPDH在所有样品中均能稳定表达,这与本试验结果相似。而ACT基因作为一种最常用的内参基因被广泛使用,且在本试验中经过GeNorm和NormFinder软件分析在不同单株和单株不同器官间也显示出了较好的稳定性。在本试验中表达相对不稳定的SAMDC和EF1-α却分别在短柄草(Brachypodium sylvaticum)逆境条件下和小麦不同器官以及不同试验条件下表现出较高的表达稳定性(袁伟等,2012)。说明这些基因在某些试验条件下缺乏稳定性。另外关于18S rRNA做内参基因使用存在一些争议,因18S rRNA末端没有poly(A)尾巴,当使用oligo-dT进行反转录或以mRNA为模板时18S rRNA不能作为内参(Stürzenbaum et al.,2001),且18S rRNA表达丰度过高,因此在qPCR数据分析时基线值对其影响过大(Vandesompele et al.,2002)。
综上所述,ACT,GAPDH,18S rRNA为相对稳定的3个内参基因。为了消除单一内参基因所引起的偏差,在一组特定的样本和试验条件下,很多试验结果证实选择2个或2个以上的内参基因可以增加结果的可靠性和准确度(Schmid et al.,2003),但基于试验成本、工作量等考虑,往往又需要较少的内参基因。基于以上综合考虑,对刺槐不同品种、不同器官间进行实时定量PCR时可以同时使用ACT和GAPDH基因作为内参对目的基因的表达进行校正。
| [1] |
孙鹏, 戴丽, 胡瑞阳, 等.2012.刺槐开花传粉及交配方式.东北林业大学学报, 40(1): 6-12.( 1) 1)
|
| [2] |
王彦杰, 董丽, 张超, 等.2012.牡丹实时定量PCR分析中内参基因的选择.农业生物技术学报, 20(5): 521-528.( 1) 1)
|
| [3] |
魏永赞, 赖彪, 胡福初, 等.2012.用于荔枝qPCR分析的内参基因克隆及稳定性分析.华南农业大学学报, 33(3):301-306.( 1) 1)
|
| [4] |
习洋, 胡瑞阳, 王欢, 等.2012.刺槐未成熟合子胚的体细胞胚胎发生和植株再生.林业科学, 48(1): 60-69.( 1) 1)
|
| [5] |
袁伟, 万红建, 杨悦俭.2012.植物实时荧光定量PCR内参基因的特点及选择.植物学报, 47(4): 427-436.( 1) 1)
|
| [6] |
袁亚男, 刘文忠.2008.实时荧光定量PCR技术的类型、特点与应用.中国畜牧兽医, 35(3): 72.( 1) 1)
|
| [7] |
赵文静, 徐洁, 包秋华,等.2010.实时荧光定量PCR中内参基因的选择.微生物通报, 37(12): 1825-1829.( 1) 1)
|
| [8] |
Bustin S A, Beaulieu J F, Huggett J, et al.2009.The MIQE guideline: Minemum information for publication of quantitative real-time PCR experience.Clinical Chemistry, 55(4): 611-622.( 1) 1)
|
| [9] |
Chang Ermei, Shi Shengqing, Liu Jianfeng, et al.2012.Selection of reference genes for quantitative gene expression studies in Platycladus orientalis (Cupressaceae) using real-time PCR.PLOS ONE, 7(3): e33278.( 1) 1)
|
| [10] |
Giulietti A, Overbergh L, Valckx D, et al.2001.An overview of real-time quantitative PCR: applications to quantify cytokine gene expression.Methods, 25(4): 386-401.( 1) 1)
|
| [11] |
Gutierrez L, Mauriat M, Guenin S, et al.2008.The lack of a systematic validation of reference genes: a serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR) analysis in plants.Plant Biotechnol J, 6(6): 609-618.( 1) 1)
|
| [12] |
Hellemans J, Mortier G, Paepe D A, et al.2007.qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data.Genome Biology, 8(2): R19.( 1) 1)
|
| [13] |
Hu R, Fan C M, Li H Y, et al.2009.Evaluation of putative reference genes for gene expression normalization in soybean by quantitative real-time RT-PCR.BMC Mol Biol, 10: 93.( 1) 1)
|
| [14] |
Huggett J, Dheda K, Bustin S, et al.2005.Real-time RT-PCR normalization: strategies and considerations.Genes Immun, 6(6): 279-284.( 1) 1)
|
| [15] |
Iskandar H M, Simpeon R S, Casu R E, et al.2004.Comparison of reference genes for quantitative real-time polymerase chain reaction analysis of gene expression in sugarcane.Plant Mol Biol Rep, 22(4): 325-337.( 1) 1)
|
| [16] |
Jain M.2009.Genome-wide identification of novel internal control genes for normalization of gene expression during various stages of development in rice.Plant Sci, 176(5): 702-706.( 1) 1)
|
| [17] |
Jian B, Liu B, Bi Y R, et al.2008.Validation of internal control for gene expression study in soybean by quantitative real-time PCR.BMC Mol Biol, 9: 59.( 1) 1)
|
| [18] | Keeler H L.1990.Our native trees and how to identify them.New York: Charles Scriber’s Sons, 97-102. |
| [19] |
Long Xiangyu, Wang Jirui, Ouellet T, et al.2010.Genome wide identification and evaluation of novel internal control genes for qPCR based transcript normalization in wheat.Plant Molecular Biology, 74(3): 307-311.( 1) 1)
|
| [20] |
Mackay I M.2004.Real-time PCR in the microbiology laboratory.Clin Microbiol Infect, 10(3):190-212.( 1) 1)
|
| [21] |
Mascia T, Santovito E, Gallitelli D, et al.2010.Evalution of reference genes for quantitative reverse transcription polymerase chain reaction normalization in infected tomato plants.Mol Plant Pathol, 11(6): 805-816.( 1) 1)
|
| [22] |
Nolan T, Hands R E, Bustin S A.2006.Quantification of mRNA using real-time RT-PCR.Nat Protoc, 1(3):1559-1582.( 1) 1)
|
| [23] |
Paolacci A R, Tanzarella O A, Porceddu E, et al.2009.Identification and validation of reference genes for quantitative RT-PCR normalization in wheat.BMC Mol Biol, 10: 11.( 1) 1)
|
| [24] |
Pfaffl M W, Horgan G W, Dempfle L.2002.Relative Expression Software Tool for group wise comparison and statistical analysis of relative expression results in real-time PCR.Nucleic Acids Res, 30(9):e36.( 1) 1)
|
| [25] |
Pfaffl M W, Tichopad A, Prgomet C, et al.2004.Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: bestkeeper-excel-based tool using pair-wise correlations.Biotechnol Lett, 26(6): 509-515.( 1) 1)
|
| [26] |
Postollec F, Falentin H, Pavan S, et al.2011.Recent advances in quantitative PCR aplications in food microbiology.Food Microbiology, 28(5): 848-861.( 1) 1)
|
| [27] |
Remans T, Smeets K, Opdenakker K, et al.2008.Normalisation of real-time RT-PCR gene expression measurements in Arabidopsis thaliana exposed to increased metal concentrations.Planta, 227(6): 1343-1349.( 1) 1)
|
| [28] |
Schmid H, Cohen C D, Henger A, et al.2003.Validation of endogenous controls for gene expression analysis in microdissected human renal biopsies.Kidney Int, 64: 356-360.( 1) 1)
|
| [29] |
Stürzenbaum S R, Kille P.2001.Control genes in quantitative molecular biological techniques: The variability of invariance.Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 130(3): 281-289.( 1) 1)
|
| [30] |
Vandesompele J, De Preter K, Pattyn F, et al.2002.Accurate normalization of real-time quantitative RT-PCR data by genometric averaging of multiple internal control genes.Genome Biology, 3(7): research0034:1-11.( 3) 3)
|
 2014, Vol. 50
2014, Vol. 50

