文章信息
- 赵亚琦, 成铁龙, 施季森, 王新民, 徐阳, 刘伟东, 陈金慧
- Zhao Yaqi, Cheng Tielong, Shi Jisen, Wang Xinmin, Xu Yang, Liu Weidong, Chen Jinhui
- 鹅掌楸属SRAP分子标记体系优化及遗传多样性分析
- Optimization of a SRAP-PCR System for Analysis of Genetic Diversity of Liriodendron
- 林业科学, 2014, 50(7): 37-43
- Scientia Silvae Sinicae, 2014, 50(7): 37-43.
- DOI: 10.11707/j.1001-7488.20140706
-
文章历史
- 收稿日期:2013-07-29
- 修回日期:2014-02-03
-
作者相关文章
2. 中国林业科学研究院科学技术处 北京 100091
2. Science and Technology Department, CAF Beijing 100091
相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)是一种基于PCR的标记(Li et al., 2001),该标记通过独特的双引物设计对基因ORFs(open reading frames)的特定区域进行扩增,上游引物对富含GC序列的外显子区域进行特异性扩增,下游引物对富含AT序列的内含子、启动子区域进行特异性扩增。因不同个体间乃至物种间的内含子、启动子与间隔区长度不同而产生多态性。相对于其他标记,SRAP标记具有简单、稳定、得率中等、易测序、便于克隆等特点(Aneja et al., 2012),在各种植物的遗传研究中得到了广泛的应用(Ferriol et al., 2003; Budak et al., 2004; Esposito et al., 2007; 孙佳琦等,2010; 唐琴等,2012)。
鹅掌楸属(Liriodendron)是木兰科(Magnoliaceae)古老属种植物,经第四纪冰期以后,现仅存鹅掌楸(L. chinense)和北美鹅掌楸(L. tulipifera)2种。鹅掌楸曾经广泛分布于我国中南部10多个省(区)的丘陵山地和亚高山地带,以及越南北部。由于大量的砍伐和人为破坏,现存的天然林已少见,呈多地孤岛状零星分布。北美鹅掌楸现存分布仍较广,在美国东部的23个州到加拿大安大略省的大湖区呈连续分布。鹅掌楸属具有独特的分类学地位,是研究系统进化、遗传多样性的理想材料。近年来,针对鹅掌楸属植物开展了RAPD,EST-SSR,ISSR,SSR等分子标记技术研究(李周岐等,2002; Xu et al., 2006; 张红莲等,2010; 占志勇等,2010; Yang et al., 2012),但有关鹅掌楸属的SRAP分子标记研究尚未见报道。本研究采用正交设计与单因素分析的方法优化了鹅掌楸属植物SRAP-PCR反应体系,并利用筛选的引物对鹅掌楸和北美鹅掌楸的不同地理种源,从分子水平上进行了遗传多样性水平分析,对鹅掌楸属不同种源的遗传结构和亲缘关系进行探讨,为进一步开展鹅掌楸属的种质创新和资源的合理利用奠定基础。
1 材料与方法 1.1 材料SRAP-PCR体系优化采用江西庐山种源材料。遗传多样性分析选用了5个鹅掌楸地理种源(江西庐山、福建武夷山、浙江松阳、浙江富阳、湖南桑植)和5个北美鹅掌楸地理种源(密苏里、路易斯安娜、佐治亚、北卡罗来纳、南卡罗来纳)。所有材料均采自南京林业大学下蜀林场鹅掌楸种源试验林。选取健康植株,采集其当年生鲜嫩叶储存于-80 ℃条件下备用。本试验所用的SRAP引物是从已公布的引物(Li et al., 2001; 林忠旭等,2003; Ferriol et al., 2003)中取正向引物和反向引物各10个(表 1),共100对引物组合。
|
|
鹅掌楸属基因组DNA的提取采用CTAB法(罗光佐等,2000)。经含溴化乙锭的1%的琼脂糖凝胶电泳检测后用NanoDrop-2000分光光度计测定浓度和质量,然后将部分DNA原液稀释至30 ng·μL-1,-20 ℃保存备用。
1.3 SRAP体系优化试验设计以江西庐山种源鹅掌楸基因组DNA为模板,以及经预试验表明能有效扩增的引物组合Me2/Em7进行体系优化试验。优化试验采用正交设计(汪成成等,2010),选用L16(45)正交表对影响反应结果的Mg2+、dNTPs、引物、模板DNA、Taq酶等因素在4个水平上进行试验(表 2)。试验结果采用何正文等(1998)的统计方法进行分析,同时对反应体系影响较大的几个因素进行单因素分析。
|
|
PCR扩增程序参考Li等(2001)提出的SRAP程序并略作修改: 94 ℃预变性5 min; 前4个循环为94 ℃1 min,35 ℃1 min,72 ℃1 min; 随后进行的29个循环为94 ℃ 1 min,52 ℃1 min,72 ℃ 1 min; 最后72 ℃延伸8 min并保存于4 ℃。扩增反应产物经8%聚丙烯酰胺凝胶电泳,银染后检测。
1.4 SRAP引物组合筛选使用3个不同地理种源的鹅掌楸属基因组DNA样品对100对引物组合通过优化过的反应体系进行筛选,从中选取条带多、多态性好的引物组合用于鹅掌楸属遗传多样性分析。
1.5 遗传多样性分析利用筛选出的引物在所建立的最佳反应体系下对鹅掌楸属10个地理种源进行多样性分析。SRAP反应扩增出的图谱中每一条带的迁移位置记为1个位点,相同迁移位置条带的有无分别记为1和0,统计100~1 500 bp区间的条带,形成1/0矩阵。记录结果采用POPGEN 32分析软件进行分析,建立聚类树状图。
2 结果与分析 2.1 鹅掌楸属植物SRAP-PCR体系的优化 2.1.1 SRAP-PCR反应正交试验结果引物组合Me2/Em7对江西庐山种源鹅掌楸的正交试验PCR产物电泳结果如图 1所示。按条带数量、清晰度及背景对PCR结果从低到高打分。条带模糊或者无条带的计1分,按顺序依次增加1分,分数越高,扩增效果越好。分别独立统计2次重复试验的分数依次为: 5,9,8,1,6,7,16,12,11,10,13,15,14,2,4,3; 以及4,7,8,1,6,7,16,11,12,10,14,15,13,2,3,5。
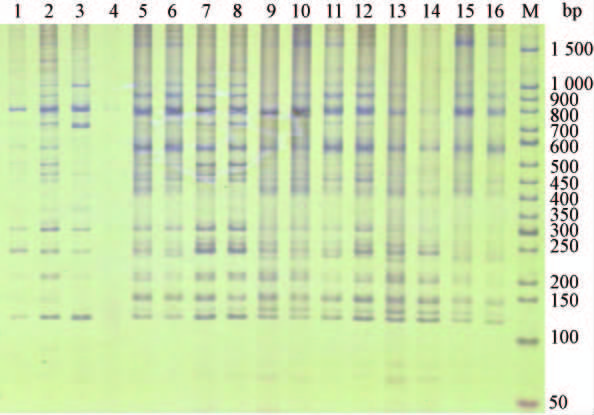
|
图 1 引物Me2 /Em7 对江西庐山种源鹅掌楸基因组DNA 的SRAP 正交试验PCR 产物电泳结果 Fig.1 Electrophoresis result showing SRAP-PCR orthogonal experiment products from L. chinense genomic DNA of Lushan,Jiangxi provenance with primer combination Me2 and Em7 50 bp DNA Ladder; 泳道1 - 16 分别与表2 编号相对应 Lane code for 1 to 16 is corresponding with Tab. 2. |
根据打分结果统计出相同因素同一水平下的试验值之和Ki,并求出相同因素不同水平间平均值的极差R,根据每一因素同一水平下的试验值之和K,对反应体系组合选优。K值越大,反应水平越好,反之则越差。R值的大小决定反应因素对反应体系影响的大小。由表 3可知,5个因素中,Mg2+ 影响程度最大,其次为Taq酶、模板DNA、dNTPs引物。最优组合与最高分值的7号组合相近,但是Mg2+、Taq酶和模板DNA浓度等因素与其不匹配且影响程度较高,所以有必要对Mg2+、Taq酶和模板DNA浓度进行单因素分析。单因素分析同样以江西庐山种源鹅掌楸基因组DNA为模板和Me2/Em7引物组合进行。
|
|
1)Mg2+浓度对反应体系的影响由图 2可以看出,当Mg2+浓度为1.25~1.75 mmol·L-1时,扩增出的条带较少; 随着浓度的升高,条带逐渐清晰; Mg2+浓度为2.75~3.50 mmol·L-1时,扩增条带丰富且清晰。综合稳定性和经济考虑,反应体系中Mg2+浓度确定为3.00 mmol·L-1。
|
图 2 Mg2 + 浓度对SRAP-PCR 反应的影响 Fig.2 The effect of Mg2 + concentration on SRAP-PCR reaction systems 50 bp DNA Ladder; 1 - 12: Mg2 + 浓度分别为1. 25,1. 50,1. 75,2. 00,2. 25,2. 50,2. 75,3. 00,3. 25,3. 50,3. 75,4. 00 mmol·L - 1 The concentrations of Mg2 + were 1. 25,1. 50,1. 75,2. 00,2. 25,2. 50,2. 75,3. 00,3. 25,3. 50,3. 75,4. 00 mmol·L - 1 . |
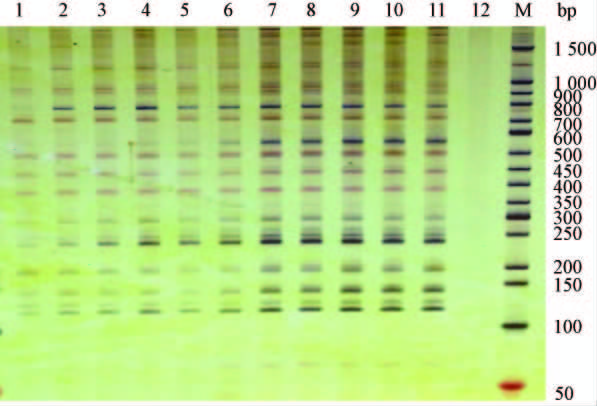
2)Taq酶浓度对反应体系的影响由图 3可知,Taq酶浓度在0.5 μmol·min-1和1.0 μmol·min-1时扩增出的条带较弱,在1.5~2.5 μmol·min-1时能扩增出清晰稳定的条带,在3.0 μmol·min-1以上时背景逐渐加深且非特异性位点增多,所以选择2.0 μmol·min-1 作为最佳Taq酶浓度。

|
图 3 Taq 酶浓度对SRAP-PCR 反应的影响 Fig.3 The effect of Taq polymerase on SRAP-PCR reaction systems 50 bp DNA Ladder; 1 - 12: Taq 酶浓度分别为0. 5,1. 0,1. 5,2. 0,2. 5,3. 0,3. 5,4. 0,4. 5,5. 0,5. 5,6. 0 μmol·min - 1 The Taq polymerase were 0. 5,1. 0,1. 5,2. 0,2. 5,3. 0,3. 5,4. 0,4. 5,5. 0,5. 5,6. 0 μmol·min - 1 . |
3)模板DNA对反应体系的影响由图 4可知,模板DNA量在45~135 ng时均能扩增出丰富稳定的条带,但是浓度过低或过高时,扩增出的条带不清晰。通过比较发现,模板DNA浓度为90 ng时条带最佳,所以选择90 ng作为反应体系的模板DNA量。

|
图 4 模板DNA 浓度对SRAP-PCR 反应的影响 Fig.4 The effect of template DNA on SRAP-PCR reaction systems 50 bp DNA Ladder; 1 - 11: 模板DNA 量分别为15,30,45,60,75,90,105,120,135,150,165 ng The template DNA were 15, 30,45,60,75,90,105,120,135,150,165 ng. |
利用优化的SRAP-PCR反应体系,对合成的100对引物进行了筛选,53对引物在3个鹅掌楸属地理种源样品上能扩增出条带。选取其中15对扩增片段多、亮度大、重复性好的引物,用于遗传多样性分析(部分扩增结果见图 5)。
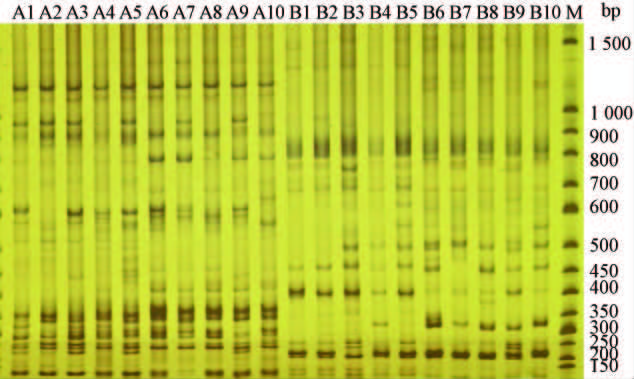
|
图 5 引物组合Me5 /Em4(A) 和Me10 /Em6(B)在鹅掌楸属植物样品中的扩增结果 Fig.5 Result of PCR amplification by SRAP primer combination Me5 /Em4(A) and Me10 /Em6( B) in samples of Liriodendron 50 bp DNA Ladder; A1 - A10 ( B1 - B10 ) : 密苏里Missouri,USA; 路易斯安娜Louisiana,USA; 佐治亚Georgia,USA; 北卡罗 来纳North Carolina,USA; 南卡罗来纳South Carolina,USA; 江西庐山Lushan,Jiangxi; 浙江松阳Songyang,Zhejiang; 福建武夷 山Wuyishan,Fujian; 浙江富阳Fuyang,Zhejiang; 湖南桑植Sangzhi,Hunan. |
用15对引物对10个鹅掌楸属地理种源样品进行扩增,共得到182个位点(表 4),其中有149个为多态性位点,多态性比例达81.87%。引物组合Me8/Em8多态位点最多,为13个; 而引物组合Me1/Em7的多态位点最少,为6个。平均位点数为12.13个,平均多态位点数为9.90个。所以选择的SRAP引物在鹅掌楸属植物上多态性丰富,能够进行遗传多样性分析。
|
|
鹅掌楸属物种水平上的等位基因数、有效等位基因数、Nei’s (1973)基因多样度、Shannon信息指数分别为1.818 7,1.533 3,0.309 0和0.457 3。鹅掌楸和北美鹅掌楸的遗传多样性水平见表 5。鹅掌楸的遗传多样性各度量值均值略高于北美鹅掌楸,但二者相差不大。这说明,鹅掌楸和北美鹅掌楸都具有一定的遗传多样性水平。
|
|
采用UPGMA法对鹅掌楸属植物的不同地理种源利用获得的SRAP多态标记进行聚类分析(图 6)。从聚类图可知,鹅掌楸属10个地理种源可分为2个聚类群: 一个类群是来源于我国5个地区的鹅掌楸;另一个类群是来源于美国密苏里、路易斯安娜、南卡罗来纳、佐治亚和北卡罗来纳的北美鹅掌楸。
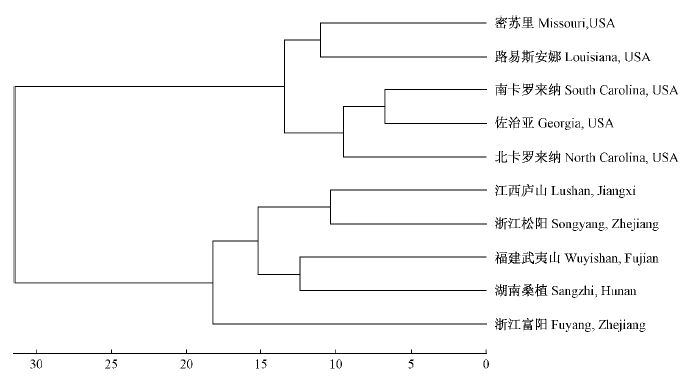
|
图 6 鹅掌楸属10 个不同地理种源的遗传距离聚类 Fig.6 Dendrogram of genetic distance for 10 provenances of Liriodendron |
影响SRAP-PCR反应的因素有很多,物种间的差异以及各反应因素之间相互作用,使得在不同因素水平间的扩增结果具有明显区别(谭碧玥等,2009),所以建立一个最优的PCR反应体系是获得高效、稳定的分子标记的前提。正交试验设计在PCR反应体系优化中的运用,可以发挥高效、经济、直观的特点,但由于正交试验设计的试验水平较少,通常不易找到最佳的试验组合(陈万胜等,2008); 单因素分析方法设置的试验水平较多,但是试验程序较繁琐,忽视了各因素间的相互影响(陈海霞等,2011)。2种方法都难以快速独立地找出PCR反应的最佳体系,所以正交设计与单因素分析相 结合在SRAP-PCR反应体系优化试验设计中是一种较好的方法。
在反应体系中Mg2+浓度变化对SRAP条带影响较大。Mg2+浓度过高会产生非特异扩增,杂带较多; 浓度过低会影响Taq酶的活性,降低引物与模板的结合效率,扩增产物减少,条带较弱(梁景霞等,2005)。本研究中,Mg2+浓度在2.75~3.50 mmol·L-1范围内,随着Mg2+用量的增多,条带逐渐清晰丰富。反应体系中Taq酶用量过高会产生大量非特异性条带且背景过深不利于读带; 用量过低则扩增产物的产量较低且背景太淡(赵杨等,2012)。本研究中,Taq酶浓度在1.5 μmol·min-1以下时扩增出的条带很弱; 在1.5~2.5 μmol·min-1时能扩增出清晰稳定的条带; 在3.0 μmol·min-1以上时背景逐渐加深且非特异性位点增多。而模板DNA浓度对PCR反应扩增的结果有一定的影响,但是通常有较宽的适用范围(李娟玲等,2011)。本研究中,模板DNA量在45~135 ng时均能扩增出丰富稳定的条带。综合各方面因素,最终确定了扩增产物多、带型清晰、重复性好的鹅掌楸属SRAP-PCR反应体系: Mg2+3.0 mmol·L-1,dNTPs 0.3 mmol·L-1,引物 0.9 μmol·L-1,Taq酶2.0 μmol·min-1,模板DNA 90 ng,10×PCR buffer 2 μL,不足部分以ddH2O补足。
3.2 鹅掌楸属遗传多样性分析鹅掌楸属的SRAP分子标记分析表明,鹅掌楸属多态性比率为81.87%,高于红花(Carthamus tinctorius)(江磊等,2013)、蒜(Allium sativum)(Chen et al., 2013)、红麻(Hibiscus cannabinus)(Xu et al., 2013)等SRAP多态性比率,同时也高于RAPD(b李周岐等,2002; 李建民等,2002)、EST-SSR(胥猛等,2008)等分子标记对鹅掌楸、北美鹅掌楸、杂交鹅掌楸的多态性比率。说明SRAP分子标记在鹅掌楸属植物具有较高的遗传多样性水平,可以运用于鹅掌楸属的遗传多样性研究。
鹅掌楸和北美鹅掌楸的Shannon信息指数分别为0.365 5和0.329 3,说明鹅掌楸地理种源间的变异略高于北美鹅掌楸。北美鹅掌楸在美国东南部成片分布、数量众多,可能使得其种群内的遗传变异程度较高,种群间的遗传分化程度相对较低。而在我国,鹅掌楸分布星散,依照地理区域可划分为“一带五岛”的分布形式,反映了一个走向濒危的物种在数量上的减少过程(郝日明等,1995)。这种生境片断化、距离隔离以及种群数量减少的分布情况有可能造成鹅掌楸较高的种群间遗传分化程度。这与李建民等(2002)得出的鹅掌楸种群内的遗传变异程度降低、而种群间的遗传分化程度加大的研究结论相符。保护一个具有复杂遗传结构的濒危物种,需要阐明不同种源间的遗传关系(朱晓琴等,1997)。SRAP分子标记有助于鹅掌楸属不同种源间遗传关系的研究。
SRAP标记的聚类分析表明,鹅掌楸属的10个不同地理种源可以聚成北美鹅掌楸和鹅掌楸2大类。与罗光佐等(2000)用RAPD标记的聚类结果不同,SRAP标记的聚类可以很好地将鹅掌楸与北美鹅掌楸区分开。SRAP标记对基因的重要组成部分ORFs进行扩增,而基因的多样性更能反映遗传资源的多样性(林忠旭等,2004)。这体现了SRAP分子标记在鹅掌楸属植物遗传多样性研究中的优越性和有效性,本研究为SRAP分子标记在鹅掌楸属植物的进一步应用奠定了良好的基础。
| [1] |
陈海霞, 彭尽晖. 2011. 绣球属植物SRAP-PCR反应体系优化及引物筛选. 中国农学通报, 27(31): 93-98.( 1) 1)
|
| [2] |
陈万胜, 王元英, 罗成刚, 等.2008. 利用正交设计优化烟草SRAP反应体系. 分子植物育种, 6(1): 177-182.( 1) 1)
|
| [3] |
郝日明, 贺善安, 汤诗杰, 等. 1995. 鹅掌楸在中国的自然分布及其特点. 植物资源与环境, 4(1):1-6.( 1) 1)
|
| [4] |
何正文, 刘运生, 陈立华, 等. 1998. 正交设计直观分析法优化PCR条件. 湖南医科大学学报, 23(4): 76-77.( 1) 1)
|
| [5] |
江磊, 李刚, 岳帅, 等. 2013. 11个红花品种遗传多样性与亲缘关系的SRAP分析. 中国油料作物学报, 35(5):546-550.( 1) 1)
|
| [6] |
李建民, 周志春, 吴开云, 等. 2002. RAPD标记研究马褂木地理种群的遗传分化. 林业科学,38(4):61-66.( 2) 2)
|
| [7] |
李娟玲, 刘国民, 曹嵩晓, 等.2011. 利用单因子和正交设计双重实验法优化鹧鸪茶RAPD-PCR反应体系. 农学学报, 1(4): 14-21.( 1) 1)
|
| [8] |
李周岐, 王章荣. 2002. RAPD标记在鹅掌楸属中的遗传研究. 林业科学, 1(1): 150-153.( 2) 2)
|
| [9] |
梁景霞, 祁建民, 吴为人, 等. 2005. 烟草DNA的提取与SRAP反应体系的建立. 中国烟草学报, 11(4): 33-38.( 1) 1)
|
| [10] |
林忠旭, 张献龙, 聂以春, 等.2003. 棉花SRAP遗传连锁图构建. 科学通报, 48(15): 1676-1679.( 1) 1)
|
| [11] |
林忠旭, 张献龙, 聂以春. 2004. 新型标记SRAP在棉花F2分离群体及遗传多样性评价中的适用性分析. 遗传学报, 31(6):622-626.( 1) 1)
|
| [12] |
罗光佐, 施季森, 尹佟明, 等.2000. 利用RAPD标记分析北美鹅掌楸与鹅掌楸种间遗传多样性. 植物资源与环境学报, 9(2): 9-13.( 2) 2)
|
| [13] |
孙佳琦, 梁建国, 石少川, 等.2010. SRAP标记在观赏植物遗传育种中的应用. 分子植物育种, 8(3): 577-588.( 1) 1)
|
| [14] |
谭碧玥, 王源秀, 徐立安. 2009. 杨树基因组SRAP扩增体系的建立与优化. 林业科技开发, 23(2): 25-29.( 1) 1)
|
| [15] |
唐琴, 曾秀丽, 廖明安, 等.2012. 大花黄牡丹遗传多样性的SRAP分析. 林业科学, 48(1): 70-76.( 1) 1)
|
| [16] |
汪成成, 文山, 董健, 等.2010. 正交设计优化日本落叶松SRAP-PCR反应体系. 沈阳农业大学学报, 41(4): 444-448.( 1) 1)
|
| [17] |
胥猛, 李火根. 2008.鹅掌楸EST-SSR引物开发及通用性分析. 分子植物育种,6(3):615-618.( 1) 1)
|
| [18] |
占志勇, 徐刚标. 2010. 鹅掌楸ISSR-PCR反应体系的建立及优化. 中南林业科技大学学报, 30(6): 80-84.( 1) 1)
|
| [19] |
张红莲, 李火根. 2010. 利用EST-SSR分子标记检测鹅掌楸种间渐渗杂交. 生物多样性, 18(2): 120-128.( 1) 1)
|
| [20] |
赵杨, 李玉璞, 代毅. 2012. 华山松SRAP-PCR反应体系的优化. 西北林学院学报, 27(5): 87-90.( 1) 1)
|
| [21] |
朱晓琴, 贺善安, 姚青菊, 等. 1997. 鹅掌楸居群遗传结构及其保护对策.植物资源与环境, 6(4):7-14.( 1) 1)
|
| [22] |
Aneja B, Yadav N R, Chawla V, et al. 2012. Sequence-related amplified polymorphism (SRAP) molecular marker system and its applications in crop improvement. Molecular Breeding, 30(4): 1635-1648.( 1) 1)
|
| [23] |
Budak H, Shearman R C, Parmaksiz I, et al. 2004. Molecular characterization of Buffalograss germplasm using sequence-related amplified polymorphism markers. Theoretical and Applied Genetics, 108(2): 328-334.( 1) 1)
|
| [24] |
Chen S, Zhou J, Chen Q, et al . 2013. Analysis of the genetic diversity of garlic (Allium sativum L.) germplasm by SRAP. Biochemical Systematics and Ecology, 50(5):139-146.( 1) 1)
|
| [25] |
Esposito M A, Martin E A, Cravero V P, et al. 2007. Characterization of pea accessions by SRAP's markers. Scientia Horticulturae, 113(4): 329-335.( 1) 1)
|
| [26] |
Ferriol M, Pico B, Nuez F. 2003. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers. Theoretical and Applied Genetics, 107(2): 271-282.( 2) 2)
|
| [27] |
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics, 103(2/3): 455-461.( 3) 3)
|
| [28] |
Xu J, Li A, Wang X, et al. 2013.Genetic diversity and phylogenetic relationship of kenaf (Hibiscus cannabinu L.) accessions evaluated by SRAP and ISSR. Biochemical Systematics and Ecology, 49(4): 94-100.( 1) 1)
|
| [29] |
Xu M, Li H, Zhang B. 2006. Fifteen polymorphic simple sequence repeat markers from expressed sequence tags of Liriodendron tulipifera. Molecular Ecology Notes, 6(3): 728-730.( 1) 1)
|
| [30] |
Yang A H, Zhang J J, Tian H, et al. 2012. Characterization of 39 novel EST-SSR markers for Liriodendron tulipifera and cross-species amplification in L. chinense (Magnoliaceae). American Journal of Botany, 99(11): e460-e464.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

