文章信息
- 李江伟, 杨琴军, 刘秀群, 季华, 张卓文
- Li Jiangwei, Yang Qinjun, Liu Xiuqun, Ji Hua, Zhang Zhuowen
- 台湾杉遗传多样性的ISSR分析
- Genetic Diversity of Taiwania cryptomerioides Detected by ISSR Analysis
- 林业科学, 2014, 50(6): 61-66
- Scientia Silvae Sinicae, 2014, 50(6): 61-66.
- DOI: 10.11707/j.1001-7488.20140608
-
文章历史
- 收稿日期:2013-07-01
- 修回日期:2013-10-10
-
作者相关文章
台湾杉(Taiwania cryptomerioides),杉科(Taxodiaceae)台湾杉属(Taiwania),是第三纪古热带植物区系孑遗植物(于永福,1995),为国家一级保护树种。它高大挺拔,四季常绿,树形优美,材质优良,是非常重要的用材树种和庭园绿化树种。曾广泛分布于欧洲和亚洲东部,由于第四纪冰期的影响,现仅残存于我国湖北、贵州、福建、台湾等地,呈星散分布(杨琴军等,2006)。国内外对台湾杉研究多集中在分类、起源(于永福,1995)、系统发育(于永福等,1996)、生态地理分布(胡玉熹等,1995)、群落、种群特征(廖凤林等,2004)、组织培养(庞慧仙等,2005)等方面,有关遗传多样性方面的研究较少,且研究结果存在较大差异。Lin等(1993)用等位酶标记研究了台湾杉4个栽培居群的遗传多样性,结果表明其遗传多样性略低于其他针叶树,且94.7%的遗传变异存在于居群内;Li等(2008)用ISSR分子标记对云南、贵州、福建、湖北的野生居群进行了研究,结果显示台湾杉具有较低的遗传多样性,且72.37%的遗传变异存在于居群间;杨琴军等(2009;2011)调查了湖北原生台湾杉资源,建立了稳定的台湾杉RAPD及ISSR反应体系,并对湖北野生居群进行了初步的遗传多样性研究,发现台湾杉具有较高水平的遗传多样性。不同研究结果的较大差异对了解台湾杉的濒危原因、进而制定科学的保护措施十分不利,因此急需开展深入研究。
ISSR(inter simple sequence repeat)技术是由加拿大蒙特利尔大学发展起来的一种基于微卫星序列的分子标记技术(Zietkiewicz et al.,1994)。它具有稳定性好、多态性高、操作简单、成本低廉等优点(Wolhf et al.,1995; Esselman et al.,1999; McGregor et al.,2000),而且不需要预先知道基因组序列信息,特别适合于对基因组序列尚不清楚的物种进行遗传变异研究(Goodwin et al.,1997; 张立荣等,2002)。本研究采用ISSR技术对湖北、贵州、福建3个省区的台湾杉4个自然居群的遗传多样性水平和遗传结构进行检测,旨在探明台湾杉自然居群的遗传变异状况,为其保护策略的制定提供科学依据。
1 材料与方法 1.1 试验材料研究材料采自湖北、贵州、福建3个省区的4个自然居群(表 1)。根据居群大小,每个居群随机选取9~20个个体,将采集的新鲜嫩叶放入装有硅胶的密封袋中干燥保存。取样时,保证不同个体间距在20 m以上,同时对所取样本的空间位置进行GPS定位。
|
|
采用改良的CTAB法(陈光富等,2008),提取台湾杉基因组DNA,用0.8%琼脂糖凝胶电泳检测DNA样品的完整性。
1.2.2 PCR反应体系的建立和优化采用杨琴军等(2011)建立的优化ISSR反应体系和PCR扩增程序对台湾杉进行试验研究。反应体系为: 在25 μL的反应体系中,含有40 ng模板DNA、1U Taq酶、0.2 mmol·L-1 dNTP、0.4 μmol·L-1 引物、1.8 mmol·L-1 Mg2+、2.5 μL10×buffer。扩增程序为: 94 ℃,4 min; 94 ℃,45 s,51~56 ℃,45 s,72 ℃,2 min,40个循环; 72 ℃,7 min。扩增在PE9600型PCR仪上完成。
1.2.3 引物筛选和最佳退火温度确定所用ISSR引物由上海英俊生物技术公司根据加拿大哥伦比亚大学(UBC)2006年公布的序列合成。利用优化的反应体系,在52 ℃退火温度条件下,对ISSR引物进行筛选,然后,再设置51,52,53,54,55,56 ℃ 6个梯度,确定最佳退火温度,选择能扩增出清晰、稳定条带的引物用于所有样品分析。
1.2.4 PCR扩增产物检测以1×TAE 为电泳缓冲液,取10 μL的扩增产物在含有0.5%EB的1.8%琼脂糖凝胶上电泳约2~3 h(5 V·cm-1),以100 bp DNA Marker作为对照。电泳结束后利用Gel-Logic 200凝胶扫描成像系统拍照并保存。
1.3 数据统计与分析相同迁移位置上有无条带分别记为“1”,“0”,获得0,1二元数据矩阵。利用POPGENE 1.31(Yeh et al.,1999)软件估算多态位点百分率(PPB,percentage of polymorphic loci),Shannon’s 多样性指数(I),Nei’s基因多样性指数(H),Nei’s遗传分化指数(GST)和居群每代迁移数(Nm)。
用AMOVA 1.55软件(张富民等,2002; Excoffier et al.,1992),对台湾杉居群间和居群内的分子变异进行分析。用NTSYS-pc2.10软件(Rohlf,2000)对居群间的遗传距离构建UPGMA聚类图。用TFPGA(Miller,1997)检测居群间的遗传距离与地理距离之间的相关性及显著性水平。
2 结果与分析 2.1 台湾杉遗传多样性从100条引物中筛选出10条引物(杨琴军等,2011),对台湾杉4个居群62个体进行扩增,扩增谱带的分子量在250~2 000 bp之间,共扩增出78条谱带,其中多态性带61条,多态条带百分率为78.21%。不同居群多态条带百分率在35.89%~65.38%之间,平均值为52.56%。
|
|
由表 3可知,台湾杉居群Nei’s遗传多样性(H)在0.126 6~0.200 6之间,平均值为0.178 3;Shannon’s信息指数(I)在0.188 1~0.304 5之间,平均值为0.267 7。在物种水平上,台湾杉的多态条带百分率(PPB)、Nei’s遗传多样性(H)、Shannon’s信息指数(I)分别为78.21%,0.249 4,0.377 8,相对其他一些濒危植物,PPB值高于华木莲(Sinomanglietia glauca)(廖文芳等,2004)、刺参(Oplopanax elatus)(Lee et al.,2002),但低于珙桐(Davidia involucrata)(张玉梅等,2012)、长叶红砂(Reaumuria trigyna)(张颖娟等,2008)等,表明台湾杉具有中等水平的遗传多样性。
|
|
从表 4可知,由Shannon信息指数估算,台湾杉总的遗传变异中,70.86%存在于居群内,29.14%存在于居群间; 根据Nei’s指数估算,台湾杉居群间的遗传分化系数GST为0.285 1。表明4个台湾杉居群的遗传变异主要来自居群内。由GST估算的居群间基因流为1.25。
|
|
利用AMOVA对台湾杉4个自然居群的遗传分化系数进行分析,结果表明: 台湾杉总的遗传变异中有28.31%存在于居群间,71.69%存在于居群内(P<0.001,表 5)。AMOVA的分析结果表明,台湾杉居群间已出现了一定程度的遗传分化。
|
|
台湾杉各居群间Nei’s遗传距离D值的变化范围为0.053 7~0.160 5,其中福建尤溪(FJYX)和贵州雷公山(GZLGS)的遗传距离最大,为0.160 5,而遗传一致度最小; 福建古田屏南(FJGP)和贵州雷公山(GZLGS)的遗传距离最小,为0.053 7,遗传一致度最大(表 6)。
|
|
根据Nei’s遗传一致度,利用UPGMA法构建居群遗传关系聚类图(图 1)。从聚类图可以看出,福建尤溪(FJYX)单独聚为一支; 贵州雷公山(GZLGS)与福建古田屏南(FJGP)优先聚在一起,然后再与湖北利川(HBLC)聚在一起。
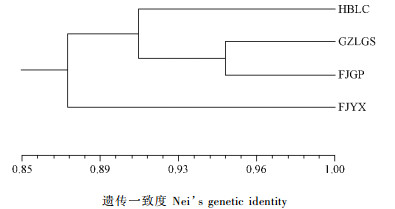 |
图 1 台湾杉居群间Nei’s遗传一致度UPGMA聚类 Fig. 1 UPGMA dendrogram for populations of T. cryptomerioides based on Nei’s genetic identity |
对台湾杉4个居群的Nei’s遗传距离与地理距离进行了Mantel统计检验,并进行1 000次随机重复,结果显示4个居群间的地理距离和遗传距离无显著相关性(r=0.043 3,P=0.435 6),即台湾杉居群遗传多样性分布没有明显的地理趋势。
3 结论与讨论 3.1 台湾杉的遗传多样性遗传多样性是物种长期进化的产物,是居群生存和发展的前提(Barrett et al.,1998)。本研究通过ISSR分析得出: 台湾杉种水平的多态条带百分率(PPB)、Nei’s遗传多样性(H)、Shannon’s信息指数(I)分别为78.21%,0.249 4,0.377 8。这与张瑞麟(2005)(ISSR,PPB=70.83%)、Lin等(1993)(等位酶,PPB=50.2%)、陈光富等(2008)(RAPD,PPB=65.81%)对台湾杉居群遗传多样性的研究结果类似,但远高于Li等(2008)用ISSR标记对台湾杉居群遗传多样性检测的结果(多态条带百分率、Nei’s基因多样性和Shannon’s信息指数分别为38.02%,0.132 6和0.198 6),Li等(2008)取样的4个自然居群有3个与本研究相同(湖北利川、贵州雷公山、福建古田屏南),但遗传多样性参数相差甚远。ISSR技术具有稳定性好、多态性高、操作简单、成本低廉等优点(Wolhf et al.,1995; Esselman et al.,1999; McGregor et al.,2000),不足之处在于稳定性易受到 Taq酶、 Mg2+、引物、退火温度等多个因素的影响。本研究通过正交试验建立了台湾杉优化的ISSR反应体系,利用该优化的反应体系筛选出了10条稳定性强、清晰度高的引物,并进一步对引物的退火温度进行了筛选,以保证试验的准确性。Li等(2008)台湾杉的反应体系是参照Ge等(1999)中红树植物的试验条件确定的,利用此反应体系,筛选出了15条引物用于试验,其中有5条引物与本研究筛选出的引物相同: 4条(AG)重复的引物和1条三碱基重复的引物,即(AG)8T,(AG)8C,(AG)8YC,(AG)8YA,(CTC)6,其他引物则与本试验不相同。试验条件的不同以及引物筛选的差异可能是导致二者研究结果差异较大的主要原因。
Hamrick等(1990)的研究表明,物种的地理分布范围和遗传多样性关联显著。一些地理分布范围狭窄的物种由于遗传漂变和基因流的限制,遗传变异水平通常较低,自然分布范围广的物种通常趋向于更高的遗传多样性(Hamrick et al.,1990; 1996)。因此一般研究认为濒危物种的遗传多样性水平较低(Hamrick et al.,1990; Li et al.,2002),但也有研究表明一些濒危物种维持了较高的遗传多样性(张玉梅等,2012; Kang et al.,2000; Zawko et al.,2001; Xue et al.,2004)。本研究中,台湾杉的遗传多样性在物种水平上丰富。由于历史原因及人为破坏等,濒危种的居群常呈片段化分布,居群规模较小甚至零星分布,但对一些长寿的木本植物(>100年)而言,由于世代较长,居群片段化的遗传效应暂时还不会反映出来(陈小勇,2000),因此,就物种而言,台湾杉仍保留着较高的遗传多样性。
在所有居群中,贵州雷公山居群的多态条带百分率最高,湖北利川的次之,福建尤溪的最低。遗传多样性较高的贵州雷公山居群,处于雷公山自然保护区内,未经历过多的人为干扰,生境相对完好; 而福建尤溪居群,野外调查仅发现9株,零星分布在房屋、农田附近,每天有大量的人畜活动,生境几乎完全破坏,与其他居群相比,植株群体处于衰退状态。因此,生境破坏可能是导致台湾杉物种濒危的一个重要原因。
3.2 台湾杉的遗传结构居群是物种进化的基本单位,而一个物种最基本的特征之一就是居群的遗传结构(王祎玲,2006)。本研究使用不同的方法对台湾杉居群的遗传结构进行分析,Nei’s基因多样性指数、Shannon信息指数以及AMOVA分析结果相似,即台湾杉的遗传变异主要来自居群内,居群内遗传变异大于居群间的遗传变异,不同居群间存在着遗传分化。这与Li等(2008)的研究结果(GST=0.723 7)不同,而与Nybom等(2000)对一些裸子植物的统计结果一致。Nybom等(2000)的研究结果表明: 一年生、自交、演替阶段早期的类群遗传变异主要发生在居群间,相比之下,寿命长、异交、演替阶段晚期的类群遗传变异主要来自居群内。台湾杉属于长寿的木本植物,生命周期长,异交为主,其居群内保持较高的遗传变异与台湾杉物种自身的生物学特性有关。Nybom(2004)对植物居群遗传结构分析表明,混合交配、自交和异交植物的GST值分别为0.40,0.65和0.27,本研究得到的台湾杉GST 值为0.283 1,这正好符合台湾杉风媒传粉、异交为主交配系统的生物学特性。
本研究中,台湾杉居群地理距离与遗传距离没有相关性(r=0.043 3,P=0.435 6),这种变异模式与UPGMA的聚类结果一致。从UPGMA聚类图上可以看出,地理距离最远的贵州雷公山(GZLGS)与福建古田屏南(FJGP)居群优先聚在一起,而地理距离最近的福建尤溪(FJYX)与福建古田屏南(FJGP)居群没有聚在一起,而是单独聚为一支。福建尤溪(FJYX)居群单独聚为一支,可能与其分布区戴云山脉和福建古田屏南(FJGP)居群分布区鹫峰山脉地理阻隔有关,而该居群样本稀少可能是其聚类异常的又一原因。Fischer(2000)认为地理距离与遗传距离之间显著不相关意味着遗传漂变在居群分化中起着重要的作用。本研究中的4个台湾杉居群地理距离相距较远,中间有山脉阻隔,即使台湾杉风媒传粉,基因交流也会受到限制。因此遗传漂变可能是导致台湾杉居群间发生遗传变异的一个重要因素。
很多因素都会影响居群的遗传结构,在影响居群遗传分化的众多因素中,基因流常常被视为使居群遗传结构均质化的主要因素之一(肖猛,2006)。基因流大的物种,居群间的遗传分化小,反之,居群间的遗传分化大(Rowe et al.,1998)。本研究中,台湾杉居群间的基因流Nm=1.253 8,表明台湾杉各居群间存在基因交流。然而现存的台湾杉居群呈零星分布,片段化严重且高度隔离,尽管其异交为主、风媒传粉的生物学特性,但其种子结实率低、萌发困难,因此其后代向外扩散的能力实际相当有限,居群间基因交流几乎不能发生。因此,推测Nm值代表的可能是片段化发生前的基因流大小,反映的是台湾杉历史上连续分布居群的遗传结构。台湾杉为第三纪古热带区系孑遗植物,曾广泛分布于欧洲和亚洲东部,因此,在其祖先时代,台湾杉居群庞大而连续,基因交流频繁畅通,遗传变异丰富。在经历第四纪冰期后,其生境遭到严重破坏,加之人类活动和环境恶化,连续的、大的居群变成了当前小的、隔离的亚居群。由此可见,台湾杉居群间的遗传分化可能是由其片段化前居群间的历史基因流形成的。
3.3 台湾杉的保护策略基于ISSR遗传多样性分析表明,台湾杉在物种水平上仍具有较高遗传多样性。这说明,尽管台湾杉为濒危孑遗物种,但是造成其珍稀、濒危的原因并非是遗传多样性的降低。野外调查发现,台湾杉自然居群多为零星分布,人类的活动使台湾杉所处生境严重片段化,台湾杉的数量和分布区大大减少。同时生境片段化将台湾杉隔离成小居群,导致自交和遗传漂变,使居群走向衰退(Hedrick et al.,2000)。因此,应切实地保护台湾杉的生存环境,禁止滥砍滥伐,并通过行政干预和立法等强制措施停止人为破坏,使居群逐渐恢复生机。
台湾杉属异花传粉植物,雌球花自然授粉率低,结籽时间较晚且数量较少,种子发芽率低,幼树生长缓慢(杨琴军等,2009)。因此有必要对台湾杉的结实特性、生殖能力以及台湾杉种子繁殖和营养繁殖进行研究,突破技术难关,为今后采种育苗、通过无性繁殖快速获得台湾杉个体创造条件。
| [1] |
陈光富,杨琴军,陈龙清.2008.台湾杉DNA提取及RAPD反应体系的建立.湖北农业科学,47(10): 1108-1121.( 2) 2)
|
| [2] |
陈小勇.2000.生境片断化对植物种群遗传结构的影响及植物遗传多样性保护.生态学报,20(5): 884-892.( 1) 1)
|
| [3] |
胡玉熹,林金星,王献溥,等.1995.中国特有植物台湾杉的生物学特性及其保护.生物多样性,3(4): 206 -212.( 1) 1)
|
| [4] |
廖凤林,李久林,吴士章,等.2004.贵州雷公山台湾杉种群结构及空间分布格居.贵州师范大学学报:自然科学版,(2): 6-9.( 1) 1)
|
| [5] |
廖文芳,夏念和,邓云飞,等.2004.华木莲的遗传多样性研究.云南植物研究,26(1): 58-64.( 1) 1)
|
| [6] |
庞惠仙,张光飞,苏文华.2005.秃杉的组织培养及植株再生.植物生理学通讯,41(2): 56-58.( 1) 1)
|
| [7] |
王祎玲.2006.七筋菇植物遗传多样性与分子系统地理学研究.西安:西北大学博士学位论文.( 1) 1)
|
| [8] |
肖猛.2006.濒危植物桃儿七(Sinopodophyllum hexandrum(Royle)Ying)的遗传多样性研究.成都:四川大学博士学位论文.( 1) 1)
|
| [9] |
杨琴军,徐辉,陈龙清,等.2006.湖北原生台湾杉资源及其保护.广西植物,26(5): 551-556.( 1) 1)
|
| [10] |
杨琴军,陈光富,刘秀群,等.2009.湖北星斗山台湾杉居群的遗传多样性研究.广西植物,29(4): 450-454.( 1) 1)
|
| [11] |
杨琴军,袁继林,付强,等.2011.台湾杉ISSR反应体系的建立及检测.华中农业大学学报,30(4): 432-437.( 2) 2)
|
| [12] |
于永福,傅立国.1996.杉科植物的系统发育分析.植物分类学报,34(2):124-141.( 1) 1)
|
| [13] |
于永福.1995.杉科植物的起源,演化及其分布.植物分类学报,33(4): 362-389.( 2) 2)
|
| [14] |
张富民,葛颂.2002.群体遗传学研究中的数据处理方法I.RAPD数据的AMOVA分析.生物多样性,10(4): 438-444.( 1) 1)
|
| [15] |
张立荣,徐大庆,刘大群.2002.SSR和ISSR分子标记及其在植物遗传育种研究中的应用.河北农业大学学报,25(1): 90-94.( 1) 1)
|
| [16] |
张瑞麟.2005.台湾杉遗传多样性之研究.高雄:国立中山大学博士学位论文.( 1) 1)
|
| [17] |
张颖娟,王玉山.2008.濒危小灌木长叶红砂种群的遗传多样性.林业科学,44(12): 43-47.( 1) 1)
|
| [18] |
张玉梅,徐刚标,申响保,等.2012.珙桐天然种群遗传多样性的ISSR标记分析.林业科学,48(8): 62-67.( 2) 2)
|
| [19] |
Barrett B A,Kidwell K K.1998.AFLP-based genetic diversity assessment among wheat cultivars from the Pacific Northwest.Crop Science,38(5): 1261-1271.( 1) 1)
|
| [20] |
Esselman E J,Li J Q,Crawford D J,et al. 1999.Clonal diversity in the rare Calamagrostis porteri ssp.insperata(Poaceae): comparative results for allozymes and random amplified polymorphic DNA(RAPD)and intersimple sequence repeat(ISSR)markers.Molecular Ecology,8(3): 443- 451.( 2) 2)
|
| [21] |
Excoffier L,Smouse P E,Quattro J M.1992.Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data.Genetics,131(2): 479-491.( 1) 1)
|
| [22] |
Fischer P.2000.Time dependent flow in equimolar micellar solutions: transient behaviour of the shear stress and first normal stress difference in shear induced structures coupled with flow instabilities.Rheologica Acta,39(3): 234-240.( 1) 1)
|
| [23] |
Ge X J,Sun M.1999.Reproductive biology and genetic diversity of a cryptoviviparous mangrove Aegiceras corniculatum(Myrsinaceae)using allozyme and intersimple sequence repeat(ISSR)analysis.Molecular Ecology,8(12): 2061-2069.( 1) 1)
|
| [24] |
Goodwin I D,Aitken E A B,Smith L W.1997.Application of inter simple sequence repeat(ISSR)markers to plant genetics.Electrophoresis,18(9): 1524-1528.( 1) 1)
|
| [25] |
Hamrick J L,Godt M J W.1990.Allozyme diversity in plant species//Brown A H D,Clegg M T,Kahler A L.Plant population genetics,breeding,and genetic resources.Sunderland,M A: Sinauer Associates,43-63.( 2) 2)
|
| [26] |
Hamrick J L,Godt M J W.1996.Effects of life history traits on genetic diversity in plant species.Philosophical Transactions of the Royal Society of London Series B: Biological Sciences,351(1345): 1291-1298.( 1) 1)
|
| [27] |
Hedrick P W,Kalinowski S T.2000.Inbreeding depression in conservation biology.Annual Review of Ecology and Systematics,31: 139-162.( 1) 1)
|
| [28] |
Kang U,Chang C S,Kim Y S.2000.Genetic structure and conservation considerations of rare endemic Abeliophyllum distichum Nakai(Oleaceae)in Korea.Journal of Plant Research,113(2): 127-138.( 1) 1)
|
| [29] |
Lee S W,Kim Y M,Kim W W,et al. 2002.Genetic variation of ISSR markers in the natural populations of a rare and endangered tree species,Oplopanax elatus in Korea.J Kor Forest Soc,91: 565-573.( 1) 1)
|
| [30] |
Li Ang,Ge Song.2002.Advances in plant conservation genetics.Biodiversity Science,10(1): 61-71.( 1) 1)
|
| [31] |
Li Zhongchao,Wang Xiaolan,Ge Xunjun.2008.Genetic diversity of the relict plant Taiwania cryptomerioides Hayata(Cupressaceae)in Mainland China.Silvae Genetica,57(4): 242-248.( 4) 4)
|
| [32] |
Lin T P,Lu C S,Chung Y L,et al. 1993.Allozyme variation in four populations of Taiwania cryptomerioides in Taiwan.Silvae Genetica,42: 278-284.( 2) 2)
|
| [33] |
McGregor C E,Lambert C A,Greyling M M,et al. 2000.A comparative assessment of DNA fingerprinting techniques(RAPD,ISSR,AFLP and SSR)in tetraploid potato(Solanum tuberosum L.)germplasm.Euphytica,113(2): 135-144.( 2) 2)
|
| [34] |
Miller M P.1997.Tools for population genetic analyses(TFPGA)1.3: A Windows program for the analysis of allozyme and molecular population genetic data.Computer Software Distributed by Author,4:157.( 1) 1)
|
| [35] |
Nybom H,Bartish I V.2000.Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants.Perspectives in Plant Ecology,Evolution and Systematics,3(2): 93-114.( 1) 1)
|
| [36] |
Nybom H.2004.Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants.Molecular Ecology,13(5): 1143-1155.( 1) 1)
|
| [37] |
Rohlf F J.2000.NTSYS 2.1: numerical taxonomic and multivariate analysis system.New York: Exeter Software.( 1) 1)
|
| [38] |
Rowe G,Beebee T J C,Burke T.1998.Phylogeography of the natterjack toad Bufo calamita in Britain: genetic differentiation of native and translocated populations.Molecular Ecology,7(6): 751-760.( 1) 1)
|
| [39] |
Wolhf K,Zietkiewicz E,Hofstra H,et al. 1995.Identification of chrysanthemum cultivars and stability of fingerprint patterns.Theoretical and Applied Genetics,91(3): 439-447.( 2) 2)
|
| [40] |
Xue Dawei,Ge Xuejun,Hao Gang,et al. 2004.High genetic diversity in a rare,narrowly endemic primrose species: Primula interjacens by ISSR analysis.Acta Botanica Sinica,46(10): 1163-1169.( 1) 1)
|
| [41] |
Yeh F C,Yang R C,Boyle T.1999.POPGENE version 1.31.A Microsoft window based free ware for population genetic analysis.Edmonton: University of Alberta.( 1) 1)
|
| [42] |
Zawko G,Krauss S L,Dixon K W,et al. 2001.Conservation genetics of the rare and endangered Leucopogon obtectus(Ericaceae).Molecular Ecology,10(10): 2389-2396.( 1) 1)
|
| [43] |
Zietkiewicz E,Rafalski A,Labuda D.1994.Genome finger printing by simple sequence repeat(SSR)anchored polymerase chain reaction amplification.Genomics,20(2): 176-183.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

