文章信息
- 谷振军, 章怀云, 张党权, 谢耀坚, 何含杰, 陈丽莉, 彭宽, 刘果, 杨丹
- Gu Zhenjun, Zhang Huaiyun, Zhang Dangquan, Xie Yaojian, He Hanjie, Chen Lili, Peng Kuan, Liu Guo, Yang Dan
- 赤桉CCoAOMT亚家族的基因克隆及可变剪接分析
- Gene Cloning and Alternative Splicing of CCoAOMT Subfamily Genes from Eucalyptus camaldulensis
- 林业科学, 2014, 50(5): 62-69
- Scientia Silvae Sinicae, 2014, 50(5): 62-69.
- DOI: 10.11707/j.1001-7488.20140508
-
文章历史
- 收稿日期:2013-09-25
- 修回日期:2014-01-08
-
作者相关文章
2. 中南林业科技大学 林业生物技术湖南省重点实验室 长沙 410004;
3. 国家林业局桉树研究开发中心 湛江 524022
2. Hunan Provincial Key Laboratory of Forestry Biotechnology Central South University of Forestry and Technology Changsha 410004;
3. China Eucalypt Research Centre Zhanjiang 524022
桉树(Eucalyptus)是目前世界上最重要的制浆造纸原料(谢耀坚等,2007),是全球三大速生材树种之一,并有望成为新型生物质能源树种(周群英等,2013)。近年来,基因组学研究取得了突破性进展。由日本Kazusa DNA研究中心(KDRI)与王子纸业(Oji Paper)合作完成了赤桉(E. camaldulensis)(2n=22,基因组约654 Mb)的基因组测序(Hirakawa et al.,2011),获得了4.2倍覆盖度的基因组草图;由美国能源部(DOE)的联合基因组学研究所(JGI)与IEuGC-EUCAGEN合作完成了巨桉(E. gr and is)(2n=22,基因组约560 Mb)和蓝桉(E. globulus)(2n=22,基因组约650 Mb)的基因组测序(Paiva et al.,2011; Myburg et al.,2011),获得了8倍覆盖度的基因组框架图。可变剪接(alternative splicing)是基因组学研究内容之一。植物基因组中约有60%的基因存在1个或多个可变剪接(Reddy et al.,2013),参与光合作用、胚胎发育、信号传导、逆境胁迫响应和开花调控等重要生理活动(Reddy,2007)。
木质素是复杂的苯丙烷单体聚合物,在植物细胞壁中作为一种填充和粘合物质,与纤维素和半纤维素之间相互紧密粘合。据报道,双子叶植物中的木质素主要由愈创木基单体(G木质素单体)和紫丁香基单体(S木质素单体)组成,并聚合形成愈创木基-紫丁香基木质素(G-S木质素)(李潞滨等,2007)。在木质素单体合成途径中,CCoAOMT(Caffeoyl-CoA O-methyltransferase,咖啡酯乙酰辅酶A氧位-甲基转移酶)亚家族是OMT(O-methyltransferase,氧位-甲基转移酶)基因家族的主要成员,可以控制双子叶植物G-S木质素的含量,及G木质素单体与S木质素单体的比例(谷振军等,2010)。而木质素的单体比例对制浆造纸过程中木质素去除难易程度具有重要影响(Weng et al.,2010; Li et al.,2011)。将木质素从纤维素和半纤维素中去除,会产生大量造纸废液并严重污染环境(Anterola et al.,2002; Wei et al.,2008)。因此,通过实施CCoAOMT基因操作去除或降低桉树木质素对于减少制浆造纸工业污染具有重要意义。研究表明,反义RNA抑制CCoAOMT基因表达的转基因烟草(Nicotiana tabacum)(Meyermans et al.,2000)和转基因杂种杨(Populus tremula ×Populus alba)(Jouanin et al.,2000; Wei et al.,2008)中,总木质素含量可降低10%以上,G木质素单体含量下降,S/G的比值升高。致使木质素单体之间(β-β,β-1,β-5和5-5)的碳-碳共价键数量减少,木质素单体之间的缩合连接点降低,因而在制浆中木质素更容易被去除(Chen et al.,2001)。基于CCoAOMT是决定木质素单体组分比例和木质素去除难易程度的关键基因,本研究通过同源克隆和RACE技术获得赤桉CCoAOMT基因的全长序列,并对该基因进行可变剪接表达分析,为深入研究桉树CCoAOMT基因的功能提供信息。
1 材料与方法 1.1 样品采集赤桉为中南林业科技大学株洲校区的15年生植株,用于提取基因组DNA和总RNA的样品均在同一单株采集。于3月初和6月中旬分别采集当年生叶片和茎段,用于克隆CCoAOMT基因的gDNA和cDNA。采集跨度8个月份的茎段和叶片样品,用于研究CCoAOMT1基因的可变剪接表达规律,采取时间分别为4月上旬、5月中旬、6月中旬、7月中旬、8月中旬、9月中旬、10月中旬、11月下旬。采集的样品洗净后,于液氮中运输,至实验室后超低温冰箱保存。
1.2 引物设计通过分析CCoAOMT亚家族的基因序列特点,依据保守DNA序列设计同源克隆引物,基于同源片段测序结果设计RACE引物,依据cDNA全长序列设计gDNA全长克隆引物(表 1)。为避免基因组污染,CCoAOMT1可变剪接的正向引物和反向引物分别设计在第1个外显子与第3个外显子。
|
|
采用改良CTAB法(刘果等,2013)提取赤桉叶片基因组DNA。赤桉茎段仔细环剥去皮后,快速刮取次生木质部材料,结合采用改良CTAB法(Sangha et al.,2010)/植物RNA试剂盒(Omega)法提取和纯化总RNA。
1.3.2 CCoAOMT基因保守片段的cDNA克隆使用Invitrogen ThermoScript kit进行cDNA第1条链的合成。以此为模板,使用CCoAOMT1-aF、CCoAOMT1-aR和CCoAOMT2-aF、CCoAOMT2-aR引物及Takara Pyrobest酶进行PCR扩增,程序为: 预变性(94 ℃,5 min); 变性(94 ℃,40 s)、退火(45 s)、延伸(72 ℃,120 s),35个循环; 总延伸(72 ℃,8 min)。扩增CCoAOMT1和CCoAOMT2基因的退火温度分别为62 ℃、58 ℃。纯化回收目的产物,连接至pMD-18T载体,转化大肠杆菌(Escherichia coli)DH5α菌株后送阳性克隆到深圳华大基因公司进行测序。
1.3.3 CCoAOMT基因全长cDNA的RACE克隆1)3’-RACE扩增使用SuperScriptTM Ⅱ逆转录酶和包含oligo(dT)的Adapter Primer(AP)锚定引物进行cDNA第1链的合成,再使用3’RACE引物分别与试剂盒通用引物AUAP进行第1轮PCR与巢式PCR扩增,获得目的基因cDNA的3’端侧翼序列。PCR程序为: 预变性(95 ℃,3 min); 变性(94 ℃,30 s)、退火(50 ℃,45 s)、延伸(72 ℃,45 s),35个循环; 总延伸(72 ℃,10 min)。
2)5’-RACE扩增 OligdT反转录后,按照Invitrogen RACE 试剂盒说明书的操作步骤,分别使用5’-RACE1引物和AAP引物进行第1轮PCR,程序为: 预变性(94 ℃,5 min); 变性(94 ℃,30 s)、退火(50 ℃,45 s)、延伸(72 ℃,45 s),35个循环; 总延伸(72 ℃,10 min)。第2轮巢式PCR使用AUAP引物与5’-RACE2引物,程序为: 预变性(94 ℃,5 min); 变性(94 ℃,30 s)、退火(58 ℃,45 s)、延伸(72 ℃,60 s),35个循环; 总延伸(72 ℃,10 min)。
1.3.4 CCoAOMT基因的gDNA克隆分别使用CCoAOMT1-gF/CCoAOMT1-gR、CCoAOMT2-gF/CCoAOMT2-gR这对特异引物,以叶片基因组DNA为模板,使用Takara LA Taq酶进行长距离PCR扩增,程序为: 预变性(95 ℃,5 min); 变性(95 ℃,40 s)、退火(60 ℃,45 s)、延伸(72 ℃,3 min),35个循环; 总延伸(72 ℃,15 min)。
1.3.5 CCoAOMT1基因的可变剪接表达分析通过改良CTAB法提取总RNA,采用DNase(RNase Free)去除残留的基因组DNA,使用Invitrogen ThermoScript kit进行cDNA第1条链的合成。以Acting2管家基因作为内参,检测赤桉CCoAOMT1基因可变剪接的时空表达,程序为: 预变性(94 ℃,2 min); 变性(94 ℃,30 s)、退火(52 ℃,30 s)、延伸(72 ℃,30 s),28个循环; 总延伸(72 ℃,1 min)。扩增产物通过5%非变性PAGE电泳进行检测。
1.3.6 生物信息学分析DNA测序结果用ContigExpress进行拼接获得完整序列,通过NCBI BLAST进行序列比对分析,采用NCBI ORF Finder寻找候选基因的开放读码框,使用NCBI Splign(Kapustin et al.,2008)预测基因组序列中的外显子区域及长度,使用NCBI CDD(Marchler-Bauer et al.,2011)和ExPASy分析氨基酸序列中的保守结构域,采用ClustalX进行氨基酸多序列比对,使用MAGE5软件(Tamura et al.,2011)最大似然法(max likelihood)构建进化树,1 000次重复抽样运算。采用Protein blast在PDB数据库中搜索同源性高且结构已测定的蛋白作为三维结构模板,并将目的蛋白氨基酸序列与模板序列同时提交至Swiss-Model(Arnold et al.,2006),获得目的蛋白初始三维结构模型,采用NAMD(Phillips et al.,2005)的CHARMM力场优化侧链构象,利用ProCheck(Laskowski et al.,1993)和Errat(Colovos et al.,1993)验证模型的合理性和评估模型构建的精炼程度; 搜索NCBI Pubchem数据库获得与目的蛋白对接的配体模型; 利用AutoDock 4.2(Morris et al.,2009)将配体与目的蛋白进行对接,进行构象聚类分析,选取最低能量构象作为配体-目的蛋白复合物的对接构象,采用PyMOL绘制三维立体结构图(Seeliger et al.,2010),使用POV-Ray进行着色渲染。
2 结果与分析 2.1 赤桉CCoAOMT基因全长克隆与序列分析 2.1.1 cDNA全长克隆以赤桉茎段韧皮部总RNA为模板,通过同源克隆法扩增出CCoAOMT1的2条不同长度的cDNA片段(电泳图略),片段较长的为794 bp(命名为CCoAOMT1-L,GenBank登录号HM106291),片段较短的为752 bp (CCoAOMT1-Δ42,GQ889423),通过3’-RACE和5’-RACE技术分别获得cDNA的3’端(212 bp)和5’端(479 bp); 采用ContigExpress软件拼接出赤桉CCoAOMT1的全长cDNA序列(1 020 bp),其ORF长度为738 bp。采用引物CCoAOMT2-aF/R扩增出1条长度为875 bp的cDNA片段(命名为CCoAOMT2,GQ889422),3’-RACE和5’-RACE技术分别获得cDNA的3’端(208 bp)和5’端(718 bp); 采用ContigExpress软件拼接出赤桉CCoAOMT2的全长cDNA序列(1 047 bp),其ORF长度为741 bp。
将所获得的赤桉CCoAOMT基因核苷酸序列翻译为氨基酸序列,并在Swiss-Prot蛋白质专家数据库中进行Blast比对,结果显示目的序列与其他植物CCoAOMT高度相似。本文克隆的赤桉EcCCoAOMT1-L(氨基酸序列登录号为ADI43381)和EcCCoAOMT1-Δ42(ACX37697)与蓝桉CCoAOMT1(UniProtKB登录号: O81185,下同)、冈尼桉(E. gunnii)CCoAOMT1(O04854)、葡萄(Vitis vinifera)CCoAOMT1(Q43237)等基因的氨基酸序列具有较高的相似性(图 1)。而本文克隆的赤桉EcCCoAOMT2(氨基酸序列登录号为ACX37696)与蓝桉CCoAOMT2(Q9SWB8)、百日草(Zinnia elegans)CCoAOMT1(Q41720)等基因的氨基酸序列具有较高的相似性。
 |
图 1 利用最大似然估计法构建赤桉与其他陆生植物CCoAOMT基因的进化树
Fig. 1 Maximum Likelihood phylogeny of CCoAOMT gene from E. camaldulensis and other plants
|
以赤桉基因组DNA为模板,分别采用CCoAOMT1-gF/R和CCoAOMT2-gF/R引物,获得了CCoAOMT1和CCoAOMT2的gDNA全长序列(图略)。序列分析结果显示,CCoAOMT1的gDNA为1 756 bp(GU109373),没有出现缺失42 bp的条带; CCoAOMT2的gDNA为1 966 bp(GU109372)。
2.2 赤桉CCoAOMT基因的外显子分析与可变剪接识别CCoAOMT1的2条cDNA序列的ClustalX比对结果表明,CCoAOMT1-Δ42除了比CCoAOMT1-L缺失42 bp外,其他区域序列完全相同。采用Splign程序进行外显子分析,结果表明,CCoAOMT1基因组序列中包含5个外显子,CCoAOMT1-Δ42的cDNA中缺失的42 bp是在第1个外显子的区域(表 2,图 2A,B)。CCoAOMT1-L是从CCoAOMT1基因可编码区域的(CDS)109 bp处开始剪切,而CCoAOMT1-Δ42则从CDS的67 bp处开始剪切(图 3),2个剪切方式都符合内含子GT-AG边界规则,且缺失42 bp的剪切方式不影响其余氨基酸的读码框顺序(图 4)。CCoAOMT2基因组序列中亦含有5个外显子(表 2,图 2C)。与CCoAMT1-L相比,CCoAOMT2在第5外显子编码序列上多出3 bp,多编码1个氨基酸,其余外显子的长度完全相同,编码的氨基酸序列也基本相同(图 4)。
|
|
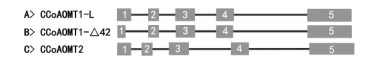 |
图 2 赤桉CCoAOMT基因组序列的外显子结构
Fig. 2 Exon profile in CCoAOMT gDNA of E. camaldulensis
|
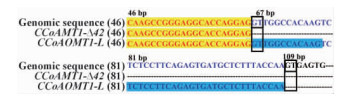 |
图 3 CCoAOMT1的第1个外显子可变剪接位点
Fig. 3 Alternative splicing site in exon 1 of CCoAOMT1
|
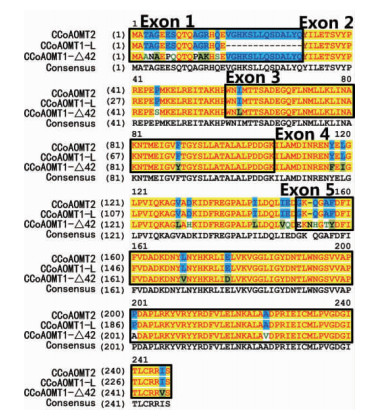 |
图 4 赤桉CCoAOMT亚家族的氨基酸序列比对
Fig. 4 Amino acid alignment of CCoAOMT subfamily
|
可变剪接的正向引物和反向引物分别设计在CCoAOMT1的第1个外显子与第3个外显子中,因而CCoAOMT1-L与CCoAOMT1-Δ42的预期扩增条带的长度分别为228 bp和186 bp。通过非变性PAGE电泳检测PCR产物(图 5),结果显示,在叶片中CCoAOMT1-L基因在4—10月都有表达,而仅在7月和8月检测到CCoAOMT1-Δ42的可变剪接表达; 在茎段中在4—11月都检测到CCoAOMT1-L基因的表达,而CCoAOMT1-Δ42则在5—9月都有可变剪接表达。
 |
图 5 赤桉叶片和茎段中CCoAOMT1基因的可变剪接表达
Fig. 5 Alternative splicing expression profile of
CCoAOMT1 in E. camaldulensis leaves and stems
|
通过NCBI的CDD程序分析保守结构域,表明赤桉CCoAOMT1-L,CCoAOMT1-Δ42和CCoAOMT2基因所编码的蛋白中都含有1个相同的结构域,属于AdoMET-MTases超家族(利用腺苷蛋氨酸的转甲基酶超家族,OMT也属于其中成员)的Methyltransfer-3亚家族。通过ExPASy预测,其N端包含1个帮助形成二聚体的结构域,C端包含1个Methyltransfer-3亚家族的结构域,其中含有3个潜在的SAM结合位点"LIDLVKVGGLI","VAPADAPLRKYV","ALAVDPRIEI",以及1个"VVDVGGGTG"二价金属阳离子绑定区域。CCoAOMT能催化转移腺苷蛋氨酸(SAM)上的甲基至相应底物的苯环上第3位或第4位、第5位上的羧基氧位。通过蛋白质三维结构同源建模,利用AUTODOCK软件将SAH 结合到蛋白质酶活催化口袋中(图 6),表明CCoAOMT蛋白质的酶活性中心能形成正确的"口袋"。结构分析结果表明,赤桉CCoAOMT与紫花苜蓿(Medicago truncatula)CCoAOMT的蛋白结构(Ferrer et al.,2005)类似,其酶活性中心SAM结合位点和底物结合位点在其蛋白质的C端,而赤桉CCoAOMT1基因的可变剪接产生的缺失发生在编码蛋白质的N端,该可变剪接不会影响酶活性中心的三维结构。
 |
图 6 赤桉CCoAOMT1的三维结构及SAM/SAH甲基供体和催化底物结合位点
Fig. 6 SAM/SAH binding site and catalytic domain basing on 3D structure of CCoAOMT1
|
抑制CCoAOMT基因的表达能降低体内木质素含量、提高木质素单体S/G的比例,使得木质素在制浆中更容易去除(Meyermans et al.,2000; Wei et al.,2008)。CCoAOMT蛋白为依靠S腺苷蛋氨酸的氧位-甲基转移酶(S-adenosyl-L-Met(SAM)-dependent OMT),其N端含有蛋白质二聚体化结构域(dimerization domain),氨基酸序列在不同物种之间保守性不高,而其C端则是氧位-甲基转移酶家族-3(OMT-3)的催化活性结构域,其一级结构和三级结构在不同物种间都高度保守(Ferrer et al.,2005)。通过分析所克隆的赤桉CCoAOMT1和CCoAOMT2基因的全长序列,二者所编码的蛋白质C端序列保持高度一致。基因外显子预测与蛋白结构分析结果显示,CCoAOMT1和CCoAOMT2蛋白的N端二聚体化结构域均是由基因的第1个外显子编码,而C端的催化活性中心则是由基因的第3、第4和第5个外显子编码。对于CCoAOMT1基因而言,第1个内含子的下游42 bp还存在1个5’端可变剪接位点(5’ splice site),使得CCoAOMT1基因能编码2种mRNA(CCoAOMT1-L和CCoAOMT1-Δ42)。由于CCoAOMT蛋白的OMT酶活性中心位于其C端,而CCoAOMT1-Δ42基因的可变剪接发生在第1个外显子上,因而这个可变剪接不会改变所编码蛋白质的C端OMT酶活中心结构,其氧位甲基转移酶的催化活性亦不会受到影响。
蛋白突变研究结果表明,植物OMT蛋白的二聚体化在酶底物识别上起到关键作用(Hoffmann et al.,2001),而异源二聚体蛋白能对其他结构类似的底物表现出OMT酶活性(Frick et al.,1999)。基于紫花苜蓿CCoAOMT的蛋白质晶体结构研究结果(Ferrer et al.,2005)和蜡样芽孢杆菌(Bacillus cereus)BcOMT2蛋白(在三维结构和功能上与紫花苜蓿CCoAOMT类似的OMT酶)的二聚体化机制研究结果(Cho et al.,2008),推测发生在N端二聚体化结构域上的可变剪接将导致赤桉CCoAOMT1-Δ42蛋白与其他CCoAOMT蛋白之间形成异源二聚体蛋白,使其能识别咖啡酰乙酰辅酶A(CCoAOMT底物)和与其结构类似的其他化合物作为催化底物,扩大木质素合成前体的来源。因而,可认为这一可变剪接方式是CCoAOMT实现蛋白功能多样性的重要机制。
与其他生物相比,植物有其特异的剪切体识别机制(Barta et al.,1986),不同基因间的内含子可变剪接识别方式和剪切组装机制差异较大(Koncz et al.,2012)。到目前为止,拟南芥(Arabidopsis thaliana)(Baek et al.,2008; Filichkin et al.,2010)、毛竹(Phyllostachys edulis)(Peng et al.,2010; 2013)、毛果杨(Populus trichocarpa)(Ko et al.,2012; Bao et al.,2013)等基因组学研究都表明CCoAOMT基因在木质部中高丰度表达,但没有关于CCoAOMT基因可变剪接的直接研究报道。为了进一步探索赤桉CCoAOMT1基因可变剪接的作用,进行了不同月份茎段和叶片中CCoAOMT1-L和CCoAOMT1-Δ42的表达分析,结果显示在叶片中仅7月和8月有可变剪接表达,在茎段中可变剪接则发生在5—9月之间,而这段时期是桉树胸径快速增长期,是一年中木质部生长轮的早材形成阶段(陈文友,2001; 施钊筑等,1990)。Yoshinaga等(1997)对木质部生长轮的亚细胞观测结果表明,在一年周期中,早材木质素中富含G木质素单体,而晚材中则富含S木质素单体。基于本文的可变剪接表达结果,推测赤桉CCoAOMT1的可变剪接与早材中G木质素单体的大量沉淀有相关性,其可能的机制是,在桉树茎段生长旺盛时期,通过CCoAOMT1-Δ42异源二聚体蛋白的结构类似底物识别功能,从而在G木质素合成前体缺乏的情况下也能维持G木质素生物合成通路的稳定。
本文获得的桉树CCoAOMT亚家族的基因全长cDNA与基因组DNA序列,是木质素单体合成与单体比例调控的关键基因,将为进一步研究细胞壁木质素沉积机制、木质素含量降低及木质素更容易去除的转基因育种研究提供理论依据和基因资源。对于目前热点的低木质素转基因林木定向育种研究而言,通过RNA干扰技术抑制CCoAOMT基因表达或敲除CCoAOMT基因都是良好的育种策略。
| [1] |
陈文友.2001.巨桉人工林树高直径年生长分析研究.四川林业科技,22(1): 73-75.( 1) 1)
|
| [2] |
谷振军,张党权,黄青云,等.2010.木质素合成关键酶基因与造纸植物转基因改良应用研究.中南林业科技大学学报,30(3): 67-74.( 1) 1)
|
| [3] |
李潞滨,刘蕾,何聪芬,等.2007.木质素生物合成关键酶基因的研究进展.分子植物育种,5(6S): 45-51.( 1) 1)
|
| [4] |
刘果,张党权,谢耀坚,等.2013.桉树Genomic-SSR和EST-SSR引物的快速筛选与通用性研究.林业科学,49(2): 127-133.( 1) 1)
|
| [5] |
施钊筑,黄连胜.1990.柠檬桉生长情况调查报告.福建林业科技,61(3): 42-47.( 1) 1)
|
| [6] |
谢耀坚,谭晓风.2007.尾叶桉遗传资源引进及其生物学入侵风险评估.中南林业科技大学学报,27(6): 169-173.( 1) 1)
|
| [7] |
周群英,陈少雄,韩斐扬,等.2013.短周期尾巨桉能源林生物量与能量特征研究.热带亚热带植物学报,21(1): 45-51.( 1) 1)
|
| [8] |
Anterola A M,Lewis N G.2002.Trends in lignin modification: a comprehensive analysis of the effects of genetic manipulations/mutations on lignification and vascular integrity.Phytochemistry,61(3): 221-294.( 1) 1)
|
| [9] |
Arnold K,Bordoli L,Kopp J,et al. 2006.The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling.Bioinformatics,22(2): 195-201.( 1) 1)
|
| [10] |
Baek J M,Han P,Iandolino A,et al. 2008.Characterization and comparison of intron structure and alternative splicing between Medicago truncatula,Populus trichocarpa,Arabidopsis and rice.Plant Mol Biol,67(5): 499-510.( 1) 1)
|
| [11] |
Bao H,Li E,Mansfield S D,et al. 2013.The developing xylem transcriptome and genome-wide analysis of alternative splicing in Populus trichocarpa(black cottonwood)populations.BMC Genomics,14: 359.( 1) 1)
|
| [12] |
Barta A,Sommergruber K,Thompson D,et al. 1986.The expression of a nopaline synthase-human growth hormone chimaeric gene in transformed tobacco and sunflower callus tissue.Plant Molecular Biology,6(5): 347-357.( 1) 1)
|
| [13] |
Chen C,Baucher M,Holst C J,et al. 2001.Biotechnology in trees: Towards improved paper pulping by lignin engineering.Euphytica,118(2): 185-195.( 1) 1)
|
| [14] |
Cho J H,Park Y,Ahn J H,et al. 2008.Structural and functional insights into O-methyltransferase from Bacillus cereus. J Mol Biol,382(4): 987-997.( 1) 1)
|
| [15] |
Colovos C,Yeates T O.1993.Verification of protein structures: patterns of nonbonded atomic interactions.Protein Sci,2(9): 1511-1519.( 1) 1)
|
| [16] |
Ferrer J L,Zubieta C,Dixon R A,et al. 2005.Crystal structures of alfalfa caffeoyl coenzyme A 3-O-methyltransferase.Plant Physiol,137(3): 1009-1017.( 3) 3)
|
| [17] |
Filichkin S A,Priest H D,Givan S A,et al. 2010.Genome-wide mapping of alternative splicing in Arabidopsis thaliana. Genome Res,20(1): 45-58.( 1) 1)
|
| [18] |
Frick S,Kutchan T M.1999.Molecular cloning and functional expression of O-methyltransferases common to isoquinoline alkaloid and phenylpropanoid biosynthesis.The Plant Journal,17(4): 329-339.( 1) 1)
|
| [19] |
Hirakawa H,Nakamura Y,Kaneko T,et al. 2011.Survey of the genetic information carried in the genome of Eucalyptus camaldulensis. Plant Biotechnology,28(5): 471-480.( 1) 1)
|
| [20] |
Hoffmann L,Maury S,Bergdoll M,et al. 2001.Identification of the enzymatic active site of tobacco caffeoyl-coenzyme A O-methyltransferase by site-directed mutagenesis.Journal of Biological Chemistry,276(39): 36831-36838.( 1) 1)
|
| [21] |
Jouanin L,Goujon T,Nadai V,et al. 2000.Lignification in transgenic poplars with extremely reduced caffeic acid O-methyltransferase activity.Plant Physiology,123(4): 1363-1374.( 1) 1)
|
| [22] |
Kapustin Y,Souvorov A,Tatusova T,et al. 2008.Splign: algorithms for computing spliced alignments with identification of paralogs.Biol Direct,3: 20.( 1) 1)
|
| [23] |
Ko J H,Kim H T,Wang I H,et al. 2012.Tissue-type-specific transcriptome analysis identifies developing xylem-specific promoters in poplar.Plant Biotechnol J,10(5): 587-596.( 1) 1)
|
| [24] |
Koncz C,Dejong F,Villacorta N,et al. 2012.The spliceosome-activating complex: molecular mechanisms underlying the function of a pleiotropic regulator.Front Plant Sci,3: 9.( 1) 1)
|
| [25] |
Laskowski R A,MacArthur M W,Moss D S,et al. 1993.PROCHECK: a program to check the stereochemical quality of protein structures.Journal of Applied Crystallography,26(2): 283-291.( 1) 1)
|
| [26] |
Li X,Chapple C.2011.Understanding lignification: Challenges beyond monolignol biosynthesis.Plant Physiology,154: 449-452.( 1) 1)
|
| [27] |
Marchler-Bauer A,Lu S,Anderson J B,et al. 2011.CDD: a Conserved Domain Database for the functional annotation of proteins.Nucleic Acids Res,39(Database issue): D225-229.( 1) 1)
|
| [28] |
Meyermans H,Morreel K,Lapierre C,et al. 2000.Modifications in lignin and accumulation of phenolic glucosides in poplar xylem upon down-regulation of caffeoyl-coenzyme A O-methyltransferase,an enzyme involved in lignin biosynthesis.J Biol Chem,275(47): 36899-36909.( 2) 2)
|
| [29] |
Morris G M,Huey R,Lindstrom W,et al. 2009.AutoDock4 and AutoDockTools4: Automated docking with selective receptor flexibility.J Comput Chem,30(16): 2785-2791.( 1) 1)
|
| [30] |
Myburg A A,Grattapaglia D,Tuskan G A,et al. 2011.Assembly and annotation of the Eucalyptus genome sequence.Plant & Animal Genome XIX Conference W226.The Eucalyptus Genome Network(EUCAGEN): 15-19.( 1) 1)
|
| [31] |
Paiva J A P,Prat E,Vautrin S,et al. 2011.Advancing Eucalyptus genomics: identification and sequencing of lignin biosynthesis genes from deep-coverage BAC libraries.BMC Genomics,12: 137.( 1) 1)
|
| [32] |
Peng Z H,Lu T,Li L,et al. 2010.Genome-wide characterization of the biggest grass,bamboo,based on 10,608 putative full-length cDNA sequences.BMC Plant Biology,10(1): 116.( 1) 1)
|
| [33] |
Peng Z H,Lu Y,Li L,et al. 2013.The draft genome of the fast-growing non-timber forest species moso bamboo(Phyllostachys heterocycla).Nat Genet,45(4): 456-461.( 1) 1)
|
| [34] |
Phillips J C,Braun R,Wang W,et al. 2005.Scalable molecular dynamics with NAMD.Journal of Computational Chemistry,26(16): 1781-1802.( 1) 1)
|
| [35] |
Reddy A S,Marquez Y,Kalyna M,et al. 2013.Complexity of the alternative splicing landscape in plants.Plant Cell,25(10): 3657-3683.( 1) 1)
|
| [36] |
Reddy A S.2007.Alternative splicing of pre-messenger RNAs in plants in the genomic era.Annu Rev Plant Biol,58: 267-294.( 1) 1)
|
| [37] |
Sangha J,Gu K,Kaur J,et al. 2010.An improved method for RNA isolation and cDNA library construction from immature seeds of Jatropha curcas L.BMC Research Notes,3(1): 126.( 1) 1)
|
| [38] |
Seeliger D,Groot B L.2010.Ligand docking and binding site analysis with PyMOL and Autodock/Vina.J Comput Aided Mol Des,24(5): 417-422.( 1) 1)
|
| [39] |
Tamura K,Peterson D,Peterson N,et al. 2011.MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Molecular Biology and Evolution,28(10): 2731-2739.( 1) 1)
|
| [40] |
Wei J H,Wang Y Z,Wang H Z,et al. 2008.Pulping performance of transgenic poplar with depressed caffeoyl-CoA O-methyltransferase.Chinese Science Bulletin,53(22): 3553-3558.( 3) 3)
|
| [41] |
Weng J K,Akiyama T,Bonawitz N D,et al. 2010.Convergent evolution of syringyl lignin biosynthesis via distinct pathways in the lycophyte Selaginella and flowering plants.Plant Cell,22(4): 1033-1045.( 1) 1)
|
| [42] |
Yoshinaga A,Fujita M,Saiki H.1997.Cellular distribution of guaiacyl and syringyl lignins within an annual ring in oak wood.Mokuzai Gakkaishi,43(5): 384-390.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

