文章信息
- 李长江, 崔晓燕, 孙帆, 张凌云
- Li Changjiang, Cui Xiaoyan, Sun Fan, Zhang Lingyun
- 青杄干旱诱导基因PwWDS1的cDNA分离与表达分析
- Isolation and Expression Analysis of PwWDS1 in Picea wilsonii
- 林业科学, 2014, 50(4): 129-136
- Scientia Silvae Sinicae, 2014, 50(4): 129-136.
- DOI: 10.11707/j.1001-7488.20140419
-
文章历史
- 收稿日期:2013-06-13
- 修回日期:2013-11-10
-
作者相关文章
2. 吉林大学植物科学学院长春 130062
2. College of Plant Science, Jilin University Changchun 130062
植物干旱诱导蛋白是植物体在遭遇干旱胁迫或缺水胁迫时被诱导表达或者表达量被上调的一类蛋白,通过直接作用或者调节其他基因的表达来使植物适应干旱逆境(Desclos et al.,2008;Urao et al.,1994)。这类蛋白在功能上分为2类:其一为功能蛋白,在细胞内直接参与保护作用,如LEA(lateembryogenesis abundant)蛋白是广泛存在于植物的一类干旱诱导蛋白。在水稻(Oryza sativa)中的研究表明,OsLEA3-1能被干旱与ABA诱导,且过表达该基因能显著增加植株的耐旱能力(Xiao et al.,2007);在欧洲油菜(Brassica napus)中发现BnLEA4-1能被非生物胁迫及ABA诱导,同时具有提高植物耐受干旱与盐胁迫的功能(Dalal et al.,2009);同样,在拟南芥(Arabidopsis thaliana)、盐芥(Thellungiella salsuginea)、牧豆树(Prosopis juliflora)等植物中也发现具有抗旱功能的LEA蛋白,研究表明LEA蛋白具有非常强的亲水能力,在水分匮乏时保持植物细胞内的水分含量(Hundertmark et al.,2008;George et al.,2009;Zhang et al.,2012)。另外,水通道蛋白(water channel protein)、离子通道蛋白(ion channel protein)、渗调蛋白(osmotin)等也在植物抵御缺水胁迫的进程中直接起到重要作用(Du et al.,2011;Goel et al.,2010;Osakabe et al.,2013)。第2类为调节蛋白,主要参与干旱胁迫的信号转导或通过调节其他基因或蛋白的表达间接地起到抗旱作用,这类蛋白主要有转录因子、磷脂酶、蛋白激酶等。在拟南芥中发现转录因子MYB96通过激活LTP3基因的表达正调控植株的抗旱能力(Guo et al.,2013);DREB(dehydration-responsive elementbinding)家族也是一类干旱诱导的转录因子,在胡杨(Populus euphratica)中分离到1个PeDREB2a基因,该基因能被干旱、盐胁迫诱导,且能增加转基因拟南芥对干旱的耐受性(Kizis et al.,2002;Zhou et al.,2012);在水稻的研究中发现,OsCDPK1编码钙依赖的蛋白激酶,过表达该基因的植株具有明显的抗旱能力(Ho et al.,2013);在拟南芥中磷脂酶PLDalpha1能通过产生磷脂酸促进叶片气孔的关闭,从而保持植物体内的水分,将该蛋白在欧洲油菜中表达,也能增加欧洲油菜的抗旱能力(Lu et al.,2013);另外,磷脂酶D(phospholipase D)也在干旱信号转导中起到重要作用(Hong et al.,2010)。
在植物抗旱过程中,植物激素脱落酸(ABA)参与部分抗旱基因表达的调控(Kizis et al.,2002;Yoshida et al.,2010),当植物体受到脱水胁迫后,ABA合成增加,继而通过信号的传导调控抗旱基因的表达,使植物体叶片气孔开度和密度降低、叶片面积减少、花期提前等(Zhao et al.,2010;Sun et al.,2011);另外,植物内源硫化氢(hydrogen sulfide)也被发现参与抗旱基因的调控,在这个过程中半胱氨酸脱硫基酶(cysteine desulfhydrase)起到重要的作用(Jin et al.,2011)。
青杄(Picea wilsonii)属于松科(Pinaceae)云杉属(Picea)植物,是多年生木本针叶树种,适应力强,耐寒、耐阴性强,为我国重要的园林绿化、造林及用材树种。在火炬松(Pinus taeda)中鉴定的LP3基因,在缺水与ABA处理后被诱导表达(Lorenz et al.,2011;Padmanabhan et al.,1997)。本研究基于青杄cDNA文库,利用RACE PCR法分离到1个干旱诱导蛋白基因,通过序列分析发现该基因与火炬松中LP3(Lorenz et al.,2011;Padmanabhan et al.,1997)具有较高的同源性,且具有ABA/WDS结构域。RT-qPCR结果表明该基因能被干旱、盐胁迫、ABA与乙烯利诱导,将其命名为PwWDS1(GenBank登录号: KJ526353)。本研究将为林木中抗逆基因的功能研究,为林木遗传育种、优质林木基因的筛选及同属珍稀濒危树种保护等提供依据。
1 材料与方法 1.1 试验材料1)植物材料青杄种子与花粉采集于中国科学院北京植物园,3年生青杄幼苗的根、茎与针叶用于组织特异表达试验,8周的青杄幼苗用于胁迫或ABA处理,于温室内培养,光周期为16 h光照/8 h黑暗,空气湿度为50%,每周定时充分浇灌自来水1次。试验所需植物材料采集或处理后用液氮处理,置于-80 ℃备用。
2)试验试剂pDONR222为文库载体,由Invitrogen公司提供,pEASY-T1载体购于全式金公司,Taq PCR Mastermix、DNA marker(DL2000)、RT-qPCR试剂盒(SYBR Green SuperReal PreMix)、大肠杆菌(Escherichia coli)感受态DH5α、RNAprepPure多糖多酚植物总RNA提取试剂盒等购于天根公司,反转录试剂盒(RevertAidTM First Str and cDNASynthesis Kit)购于Fermentas公司。其他试剂均购于AMRESCO公司。
1.2 试验方法1)PwWDS1基因cDNA的分离青杄的全长cDNA文库由英潍捷基(Invitrogen)公司完成,构建于pDONR222载体上,作为RACE PCR的模板(李长江等,2012;张盾等,2012)。5’-RACE PCR引物为5-race-f: 5 ’-CAGTCTTAAGCTCGGGCCCCA-3’与5-race-r: 5 ’-CATGCTTCTCATACAGTGCATA-3 ’;3 ’-RACE PCR引物为3-race-f: 5’-TGCTCTGTGGGTGTCAAACAAA-3’与3-race-r: 5’-CTGCCAGGAAACAGCTATGAC-3’。经PCR、胶回收、测序后获得PwWDS1的末端序列,与EST序列拼接后获得PwWDS1全长cDNA序列,利用WDS1-f: 5’-ATGTCTGAAGGGAATCGTCACC-3’与WDS-r: 5’-TTAGAAGAGATGGTGGCGGTGC-3’,通过PCR克隆得到PwWDS1的完整编码框序列,构建于pEASY-T1载体上。
2)生物信息学分析运用DNAMAN软件进行核酸序列拼接及翻译,运用ProtParam工具(http://web.expasy.org/protparam/)分析蛋白的氨基酸组成及理化特性。利用TMHMM工具(http://www.cbs.dtu.dk/services/TMHMM-2.0/)进行蛋白跨膜预测;利用ProtScale工具(http://web.expasy.org/cgi-bin/protscale/protscale.pl)进行蛋白疏水性分析;运用GOR4工具(http://gor.bb.iastate.edu/)进行蛋白的二级结构分析;多序列比对由ClustalX工具完成,系统发育树由MEGA5构建完成,构建系统树计算方法为邻位相连法,bootstrap检测设置重复为1 000次。
3)组织特异表达试验用于试验的3年生青杄的根、茎、针叶、种子及花粉等总RNA的提取利用天根公司的植物RNA提取试剂盒。运用Fermentas公司的反转录试剂盒合成cDNA第1条链。均一化浓度之后在StepOnePlus Real Time RT-PCR仪器与S1000TM Thermal Cycler PCR仪上进行RT-qPCR与半定量RT-PCR检测基因在不同组织的相对表达量,所用特异引物为RT-PwWDS1-F: 5 ’-GAAAGGAGGAGGACGACGATAA-3’与RT-PwWDS1-F: 5’-GGTAATCAGAGCCGGCGTTATA-3’,内参基因为青杄延伸因子EF1-α(Yu et al.,2011),引物为EF1-α-F: 5 ’-AACTGGAGAAGGAACCCAAG-3 ’与EF1-α-R: 5’-AACGACC CAATGGAGGATAC-3’;试验进行3次重复。
4)种子萌发试验种子萌发试验采用水培法(李长江等,2013)。将吸水滤纸铺在培养皿中,青杄种子分散放入湿润的滤纸上培养,试验过程中保持培养皿底部水分充足。将培养皿置于25 ℃光照培养箱中,光周期为16 h光照/8 h黑暗,培养12天,分别在培养0,2,4,6,8,10,12天时采样,并置于-80 ℃保存备用。提取各时期萌发幼体总RNA后,经反转录、半定量RT-PCR与RT-qPCR等试验检测各个时期PwWDS1的表达量,各个时期取样、半定量RT-PCR与RT-qPCR试验均设置3次重复。
5)逆境与激素响应试验逆境与激素处理参考文献(Yamaguchi-Shinozaki et al.,1994;Yu et al.,2012),略有改动。干旱试验中将8周幼苗置于吸水纸上3,6,12 h;盐胁迫试验中将8周幼苗的根浸入100 mmol·L-1 NaCl溶液中,分别于3,6,12 h取样;激素处理中将8周幼苗的根分别浸入100 nmol·L-1ABA、100 nmol·L-1乙烯利溶液中,分别于3,6,12 h取样。所有试验的对照组均为正常生长的植株。所有试验进行3次重复。材料取样后进行液氮冻存,于-80 ℃备用。
2 结果与分析 2.1 青杄WDS1的克隆PwWDS1全长cDNA序列共1 077 bp,在第6 bp处发现起始密码子ATG,在627 bp处发现终止密码子TAA,编码207个氨基酸。3’非编码区为426 bp,在序列末端发现Poly(A)22尾巴(图 1A),将翻译的氨基酸序列在NCBI中CDD工具检索,结果表明该蛋白在118—180 aa处具有ABA/WDS结构域(Bit Score: 66.72;E-value:6.14e-15)(图 1B)。
 |
图 1 PwWDS1的核酸序列及其蛋白的保守结构域
Fig. 1 Nucleotide sequence of PwWDS1 and conserved domain of PwWDS1
A: PwWDS1的核酸序列与蛋白的氨基酸序列; B: PwWDS1 的保守域预测。 A: Full length cDNA sequence and deduced protein amino acid sequence of PwWDS1; B: Conserved domain analysis of PwWDS1. |
1)理化性质预测 Protpapram工具分析蛋白的分子质量为22.76 kDa,理论等电点为5.02。蛋白的分子式预测为C950H1400N298O356 S2,其中甘氨酸(Gly)含量最多,为14.0%,丙氨酸(Ala)含量10.6%,丝氨酸(Ser)含量10.1%,苏氨酸(Tyr)含量9.7%。蛋白的不稳定指数为42.48,预测蛋白比较稳定。
利用Protscale工具预测蛋白的疏水性,结果发现蛋白的大部分序列,尤其是N端的氨基酸序列,具有较强的亲水性,蛋白的疏水分值最小为-3.700(Arg9,Lys10),最大值为1.522(Ala171),推断蛋白为亲水性蛋白(图 2A)。SignalP工具预测蛋白没有信号肽结构域,TMHMM工具预测蛋白没有跨膜结构(图略)。利用FoldIndex工具进行蛋白固有无序化预测,结果说明蛋白的无序化序列较多,推测在生理环境下蛋白的动态活性较大(图 2B)。
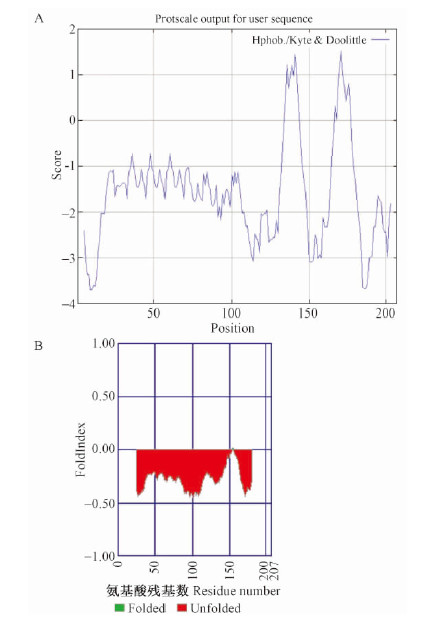 |
图 2 PwWDS1的疏水性分析(A)与固有无序化分析(B)
Fig. 2 Hydrophobic analysis(A) and intrinsically
disordered protein analysis( B) of PwWDS1
|
2)多序列比对及系统树分析运用NCBI上的Blast工具进行同源检索,发现在北美云杉(Piceasitchensis)、火炬松(Pinus taeda)、玉米(Zea mays)、毛果杨(Populus trichocarpa)等物种中的同源基因(表 1)。利用ClustalX工具进行PwWDS1与其同源的蛋白氨基酸序列比对,结果发现蛋白的N端(1—20 aa)具有较多的组氨酸(His),不同物种间蛋白的C端具有非常高的相似性,ABA/WDS结构域主要位于蛋白的C端,且含有大量的组氨酸、赖氨酸(Lys)、丙氨酸(Ala)、谷氨酸(Glu)等(图 3)。
|
|
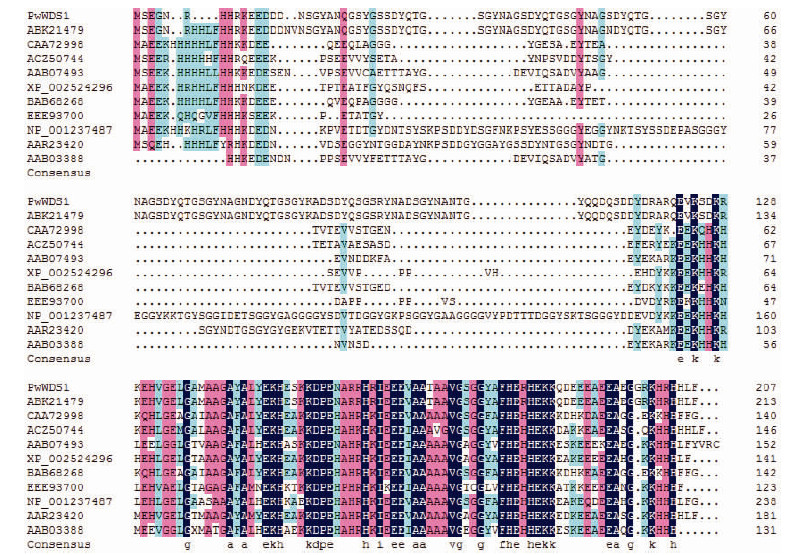 |
图 3 PwWDS1与同源蛋白的氨基酸多序列比对分析
Fig. 3 Multiple sequences alignment of PwWDS1 and homological proteins
相似性: 黑色= 100% ; 粉色≥75% ; 蓝色≥50% Conserved percent: Black = 100% ; Pink≥75% ; Blue≥50% . |
利用MEGA软件进行系统发育树分析,发现PwWDS1与北美云杉的ABK21479、银杏的AAR23420聚为一簇(图 4)。
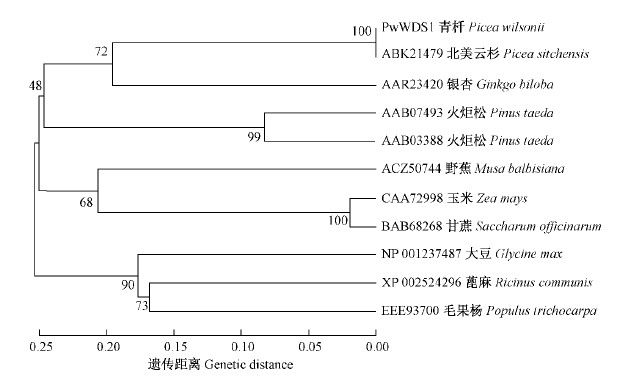 |
图 4 PwWDS1的系统树分析
Fig. 4 Phylogenetic tree analysis of PwWDS1
|
3)二级结构预测运用GOR4工具进行二级结构预测,发现蛋白有36.71%的α-螺旋结构(α-helix),主要位于C端;含有60.39%的无规则卷曲(r and om coil),主要位于蛋白的N端;还有2.90%的延伸链(extended str and )结构(图略)。
2.3 组织表达分析利用RT-qPCR与半定量RTPCR检测PwWDS1在青杄各组织的表达及在种子萌发过程中的表达情况,结果表明PwWDS1主要在青杄的针叶与茎中表达(图 5A);在种子萌发过程中,随着子叶的生长,PwWDS1的表达逐步增加,且在种子萌发10天与12天的表达量较高(图 5B)。半定量RT-PCR与RT-qPCR的结果一致。
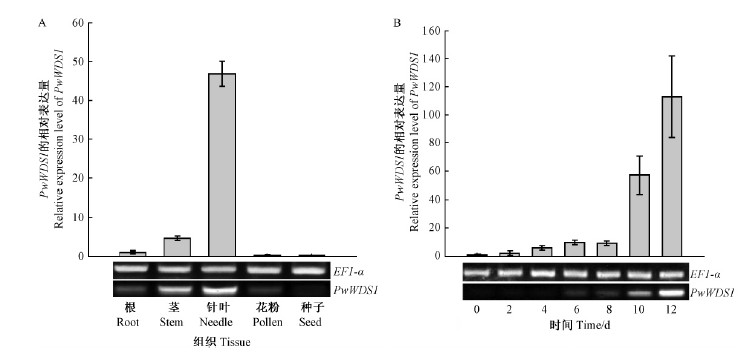 |
图 5 PwWDS1的表达模式
Fig. 5 Expression pattern of PwWDS1
A: PwWDS1的组织特异性表达; B: PwWDS1在种子萌发过程中的表达。所有试验重复3 次。 A: Tissue specific expression of PwWDS1; B: Expression level of PwWDS1 during the seed germination. Values are means ± SD from three independent assays. |
由于PwWDS1具有ABA/WDS结构域,为了验证其参与逆境的响应,对8周生幼苗进行干旱、盐胁迫、ABA与乙烯利处理,提取整株幼苗的RNA后,进行反转录、RTPCR试验。结果表明在上述处理下PwWDS1的表达量均上调。干旱或ABA处理6 h后PwWDS1的表达量急剧升高(图 6A,C);盐胁迫处理下PwWDS1在12 h的表达量最高(图 6B);乙烯利处理后的幼苗在12 h时PwWDS1的表达量较多(图 6D)。RT-qPCR与半定量RT-PCR结果一致(图 6)。
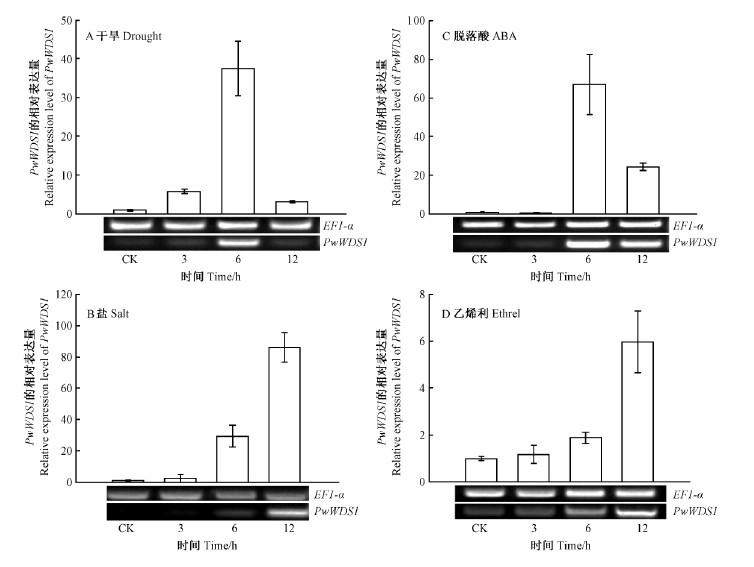 |
图 6 PwWDS1在逆境与激素处理下的表达模式
Fig. 6 Expression patterns of PwWDS1 under different stress treatments
所有试验重复3 次。All values are means ± SD from three independent experiments. |
本研究成功分离了青杄干旱诱导蛋白PwWDS1基因(GenBank登录号: KJ526353),基于其蛋白氨基酸组成进行了生物信息学分析。运用半定量RTPCR与RT-qPCR技术检测了PwWDS1在青杄不同组织、种子萌发不同时期以及在ABA、干旱与盐胁迫下的表达情况。结果显示PwWDS1是一个由207aa组成的蛋白,含有典型的ABA/WDS结构域,且N端具有亲水性较强的氨基酸组成,这与LEA蛋白的特征相似,其蛋白组成也具有较多的亲水性氨基酸,从而在缺水胁迫下有效地保持植物细胞内的水分(Hundertmark et al.,2008;George et al.,2009;Zhang et al.,2012),表明其可能是一个干旱诱导蛋白,参与了植物响应干旱逆境胁迫的反应过程。通过blast检索发现其氨基酸序列与北美云杉的ABK21479高度同源,二者相似性达到96.62%。进化树分析也表明二者聚为一簇,由于青杄与北美云杉均为松科云杉属植物,推测2树种在进化上有共同的祖先。
叶是植物进行光合作用与呼吸作用的主要场所,也是蒸腾作用的主要器官,植物体的水分主要通过叶片的气孔输送到体外。对拟南芥的研究表明,WRKY57,ATHB6等抗逆基因主要在叶中表达,通过调节ABA响应元件、关闭气孔等发挥抵御干旱的作用(Jiang et al.,2012;Soderman et al.,1999),而相关的研究在木本植物中则报道较少。在本研究中,PwWDS1基因主要表达于针叶或者正在生长的子叶中(图 5),这暗示着PwWDS1可能参与叶片中水分的保持或者气孔的调控。已有研究表明ABA参与了部分干旱信号的调控(Kizis et al.,2002;Yoshida et al.,2010)。在本研究的干旱处理与ABA处理试验中,PwWDS1在6 h表达量均上调,这表明PwWDS1可能参与ABA调控的干旱信号通路。另外,盐胁迫下PwWDS1的表达量在12 h上调,在乙烯利的处理下也是在12 h具有较高的表达量。在拟南芥、番茄(Lycopersicum esculentum)中的研究表明,乙烯信号通路参与调控植物对盐胁迫的响应(Cao et al.,2008;Poor et al.,2013)。这暗示PwWDS1基因可能参与了青杄乙烯调控的盐信号通路过程。
已有研究证明植物体不同逆境信号存在交互作用,例如在拟南芥中发现的HARDY基因、水稻中的OsHsfA7、蒺藜苜蓿(Medicago truncatula)中的MtCaMP1等具有同时提高植物盐胁迫与干旱胁迫耐受性的作用(Karaba et al.,2007;Liu et al.,2013;Wang et al.,2013),而ABA与乙烯信号通路的交互作用在植物的种子萌发、成花、果实的成熟、逆境响应等过程中起到重要作用(Arc et al.,2013;Wilmowicz et al.,2008;Zegzouti et al.,1997;Zhang et al.,2009)。本研究中PwWDS1基因能同时被干旱、盐胁迫、ABA与乙烯利诱导,表明其同时参与干旱与盐胁迫的信号通路,且受到ABA与乙烯的调控,但该基因被不同信号途径诱导时间存在差异。今后将在模式植物及烟草中对该基因开展研究,进一步验证其抗逆功能,并对其参与青杄木本植物的抗逆机制进行研究。本研究为林木逆境信号通路的研究、不同植物激素间交联的研究提供了新的思路。
| [1] |
李长江, 曹一博, 张凌云. 2012. 青杄PSAK的克隆及生物信息学分析. 生物技术, 22(3): 4-9.( 1) 1)
|
| [2] |
李长江, 孙帆, 张通, 等. 2013. 青杄PwPSAF基因的克隆与组织表达分析. 林业科学, 49(10): 40-47.( 1) 1)
|
| [3] |
张盾, 刘亚静, 李长江, 等. 2012. 青杄均一化cDNA文库构建及EST序列分析. 生物技术通报, (6): 71-76.( 1) 1)
|
| [4] |
Arc E, Sechet J, Corbineau F, et al. 2013. ABA crosstalk with ethylene and nitric oxide in seed dormancy and germination. Frontiers in Plant Science, 4: 63.( 1) 1)
|
| [5] |
Cao Y R, Chen S Y, Zhang J S. 2008. Ethylene signaling regulates salt stress response: An overview. Plant Signaling & Behavior, 3 (10): 761-763.( 1) 1)
|
| [6] |
Dalal M, Tayal D, Chinnusamy V, et al. 2009. Abiotic stress and ABA-inducible group 4 LEA from Brassica napus plays a key role in salt and drought tolerance. Journal of Biotechnology, 139(2): 137-145.( 1) 1)
|
| [7] |
Desclos M, Dubousset L, Etienne P, et al. 2008. A proteomic profiling approach to reveal a novel role of Brassica napus drought 22 kD/water-soluble chlorophyll-binding protein in young leaves during nitrogen remobilization induced by stressful conditions. Plant Physiology, 147(4): 1830-1844.( 1) 1)
|
| [8] |
Du Q S, Fan X W, Wang C H, et al. 2011. A possible CO2 conducting and concentrating mechanism in plant stomata SLAC1 channel. PloS One, 6(9): e24264.( 1) 1)
|
| [9] |
George S, Usha B, Parida A. 2009. Isolation and characterization of an atypical LEA protein coding cDNA and its promoter from drought-tolerant plant Prosopis juliflora. Applied Biochemistry and Biotechnology, 157(2): 244-253.( 2) 2)
|
| [10] |
Goel D, Singh A K, Yadav V, et al. 2010. Overexpression of osmotin gene confers tolerance to salt and drought stresses in transgenic tomato (Solanum lycopersicum L.). Protoplasma, 245(1-4): 133-141.( 1) 1)
|
| [11] |
Guo L, Yang H, Zhang X, et al. 2013. Lipid transfer protein 3 as a target of MYB96 mediates freezing and drought stress in Arabidopsis. Journal of Experimental Botany, 64(6): 1755-1767.( 1) 1)
|
| [12] |
Ho S L, Huang L F, Lu C A, et al. 2013. Sugar starvation-and GA-inducible calcium-dependent protein kinase 1 feedback regulates GA biosynthesis and activates a 14-3-3 protein to confer drought tolerance in rice seedlings. Plant Molecular Biology, 81(4/5): 347-361.( 1) 1)
|
| [13] |
Hong Y, Zhang W, Wang X. 2010. Phospholipase D and phosphatidic acid signalling in plant response to drought and salinity. Plant, Cell & Environment, 33(4): 627-635.( 1) 1)
|
| [14] |
Hundertmark M, Hincha D K. 2008. LEA (late embryogenesis abundant) proteins and their encoding genes in Arabidopsis thaliana. BMC Genomics, 9: 118.( 2) 2)
|
| [15] |
Jiang Y, Liang G, Yu D. 2012. Activated expression of WRKY57 confers drought tolerance in Arabidopsis. Molecular Plant, 5(6): 1375-1388.( 1) 1)
|
| [16] |
Jin Z, Shen J, Qiao Z, et al. 2011. Hydrogen sulfide improves drought resistance in Arabidopsis thaliana. Biochemical and Biophysical Research Communications, 414(3): 481-486.( 1) 1)
|
| [17] |
Karaba A, Dixit S, Greco R, et al. 2007. Improvement of water use efficiency in rice by expression of HARDY, an Arabidopsis drought and salt tolerance gene. Proceedings of the National Academy of Sciences of the United States of America, 104(39): 15270-15275.( 1) 1)
|
| [18] |
Kizis D, Pages M. 2002. Maize DRE-binding proteins DBF1 and DBF2 are involved in rab17 regulation through the drought-responsive element in an ABA-dependent pathway. The Plant Journal, 30(6): 679-689.( 3) 3)
|
| [19] |
Liu A L, Zou J, Liu C F, et al. 2013.Over-expression of OsHsfA 7 enhanced salt and drought tolerance in transgenic rice. BMB Reports, 46(1): 31-36.( 1) 1)
|
| [20] |
Lorenz W W, Alba R, Yu Y S, et al. 2011. Microarray analysis and scale-free gene networks identify candidate regulators in drought-stressed roots of loblolly pine (P. taeda L.). BMC Genomics, 12: 264.( 2) 2)
|
| [21] |
Lu S, Bahn S C, Qu G, et al. 2013. Increased expression of phospholipase Dalpha1 in guard cells decreases water loss with improved seed production under drought in Brassica napus. Plant Biotechnology Journal, 11(3): 380-389.( 1) 1)
|
| [22] |
Osakabe Y, Arinaga N, Umezawa T, et al. 2013. Osmotic stress responses and plant growth controlled by potassium transporters in Arabidopsis. The Plant Cell, 25(2): 609-624.( 1) 1)
|
| [23] |
Padmanabhan V, Dias D M, Newton R J. 1997. Expression analysis of a gene family in loblolly pine (Pinus taeda L.) induced by water deficit stress. Plant Molecular Biology, 35(6): 801-807.( 2) 2)
|
| [24] |
Poor P, Kovacs J, Szopko D, et al. 2013. Ethylene signaling in salt stress-and salicylic acid-induced programmed cell death in tomato suspension cells. Protoplasma, 250(1): 273-284.( 1) 1)
|
| [25] |
Soderman E, Hjellstrom M, Fahleson J, et al. 1999. The HD-Zip gene ATHB 6 in Arabidopsis is expressed in developing leaves, roots and carpels and up-regulated by water deficit conditions. Plant Molecular Biology, 40(6): 1073-1083.( 1) 1)
|
| [26] |
Sun L, Wang Y P, Chen P, et al. 2011. Transcriptional regulation of SlPYL, SlPP 2 C, and SlSnRK 2 gene families encoding ABA signal core components during tomato fruit development and drought stress. Journal of Experimental Botany, 62(15): 5659-5669.( 1) 1)
|
| [27] |
Urao T, Katagiri T, Mizoguchi T, et al. 1994. Two genes that encode Ca2+-dependent protein kinases are induced by drought and high-salt stresses in Arabidopsis thaliana. Molecular & General Genetics: MGG, 244(4): 331-340.( 1) 1)
|
| [28] |
Wang T Z, Zhang J L, Tian Q Y, et al. 2013. A Medicago truncatula EF-Hand family gene, MtCaMP 1 , is involved in drought and salt stress tolerance. PloS One, 8(4): e58952.( 1) 1)
|
| [29] |
Wilmowicz E, Kesy J, Kopcewicz J. 2008. Ethylene and ABA interactions in the regulation of flower induction in Pharbitis nil. Journal of Plant Physiology, 165(18): 1917-1928.( 1) 1)
|
| [30] |
Xiao B, Huang Y, Tang N, et al. 2007. Over-expression of a LEA gene in rice improves drought resistance under the field conditions. Theor Appl Genet, 115(1): 35-46.( 1) 1)
|
| [31] |
Yoshida T, Fujita Y, Sayama H, et al. 2010. AREB1, AREB2, and ABF3 are master transcription factors that cooperatively regulate ABRE-dependent ABA signaling involved in drought stress tolerance and require ABA for full activation. The Plant Journal, 61(4): 672-685.( 2) 2)
|
| [32] |
Yamaguchi-Shinozaki K, Shinozaki K. 1994. A novel cis-acting element in an Arabidopsis gene is involved in responsiveness to drought, low-temperature, or high-salt stress. The Plant Cell, 6(2): 251-264.( 1) 1)
|
| [33] |
Yu Y, Li Y, Huang G, et al. 2011. PwHAP5, a CCAAT-binding transcription factor, interacts with PwFKBP12 and plays a role in pollen tube growth orientation in Picea wilsonii. Journal of Experimental Botany, 62(14): 4805-4817.( 1) 1)
|
| [34] |
Yu Y, Zhang H, Li W, et al. 2012. Genome-wide analysis and environmental response profiling of the FK506-binding protein gene family in maize (Zea mays L.). Gene, 498(2): 212-222.( 1) 1)
|
| [35] |
Zegzouti H, Jones B, Marty C, et al. 1997. ER 5 , a tomato cDNA encoding an ethylene-responsive LEA-like protein: characterization and expression in response to drought, ABA and wounding. Plant Molecular Biology, 35(6): 847-854.( 1) 1)
|
| [36] |
Zhang M, Yuan B, Leng P. 2009. The role of ABA in triggering ethylene biosynthesis and ripening of tomato fruit. Journal of Experimental Botany, 60(6): 1579-1588.( 1) 1)
|
| [37] |
Zhang Y, Li Y, Lai J, et al. 2012. Ectopic expression of a LEA protein gene TsLEA 1 from Thellungiella salsuginea confers salt-tolerance in yeast and Arabidopsis. Molecular Biology Reports, 39(4): 4627-4633.( 2) 2)
|
| [38] |
Zhao L, Hu Y, Chong K, et al. 2010. ARAG 1 , an ABA-responsive DREB gene, plays a role in seed germination and drought tolerance of rice. Annals of Botany, 105(3): 401-409.( 1) 1)
|
| [39] |
Zhou M L, Ma J T, Zhao Y M, et al. 2012. Improvement of drought and salt tolerance in Arabidopsis and Lotus corniculatus by overexpression of a novel DREB transcription factor from Populus euphratica.Gene, 506(1): 10-17.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

