文章信息
- 郭伟, 卢孟柱
- Guo Wei, Lu Mengzhu
- 毛白杨和烟草次生维管发育相关NAC转录因子的比较
- Comparison of NAC Transcription Factor Involved in Secondary Vascular Development between Populus tomentosa and Tobacco
- 林业科学, 2014, 50(3): 22-30
- Scientia Silvae Sinicae, 2014, 50(3): 22-30.
- DOI: 10.11707/j.1001-7488.20140304
-
文章历史
- 收稿日期:2013-05-09
- 修回日期:2013-05-20
-
作者相关文章
木材是世界上最重要的天然资源之一,具有代替化石资源的潜力(Plomion et al.,2001)。木材是次生维管发育的产物,主要由具有次生细胞壁的纤维和管状细胞组成(Plomion et al.,2001)。木材的形成源于顶端分生组织及维管形成层细胞的连续分裂与分化,是具有高度时空调控性的次生发育过程,包括6个主要的生物学过程: 维管形成层母细胞的分化、细胞分裂、细胞扩张、次生细胞壁的合成、细胞程序化死亡和心材形成(Groover et al.,2006; Groover,2005; Plomion et al.,2001)。因此,研究木材形成的分子机制对于丰富次生维管发育的理论基础,了解木材形成的生物学基础,采用分子育种手段定向培育材性优良的树木新品种具有重要价值。
木本植物的研究难度比较大,所以大多数研究工作是在草本植物中开展的,导致林木木材形成的调控机制研究仍然属于初始阶段。因此,对于木本植物维管形成层的起始、维持及分化的分子决定机制方面仍然需要大量的研究工作(Farrokhi et al.,2006; Somerville,2006)。随着基因组学和功能基因组学的飞速发展,研究人员在拟南芥(Arabidopsis thaliana)(Ehlting et al.,2005; Oh et al.,2003; Zhao,2005)、百日草(Zinnia elegans)(Demura et al.,2007)、毛白杨(Populus tomentosa)(Wang et al.,2009)、毛果杨(Populus trichocarpa)(Dharmawardhana et al.,2010)、杂交山杨(Populus tremula×Populus tremuloides)(Hertzberg et al.,2001)、刺槐(Robinia pseudoacacia)(Yang et al.,2004)、海岸松(Pinus pinaster)(Paiva et al.,2008)、冈尼桉(Eucalyptus gunnii)(Foucart et al.,2006)和白云杉(Picea glauca)(Pavy et al.,2008)中进行了维管发育的相关研究。杨树被证实是一种进行维管发育研究的木本模式植物,其茎的生长具有典型的次生维管发育特征(Robischon et al.,2011)。研究人员利用cDNA基因芯片和蛋白表达谱等技术从杨树维管发育过程中鉴定了大量的功能基因,迫切地需要对它们进行功能分析(Dharmawardhana et al.,2010; Du et al.,2006; Wang et al.,2009)。其中,NAC转录因子是一类重要的参与木质部分化的调控因子。
NAC(NAM,ATAF1/2和CUC2)结构域蛋白家族是最大的植物特异的转录因子家族(Fang et al.,2008; Ooka et al.,2003)。NAC蛋白通常在N末端具有1个保守的NAC结构域,该结构域由160个被划分为5个亚结构域的氨基酸残基组成(Kikuchi et al.,2000; Ooka et al.,2003)。NAC结构域中的1个60个氨基酸残基的序列区域包含1个特异的TF折叠,由外部的螺旋元件和内部的扭曲折叠构成(Duval et al.,2002)。NAC结构域定位在细胞核,能够结合DNA,并且与其他NAC蛋白形成同源二聚体或异源二聚体(Duval et al.,2002; Jensen et al.,2010)。而NAC蛋白的C末端具有高度的特异性,导致具有不同的转录调节活性(Zhong et al.,2010)。研究发现NAC结构域蛋白在木质素形成、纤维素的发育和维管植物木材形成中起到重要的作用(Mitsuda et al.,2008; Zhao et al.,2008; Zhong et al.,2010; 2008)。尽管对NAC转录因子基因的功能分析取得了很多进展,但NAC基因家族庞大,大部分成员功能仍然未知,特别是在杨树中进行功能分析的NAC家族基因非常有限。缺乏高效的基因功能鉴定手段是制约因素之一。
相对较大的体积和较慢的生长速度使得杨树在研究基因的详细功能时存在困难,需要找到更容易进行研究的模式植物。拟南芥虽然体积比较小、生长速度比较快、遗传转化比较容易,但是却不具备典型的次生生长特征,同时缺少某些木材形成的基因和相关的生物学过程(Nieminen et al.,2004)。烟草(Nicotiana tabacum)作为一种具有典型的次生维管发育的一年生草本植物(Cook et al.,2012; Damiani et al.,2005; Guillaumie et al.,2010),具有较高的遗传转化效率、较快的生长速度、较短的生命周期(Al-Ahmad et al.,2004),并且烟草的病毒诱导基因沉默(virus induced gene silencing,VIGS)系统为研究者进行大量的基因功能研究提供了平台(Gossele et al.,2002; Huang et al.,2011; Tao et al.,2004; Zhu et al.,2010)。这一平台实际为大规模筛选、鉴定相关基因提供了有效手段。
本文将烟草维管组织转录组数据中的NAC基因与毛果杨基因组数据库进行比对,得到了43对直系同源基因,通过基因表达谱测序(RNA-seq)和相对荧光定量PCR对烟草和毛白杨NAC同源基因的表达异同进行了分析,旨在进一步通过烟草VIGS系统进行杨树次生维管发育相关基因的功能研究,揭示木材形成过程中NAC转录因子的调控机制。
1 材料与方法 1.1 植物材料烟草(Nicotiana tabacum cv. SR1)和毛白杨为中国林业科学研究院林业研究所生物技术室保存并繁殖。植株种植在直径为15 cm的塑料花盆(内含50%的营养土+50%的蛭石)中,置于温室中培养,每隔5 天用自来水浇1次。
分别选取生根期(rooting stage,4片展开叶)、抽薹期(stem bolting stage,6片展开叶)、团棵期(rosette stage,8片展开叶)、旺长期(vigorously growing stage,14片展开叶)和现蕾期(flower budding stage,20片展开叶)的烟草的第1-4,1-6,1-8,1-8,1-8节间进行取材,作为烟草转录组高通量测序的试验材料。
分别对旺长期(vigorously growing stage,14片展开叶)的烟草和扦插4个月的毛白杨的顶端分生组织、第1节间、第2节间、第3节间、第4节间、第5节间、第6节间、从顶端起第5片展开叶、根进行取材,分别标记为TS,TIN1,TIN2,TIN3,TIN4,TIN5,TIN6,TL,TR和PS,PIN1,PIN2,PIN3,PIN4,PIN5,PIN6,PL,PR,作为基因表达谱高通量测序和相对荧光定量PCR的试验材料。
取材时间是16:00—17:00。每个样本均为取自5个植株的混合样本,液氮速冻后放置在-80 ℃的超低温冰箱里储存。
1.2 试验方法 1.2.1 烟草转录组的高通量测序选取生根期、抽薹期、团棵期、旺长期和现蕾期的烟草的节间,用Qiagen的RNA提取试剂盒(RNeasy Plant Mini kit)提取RNA,用Qiagen的RNase-free DNaseⅠ去除DNA,通过Agilent 2100 Bioanalyzer对RNA样本进行检测。cDNA文库构建、高通量测序(Illumina HiSeqTM 2000)和数据处理由华大基因公司完成,通过SOAPdenovo软件对short reads进行组装。
1.2.2 烟草与毛白杨之间的NAC直系同源基因的鉴定通过基因注释与NAC保守基序进行本地BLASTP搜索相结合的方法在烟草维管发育的转录组数据库中对NAC基因进行搜索,然后通过BLASTX程序(E-value≤1e-10)比对烟草基因组数据库(http://solgenomics.net/,v0.4.4)得到全长蛋白序列,然后通过BLASTP程序(E-value≤1e-10)比对毛果杨基因组数据库(http://www.phytozome.net/,版本v3.0)。
1.2.3 基因表达谱(RNA-seq)高通量测序选取烟草的3个样本(TIN1,TIN2,TIN3)和毛白杨的3个样本(PIN1,PIN2,PIN3),RNA提取、RNA检测、cDNA文库构建、高通量测序方法同1.2.1。基因表达水平(RPKM)的归一化计算通过ERANGE软件(http://woldlab.caltech.edu/gitweb/,版本4.0)完成(Mortazavi et al.,2008)。
1.2.4 相对荧光定量PCR表达分析提取烟草和毛白杨不同组织样本(TS,TIN1,TIN2,TIN3,TIN4,TIN5,TIN6,TL,TR和PS,PIN1,PIN2,PIN3,PIN4,PIN5,PIN6,PL,PR)的RNA,并且进行DNA去除(同上),取800 ng RNA为模板,用Invitrogen的反转录试剂盒(Superscript Ⅲ First-Str and Synthesis System)进行cDNA的合成,并且稀释20倍作为荧光定量 PCR反应的模板。
反应在ABI 7500实时定量PCR仪上进行,方法参照《美国应用生物系统公司7300/7500实时定量PCR仪相对定量实验入门指南》和荧光定量试剂盒SYBR® Premix Ex TaqTM(TaKaRa)说明书。反应体系为: 2 μL cDNA,10 μL 2  Premix Ex TaqⅡ(Tli RnaseH Plus),0.4 μL 50
Premix Ex TaqⅡ(Tli RnaseH Plus),0.4 μL 50  Reference Dye Ⅱ,上下游特异引物(表 1)各0.8 μL,双蒸水6 μL。采用两步法标准反应程序: 1)95 ℃ 30 s; 2)95 ℃ 5 s,60 ℃ 34 s,40个循环。每个试验设4次重复,选取NtEF-1α(Accession number: AF120093)and PtUBQ(Accession number: BU879229)分别作为烟草和毛白杨的内参基因。利用Sequence Detection Software软件(2-ΔΔCt法)(Livak et al.,2001)和SigmaPlot 12.0 软件进行数据分析。
Reference Dye Ⅱ,上下游特异引物(表 1)各0.8 μL,双蒸水6 μL。采用两步法标准反应程序: 1)95 ℃ 30 s; 2)95 ℃ 5 s,60 ℃ 34 s,40个循环。每个试验设4次重复,选取NtEF-1α(Accession number: AF120093)and PtUBQ(Accession number: BU879229)分别作为烟草和毛白杨的内参基因。利用Sequence Detection Software软件(2-ΔΔCt法)(Livak et al.,2001)和SigmaPlot 12.0 软件进行数据分析。
|
|
烟草维管组织转录组测序产生了51 Mb的short reads和4.6 Gb的核苷酸数据,通过 SOAPdenovo组装得到了170 321条unigene(另文发表)。通过基因注释与NAC保守基序进行本地BLASTP搜索相结合的方法在烟草维管组织转录组数据库中鉴定到了43个NAC转录因子家族成员基因。然后将烟草的NAC基因的蛋白序列作为探针在毛果杨基因组数据库中进行BLASTP搜索,选择比对结果中分值(Score)最高并且E-value<e-10的序列作为烟草NAC基因在毛白杨中的直系同源基因(表 2),并且根据所对应的拟南芥的基因名称对烟草和毛白杨基因进行命名。
|
|
运用Clustal X 1.83软件对烟草和毛白杨的NAC基因的蛋白全长序列进行多重序列比对,用MEGA 5.10软件采用Neighbor-Joining(NJ)方法进行了系统进化树的构建(图 1)。如图所示,86个烟草和毛白杨的NAC基因划分为19个亚组(S1-19),且烟草和毛白杨的直系同源基因在系统进化树中属于同一个亚组。
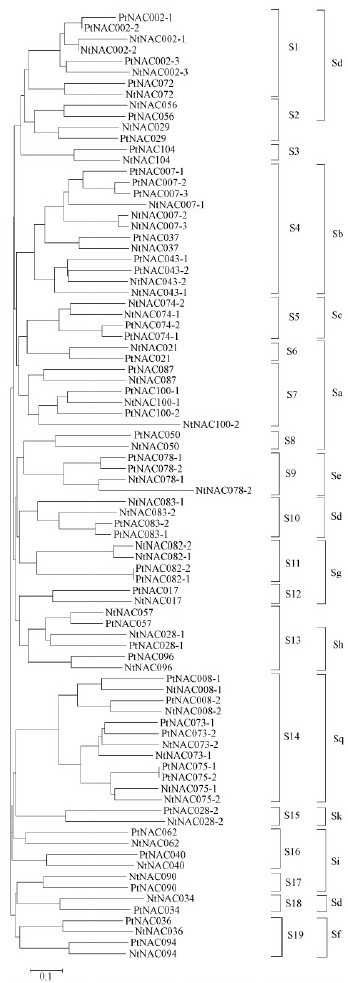 |
图 1 烟草和毛白杨的NAC基因的系统树分析
Fig. 1 Phylogenetic tree of NAC gene in tobacco and
Populus tomentosa
|
Hu等(2010)根据毛果杨基因组v2.0版本,将毛果杨的NAC基因分为18个亚组(Sa-Sr),其中Sb,Sc,Se,Sd,Sq,Sk,Sd和Sf亚组分别对应本研究中毛白杨(比对毛果杨基因组v3.0版本)和烟草的S4,S5,S9,S10,S14,S15,S18和S19亚组。而S2中的NAC029、S3中的NAC104和S13中的NAC057在毛果杨基因组v2.0版本中没有出现,是因为毛果杨基因组v3.0版本具有比v2.0版本更多的序列信息。S6,S7和S8对应毛果杨基因组v2.0版本中的Sa,S11和S12对应Sg,S16和S17对应Si。毛果杨基因组2个版本的不同造成了系统进化树中不同的聚类方式,从而导致亚组分类的不同。
2.3 烟草和毛白杨的NAC同源基因在维管发育过程中的表达分析在早期的研究中,通过组织切片观察,发现旺长期的烟草和扦插4个月的毛白杨在1-6节间具有相似的维管发育过程(数据未提供,另文发表)。分别对旺长期的烟草和扦插4个月的毛白杨的第1-3节间的样本进行了高通量测序,得到了木质化过程中相关基因的表达谱。根据NAC基因的表达值(RPKM)制作热图(heatmap)(图 2)。在43对烟草和毛白杨同源对应的NAC基因中,NAC028-1,NAC029,NAC036,NAC040,NAC043-2,NAC050,NAC056,NAC062,NAC072,NAC073-1,NAC073-2,NAC074-2,NAC100-213对基因在烟草和毛白杨的3个节间中具有相似的表达模式。然而,NAC002-3,NAC007-3,NAC008-1,NAC017,NAC021,NAC028-2,NAC037,NAC043-1,NAC057,NAC075-1,NAC075-2,NAC082-1,NAC082-2,NAC094,NAC100-1和NAC104 16对NAC同源基因中的毛白杨基因在3个节间均没有发生表达,而相应的烟草的同源基因却发生了表达,说明烟草和毛白杨在进化过程中发生了组织特异表达的改变。NAC002-1,NAC002-3,NAC007-1,NAC008-2,NAC017,NAC028-2,NAC037,NAC043-1,NAC074-1,NAC078-1,NAC078-2,NAC082-1,NAC082-2,NAC083-1,NAC083-2和NAC104 16个基因在烟草和毛白杨中的表达模式截然不同,在毛白杨中表达量比较低,而在烟草中表达量却比较高,说明这16个基因更多参与了烟草维管组织发育的调控。
 |
图 2 烟草和毛白杨NAC直系同源基因在前3个节间的表达分析
Fig. 2 Expression analysis of the NAC orthologous genes in the
first three internodes of tobacco and Populus tomentosa
|
选择8对在烟草和毛白杨中都发生表达并且在杨树中尚未见研究报道的NAC基因,在旺长期的烟草和扦插4个月的毛白杨的第1-6节间以及顶端分生组织、叶片、根中通过荧光定量PCR进行表达分析(图 3)。结果表明荧光定量PCR与基因表达谱的结果具有相同的变化趋势,说明建立在高通量测序基础上的基因表达谱测序具有较高的准确性。这8对NAC同源基因在烟草和毛白杨的不同组织中的表达模式也基本一致。
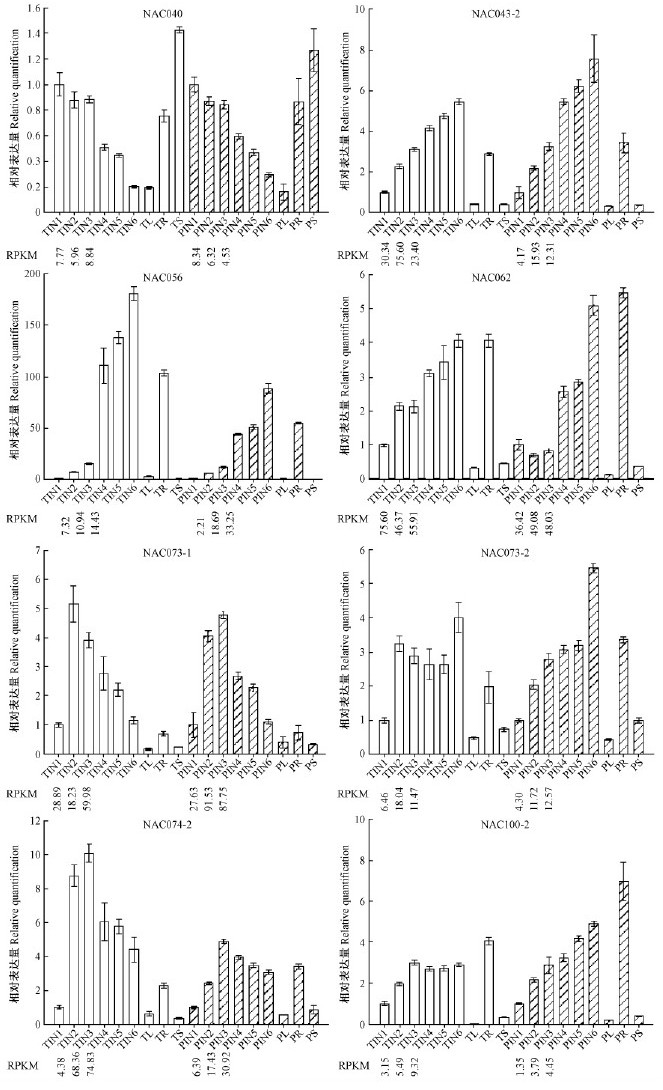 |
图 3 烟草和毛白杨的NAC直系同源基因在不同组织的表达分析
Fig. 3 Expression analysis of the NAC orthologous genes in a range of tissues of tobacco and Populus tomentosa
|
NAC043-2,NAC056,NAC062,NAC073-2和NAC100-2的同源基因在烟草和毛白杨的顶端分生组织(SAM)和叶片中具有最低的表达量,在根中具有比较高的表达量,在远离顶端的节间中的表达量比靠近顶端的节间高,说明其随着茎的木质化程度提高表达水平也相应提高。NAC040同源基因的表达模式与NAC043-2,NAC056,NAC062,NAC073-2和NAC100-2相反,在远离顶端的节间比靠近顶端的节间表达量低,同时在顶端分生组织具有最高的表达量,说明NAC040的表达量变化与茎的木质化程度成反比,可能更多参与了初生维管组织的形成。而NAC073-1和NAC074-2同源基因在烟草和毛白杨的不同节间中的表达量先升高后降低,在第1和第6节间、顶端分生组织、叶片和根中的表达量较低,在维管组织分化的过程(第2-5节间)中具有较高的表达量,推测它们更多参与初生到次生生长的转换。值得注意的是,NAC073-1和NAC073-2虽然具有较高的同源性(图 1),但是却具有不同的表达模式,推测这2个基因在进化过程中组织表达或执行的功能发生了分化。
3 讨论杨树作为一种研究木材形成的模式植物,具有典型的次生维管系统发育过程(Groover et al.,2006; Groover,2005; Plomion et al.,2001)。随着生命科学和生物技术的快速发展,毛果杨基因组测序的完成,获得了大量有关木材形成过程的相关基因。但是数据库中的注释信息(基因结构、推定的功能等)是通过与拟南芥基因组进行比对,根据拟南芥的同源基因、同源基因家族和蛋白结构域的信息进行推测。而拟南芥本身不具备典型的次生生长特征,缺少某些木材形成基因和相关的生物学过程(Nieminen et al.,2004)。很显然,这些基因的功能需要进一步鉴定。
本文将烟草维管组织转录组数据库中的NAC转录因子基因与毛果杨基因组数据库进行比对,得到43对NAC同源基因(表 2)。通过对烟草和毛白杨的第1-3节间的样本进行基因表达谱高通量测序,得到每个同源基因在不同节间的表达量,找到了13对表达模式相似的同源基因。选择8对在烟草和毛白杨中都发生表达并且在毛白杨中没有研究的NAC基因,在烟草和毛白杨的第1-6节间、根、叶和顶端分生组织中通过相对荧光定量PCR进行表达分析,结果与表达谱一致,并且这8对NAC同源基因在烟草和毛白杨中具有相似的表达模式,说明通过研究烟草的NAC转录因子来揭示杨树的维管发育相关基因的作用具有可行性。
NAC043-2,NAC056,NAC062,NAC073-2和NAC100-2的同源基因在烟草和毛白杨的远离顶端的节间中的表达量比靠近顶端的节间高,说明随着茎的木质化程度的提高而提高。其中,NST1/ANAC043(NAC Secondary Wall Thickening Promoting Factor 1),NST2/ANAC066和NST3/SND1(Secondary Wall-associated NAC Domain Protein)/ANAC012在控制次生细胞壁合成中起作用,并且功能部分冗余(Mitsuda et al.,2007; 2008; 2005; Zhong et al.,2007),异位表达后能够导致转基因植株不同组织的次生细胞壁加厚(Mitsuda et al.,2005; Zhong et al.,2007)。NST1/ANAC043和NST3/SND1/ANAC012在调节拟南芥花序茎的束间纤维和胚轴的次生木质部的次生细胞壁加厚中功能冗余(Mitsuda et al.,2007; 2008)。SND2/ANAC073和SND3/ANAC010在调节纤维次生细胞壁的形成中起作用(Zhong et al.,2008),SND2经被证实是 NST3/SND1的下游靶基因,而SND3也被推测是NST3/SND1的下游靶基因(Zhong et al.,2008)。而目前却没有关于NAC056,NAC062和NAC100的功能研究的相关报道,但是根据表达模式推测它们在维管发育过程中能够行使功能,需要进一步加以阐明。NAC073-1和NAC074-2同源基因在烟草和毛白杨的维管组织分化(第2-5节间)中具有较高的表达量,其中SND2/ANAC073在调节纤维次生细胞壁形成中起作用(Zhong et al.,2008),ANAC074在拟南芥的木质部中表达上调,可以推测NAC073-1和NAC074-2都能够在维管分化中行使功能。根据表达模式可以推测这8对NAC基因都能够在维管发育中起到重要作用,因此有必要利用烟草的研究系统对这8对基因进行深入的功能鉴定。
采用烟草病毒诱导的基因沉默(virus induced gene silencing,VIGS)系统来鉴定杨树次生维管系统发育相关的基因是本研究的目的之一。该技术是一种能够快速通过RNAi来介导基因沉默的技术,不需要对植物进行遗传转化,能够对植物基因功能进行快速分析(Gossele et al.,2002; Huang et al.,2011; Tao et al.,2004; Zhu et al.,2010)。烟草的VIGS系统是一个快速、可靠和强大的系统,并且在研究植物细胞壁形成相关基因的功能中得到了成功的应用(Zhu et al.,2010)。纤维素、木聚糖和木质素合成相关基因在通过VIGS介导的烟草植株中与在其他不同的物种中不同的基因沉默系统具有相似的表型(Zhu et al.,2010)。因此,上述获得的烟草和毛白杨NAC同源基因可以通过烟草相应的基因,采用烟草VIGS技术,研究它们在次生维管系统发育中的作用。
| [1] |
Al-Ahmad H, Galili S, Gressel J. 2004. Tandem constructs to mitigate transgene persistence: tobacco as a model. Molecular Ecology, 13(3):697-710.( 1) 1)
|
| [2] |
Cook C M, Daudi A, Millar D J, et al. 2012. Transcriptional changes related to secondary wall formation in xylem of transgenic lines of tobacco altered for lignin or xylan content which show improved saccharification. Phytochemistry, 74:79-89.( 1) 1)
|
| [3] |
Damiani I, Morreel K, Danoun S, et al. 2005. Metabolite profiling reveals a role for atypical cinnamyl alcohol dehydrogenase CAD1 in the synthesis of coniferyl alcohol in tobacco xylem. Plant Molecular Biology, 59(5):753-769.( 1) 1)
|
| [4] |
Demura T, Fukuda H. 2007. Transcriptional regulation in wood formation. Trends Plant Sci, 12(2):64-70.( 1) 1)
|
| [5] |
Dharmawardhana P, Brunner A M, Strauss S H. 2010. Genome-wide transcriptome analysis of the transition from primary to secondary stem development in Populus trichocarpa. BMC Genomics, 11:150.( 2) 2)
|
| [6] |
Du J, Xie H L, Zhang D Q, et al. 2006. Regeneration of the secondary vascular system in poplar as a novel system to investigate gene expression by a proteomic approach. Proteomics, 6(3):881-895.( 1) 1)
|
| [7] |
Duval M, Hsieh T F, Kim S Y, et al. 2002. Molecular characterization of AtNAM: a member of the Arabidopsis NAC domain superfamily. Plant Molecular Biology, 50(2):237-248.( 1) 1)
|
| [8] |
Ehlting J, Mattheus N, Aeschliman D S, et al. 2005. Global transcript profiling of primary stems from Arabidopsis thaliana identifies candidate genes for missing links in lignin biosynthesis and transcriptional regulators of fiber differentiation. The Plant Journal, 42(5):618-640.( 1) 1)
|
| [9] |
Fang Y, You J, Xie K, et al. 2008. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice. Mol Genet Genomics, 280(6):547-563.( 1) 1)
|
| [10] |
Farrokhi N, Burton R A, Brownfield L, et al. 2006. Plant cell wall biosynthesis: genetic, biochemical and functional genomics approaches to the identification of key genes. Plant Biotechnology Journal, 4(2):145-167.( 1) 1)
|
| [11] |
Foucart C, Paux E, Ladouce N, et al. 2006. Transcript profiling of a xylem vs phloem cDNA subtractive library identifies new genes expressed during xylogenesis in Eucalyptus. The New Phytologist, 170(4):739-752.( 1) 1)
|
| [12] |
Gossele V, Fache I, Meulewaeter F, et al. 2002. SVISS—a novel transient gene silencing system for gene function discovery and validation in tobacco plants. The Plant Journal, 32(5):859-866.( 2) 2)
|
| [13] |
Groover A, Robischon M. 2006. Developmental mechanisms regulating secondary growth in woody plants. Current Opinion in Plant Biology, 9(1):55-58.( 2) 2)
|
| [14] |
Groover A T. 2005. What genes make a tree a tree? Trends Plant Sci, 10(5):210-214.( 2) 2)
|
| [15] |
Guillaumie S, Mzid R, Mechin V, et al. 2010. The grapevine transcription factor WRKY2 influences the lignin pathway and xylem development in tobacco. Plant Molecular Biology, 72(1/2):215-234.( 1) 1)
|
| [16] |
Hertzberg M, Aspeborg H, Schrader J, et al. 2001. A transcriptional roadmap to wood formation. Proceedings of the National Academy of Sciences of the United States of America, 98(25):14732-14737.( 1) 1)
|
| [17] |
Hu R, Qi G, Kong Y, et al. 2010. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa. BMC Plant Biol, 10:145.( 1) 1)
|
| [18] |
Huang C J, Zhang T, Li F F, et al. 2011. Development and application of an efficient virus-induced gene silencing system in Nicotiana tabacum using geminivirus alphasatellite. Journal of Zhejiang University Science B, 12(2):83-92.( 2) 2)
|
| [19] |
Jensen M K, Kjaersgaard T, Nielsen M M, et al. 2010. The Arabidopsis thaliana NAC transcription factor family: structure-function relationships and determinants of ANAC019 stress signalling. The Biochemical Journal, 426(2):183-196.( 1) 1)
|
| [20] |
Kikuchi K, Ueguchi-Tanaka M, Yoshida K T, et al. 2000. Molecular analysis of the NAC gene family in rice. Molecular & General Genetics, 262(6):1047-1051.( 1) 1)
|
| [21] |
Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔ Ct Method. Methods, 25(4):402-408.( 1) 1)
|
| [22] |
Mitsuda N, Iwase A, Yamamoto H, et al. 2007. NAC transcription factors, NST1 and NST3, are key regulators of the formation of secondary walls in woody tissues of Arabidopsis. The Plant Cell, 19(1):270-280.( 2) 2)
|
| [23] |
Mitsuda N, Ohme-Takagi M. 2008. NAC transcription factors NST1 and NST3 regulate pod shattering in a partially redundant manner by promoting secondary wall formation after the establishment of tissue identity. The Plant Journal, 56(5):768-778.( 1) 1)
|
| [24] |
Mitsuda N, Seki M, Shinozaki K, et al. 2005. The NAC transcription factors NST1 and NST2 of Arabidopsis regulate secondary wall thickenings and are required for anther dehiscence. The Plant Cell, 17(11):2993-3006.( 1) 1)
|
| [25] |
Mortazavi A, Williams B A, McCue K, et al. 2008. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 5(7):621-628.( 1) 1)
|
| [26] |
Nieminen K M, Kauppinen L, Helariutta Y. 2004. A weed for wood? Arabidopsis as a genetic model for xylem development. Plant Physiol, 135(2):653-659.( 2) 2)
|
| [27] |
Oh S, Park S, Han K H. 2003. Transcriptional regulation of secondary growth in Arabidopsis thaliana. Journal of Experimental Botany, 54(393):2709-2722.( 1) 1)
|
| [28] |
Ooka H, Satoh K, Doi K, et al. 2003. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana. DNA Research, 10(6):239-247.( 2) 2)
|
| [29] |
Paiva J A, Garces M, Alves A, et al. 2008. Molecular and phenotypic profiling from the base to the crown in maritime pine wood-forming tissue. The New Phytologist, 178(2):283-301.( 1) 1)
|
| [30] |
Pavy N, Boyle B, Nelson C, et al. 2008. Identification of conserved core xylem gene sets: conifer cDNA microarray development, transcript profiling and computational analyses. The New Phytologist, 180(4):766-786.( 1) 1)
|
| [31] |
Plomion C, Leprovost G, Stokes A. 2001. Wood formation in trees. Plant Physiology, 127(4):1513-1523.( 3) 3)
|
| [32] |
Robischon M, Du J, Miura E, et al. 2011. The Populus class III HD ZIP, popREVOLUTA, influences cambium initiation and patterning of woody stems. Plant Physiol, 155(3):1214-1225.( 1) 1)
|
| [33] |
Somerville C. 2006. Cellulose synthesis in higher plants. Annual Review of Cell and Developmental Biology, 22:53-78.( 1) 1)
|
| [34] |
Tao X, Zhou X. 2004. A modified viral satellite DNA that suppresses gene expression in plants. The Plant Journal, 38(5):850-860.( 2) 2)
|
| [35] |
Wang M, Qi X, Zhao S, et al. 2009. Dynamic changes in transcripts during regeneration of the secondary vascular system in Populus tomentosa Carr. revealed by cDNA microarrays. BMC Genomics, 10:215.( 2) 2)
|
| [36] |
Yang J, Kamdem D P, Keathley D E, et al. 2004. Seasonal changes in gene expression at the sapwood-heartwood transition zone of black locust (Robinia pseudoacacia) revealed by cDNA microarray analysis. Tree Physiology, 24(4):461-474.( 1) 1)
|
| [37] |
Zhao C. 2005. The xylem and phloem transcriptomes from secondary tissues of the Arabidopsis root-hypocotyl. Plant Physiology, 138(2):803-818.( 1) 1)
|
| [38] |
Zhao C, Avci U, Grant E H, et al. 2008. XND1, a member of the NAC domain family in Arabidopsis thaliana, negatively regulates lignocellulose synthesis and programmed cell death in xylem. The Plant Journal, 53(3):425-436.( 1) 1)
|
| [39] |
Zhong R, Lee C, Ye Z H. 2010. Functional characterization of poplar wood-associated NAC domain transcription factors. Plant Physiol, 152(2):1044-1055.( 2) 2)
|
| [40] |
Zhong R, Lee C, Zhou J, et al. 2008. A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis. The Plant Cell, 20(10):2763-2782.( 3) 3)
|
| [41] |
Zhong R, Richardson E A, Ye Z H. 2007. Two NAC domain transcription factors, SND1 and NST1, function redundantly in regulation of secondary wall synthesis in fibers of Arabidopsis. Planta, 225(6):1603-1611.( 1) 1)
|
| [42] |
Zhu X, Pattathil S, Mazumder K, et al. 2010. Virus-induced gene silencing offers a functional genomics platform for studying plant cell wall formation. Molecular Plant, 3(5):818-833.( 3) 3)
|
 2014, Vol. 50
2014, Vol. 50

