文章信息
- 程晓甜, 阿地力·沙塔尔, 张伟, 李新泉
- Cheng Xiaotian, Adil Sattar, Zhang Wei, Li Xinquan
- 枣实蝇4个地理种群的线粒体Cytb基因序列分析
- Sequence Analysis of Mitochondrial Cytb Gene of Four Geographic Populations of Carpomya vesuviana
- 林业科学, 2014, 50(3): 144-150
- Scientia Silvae Sinicae, 2014, 50(3): 144-150.
- DOI: 10.11707/j.1001-7488.20140321
-
文章历史
- 收稿日期:2013-04-15
- 修回日期:2013-09-21
-
作者相关文章
2. 新疆出入境检验检疫局技术中心 乌鲁木齐 830083
2. Xinjiang Entry-Exit Inspection and Quarantine Bureau Urumqi 830083
新入侵重大检疫性害虫枣实蝇(Carpomya vesuviana )于2007年首次在吐鲁番地区发现,对该地区枣(Zizyphus jujuba)产业造成了毁灭性损失,且疫情有向其他地区扩散的趋势。枣实蝇原产印度,现已扩散至阿富汗、塔吉克斯坦、泰国、意大利等国家(张润志等,2007)。枣实蝇以成虫产卵和幼虫蛀食危害枣果,成虫产卵于果实表面,致使产卵孔周围组织发育停止,形成凹陷或畸形。幼虫孵化后蛀食果肉导致枣果早熟和腐烂,严重影响枣果品质及产量。在印度枣实蝇是枣树最具毁灭性的害虫,已成为影响枣树成功种植的限制因子(Lakra et al.,1983)。国内最早由陈乃中(1998)对枣实蝇形态进行了初步报道,张润志等(2007)以及吴佳教等(2008)对枣实蝇以及咔实蝇属的形态特征作了详尽报道。然而有关枣实蝇分子方面仅有阿地力·沙塔尔等(2012)对枣实蝇不同地理种群的亲缘关系研究。因此对枣实蝇4个地理种群的线粒体Cytb基因序列分析,再次分析枣实蝇不同地理种群的亲缘关系,从而更有力地说明枣实蝇4个不同地理种群的亲缘关系,并为进一步设计特异性引物及研究枣实蝇快速鉴定奠定基础。
动物mtDNA 属母系遗传,为双链闭环结构,基因序列组成相对保守,无重组和单拷贝,并易于检测。昆虫mtDNA 平均进化速率是单拷贝核DNA的1~2 倍,长度为1 514~1 613 kb,包括12~13 个编码蛋白质的基因。由于其结构和进化上的特点,mtDNA 多被作为研究物种进化的重要分子标记(张亚平等,1992; 施伟等,2004; Moritz et al.,1987)。Cytb基因在线粒体基因组中进化速度适中,一个较短的片段就可以将种下水平以及科的水平的系统发育信息包含在内,适合研究种内到种间甚至科间的系统发育关系(Meyer et al.,1990)。在mtDNA各基因中,Cytb基因的结构和功能研究的比较清楚(Moritz et al.,1987),因其碱基序列稳定性较高而常用于动物类群的系统进化和分类鉴定研究(任竹梅等,2003)。在研究瓜实蝇等8种实蝇在COⅠ与Cytb基因上的同源性时发现,总体在COⅠ基因上的同源性高于Cytb基因,COⅠ基因比Cytb基因要保守,Cytb基因的进化突变速率要快于COⅠ基因(胡剑,2008)。
本研究以吐鲁番市、KSS、HSX和HMS 4个地方枣实蝇作为研究对象,对4个不同地理种群的枣实蝇mtDNA Cytb的序列特征及各种群间的遗传分化水平和进化关系进行了分析,探讨了利用Cytb 基因序列开展枣实蝇分子鉴定的可行性,为下一步构建枣实蝇的快速分子鉴定和检测技术积累了基础资料。
1 材料与方法 1.1 标本来源试验材料为用100%酒精浸泡的标本(蛹),并进行形态鉴定。标本的采集地点、个体数量和时间见表 1。
|
|
1)引物参照 Simon(1994)设计的通用引物,并由北京华大中生科技发展有限公司负责合成,引物如表 2所示。
|
|
2)酶及其他试剂 动物组织基因组DNA提取试剂盒、琼脂糖、PCR反应体系试剂(10×buffer、 dNTP和Mg2+)、2 000 bp Ladder Marker Taq、酶、EB均购自北京庄盟国际生物基因科技有限公司。
1.3 主要仪器Biometra定量梯度PCR仪(华粤)、TGL-1613台式高速离心机(上海)、DYY-12C多功能电泳仪(北京市六一仪器厂)、D56-26M型数字图像分析仪(北京市六一仪器厂)、Thermo Nanodrop2000超微量紫外分光光度计(北京科誉兴业科技发展有限公司)、SANYOMLS-3750全自动高压灭菌仪(上海中庸检验设备有限公司)、XMTD-4000水浴锅(北京市永光明医疗仪器厂)、超低温冰箱MDF -382 E(N)SANYO(上海中庸检验设备有限公司)。
1.4 试验方法1)枣实蝇基因组DNA的提取 应用北京庄盟国际生物基因科技有限公司研发的动物组织基因组DNA提取试剂盒的相应方法来提取基因组DNA,按操作技术指南的步骤进行提取(阿地力·沙塔尔等,2012)。
2)目标片段PCR扩增 用引物CB1/CB2对4个地区枣实蝇进行PCR扩增。
PCR反应体系: DNA200 ng,TaqDNA酶2 U,10×PCR buffer5 μL,2.5 mmol·L-1 dNTP 3 μL,25 mmol·L-1 Mg2+ 4 μL,5.0 mmol·L-1正反引物各1.0 μL,加水至总体积50 μL,阴性对照为ddH2O。
PCR扩增条件: 先94 ℃预变性3 min,再94 ℃变性1 min,44 ℃退火1 min,72 ℃,90 s,36 个循环,最后72 ℃,6 min。在数字图像分析仪上取PCR产物5 μL在含溴化乙锭的1.5%琼脂凝胶上电泳30 min(90 V)检测结果(朱振华等,2005)。
3)序列测定及数据分析 (1)测序 将扩增带亮度好、清晰且无非特异扩增的PCR产物进行测序,正反链双向同时测定。测序由北京华大中生科技发展有限公司完成。
(2)枣实蝇Cytb基因序列比较分析 用Chromas软件校对测序之后序列的正反双链,根据测序的荧光图谱对其进行修正。所得Cytb基因片段来源于线粒体的编码区,通过无脊椎动物密码子-反密码子系统对其进行翻译,未发现终止密码子,所以所测序列不是假基因。用Clustal X 多序列对位排列程序对同源序列进行排序,并人工核对校正。分子遗传学分析采用软件MEGA4:不同地理种群枣实蝇的进化分歧矩阵根据Kimura 2-paramter 模型计算得出; 构建分子系统发育树用未加权组对算术平均法(UPGMA)和邻接法(NJ),其中遗传距离用P-distance模型。系统树各分支的置信度(boostrap)均进行1 000次的重复检验(张长禹,2007)。
2 结果与分析 2.1 PCR扩增Cytb基因由图 1可见,有一清晰、明亮的约500 bp的电泳条带,没有出现杂带,与预期的结果相符合,从而说明PCR产物与引物设计的要求相符合。
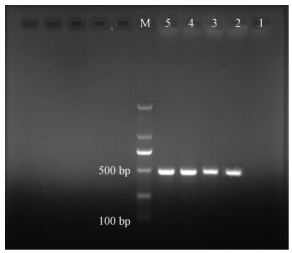 |
图 1 CB1/CB2引物PCR模板DNA质量电泳分析
Fig. 1 Agarose gel electrophoresis analysis(GEA)of the quality of DNA template with primer set CB1/CB2
|
对Cytb基因序列采用双向测序,并且重复测定样品。从序列图分析可看出,在500 bp的范围内的信号稳定、清晰,由此可说明送测的样品效果较好。
2.3 数据处理及分析1)Cytb基因序列的校正 4个地理种群枣实蝇个体的Cytb基因序列用软件Clustal X和DNAMAN加以排序比较,从474 bp中可以得到469 bp比较完整的序列片段。为了验证Cytb基因片断的正确性,将转换成FASTA格式后的序列在NCBI数据库中(http://www.ncbi.nih.gov/BLAST/)进行Blastn同源相似性搜索。分析结果表明,试验所测得的序列为Cytb基因的部分序列(张长禹,2007)。
2)4个地区枣实蝇Cytb基因序列 利用引物CB1/CB2分别测定了KSS、吐鲁番市、HMS及HSX 4个地区枣实蝇的mtDNA Cytb基因约500 bp片段序列,共获得Cytb基因序列4条,片段长度为469 bp,其中吐鲁番市枣实蝇Cytb基因序列已录入GenBank数据库(序列号: JX101439)。
3)Cytb部分序列组成的分析 用Mega4.0软件将比对结果进行格式转换后,然后分析2个引物扩增所得的片段。其扩增获得的片段为mtDNA Cytb编码区的片段,其长度为468 bp。对所得的Cytb部分序列进行编码,可知前一氨基酸的第三位密码子是469 bp序列的第1个碱基,所以将第1个碱基去除再进行核苷酸序列翻译,从而进行分析的有468 个碱基(Simon et al.,1994)。扩增的Cytb部分序列中,A+T含量在第3位点占55.3%,在第1位点为64.8%,而在第2位点含量最高为93.1%,G+C在第2位点含量最低仅占6.9%,而在第3位点为44.7%(表 3)。
|
|
4)Cytb基因碱基组成及特殊位点的分析 对4条已经测序的Cytb基因蛋白编码区468 bp进行排列,其Cytb基因序列碱基组成如表 4所示。由表可知: 4个地方枣实蝇DNACytb片段长度均为468 bp。mtDNA Cytb基因的碱基T,C,A,G的平均含量分别为38.4%,15.3%,32.6%,11.7%,KSS枣实蝇的A+T含量最低为70.8%,吐鲁番市枣实蝇和HSX枣实蝇的A+T含量最高达71.2%。总体来讲,A+T含量较高,G+C的含量偏低,A+T的平均含量为71.0%,G+C的平均含量为29.0%。
|
|
Mega4.0软件的分析结果如表 5所示: 在Cytb基因序列中有41个总变异位点,有428个保守位点,有2个简约信息位点。但在第2位点发生的变异占多数,是总变异位点的62.85%。共编码了156个氨基酸,其中发生变异的有27个,占氨基酸总数的17.31%(孙娜等,2010)。
|
|
5)4个不同地区的枣实蝇Cytb基因碱基转换和颠换频率 如表 6所示,在长度为468 bp的Cytb基因序列中平均有21个位点发生了替换,其中转换数为15个,颠换数为6个,其中C与T之间的转换率(85.7%)大于A与G之间的转换率(14.3%),而T与A之间的颠换率(80%)远大于其他碱基之间的颠换率,T与G之间的颠换率0,A与C之间的颠换率20%。转换和颠换主要发生在第2和第3位点。
|
|
6)Cytb基因序列密码子的使用和氨基酸的组成分析 通过Mega4.0使用无脊椎动物线粒体密码表,统计4个不同地区的枣实蝇的密码子使用频率,从而推断mtDNA Cytb编码区的密码子的使用频率(表 7)以及氨基酸组成(表 8)。从表 7和8可知,密码子UUC,UUG,CAG等使用频率较高,分别是13.5%,9.0%,9.0%。GUU,GUC,GUA,UCC,CCU等未出现。由氨基酸片断的分析数据可知,Arg和Cys使用频率为0,Phe,Leu,Gln等使用频率较高分别是12.06%,22.70%,12.77%,这与孙娜等(2010)的研究结果相符。
|
|
|
|
7)4个地区枣实蝇遗传距离 遗传距离最近的是HMS和HSX枣实蝇,为0.011;遗传距离最远的为KSS枣实蝇与吐鲁番市的枣实蝇,为0.082。通过 BLAST方法检索国外有代表性的咔实蝇属mtDNA Cytb基因序列,其标本名称及其登录号分别为樱桃绕实蝇Rhagoletis cerasi FJ571369.1,蔷薇咔实蝇C. schineri R141 FJ571370.1,蔷薇咔实蝇C. schineri R248 FJ571371.1,与本试验中4个地区的枣实蝇进行比对,结果见表 9(阿地力·沙塔尔等,2012)。
|
|
8)聚类分析 以同属于双翅目的樱桃绕实蝇、蔷薇咔实蝇isolate R141和蔷薇咔实蝇isolate R248为外群,利用邻接法(NJ)和未加权组对算术平均法(UPGMA)构建分子系统聚类图(图 2,3)。由聚类分析可知: 由2种方法得到的聚类图的拓扑结构基本一致,相同种类不同个体的实蝇都各自聚为一支,一般都在90%以上,置信度较高。在系统树中,吐鲁番市、HSX和HMS枣实蝇先聚在一起,然后与分别来自2个不同地方的蔷薇咔实蝇聚为一支,再与KSS枣实蝇相聚。最后,整个咔实蝇属的这2种实蝇再与樱桃绕实蝇聚在一起。对外群分析发现,2种方法构建的聚类图均能够把樱桃绕实蝇与枣实蝇分开,且外群亲缘关系基本符合其阶元划分,从而可知外群选择的正确性。2种聚类图表明KSS枣实蝇与吐鲁番市、HSX、HMS枣实蝇亲缘关系较远。
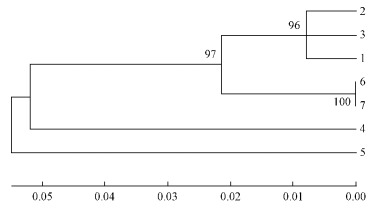 |
图 2 利用NJ法构建mtDNA Cytb基因序列的聚类图
Fig. 2 Dendrogram based on NJ analysis of mtDNA Cytb
|
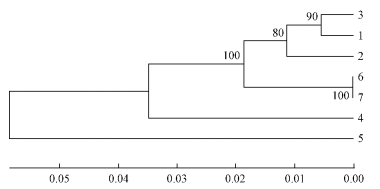 |
图 3 利用UPGMA法构建mtDNA Cytb基因序列的聚类图
Fig. 3 Dendrogram based on UPGMA analysis of mtDNA Cytb
|
本研究测定的4个不同地理种群的枣实蝇mtDNA Cytb部分序列基因的碱基T,C,A,G的平均含量分别为38.4%,15.3%,32.6%,11.7%,吐鲁番市枣实蝇和HSX枣实蝇的A+T含量最高为71.2%,KSS枣实蝇的A+T含量最低为70.8%,A+T的平均含量为71.0%,而G+C的平均含量为29.0%,从而可以看出A+T的含量明显地高于G+C的含量,这与昆虫线粒体DNA的A+T偏倚性结论(Lunt et al.,1996)一致。
在特异性位点上Cytb序列 A+T含量在第3位点占55.3%,而在第2位点最高含量到93.1%,在第1位点为64.8%;G+C在第3位点占44.7%,在第2位点含量最低,仅6.9%。所以,密码子的碱基使用频率存在明显的偏向性,与典型的昆虫线粒体DNA碱基组成一致(Simon et al.,1994)。Cytb基因序列的总变异位点为41个,简约信息位点为2个,428个保守位点,但在第2位点发生变异的为多数,占总变异位点62.85%;共编码156个氨基酸,其中有27个发生变异,占总氨基酸的17.31%。
3.2 对碱基替换的讨论替换现象会发生在DNA序列进化过程中,包括转换和颠换。转换(transition)是指一个嘌呤被另一个嘌呤,或者是一个嘧啶被另一个嘧啶替代;而碱基颠换(transversion)则是在碱基置换中嘌呤与嘧啶之间的替代。
在长度为468 bp的Cytb基因序列中平均有21个位点发生了替换,其中转换数为15个,颠换数为6个,其中C与T之间的转换率(85.7%)大于A与G之间的转换率(14.3%);而T与A之间的颠换率(80%)远大于其他碱基之间的颠换率,T与G之间的颠换率0,A与C之间的颠换率20%。颠换和转换主要发生的部位是第2及第3位点。颠换主要以T与A之间为主,转换主要以T与C之间为主,且颠换率低于转换率,在密码子第3位点发生的颠换占绝大多数,这与密码子第3位点的A+T含量高可能有关。这也与脊椎动物普遍结论相一致,是进化分歧时间较短的表现(IrWin et al.,1991)。关于氨基酸序列变异与高A+T含量是否存在显著的相关性仍存在着争议(刘运强等,2001; Hugall et al.,1997)。
3.3 氨基酸组成和密码子使用频率特征对4个不同地区的枣实蝇的密码子使用频率进行统计,推断出密码子UUC,UUG,CAG等使用频率较高,分别是13.5%,9.0%,9.0%。GUU,GUC,GUA,UCC,CCU等未出现。在氨基酸片断的分析数据中Arg和Cys使用频率为0。Phe,Leu,Gln等使用频率较高,分别是12.06%,22.70%,12.77%。
3.4 遗传距离遗传距离最近的是HMS和HSX枣实蝇,为0.011,遗传距离最远的为KSS枣实蝇与吐鲁番市的枣实蝇,为0.052。
3.5 建树方法以同属于双翅目的樱桃绕实蝇、蔷薇咔实蝇isolate R141、蔷薇咔实蝇isolate R248为外群,利用未加权组对算术平均法(UPGMA)和邻接法(NJ)构建分子系统聚类图(图 2,3)。由聚类分析可知2两种方法得到的聚类图的拓扑结构基本一致。在系统树中,吐鲁番市,HSX和HMS枣实蝇先聚在一起,然后与分别来自2个不同地方的蔷薇咔实蝇聚为一支,整个咔实蝇属的2种实蝇再与樱桃绕实蝇聚在一起。对外群分析发现,2种方法构建的聚类图均能够把樱桃绕实蝇与枣实蝇分开,且外群亲缘关系基本符合其阶元划分,可以看出本试验所选的外群是正确性。2种聚类图表明KSS枣实蝇与吐鲁番市、HSX、HMS枣实蝇亲缘关系较远,这与阿地力·沙塔尔等(2012)的研究结果一致。
在Cytb基因上的遗传分化及进化突变速率要比COI基因上的快一些,这与Simon等(1994)的研究结果一致。因此,在研究枣实蝇的地理种群结构中,以Cytb基因作为分子标记比COI基因可能要灵敏,但区分不会太大。要探测枣实蝇种群因地理条件、寄主、气候等因素而呈现出的进化上的细微分化,仍需结合SSR(简单序列重复)等方法。
| [1] |
阿地力·沙塔尔,张伟,程晓甜,等. 2012.枣实蝇不同地理种群的亲缘关系研究.林业科学,48(6):136-140.( 4) 4)
|
| [2] |
陈乃中. 1998.具有检疫意义的果实害虫-实蝇科(部分属种).植物检疫,12(5):298-301.( 1) 1)
|
| [3] |
胡剑. 2008.瓜实蝇的亲缘地理学和生态学研究.广州:中山大学博士学位论文.( 1) 1)
|
| [4] |
刘运强,廖顺尧,鲁成,等. 2001.动物线粒体编码序列相邻碱基组成对核苷酸替换的影响.西南农业大学学报,23(3):270-272.( 1) 1)
|
| [5] |
任竹梅,马恩波,郭亚平. 2003.不同地域小稻蝗mtDNA部分序列及其相互关系.昆虫学报,46(1):51-57( 1) 1)
|
| [6] |
施伟,叶辉. 2004.云南桔小实蝇五个地理种群的遗传分化研究.昆虫学报,47(3):384-388.( 1) 1)
|
| [7] |
孙娜. 2010.金龟子Cytb、COI序列比较及在分子系统学研究中的作用.沈阳:沈阳大学硕士学位论文.( 2) 2)
|
| [8] |
吴佳教,陈乃中. 2008.Carpomya属检疫性实蝇.植物检疫,22(1):32-34.( 1) 1)
|
| [9] |
张长禹. 2007.五种实蝇的分子系统发育分析与分子快速鉴定研究.武汉:华中农业大学硕士学位论文.( 2) 2)
|
| [10] |
张润志,汪兴鉴,阿地力·沙塔尔,等. 2007.检疫性害虫枣实蝇的鉴定与入侵威胁.昆虫知识,44(6):928-930.( 2) 2)
|
| [11] |
张亚平,施立明. 1992.动物线粒体DNA多态性的研究概况.动物学研究,13(3):289-298.( 1) 1)
|
| [12] |
朱振华,叶辉,张智英. 2005.基于mtDNA Cytb的六种果实蝇的分子鉴定.昆虫学报,48(3):386-390.( 1) 1)
|
| [13] |
Hugall A,Stanton J,Moritz C. 1997.Evolution of the AT-rich mitochondrial DNA of the root knot nematode Meloidogyne hapla.Mol Biol Evol,4(1):40-48.( 1) 1)
|
| [14] |
IrWin D M,Kocher T D,Wilson A C. 1991.Evolution of the cytochrome bgene of mammals.Mol Evol,32(2):128-144.( 1) 1)
|
| [15] |
Lakra R K, Singh Z. 1983.Oviposition behavior of Ber Fruitfly Carpomya vesuviana and relationship between its incidence and ruggedness in fruits in Haryana India. Indian Journal of Entomology,45(1):48-59.( 1) 1)
|
| [16] |
Lunt D H,Zhang D X,Szymura J M, et al.1996.The insect cytochrome oxidase I gene:evolutionary patterns and conserved primers for phylogenetic studies.Insect Mol Biol, 47(3):361-366.( 1) 1)
|
| [17] |
Meyer A,Wi1son A C. 1990.Origin of tetrapods inferred from their mitochondrial DNA affiliation to lungfish.Mol Evol,31(5):359-364.( 1) 1)
|
| [18] | Motitz C, Dowling T E,Brown W M. 1987.Evolution of animal mitochondrial DNA:Relevance for population biology and systematics.Ann Rev Ecol Syst,18(6):269-292. |
| [19] |
Simon C, Frati F, Beckenback A, et al.1994.Evolution,weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers.Annms of the Entomological Society of America,87(6):651-701.( 4) 4)
|
 2014, Vol. 50
2014, Vol. 50

