文章信息
- 于存, 池玉杰
- Yu Cun, Chi Yujie
- 一色齿毛菌锰过氧化物酶(MnP)活性检测与MnP1基因的克隆
- Detection on Manganese Peroxidase Activity and Cloning of Cu-mnp1 of Cerena unicolor
- 林业科学, 2014, 50(3): 109-116
- Scientia Silvae Sinicae, 2014, 50(3): 109-116.
- DOI: 10.11707/j.1001-7488.20140315
-
文章历史
- 收稿日期:2013-05-12
- 修回日期:2013-08-01
-
作者相关文章
白腐菌在分解木质素的过程中通过分泌能够降解木质素的酶系统,氧化与分解木质素,这些酶系统主要包括锰过氧化物酶(manganese peroxidase,MnP)、木质素过氧化物酶(lignin peroxidase,LiP)和漆酶(laccase)等。其中MnPs(EC 1.11.1.13)是一类依赖H2O2的含Fe3+和血红素辅基的糖基化细胞外过氧化物酶,分子质量在38~62.5 kDa之间,仅广泛存在于多孔菌目(Polyporales)、伞菌目(Agaricales)、革菌目(Corticiales)、刺革菌目(Hymenochaetales)的白腐担子菌中,到目前为止在其他微生物中并未发现有MnP的相关报道(Janusz et al.,2013)。在白腐菌降解木质素的非特异性细胞外氧化酶中,MnP最常见,对降解木质素结构起着至关重要的作用,被认为是木质素降解的关键酶(Deguchi et al.,1998; Kirk et al.,1998)。
自从Masaaki等(1984)首次从黄孢原毛平革菌(Phanerochaete chrysosporium)的木质素裂解培养液中分离检测到MnP以来,由于其在生物制浆、生物降解等方面的潜在应用,被人们广泛关注。MnP通常以多种同工酶的形式存在,白腐菌在菌丝营养生长的不同阶段会分泌一系列相近的MnPs同工酶,一般高水平的液体培养基中至少存在3种及以上的异构体。随着白腐菌分子生物学研究的迅速发展,越来越多的MnPs同工酶基因已被克隆(Alic et al.,1997; Li et al.,1999; Giardina,2000; Maeda et al.,2001; Johansson et al.,2002; Hilden et al.,2005; Nagai et al.,2007)。
一色齿毛菌(Cerrena unicolor)又名单色下皮黑,是一种生长在多种阔叶树枯立木、倒木和衰弱活立木上的多孔菌科(Polyporaceae)白腐菌。对该种MnP基因的研究国内外尚属空白。由于MnP 底物的多样性及酶本身结构、基因编码、调控表达的复杂性,对于MnP的分子生物学、高效表达体系的构建等研究尚有待深入。因此,研究白腐菌MnP的基因结构具有重要意义,可为进一步深入研究MnP降解芳香族化合物的机制与mnp基因表达调控之间的关系提供基础依据,对进一步构建优良工程菌株也具有重要意义。
1 材料与方法 1.1 菌种来源和培养基一色齿毛菌菌株CB1由采自于长白山的子实体上分离得到,PDA斜面上的菌种保存于4 ℃冰箱。试验时挑取小块试管内的菌种,接种于PDA平板培养基上,于28 ℃下培养,经4~5天菌丝长满平板。
产酶基础培养基(L-1): 低氮天冬酰胺-琥珀酸培养基(LNAS,low nitrogen asparagine succinic acid),是一种营养成分有限的基础产酶培养基,参见闫洪波(2009)。
1.2 产酶培养方式与酶活性测定方法产酶培养方式与酶活性测定方法均按照闫洪波(2009)进行。采用3种不同培养方式进行菌种培养: A. LNAS培养基不含Mn2+,即矿物元素溶液中不添加MnSO4·H2O; B. LNAS培养基含Mn2+(2.67×10-3 mmol·L-1),即矿物元素溶液中添加MnSO4·H2O; C. LNAS培养基含Mn2+(2.67×10-3 mmol·L-1),并加入2 g大青杨(Populus ussuriensis)木屑为产酶底物。每项试验设3个重复。培养至3,5,7,9,11,13,15,17,19,21天提取培养液即胞外酶液,测定酶活后,绘制产酶活性时间曲线图,利用SPSS软件数据处理系统对3种不同的培养方式产生的酶活性进行方差分析。
1.3 Cu-mnp1 基因克隆 1.3.1 基因组DNA的提取与Cu-mnp1 基因DNA片段的扩增从PDA斜面培养基上取少量菌丝接种于PDA平板培养基上,在28 ℃下培养4天后,刮取长势旺盛的三皿平板上的新鲜菌丝体,使用TIANGEN公司的DNAquick_快捷型植物基因组DNA提取系统及相关方法提取真菌基因组DNA。将所得DNA用1.0%琼脂糖凝胶电泳和Eppendof公司的核酸测定仪检测DNA的大小、纯度及浓度,然后将DNA稀释到终浓度为100 ng·μL-1用于PCR反应。
根据GenBank已报道的多种担子菌不同MnP基因序列,用序列对比软件ClustalX 2.0进行同源比对查找保守区,设计出上游和下游2条PCR引物1F,1R(表 1),以一色齿毛菌基因组DNA为模板,用于扩增其MnP1基因(Cu-mnp1)的DNA片段。25 μL反应体系组成为: 10×PCR Buffer 2.5 μL,2.5 mmol·L-1 dNTP 4 μL,10 μmol·L-1上下游引物各0.5 μL,5 U·μL-1 rTaq DNA 聚合酶0.25 μL,模板DNA 0.5 μL,然后往反应体系内补足ddH2O至25 μL。反应条件为: 94 ℃预变性3 min; 94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,30个循环; 72 ℃延伸10 min。扩增完毕,取样用1.0%的琼脂糖凝胶电泳检测,观察电泳图确定目的条带。
1.3.2 染色体步移法(genome walking)扩增Cu-mnp1 全长DNA基因根据已克隆到的Cu-mnp1基因片段,在 5' 端设计了2次3个巢式特异性引物SP1,SP2和SP3,在3' 端设计了1次3个巢式特异性引物SP1,SP2和SP3(表 1),设计方向为需要扩增的未知区域方向,以一色齿毛菌基因组DNA为模板,利用TaKaRa公司的染色体步移试剂盒(Genome Walking Kit)及相关方法克隆该基因的5'上游和3'下游序列,以获得全长DNA基因。引物设计要求、PCR的反应体系、扩增的温度条件和循环数等见http://www.takara.com.cn/?action=Page&Plat=pdetail&newsid=289&subclass=1。扩增完毕后,取第2,3轮PCR反应液各2 μL,用1.0%琼脂糖凝胶进行电泳检测,观察电泳图确定目的条带。
|
|
使用凝胶回收试剂盒(Gel Extraction Kit,E.Z.N.A)回收各个环节中PCR产物的目的片段,将目的片段连接到PMD18-T载体(TaKaRa)并转化至大肠杆菌(Escherichia coli)Top10感受态细胞中,进行蓝白斑筛选后所得菌液交给上海英俊生物技术公司进行双向测序。每个PCR产物都是2个以上克隆进行测序以核准编码。测序结果用BLAST进行比对分析,将Cu-mnp1基因组DNA片段与染色体步移获得的5'上游和3'下游序列进行比对并拼接,从而获得Cu-mnp1全长DNA基因。
1.4 Cu-mnp1 全长DNA基因生物信息学分析通过RNA剪接位点(内含子/外显子)Augustus预测网站(http://augustus.gobics.de/)预测内含子和外显子的数量、位置,并通过在NCBI上进行同源序列比对进行核对与校正。外显子核准后,通过NCBI的ORF Finder(http://www.ncbi.nlm.nih.gov/projects/gorf/)查找该基因的读码框ORF,分析起始密码子、终止密码子。使用BioEdit分析软件对DNA和 cDNA核苷酸序列进行碱基成分分析,将cDNA核苷酸序列翻译为氨基酸序列,并对其氨基酸组成进行分析。利用Neural Network Promoter Prediction(http://www.Fruitfly.org/seq_tools /promoter.html)对Cu-mnp1 5'启动子区进行结构分析。使用NCBI的BLAST和Conserved Domain Database(CDD)软件(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析蛋白序列的基本信息、保守结构域。采用Signal Scan(http://www-bimas.cit.nih.gov/ molbio/signal/)和SignalP 4.0(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白信号肽。使用CLUSTAL 2.0在线(http://www.ebi.ac.uk/Tools/clustalw2/index.html)和MEGA 4.0软件对多种白腐菌的MnP同工酶进行蛋白序列比对,分析保守氨基酸及金属结合位点等信息,与其他白腐菌MnP的异同,采用NJ法构建系统发育树。使用swissmodel的自动建模模式(http://swissmodel.expasy.org/)以及NCBI的Vector Alignment Search Tool(VAST)进行蛋白质三维结构的预测以及功能位点的模拟。
2 结果与分析 2.1 3种培养方式降解木质素的过氧化物酶活性检测一色齿毛菌菌株CB1在21天的培养过程中,经以藜芦醇为底物的测定方法,在3种不同的培养方式中于310 nm处均未检测到LiP的活性; 但3种培养方式,经以2,6-二甲氧基苯酚(2,6-DMP)为底物,于470 nm处都检测到了MnP的活性。在培养方式A的培养液中不含Mn2+,提取的酶液检测到了MnP的活性,9天达到最高为15.07 U·L-1,随后出现下降,然后在较低的水平保持稳定; 在培养方式B的培养液中含Mn2+,提取的酶液检测到了MnP的活性,也是在9天达到最高为16.05 U·L-1,随后出现下降,然后在较低的水平保持稳定。A、B 2种培养方式在第3天都可以检测到MnP的活性,2种培养方式的酶活性曲线较相似,仅仅是在培养液中含有Mn2+比不含有Mn2+的MnP酶活性稍高一点,但Mn2+不是一色齿毛菌产生MnP的必要因子。在培养方式C中,提取的酶液也是在第3天就检测到MnP的活性,但第3天就出现一个高峰,有较高的酶活分泌,随后第5天出现分泌量下降的趋势,在第7天又上升达到最大分泌量,酶活性为18.4 U·L-1,随后出现下降,然后在较低的水平保持稳定。对比A、B、C 3种培养过程中MnP的产生结果,表明在培养液中添加底物木屑可以提高MnP的产生量,且最大酶活分泌时间提前2天,底物对于一色齿毛菌的MnP可产生诱导作用。 21天内一色齿毛菌在3种培养方式中产生MnP 的情况见图 1,3组重复数据的误差线标注其上。
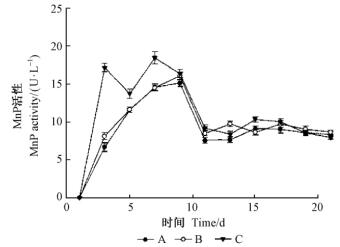 |
图 1 一色齿毛菌在3种培养方式下MnP活性随时间的变化
Fig. 1 MnP activity of 3 kinds of culture methods
|
方差分析结果表明,培养时间的显著性水平Sig.值=0.00<0.05,表明不同培养时间之间的MnP活性差异显著; 而培养方式的显著性水平Sig.值=0.03<0.05,表明3种不同培养方式之间MnP的活性有差异,但差异不如不同培养时间之间的MnP活性显著(表 2)。培养方式的多重比对结果(表 3)显示,在P=0.05时培养方式A与培养方式C之间差异显著,即添加木屑对MnP的影响是显著的。
|
|
|
|
以基因组DNA为模板,用引物1F和1R扩增到一个598 bp的目的基因片段(图 2a-2)。将该片段序列登录到NCBI网站进行BLAST同源序列比对后,发现其具有MnP特性,与其他白腐菌的MnP具有最大同源性,其中与日本刺皮菌(Echinodontium japonicum)mnp(AF218412.1)、南方灵芝(Ganoderma australe)mnp(DQ267753.1)、糙皮侧耳(Pleurotus ostreatus)mnp(JN020144.1)等MnP基因相似性在67%~84%之间,因此确定该序列即为一色齿毛菌锰过氧化物酶1片段基因,将其命名为Cu-mnp1。
 |
图 2 Cu-mnp1全长DNA基因克隆(M: DL2000 Marker; APx: AP1-4)
Fig. 2 Cloning of full-length DNA gene Cu-mnp1(M: DL2000 Marker; APx: AP1-4)
a.Degenerate PCR product of Cu-mnp1 Lane 1: Not-target fragment; Lane 2: Target fragment. b.First 5' Genome Walking PCR product Lane 1,4,7,10: 5'-SP1+APx; Lane 2,5,8,11: 5'-SP2+APx; Lane 3,6,9,12: 5'-SP1+APx.c.3' Genome Walking PCR product Lane 1,2,3,4: 3'-SP1+APx; Lane 5,6,7,8: 3'-SP2+APx; Lane 9,10,11,12: 3'-SP3+APx. d.Second 5' Genome Walking PCR product Lane 1,2,3,4: 5'UTR-SP1+APx; Lane 5,6,7,8: 5'UTR-SP2+APx; Lane 9,10,11,12: 5'UTRSP3+APx. |
通过筛选5' 端第1次和3' 端染色体步移第1~3轮PCR产物的目的片段,经测序和BLAST比对分析,在5'和3' 端分别获得了一个905 bp和1 547 bp的目的序列(图 2b-1、图 2c-11上带),与Cu-mnp1 部分序列分别有244 bp和99 bp重叠区域,表明所得序列即为基因Cu-mnp1片段的5'端和3'端侧翼序列。3部分拼接后得到了2 680 bp的全长DNA基因。在此基础上,又在5' 端进行了第2次染色体步移,获得了一个1 623 bp的目的序列(见图 2d-12亮带),与2 680 bp的全长DNA基因5' 端序列有35 bp的重叠,并具有基因启动子序列的特征,表明所得序列即为全长DNA基因Cu-mnp1 5' 端的启动子序列。2部分拼接后得到了4 268 bp包括启动子区域的全长DNA基因。Cu-mnp1的基因组碱基序列包括5'启动子区域、外显子、内含子和3' 下游非编码区。
结合Augustus与BLAST比对,全面分析了Cu-mnp1全长DNA基因内含子和外显子的位置与数量,获得了cDNA序列信息。将该基因组DNA与cDNA基因序列分别提交到NCBI 的GenBank上,登录号分别为JQ782578.1(DNA)和JQ782579.1(cDNA)。对Cu-mnp1全长DNA基因序列结构分析的结果表明,该基因含有1 909 bp的完整开放阅读框(open reading frame,ORF)、起始密码子ATG、终止密码子TAA、5'非翻译区1 768 bp、3'非翻译区591 bp。该基因含有16个外显子、15个内含子,外显子长度在9~241 bp之间,内含子长度在50~58 bp之间。所有内含子剪切位点均符合真核生物普遍遵循的GT-AG规则。从ATG到TAA的cDNA序列G-C含量占52.38%,而DNA序列由于加入了内含子导致了G-C含量下降了4.19%。在cDNA水平上进行BLAST比对分析,Cu-mnp1与猴头菇(Hericium erinaceum)CB1 mnp2基因(JQ248599.1)的序列相似性最高为70%,与绵皮孔菌(Spongipellis sp.)mnp1(AB244274.1)、扁芝(Ganoderma applanatum)mnp1(AB035734.1)、云芝(Trametes versicolor) mnp(AJ745080.3)、猴头菇mnp1(HM116841.3)等相似性达到67%~69%,与T. versicolor的manganese-repressed peroxidase(mrp)(AF008585.1)、毛缘齿耳(Steccherinum fimbriatum)的过氧化物酶基因(HM480284.1)也有较高的相似性。
对基因Cu-mnp1 5' 非翻译区序列进行分析的结果表明,该区域包含了调控真核基因转录的基本元件。在翻译起始位点上游区-115~-65 bp之间存在推定的基础启动子区域,其序列为: 5'-tcctgcctc gtataaatacgtcaagaaatgcaggggaggacctcagtgctc-3',其中存在1个TATA-Box位于-105~-100 bp之间,推定的转录起始点位于-74 bp处。1个CAAT-Box位于-1 613~-1 609 bp,还有5个反向的CAAT-Box(-168,-424,-738,-1 410,-1 538)。还发现了一些重要的作用元件,包括1个转录因子结合位点SP1(-420,gggcgg)、2个转录激活蛋白1结合位点AP1(-1 595,tgactaa; -1 561,tgactca)、3个热激元件HSE符合5'-NTTCNNGAAN-3'(heat-shock element,-1 654,-1 015,-572)、3个异型生物质响应元件XRE(Xenobiotic response element,-1 599,-1 020,-632)等。
2.3 推定的一色齿毛菌锰过氧化物酶1(Cu-MnP1)的特征与系统发育分析基因Cu-mnp1在去掉内含子后,在cDNA水平上,含有1 092 bp的完整开放阅读框ORF,编码了363个氨基酸的蛋白质多肽前体(GenBank登录号AFK91529.1)。经信号肽预测,这个蛋白质多肽前体包含了1个19-aa的信号肽(其中包含有N-末端的MAF短序列),以及1个6-aa的中间前导短肽。这样成熟的一色齿毛菌锰过氧化物酶1(Cu-MnP1)的蛋白质长度为338个氨基酸。预测成熟蛋白分子质量为35.78 kDa,pI为4.89,带负电荷残基(Asp+Glu)有40个,带正电荷残基(Arg+Lys)有24个,总原子数为4 961个,分子式为C1 584H2 442N432O491S12。与催化结构、晶体结构和功能有关的氨基酸残基与其他白腐菌的MnP相同,都是保守的。主要包括: 1)3个与血红素连接的氨基酸R43,H47,H175; 2)3个与Mn2+连接的氨基酸E36,E40和H182; 3)5个与血红素远侧末端的Ca2+连接的氨基酸 S176,D193,T195,S198,D200,4个与近侧末端的Ca2+连接的氨基酸D48,G66,D68,S70; 4)构成4个二硫桥的8个半胱氨酸残基C3:C15,C14:C284,C34:C120和C248:C314。
Cu-MnP1蛋白三维结构的预测以及功能位点的模拟图如图 3。图中A,B,C为三级结构的不同侧面。从图 3可看出Cu-MnP1蛋白是一个含Fe3+,Ca2+和Mn2+金属离子结合位点,包含有10个主螺旋的高聚物蛋白。
 |
图 3 Cu-MnP1蛋白的三级结构与模拟功能位点
Fig. 3 The third-class structure and putative function sites of Cu-MnP1
|
使用BLAST和CLUSTAL 2.0在线对Cu-MnP1和多种白腐菌的过氧化物酶进行氨基酸序列同源性比对的结果表明: Cu-MnP1与污叉丝孔菌(D. squalens)的MnP(EJF56126.1)相似性最高为75%(覆盖度100%),与该种的LiP(EJF59849.1)相似性为71%(覆盖度100%),与T.versicolor FP-101664 SS1的多功能过氧化物酶VP(Versatile Peroxidase,EIW53183.1)相似性为69%(覆盖度100%),另外与T. versicolor的MnP(AAT90348.1)、D. squalens的VP(EJF56475.1)、H. erinaceum的MnP2(AFD50189.1)和MnP1(ADK26471.3)、肺形侧耳(P. pulmonarius)的VP(AFM93767.1)、P. ostreatus 的MnP3(ACM47219.1)的相似性在60%~65%之间。对来自10种白腐菌的21个MnP同工酶氨基酸序列进行系统进化树分析的结果表明,多个MnPs明显分为2个进化分枝(图 4),在进化树的上半部分即第1分枝的MnPs都含有5个二硫键,属于长的MnP称为MnP1; 在进化树的下半部分即第2分枝的MnPs都含有4个二硫键,属于短的MnP,称为MnP2。Cu-MnP1由于含有4个二硫键,在系统发育过程中与短MnPs(MnP2)遗传距离较近并聚类在一起。
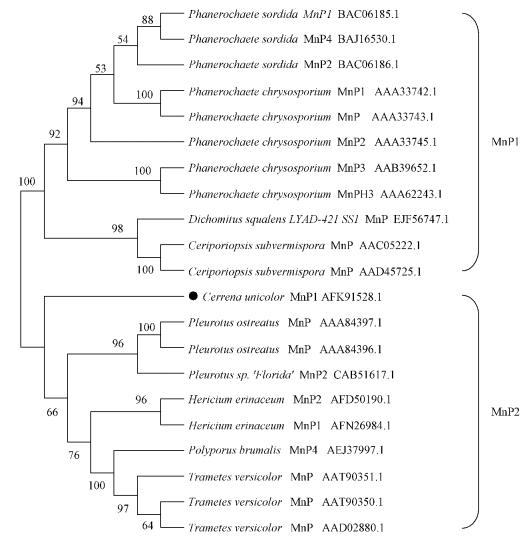 |
图 4 来源于10种白腐菌的21个MnPs的NJ系统发育树
Fig. 4 Neighbour joining tree of 21 complete MnPs from 10 basidiomycetes based on realigned sequences
Main groups MnP1 and MnP2 are depicted. Cu-MnP1 is indicated as ●. Bootstrap values are from 1 000 replications. |
本研究采用3种不同培养方式对一色齿毛菌降解木质素的过氧化物酶系统MnP和LiP的活性进行了检测; 在此基础上,采用常规PCR和染色体步移技术,克隆到了一色齿毛菌菌株CB1的MnP1基因Cu-mnp1。得出以下结论: 一色齿毛菌可产生MnP,但不产生LiP。对比3种不同培养方式下酶活性测定结果可以看出,在缺乏Mn2+的情况下也有微量的MnP产生; 在培养液内添加酶作用的底物木屑,可以提高MnP的分泌量。基因Cu-mnp1的5'启动子区域包含了调控真核基因转录的基本元件TATA-Box,CAAT-Box,Inverted CAAT-Box以及其他重要的顺式作用元件SP1,AP1,HSE,XRE等; Cu-MnP1蛋白的三维结构与功能位点具有与黄孢原毛平革菌MnP类似的特征; 对多条MnP蛋白进行系统进化分析的结果表明MnP可分为两大类群,其中Cu-MnP1归属于第2类含有4个二硫键的短MnP类群。
尹立伟等(2010)对灰树花、红平菇、杏鲍菇和乳白耙齿菌的MnP活性随时间变化的研究结果显示,在11~15天MnP活性达到最高。本研究中一色齿毛菌在相同培养条件下,在第7~9天MnP活性达到最高,MnP最大酶活分泌时间提前,推测原因可能与一色齿毛菌菌丝生长速度较快和诱导代谢提前有关。另外,对于灰树花和红平菇,Mn2+是产MnP必要因子,而一色齿毛菌在缺乏Mn2+的情况下也有微量的MnP产生。
在mnp启动子的研究中,发现一些mnp基因5'端上游启动子区域包含有几个共同的响应元件如SP1,AP2,HSE,MRE和XRE等,在不同来源的mnps之间也存在着不同的调控元件(Tello et al.,2000; Pease et al.,1989; Lobos et al.,1998; Hilden et al.,2005)。本研究中Cu-mnp1的5'启动子区域包含了调控真核基因转录的基本元件以及其他重要的顺式作用元件SP1,XRE,HSE等,但是却没有很多mnp基因启动子区域存在的金属响应因子(metal response element,MRE),这与Le-mnp1 (Nagai et al., 2007)和Ds-mnp(Li et al.,1999)相一致,这种情形在mnp基因中并不常见。推测一色齿毛菌MnP酶活不受Mn2+的调控可能与不存在该元件有关。
MnP从进化的观点来看,可以分出2类。第1类的预蛋白序列较长,一般含有376~395个氨基酸(平均383aa),含有5个二硫桥,其中第5个二硫桥用于稳定较长的C末端。很多较早研究的MnPs属于这类典型的或称作长型MnP(MnP1),它们形成了一个单系的类群并较早地从真菌过氧化物酶的系统发育树中分支出去(Lankinen et al.,2005; Morgenstern et al.,2010)。第2类MnP来源于木质素过氧化物酶(LiP)和多功能过氧化物酶(VP),具有较短的预蛋白(一般含有354~365aa,平均为363aa),含有4个二硫桥与LiP相似,具有MnP和LiP 2种特性(Sundaramoorthy et al.,1994),称作短MnP(MnP2)。随着研究的深入,一些短MnPs较多地被克隆出来,如Cu-MnP1由于含有4个二硫键而属于短MnP,系统发育分析也表明Cu-MnP1与短MnPs遗传距离较近并聚类在一起。氨基酸序列同源性比对的结果也表明,Cu-MnP1不仅与一些MnPs有较高的相似性,也与部分LiPs和VPs具有类似的相似性。
| [1] |
闫洪波. 2009. 偏肿拟栓菌锰过氧化物酶cDNA基因克隆及在毕赤酵母中的表达.哈尔滨:东北林业大学博士学位论文. ( 1) 1)
|
| [2] |
尹立伟,池玉杰,王雪童.2010.灰树花的系统发育分析和主要木质素降解酶的测定.林业科学研究,23(4):574-580.( 1) 1)
|
| [3] |
Alic M,Akileswaran L,Gold M H.1997.Characterization of the gene encoding manganese peroxidase isozyme 3 from Phanerochaete chrysosporium.Biochimica et Biophysica Acta,1338(1):1-7.( 1) 1)
|
| [4] |
Deguchi T, Kitaoka Y, Kakezawa M, et al.1998.Purification and characterization of a nylon-degrading enzyme. Appl Environ Microbiol,64(4):1366-1371.( 1) 1)
|
| [5] |
Giardina P, Palmieri G, Fontanella B, et al.2000.Manganese peroxidase isoenzymes produced by Pleurotus ostreatus grown on wood sawdust.Archives of Biochemistry and Biophysics,376(1):171-179.( 1) 1)
|
| [6] |
Hilden K, Martinez A T, Hatakka A, et al.2005. The two manganese peroxidases Pr-MnP 2 and Pr-MnP 3 of Phlebia radiata, a lignin-degrading basidiomycete,are phylogenetically and structurally divergent. Fungal Genetics and Biology,42(5):403-419.( 2) 2)
|
| [7] |
Janusz G, Kucharzyk K H, Pawlik A, et al.2013.Fungal laccase, manganese peroxidase and lignin peroxidase: gene expression and regulation.Enzyme and Microbial Technology, 52(1):1-12.( 1) 1)
|
| [8] |
Johansson T, Nyman P O, Cullen D.2002.Differential regulation of mnp 2 , a new manganese peroxidase-encoding gene from the ligninolytic fungus Trametes versicolor PRL 572.Appl Environ Microbiol,68(4):2077-2080.( 1) 1)
|
| [9] |
Kirk T K, Cullen D. 1998. Enzymology and molecular genetics of wood degradation by white-rot fungi//Young R A, Akhtar M.Environmentally friendly technologies for the pulp and paper industry. John Wiley and Sons, New York, 273-307.( 1) 1)
|
| [10] | Kuwahara M, Glenn J K, Morgan M A, et al. 1984.Separation and characterization of two extracelluar H2O-dependent oxideses from ligninolytic cultures of Phanerochaete chrysosporium. FEBS Lett,169(2):247-250. |
| [11] |
Lankinen P,Hildén K,Aro N,et al.2005.Manganese peroxidase of Agaricus bisporus: grain bran-promoted production and gene characterization.Appl Microbiol Biotechnol, 66(4): 401-407.( 1) 1)
|
| [12] |
Li D, Li N, Ma B, et al.1999.Characterization of genes encoding two manganese peroxidases from the lignin-degrading fungus Dichomitus squalens. Biochimica et Biophysica Acta,1434(2):356-364.( 2) 2)
|
| [13] |
Lobos S, Larrondo L, Salas L, et al. 1998.Cloning and molecular analysis of a cDNA and the Cs-mnp 1 gene encoding a manganese peroxidase isoenzyme from the lignin-degrading basidiomycete Ceriporiopsis subvermispora.Gene, 206(2):185-193.( 1) 1)
|
| [14] |
Maeda Y,Kajiwara S,Ohtaguchi K.2001.Manganese peroxidase gene of the perennial mushroom Elfvingia applanata: cloning and evaluation of its relationship with lignin degradation.Physics in Medicine and Biology,23(2):103-109.( 1) 1)
|
| [15] |
Morgenstern I, Robertson D L, Hibbett D S.2010.Characterization of three mnp genes of Fomitiporia mediterranea and report of additional class Ⅱ peroxidases in the order Hymenochaetales.Appl Environ Microbiol,76(19):6431-6440.( 1) 1)
|
| [16] |
Nagai M,Sakamoto Y,Nakade K,et al.2007.Isolation and characterization of the gene encoding a manganese peroxidase from Lentinula edodes.Mycoscience,48(2):125-130.( 2) 2)
|
| [17] |
Pease E A, Andrawis A, Tien M. 1989.Manganese-dependent peroxidase from Phanerochaete chrysosporium. Primary structure deduced from cDNA sequence.Journal of Biological Chemistry,264(23):13531-13535.( 1) 1)
|
| [18] |
Sundaramoorthy M, Kishi K, Gold M H, et al. 1994.The crystal structure of manganese peroxidase from Phanerochaete chrysosporium at 2.06-A resolution. J Biol Chem, 269(52): 32759-32767.( 1) 1)
|
| [19] |
Tello M,Corsini G, Larrondo L F, et al.2000.Characterization of three new manganese peroxidase genes from the ligninolytic basidiomycete Ceriporiopsis subvermispora.Biochimica et Biophysica Acta, 1490(1/2): 137-144.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

