文章信息
- 王文婷, 张东, 胡德夫, 初红军, 曹杰, 葛炎, 艾尔肯·吉力力, 李凯
- Wang Wenting, Zhang Dong, Hu Defu, Chu Hongjun, Cao Jie, Ge Yan, Aierken Jilili, Li Kai
- 新疆普氏野马马胃蝇蛆病主要病原体黑腹胃蝇溯源
- Analysis of the Main Etiology of Gasterophilosis in Przewalski's Horse in Xinjiang
- 林业科学, 2014, 50(11): 90-95
- Scientia Silvae Sinicae, 2014, 50(11): 90-95.
- DOI: 10.11707/j.1001-7488.20141112
-
文章历史
- 收稿日期:2014-04-17
- 修回日期:2014-05-29
-
作者相关文章
2. 新疆阿勒泰林业局 阿勒泰 836599;
3. 新疆野马繁殖研究中心 乌鲁木齐 831700
2. Altar Forestry Bureau of Xinjiang Altay 836599;
3. Xinjiang Research Center for Breeding Przewalski's Horse Urumqi 831700
马胃蝇在分类上属于双翅目(Diptera)环裂亚目(Cyclorrhapha)胃蝇科(Gasterophilidae)胃蝇属(Gasterophilus),其幼虫主要寄生于马科动物的消化道,对猪、狗、鸟和人类也偶有侵袭(Zumpt,1965; Soulsby,1982)。 现今全球共记载9种马胃蝇(Zumpt,1965),在中国新疆地区已发现其中的6种,即黑腹胃蝇(G. pecorum)、肠胃蝇(G. intestinalis)、鼻胃蝇(G. nasalis)、黑角胃蝇(G. nigricornis)、红尾胃蝇(G. haemorrhoidalis)和裸节胃蝇(G. inermis)(李凯等,2007)。
黑腹胃蝇雌蝇将卵产在植物的叶片和茎上,其他种马胃蝇将卵产在宿主的毛发、前肢、嘴唇、面部以及颌间等处(Sánchez-Andrade et al.,2010)。孵化后的1龄幼虫随宿主的采食进入口腔,蜕皮变成2龄幼虫后,迁移至消化道特定部位,并蜕皮成3龄幼虫。3龄幼虫在胃肠道间附着8~10个月(Coles et al.,2000)。
马胃蝇幼虫能引起马科动物胃肠道内的蝇蛆病。马胃蝇蛆病会影响吞咽,引起胃肠溃疡、内脏障碍物和肠扭结,导致贫血、腹泻、疝气、腹膜炎以及胃与十二指肠穿孔等疾病(Soulsby,1982; Sequeira et al.,2001)。
马胃蝇蛆病普遍发生于马科动物群体中(Edwards,1982;Hatch et al.,1976;Sweeney,1990;王善志等,1991;王明福,1998)。普氏野马(Equus przewalskii)作为再引入物种自2001年放归至祖居地新疆卡拉麦里有蹄类自然保护区(以下简称卡山保护区)以来,年年感染马胃蝇蛆病。卡山保护区常年生活着蒙古野驴(E.hemionus),同时是当地牧民传统的冬牧场,每年的10月下旬至次年的4月中下旬,牧民的家马(E.caballus)等家畜广布于卡山保护区内外,为避免普氏野马与家马打斗、相互圈群等意外发生,每年的10月下旬至次年的3月中下旬即家马聚集时期将野放普氏野马临时圈养,期间对所有野马进行驱虫保健。近年监测结果显示,虽然每年给野马驱虫,但次年感染情况依然普遍,野马感染率为100%(刘善辉,2012)。中国新疆野放野马群体感染马胃蝇的情况较其他国家和地区复杂得多,6种胃蝇集中出现在一个物种(野马)甚至出现在同一个体上,在国内外尚属首例(李凯等,2007)。普氏野马感染马胃蝇蛆病与同域生活的其他马科动物即家马和野驴密切相关。研究表明,普氏野马感染程度(马胃蝇寄生数量/匹马)明显高于同域其他马科动物,且黑腹胃蝇占感染数量的绝大多数(刘善辉,2012)。这一感染状况有别于世界其他地区报道的肠胃蝇、鼻胃蝇为优势种的研究情况(Horak et al.,1984;Otranto et al.,2005a)。
为探究普氏野马马胃蝇蛆病主要感染源,本文利用线粒体基因编码的细胞色素氧化酶亚基I(COI)进行研究。线粒体COI基因已被证明是研究昆虫种内和种间遗传结构分化和系统进化特别合适的分子标记(Lunt et al.,1996),在昆虫分子生物学研究中大量使用。线粒体COI基因的高变区已被用于研究狂蝇科18个种的分子鉴定及遗传分化(Otranto et al.,2003)。同时,基于线粒体COI基因对马胃蝇5个种的分子鉴定和系统发育研究发现,线粒体COI基因的分子鉴定结果与形态学分类结果相一致,肯定了COI基因在解决马胃蝇种间系统发生问题中起的作用(Otranto,2005b)。依据COI基因对意大利和波兰的鼻胃蝇和肠胃蝇的分子比较研究进一步证明COI基因是分析胃蝇遗传结构地理水平差异的有效工具(Pawlas-Opiela et al.,2010)。
本研究通过对新疆荒漠草原黑腹胃蝇的母系来源和种群遗传结构进行分析,以阐明新疆地区黑腹胃蝇的传染源,探讨当地3种宿主感染黑腹胃蝇程度差异的原因,为当地寄生虫控制及普氏野马保护提供科学指导和依据。
1 材料与方法 1.1 幼虫采集本研究所用的黑腹胃蝇3龄幼虫样本于2013年1月采卡山保护区。分别在野马(3匹)、家马(3匹)和野驴(1只)3种宿主体内各采集了116,31,48 只黑腹胃蝇幼虫。感染野马和家马的马胃蝇幼虫通过驱虫保健获得,感染野驴的马胃蝇幼虫通过对保护区同期意外倒毙的野驴尸检获得。所有采集样本用无水乙醇浸泡保存,经形态学分类鉴定(Zumpt,1965),置于-80 ℃冰箱保存。
1.2 DNA提取黑腹胃蝇基因组DNA的提取采用传统的酚氯仿抽提法(Sambrook et al.,2001)并稍加修改。所得DNA用紫外分光光度计检测,并于-80 ℃冰箱保存。
1.3 PCR扩增及测序PCR参照Lunt等(1996)用正引物UEA7 5’-TACAGTTGGAATAGACGTTGATAC-3’和反引物UEA10 5’-TCCAATGCACTAATCTGCCATATTA-3’扩增线粒体COI基因的目的区段。PCR扩增在0.2 mL PCR管中进行,反应体系为50 μL [2 mmol\5L-1 MgCl2,0.2 mmol\5L-1 dNTPs,5 μL 10×PCR buffer,0.25 μL Taq DNA聚合酶(5 U·μL-1)(Biomed,北京,中国),10 μmol\5L-1正反引物各0.5 μL,DNA模板3 μL]。PCR扩增程序为: 94 ℃预变性5 min;95 ℃ 1 min,58 ℃退火1 min,72 ℃延伸1 min,反应35个循环;72 ℃延伸10 min,16 ℃ 1 min。反应结束后,取3 μL PCR产物于1.5%的琼脂糖凝胶电泳检测。PCR产物经纯化后在ABI 3130XL型自动分析仪上进行测序。
1.4 核苷酸序列分析对所得COI基因序列通过整理去除两端序列。在Mega5.0(Tamura et al.,2011)中利用Clustal W进行多序列同源比对,辅以人工校对,并分析核苷酸组成及可变位点。应用软件DnaSP 5.0(Librado et al.,2009)计算种群间的单倍型多样性Hd、核苷酸多样性指数π、种群特征固定系数Fst等分子遗传学参数,计算种群间的基因流Nm,并进行中性检验(Tajima,1989)。通过MEGA 5.0软件,并基于Kimura two-parameter(K2P)模型,计算种群间遗传距离,采用NJ邻接法(Neighbor-joining)构建单倍型系统发生树(Saitou et al.,1987)。系统树各分支的置信度(bootstrap)均进行1 000次重复检验(Felsenstein,1985)。利用软件ARLEQUIN 3.5(Excoffier et al.,2010)中的分子变异分析(AMOVA)对黑腹胃蝇的种群遗传结构进行分析。
2 结果与分析 2.1 COI基因序列分析将总计195条COI序列进行比对,用于分析的序列总长为593 bp。序列相似度为99.52%。核苷酸平均含量为T 35.2%,C 20.9%,A 29.3%,G 14.7%,A+T含量为64.5%,显示出很强的偏向性。这与Otranto(2005b)对胃蝇属的研究结果(平均含量65%)一致。片段中无插入和缺失,含有527个保守位点,66个可变位点,其中单一位点19个,简约信息位点47个。在所有可变位点中,88%为核苷酸转换,12%为核苷酸颠换,总体转换/颠换比值为7.3。本研究共定义了57种单倍型: H1—H57(表 1)(单倍型序列已提交GenBank,登录号为KF844302—KF844358)。总体核苷酸多样性π为0.008 06,单倍型多样性Hd为0.936。
|
|
采用邻接法(NJ)构建黑腹胃蝇COI基因57种单倍型之间的系统发育树(图 1),显示出6个主要的簇。簇1占感染野马的黑腹胃蝇总数的63.79%,占感染野驴的黑腹胃蝇总数的60.42%。簇1和2分别占感染家马的黑腹胃蝇总数的35.48%和41.94%(表 2)。
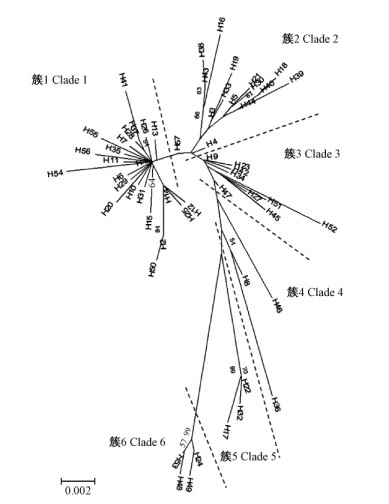 |
图 1 利用邻接法构建的黑腹胃蝇195个个体COI基因57个单倍型之间的系统发育树
Fig. 1 Phylogenetic tree based on the mtCOI gene of the 57
mitochondrial haplotypes from 195 individual
G.pecorum larvae using the neighbor-joining method
各分支上的数字为bootstrap 1 000 次循环的自举检验值。图中仅显示大于50%的bootstrap置信度值。 The confidence level was calculated with 1 000 bootstrap replicates. Probabilities are given at each node, but only values over 50% are presented. |
|
|
分别按3种宿主和6个簇统计195条序列的单倍型数、核苷酸多样性和单倍型多样性,并采用Tajima’s D值对COI基因序列做中性检验(表 3,4)。3种宿主的黑腹胃蝇种群单倍型多样性均很高(0.907 53~0.949 47),且种群整体没有出现群体扩张。6个簇的单倍型多样性范围为0.345 45~1,其中簇1,2,4,6的单倍型多样性较高,簇3和5的单倍型多样性相对较低。簇1和3出现过明显的群体扩张和持续增长,其余4簇未出现明显的群体扩张和持续增长,群体大小保持相对稳定。平均遗传距离显示,黑腹胃蝇在3种宿主间的平均遗传距离分别为0.008(普氏野马和家马)、0.009(普氏野马和野驴)和0.009(家马和野驴),在宿主间遗传距离没有明显差异,在6个簇之间遗传距离差异较大,范围在0.006~0.027之间(表 5)。黑腹胃蝇在3种宿主之间的固定系数(Fst)分别为0.033 53(普氏野马和家马),0.001 15(普氏野马和野驴)和0.021 11(家马和野驴),基因流(Nm)分别为7.206 01(普氏野马和家马),217.141 3(普氏野马和野驴)和11.592 73(家马和野驴),显示黑腹胃蝇在3种宿主之间的遗传分化程度较低(Fst<0.05,Nm>4),表明感染这3种宿主的黑腹胃蝇群体存在基因交流,其中,感染野马和野驴的黑腹胃蝇群体之间遗传分化程度最低,Nm值最大,表明感染野马和野驴的黑腹胃蝇群体基因交流最为频繁。6个簇之间黑腹胃蝇的固定系数(Fst)范围为0.357 05~0.916 67,基因流(Nm)范围0.022 726~0.450 182(表 6)。各簇间的基因交流较少(Nm<4),遗传分化程度很大(Fst>0.25)。在所有57种单倍型中,有5种共享单倍型(H1,H5,H9,H27,H28),其中H1为主导单倍型。分子变异分析(AMOVA)显示,黑腹胃蝇绝大部分(98.49%)变异来自同种宿主内部,显著大于在3种宿主之间的变异(1.51%),而在同一簇内部的变异(31.66%)小于不同簇之间的变异(68.34%)。
|
|
|
|
|
|
|
|
由于线粒体DNA具有严格的母系遗传特性,因此来源于相同母系的个体拥有相同的单倍型(Prabhakar et al.,2012)。这一独特性质使线粒体DNA成为研究系统发育及亲缘关系的有效分子标记。本研究通过对新疆地区黑腹胃蝇线粒体COI基因的分析发现,感染当地3种宿主的黑腹胃蝇均展现出很高的单倍型多样性,表明寄生于同一宿主个体中的黑腹胃蝇来自多个母本,推断新疆地区的黑腹胃蝇种群来源于具有一定程度遗传分化的群体。
本研究中,黑腹胃蝇在3种宿主间较低的遗传分化程度及AMOVA结果表明其在宿主间遗传多样性较低,在不同种宿主间未发生重大变异,且黑腹胃蝇在不同宿主间基因交流很频繁,足以抵制宿主间的差异造成的遗传漂变,即在不同种宿主间未形成生殖隔离。研究中获得5个共享单倍型,其中包括主导单倍型H1,这可能是由宿主的运动造成的。另有41种单倍型为单一宿主所特有,表明黑腹胃蝇在这3种宿主中可能存在一定程度的分化及具有一定程度宿主特异性。
宿主的运动对寄生虫的基因流起着关键的、决定性作用(Gómez-Díaz et al.,2012; McCoy et al.,2012)。寄生虫的种群遗传结构主要取决于寄生虫种群大小及群体间基因流(Blouin et al.,1995; Hawdon et al.,2001),这些因素很大程度上受宿主的活动区域及寄生虫生活史特征影响(Blouin et al.,1995; Wu et al.,2009)。本研究中,黑腹胃蝇的聚类分支模式可清晰地反映出其宿主的运动空间分为进行南北大尺度迁徙的家马类型和常年滞留卡山保护区的以野驴、野马为载体的荒漠草原类型。在卡山保护区黑腹胃蝇群体中,感染野马和野驴的黑腹胃蝇种群各簇所占的比例近似,系统发育和聚类模式相似,显示出非常相似的种群遗传结构,且感染这2种宿主的黑腹胃蝇群体间基因交流最频繁。这表明感染普氏野马和野驴的黑腹胃蝇近似为同一群体。这一结果可解释为,在黑腹胃蝇繁殖季节,野马和野驴活动区域存在着较大重叠。野马和野驴活动区域的高度重叠为黑腹胃蝇在这2种宿主间频繁的基因交流提供了很大机会。该结论首次明确了以黑腹胃蝇为优势种、导致普氏野马每年重度感染马胃蝇蛆病的主要病原体携带者为蒙古野驴,同时也反映出当地黑腹胃蝇传播盛期较为滞后,即在家马离开冬牧场之后发生的。本研究首次报道了黑腹胃蝇的种群遗传结构,结果反映出黑腹胃蝇与当地马科动物间的密切关系,研究结论为这一寄生虫的进一步研究及防控提供了战略指导,对普氏野马的保护和管理有重要意义。
| [1] |
李凯,吴专,胡德夫,等.2007.中国区普氏野马(Equus przewalski)胃蝇蛆病新病原体(Gasterophilus spp.)报道.畜牧兽医学报,38(8): 837-840.( 2) 2)
|
| [2] |
刘善辉.2012.马胃蝇形态学、流行病学调查及COI、16S rRNA基因系统发育研究.北京:北京林业大学硕士学位论文.( 2) 2)
|
| [3] |
王明福.1998.胃蝇科//薛万琦,赵建铭.中国蝇类.第2册.沈阳:辽宁科学技术出版社,2207-2215.( 1) 1)
|
| [4] |
王善志,徐显曾.1991.新疆马胃蝇种类观察及其地理分布.中国兽医科技,21(11): 14-16.( 1) 1)
|
| [5] |
Blouin M S,Yowell C A,Courtney C H,et al. 1995.Host movement and the genetic structure of populations of parasitic nematodes.Genetics,141(3):1007.( 1) 1)
|
| [6] |
Coles G,Pearson G.2000.Gasterophilus nasalis infection: prevalence and pathological changes in equids in south-west England.Veterinary Record,146(8): 222-223.( 1) 1)
|
| [7] |
Edwards G T.1982.The prevalence of Gasterophilus intestinalis in horses in Northern England and Wales.Veterinary Parasitology,11(2):215-222.( 1) 1)
|
| [8] |
Excoffier L,Lischer H E.2010.Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows.Molecular Ecology Resources,10(3):564-567.( 1) 1)
|
| [9] |
Felsenstein J.1985.Confidence limits on phylogenies: an approach using the bootstrap.Evolution,39(4):783-791.( 1) 1)
|
| [10] |
Gómez-Díaz E,Morris-Pocock J,González-Solís J, et al.2012.Trans-oceanic host dispersal explains high seabird tick diversity on Cape Verde islands.Biology Letters,8(4): 616-619.( 1) 1)
|
| [11] |
Hatch C,McCaughey W J,O'Brien J J.1976.The prevalence of Gasterophilus intestinalis and G.nasalis in horses in Ireland.Veterinary Record,98(14): 274-276.( 1) 1)
|
| [12] |
Hawdon J,Li T,Zhan B, et al.2001.Genetic structure of populations of the human hookworm,Necator americanus, in China.Molecular Ecology,10(6): 1433-1437.( 1) 1)
|
| [13] |
Horak I,Biggs H,Reinecke R.1984.Arthropod parasites of Hartmann's mountain zebra,Equus zebra hartmannae,in South West Africa/Namibia.The Onderstepoort Journal of Veterinary Research,51(3): 183.( 1) 1)
|
| [14] |
Librado P,Rozas J.2009.DnaSP v5: a software for comprehensive analysis of DNA polymorphism data.Bioinformatics,25(11): 1451-1452.( 1) 1)
|
| [15] |
Lunt D,Zhang D X,Szymura J,et al. 1996.The insect cytochrome oxidase I gene: evolutionary patterns and conserved primers for phylogenetic studies.Insect Molecular Biology,5(3): 153-165.( 2) 2)
|
| [16] |
McCoy K,Beis P,Barbosa A,et al. 2012.Population genetic structure and colonisation of the western Antarctic Peninsula by the seabird tick Ixodes uriae.Marine Ecology Progress Series,459: 109-120.( 1) 1)
|
| [17] |
Otranto D,Milillo P,Capelli G,et al. 2005a.Species composition of Gasterophilus spp. (Diptera,Oestridae)causing equine gastric myiasis in southern Italy: Parasite biodiversity and risks for extinction.Veterinary Parasitology,133(1): 111-118.(  1) 1)
|
| [18] |
Otranto D,Traversa D,Guida B,et al. 2003.Molecular characterization of the mitochondrial cytochrome oxidase I gene of Oestridae species causing obligate myiasis.Medical and Veterinary Entomology,17(3): 307-315.( 1) 1)
|
| [19] |
Otranto D,Traversa D,Milillo P,et al. 2005b.Utility of mitochondrial and ribosomal genes for differentiation and phylogenesis of species of gastrointestinal bot flies.Journal of Economic Entomology,98(6): 2235-2245.( 2) 2)
|
| [20] |
Pawlas-Opiela M,Wojciech Ł,Sołtysiak Z,et al. 2010.Molecular comparison of Gasterophilus intestinalis and Gasterophilus nasalis from two distinct areas of Poland and Italy based on cox 1 sequence analysis.Veterinary Parasitology,169(1): 219-221.( 1) 1)
|
| [21] |
Prabhakar C,Mehta P,Sood P,et al. 2012.Population genetic structure of the melon fly,Bactrocera cucurbitae(Coquillett)(Diptera: Tephritidae)based on mitochondrial cytochrome oxidase(COI)gene sequences.Genetica,140(1-3): 83-91.( 1) 1)
|
| [22] |
Saitou N,Nei M.1987.The neighbor-joining method: a new method for reconstructing phylogenetic trees.Molecular Biology and Evolution,4(4): 406-425.( 1) 1)
|
| [23] |
Sambrook J,Russell D W.2001.Molecular Cloning: A Laboratory Manual(3-volume set).Cold Spring Harbor Laboratory Press,Cold Spring Harbor.( 1) 1)
|
| [24] |
Sánchez-Andrade R,Cortiñas F,Francisco I,et al. 2010.A novel second instar Gasterophilus excretory/secretory antigen-based ELISA for the diagnosis of gasterophilosis in grazing horses.Veterinary Parasitology,171(3): 314-320.( 1) 1)
|
| [25] |
Sequeira J,Tostes R,Oliveira-Sequeira T.2001.Prevalence and macro-and microscopic lesions produced by Gasterophilus nasalis(Diptera: Oestridae)in the Botucatu Region,SP,Brazil.Veterinary Parasitology,102(3): 261-266.( 1) 1)
|
| [26] |
Soulsby E.1982.Helminths,Arthropods and Protozoa of Domesticated Animals.7th ed.Bailliere Tindall,London.( 2) 2)
|
| [27] |
Sweeney H J.1990.The prevalence and pathogenicity of Gasterophilus intestinalis larvae in horses in Ireland.Irish Veterinary Journal,43(3): 67-73.( 1) 1)
|
| [28] |
Tajima F.1989.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism.Genetics,123(3): 585-595.( 1) 1)
|
| [29] |
Tamura K,Peterson D,Peterson N,et al. 2011.MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Molecular Biology and Evolution,28(10): 2731-2739.( 1) 1)
|
| [30] |
Wu S G,Wang G T,Xi B W,et al. 2009.Population genetic structure of the parasitic nematode Camallanus cotti inferred from DNA sequences of ITS1 rDNA and the mitochondrial COI gene.Veterinary Parasitology,164(2): 248-256.( 1) 1)
|
| [31] |
Zumpt F.1965.Myiasis in Man and Animals in the Old World: Textbook for Physicians,Veterinarians and Zoologist.Butterworths,University of Michigan.( 2) 2)
|
 2014, Vol. 50
2014, Vol. 50

