文章信息
- 赵鹏, 周惠娟, 刘占林, 呼东峰, 党萌, 李忠虎, 王玛丽, 赵桂仿
- Zhao Peng, Zhou Huijuan, Liu Zhanlin, Hu Dongfeng, Dang Meng, Li Zhonghu, Wang Mali, Zhao Guifang
- 胡桃属植物分子系统发育和生物地理研究进展
- A Review of Research Progress on Molecular Phylogeny and Biogeography in Juglans
- 林业科学, 2014, 50(11): 147-157
- Scientia Silvae Sinicae, 2014, 50(11): 147-157.
- DOI: 10.11707/j.1001-7488.20141120
-
文章历史
- 收稿日期:2014-01-13
- 修回日期:2014-04-29
-
作者相关文章
2. 美国农业部 普渡大学森林与自然资源系 美国西拉法叶市 47907;
3. 陕西省环境监测中心站 西安 710054
2. United States Department of Agriculture Department of Forestry and Natural Resources, Purdue University West Lafayette, IN 47907, USA;
3. Shaanxi Environmental Monitoring Station Xi'an 710054
胡桃属(Juglans)隶属胡桃科(Jugl and aceae),约有21~23个种,为胡桃科内较为进化的属,该属植物在全球分布十分广泛,为欧洲、亚洲和美洲间断分布植物类群(图 1,Aradhya et al.,2007)。其多数成员为重要的树种资源,有着重要的食用、材用、药用和生态价值,在果仁品质、木材质量和古玩艺术雕刻等方面的研究备受关注。大量研究表明,核桃(J. regia)果仁具有很高的抗氧化能力(Damasceno et al.,2011),且含有很高浓度的Omega-3脂肪酸和蛋白质等营养成分,对人类健康非常有利(Ros et al.,2006; Vinson et al.,2012)。
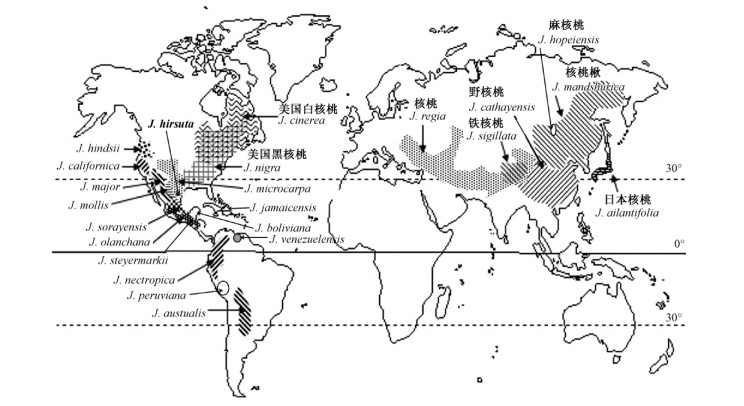 |
图 1 胡桃属植物的世界分布范围(Aradhya et al.,2007) Fig. 1 Distribution of Juglans species in the world(Aradhya et al.,2007) |
我国是世界胡桃属植物分布中心和起源地之一(图 1,Aradhya et al.,2007),是重要的核桃产区,种质资源丰富(Wang et al.,2008; 赵鹏,2011),分布十分广泛,其中包括大量人工栽培和野生胡桃属植物资源(路安民等,1990)。主要有以下5个种: 核桃、泡核桃(J. sigillata)、麻核桃(J. hopeiensis)、胡桃楸(J. m and shurica)和野核桃(J. cathayensis)(匡可任等,1979; 奚声珂,1987)。除了天然分布的胡桃属植物外,J. sieboldiana、美国黑核桃(J. nigra)、J. hindsii、J. microcarpa及J. hindsii×J. regia等在我国也有栽培(奚声珂,1987; 郗荣庭等,1996)。
近20多年来,随着分子生物学技术的迅猛发展,DNA分子水平的遗传标记被广泛用于解决系统进化、基因资源保护和遗传育种中的科学问题,帮助人们深刻理解和操作一些重要经济植物的复杂基因。同工酶技术和各种分子标记(RAPD,RFLP,SSR,ISSR,ITS,cpDNA)都曾被用于胡桃属亲缘关系(Potter et al.,2002a; Pollegioni et al.,2009)、系统发育(Aradhya et al.,2007)、谱系地理(Stanford et al.,2000; Bai et al.,2007)和遗传多样性(Woeste et al.,2002; Wang et al.,2008; Ross-Davis et al.,2008; 王滑等,2007)等方面的研究(Malvolti et al.,1994; Mohsenipoor et al.,2009; Zhao et al.,2011)。目前,根据文献报道,尽管胡桃属植物系统发育和生物地理学方面做了较多的研究工作,属内系统发育关系比较清楚,但关于胡桃属植物分组及个别种的归属和定论等问题有些争议,仍需进一步深入研究探讨。此外,世界范围胡桃属植物的起源和系统发育仍然需要利用形态学、孢粉学、化石和分子生物学等证据和方法进行综合验证和研究。
1 起源分布与系统发育胡桃科全球共包含有8个属,即胡桃属、山核桃属(Carya)、化香树属(Platycarya)、黄杞属(Englehardia)、青钱柳属(Cyclocarya)、枫杨属(Pterocarya)、果黄杞属(Alfaroa)及Oreomunnea属,共60余种,广泛分布在亚洲、欧洲和美洲; 中国有7属25种,分布在全国各地(Lu et al.,1999),均为落叶乔木,具有大型羽状复叶,叶长一般达20~100 cm(Polechko et al.,1986; Angiosperm Phylogeny Group,2009; Manos et al.,2001)。克朗奎斯特分类法单独将胡桃目(Jugl and ales)分出,属于金缕梅亚纲(Hamameliidae)(Cronquist,1988),2003年的APG Ⅱ分类法将胡桃科根据基因亲缘关系列入壳斗目(Fagales)(Angiosperm Phylogeny Group,2003)。胡桃属植物约有21~23种,约占胡桃科的35%(Dandekar et al.,2005; Aradhya et al.,2007),其系统发育方面的研究一直受到生物学家们的关注。
1.1 形态学分类最早进行胡桃属植物系统分类研究的科学家为Dode,他根据叶和花的形态将胡桃属植物分为黑胡桃组(Sect. Rhysocaryon)、胡桃楸组(Sect. Cardiocaryon)、灰胡桃组(Sect. Trachycaryon)和胡桃组(Sect. Dioscaryon或Sect. Juglans)4个组(Dode,1909a; 1909b)。Manning(1978)根据果实和核仁的形态特征也将胡桃属植物分为上述4个组(图 2a)。在形态学方面,胡桃属植物4组有着明显的差异,前3组(黑胡桃组、胡桃楸组、灰胡桃组)果实成熟后均有丰厚不开裂果皮,而第4组(胡桃组)果实成熟后有开裂的果皮(Dode,1909a; 1909b; Manning,1978)。路安民(1982)发表的“论胡桃科植物的地理分布”一文中也将胡桃属植物分为上述4组,其中,胡桃组和胡桃楸组分布于欧亚,黑胡桃组和灰胡桃组分布于北美到南美。之后人们普遍按照4组的分类方法对胡桃属植物进行研究。
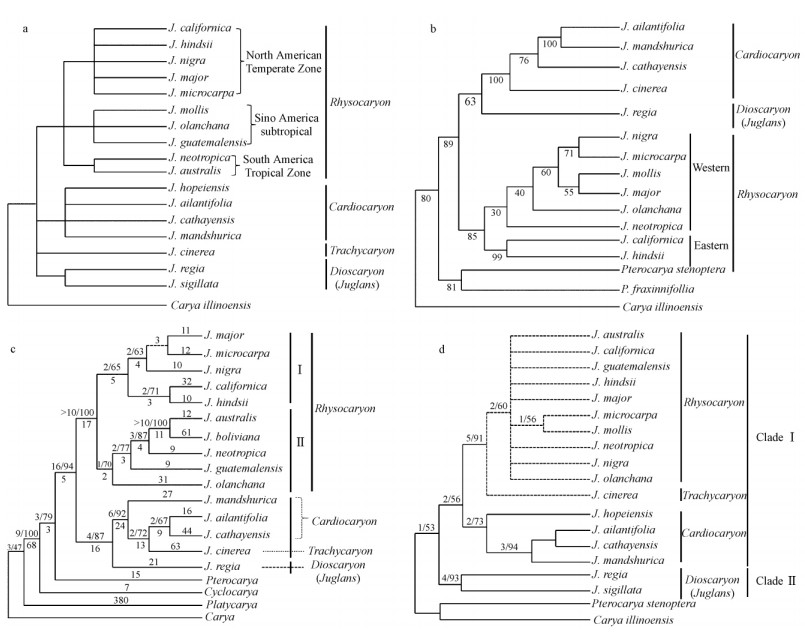 |
图 2 形态与分子数据构建胡桃属植物系统发育树
Fig. 2 Phylogenetic tree of Juglans using morphology and molecular data
a. 形态学分类特征构建的胡桃属植物系统发育树(Manning,1978); b. RFLP片段数据构建的胡桃属植物系统发育树(Fjellstrom et al.,1995); c. MatK+ITS片段数据构建的胡桃属植物系统发育树(Stanford et al.,2000); d. 基于7套叶绿体DNA片段序列数据构建的胡桃属植物系统发育树(Aradhya et al.,2007)。图中数字表示系统发育树分支支持率; 虚线表示新世界,实线表示旧世界。
a. Phylogenetic tree of Juglans using morphology and phenotype data(Manning,1978); b. Phylogenetic tree of Juglans using RFLP fragment data(Fjellstrom et al.,1995); c. Phylogenetic tree of Juglans using MatK+ITS data(Stanford et al.,2000); d. Phylogenetic tree of Juglans using seven chloroplast DNA sequence data(Aradhya et al.,2007). The number indicates that approval rating of phylogenetic tree; The dotted line indicates that New World,and the solid line indicates that Old World. |
Manchester(1987)根据叶的形态结构、木材解剖结构、花粉和果实形态学和化石证据将胡桃属植物分为3组,由于化石形态特点一致,将灰胡桃组和胡桃楸组合并为一组。从地理分布特点来看,胡桃楸组的4个种均分布在亚洲,而灰胡桃组的美国白核桃(J. cinerea)却天然分布在北美地区。这一不符合植物自然地理分布特征的化石证据结论使得人们对胡桃属植物系统发育的研究产生了争议(Manning,1978; Manchester,1987)。
1.3 分子系统发育学随着分子生物学的发展,人们开始利用分子方法对植物系统发育进行研究(Baldwin,1992)。Fjellstrom等(1995)利用限制片段长度多态性(RFLPs,restriction fragment length polymorphisms)对胡桃属13个类群和3个外类群植物(枫杨 Pterocarya stenoptera、P. fraxinnifollia及美国山核桃 Carya illinoensis)的系统发育进行研究,结果与公认的形态学分组不同,将胡桃属植物分为3组(图 2b): 第1组为黑胡桃组,分成东西分布地2支; 第2组为胡桃楸组和灰胡桃组的合并组; 第3组为胡桃组,并仅有核桃1个种(图 2b)。该研究没有对我国分布的泡核桃和麻核桃的系统地位进行分析,不仅将美国白核桃的系统位置归到了胡桃组和胡桃楸组之间,同时将胡桃楸和日本核桃(J. ailantifolia)归为一支,并提出了美国白核桃与我国分布的野核桃亲缘关系近、胡桃楸和日本核桃亲缘关系近(图 2b)的观点。由于有证据表明北美地区分布的美国白核桃与欧亚分布的胡桃组和胡桃楸组亲缘关系近(Fjellstrom et al.,1995),加之化石证据表明美国白核桃起源于北美(Manchester,1987),因此认为胡桃属植物可能起源于美洲,推测胡桃属植物在历史上经历了洲际迁移事件(Fjellstrom et al.,1995)。
为了进一步研究胡桃属植物系统发育关系,Stanford等(2000)利用双亲遗传的核糖体内转录间隔区(ITS)核基因片段和单亲遗传的叶绿体MatK基因片段对13个胡桃属植物的系统发育与生物地理关系进行了研究,结果可将胡桃属植物分为不同的两大支: 黑胡桃组(包括10个胡桃属植物)为一支,其他3个组胡桃楸组、灰胡桃组和胡桃组为一支(图 2c)。这一结果与形态特征(Manning,1978)、化石(Manchester,1987)和RFLP(Fjellstrom et al.,1995)分组有些类似,也将北美地区的美国白核桃归于欧亚地区分布组中,位于胡桃楸组和胡桃组之间。北美地区分布的美国白核桃与欧亚大陆分布的胡桃组和胡桃楸组亲缘关系近,化石证据表明该属植物在进化过程中有迁移和基因交流的经历(Manchester,1987),然而胡桃属植物为风媒植物,花粉主要通过风来传播,传播距离可能比较远,而种子主要靠动物和人类来传播(Gunn et al.,2010)。因此,如何进行长远距离的种间基因流及不同物种间分子序列相似的原因有待进一步研究(Manchester,1987)。目前,科学界解释为现代被子植物的分布格局显示欧亚大陆和美洲之间有间断分布现象是由于历史中欧亚大陆和美洲之间有路桥相连接,植物可以通过路桥进行基因交流(Wu,1983; Wen,1999; Xu et al.,2010; Wang et al.,2013)。
随机扩增多态性DNA分子标记(R and omly Amplified Polymorphic DNA,RAPD),呈显性遗传(Williams et al.,1990),方法简单快捷,不需要任何前期DNA模板信息(Zhao et al.,2011; Zhao et al.,2014)。Orel等(2003)利用形态学、线粒体DNA和RAPD分子标记技术,对胡桃科胡桃属8种植物、山核桃属美国山核桃和枫杨属越南枫杨(Pterocarya stenoptera var. tonkinensis)种间及属间的系统发育关系进行了分析,但仍有3个问题值得进一步探讨: 1)该文献指出美洲4个胡桃属植物J. australis、J. neotropica、J. olanchana和美国黑核桃亲缘关系近,与前人的研究结论一致,但没有对灰胡桃组系统发育地位和地理起源分布有争议的物种(美国白核桃)进行研究; 2)经典形态学分类研究表明我国分布的泡核桃应归于胡桃组,但是该文献将泡核桃分成单系支,且日本核桃与核桃亲缘关系近; 3)将胡桃属植物地理分布明显分为美洲大陆和欧亚大陆两大支,这就与前面提到的亚洲-美洲之间存在间断分布(Manning,1978; Stanford et al.,2000)有冲突。
由于胡桃属植物系统发育关系仍然需要进一步研究,因此,Aradhya等(2007)利用7套叶绿体基因序列,结合ITS序列(Stanford et al.,2000)和RFLP片段序列(Fjellstrom et al.,1995)分析探讨了核桃属植物的起源、进化时间以及迁移路线等问题,根据这些分子数据将胡桃属植物分成2组,即黑胡桃组、胡桃楸组和灰胡桃组合并为一组,胡桃组单系发育为一组(图 2d)。与形态学分类相比,原来单系灰胡桃组(美国白核桃)归到了黑胡桃组且5套叶绿体基因对此分类的支持率大于90%; 黑胡桃组和灰胡桃组归为一支,胡桃楸组和胡桃组归为一支,这两大支胡桃属植物正好与地理分布格局形成了旧世界和新世界2部分(Aradhya et al.,2007)(图 2d)。美国白核桃的现代地理分布似乎可以用间断分布解释清楚了,而为什么该种种群数量在北美不断减少(Hoban et al.,2010),现代该种在欧亚大陆为什么没有分布?这些科学问题仍需进一步探讨。
1.4 我国胡桃属植物系统发育研究除了世界范围胡桃属植物系统发育的研究以外,我国分布的5个胡桃属植物系统发育也有研究报道。关于我国特有种泡核桃的系统地位,Dode(1909a; 1909b)根据形态差异将其定为新种; 同工酶分析,却认为泡核桃与核桃种下的生态型平行,不宜划为另一个种(杨自湘等,1989); RAPD和EST-SSR标记分析结果认为,泡核桃可作为一个独立的种(吴燕民等,2000; 齐建勋等,2011); 结合ITS、RFLP与cpDNA基因序列分析结果也表明核桃和泡核桃为2个种,且支持率为92%(Aradhya et al.,2007)。因此,除了采用同工酶外,利用形态学、RAPD、EST-SSR、cpDNA及ITS等手段均支持我国特有种泡核桃为胡桃属的一个物种。
我国特有种麻核桃的系统发育地位一直争议较大,该种最早经中国植物分类学家胡先骕教授命名为新种(曲泽洲,1990)。由于其具有大型、长椭圆形或卵状椭圆形的叶,果序仅1~3个果而近似于核桃; 但内果皮多棱脊、具空隙,且隔膜厚与胡桃楸相像; 为此认为其为2个种的杂交种(胡先骕,1934; Rehder,1940)。然而,根据其花药有毛,幼叶、幼枝密被短柔毛及星芒状毛而后来毛被脱落,以及果实等形态特点认为该种更接近于胡桃楸,将该种作为胡桃楸的一个变种可能更为恰当,《中国植物志》二十一卷中,将麻核桃归为胡桃属的胡桃楸组(匡可任等,1979)。随着分子生物学的发展,利用同工酶技术也证实了麻核桃的酶谱是核桃和胡桃楸的互补酶谱(成锁占等,1987)。小孢子发生及核型研究表明,麻核桃花粉孔数目与花粉体积均居于核桃和胡桃楸之间,麻核桃花粉母细胞(PMC)的减数分裂过程极不正常(穆英林等,1990)。RAPD分子标记对麻核桃系统地位的分析结果显示,胡桃楸×核桃的天然杂交是麻核桃形成的主要机制,在麻核桃的起源中,胡桃楸的遗传贡献率大于核桃,结果赞成传统分类学对麻核桃分类地位的划分,证实麻核桃是核桃与胡桃楸的杂交种(吴燕民等,2000),这一结论与Aradhya等(2007)用cpDNA序列分析的结果(图 2d)并不一致。《Flora of China》将我国胡桃属植物归并为3个种: 核桃、泡核桃和胡桃楸(包括野核桃),关于麻核桃的归属问题在此未见详细的描述(Lu et al.,1999)。因此,关于麻核桃的系统地位还需进一步探讨的是: 麻核桃到底是不是核桃和胡桃楸的杂交种; 麻核桃因其形态特征表现更原始,是否可以推断该物种起源更早; 该物种在胡桃属植物分子系统发育树中的位置如何; 麻核桃与美国白核桃在进化历史中有什么关系; 麻核桃是不是可以也作为植物生物地理间断分布研究?
除此之外,我国特有种野核桃与胡桃楸的系统地位也有争议,如,《中国植物志》与《Flora of China》不一致,即《中国植物志》起初将野核桃与胡桃楸定为2个种,《Flora of China》根据野核桃与胡桃楸形态特征相似,将野核桃归到胡桃楸中(匡可任等,1979; Lu et al.,1999)。而国外学者基于形态学与各种遗传标记(如cpDNA、核基因ITS)均支持我国特有种野核桃与胡桃楸为2个种(Dode,1909a; 1909b; Manning,1978; Stanford et al.,2000; Aradhya et al.,2007)。如上所述,我国胡桃属植物5个种的系统发育地位仍需进一步详细研究。
1.5 基于基因数据库序列的系统发育研究随着测序技术的不断发展和更新,专家和学者们将已经检测出的胡桃属植物基因序列和数据共享到美国国家生物技术信息中心(national center biolotechnology information,NCBI,http://www.ncbi.nlm.nih.gov/)等基因库,即公共DNA序列库(GenBank)中。研究人员可从该数据库中获取各种资源共享的生物信息,并进行一系列的分析研究,大大加快了基因组学领域的研究速度。事实上,这些数据库已是核桃和其他许多经济植物基因数据重要的信息来源。截止2013年底,NCBI数据中共有1 822条胡桃属植物核苷酸序列(nucleotide),其中890条序列为核桃数据,214条为美国黑核桃。49 340条GSS(genome survey sequences)序列,21 375条EST(expressed sequence tag sequences)序列,其中21 294条序列与核桃有关(99.62%),81条美国黑核桃数据(0.38%)。另外有1条SRA(high-throughput DNA and RNA sequence read archive)美国黑核桃数据约2 Gb,151条PopSet(sequence sets from phylogenetic and population studies),465条蛋白质序列(protein sequences),其中核桃数据有296条(63.66%),美国黑核桃数据为57条(12.26%)(表 1)。
|
|
笔者根据NCBI基因库公布的ITS序列对16个胡桃属植物、外类群山核桃属美国山核桃及壳斗科(Fagaceae)板栗(Castanea mollissima)进行聚类分析。结果显示: 胡桃属植物可分成3组,即黑胡桃组、胡桃组和胡桃楸组,有趣的是美国白核桃仍归到胡桃楸组,并与日本核桃、胡桃楸和野核桃亲缘关系近; 核桃单独为一组,且胡桃组位于黑胡桃组与胡桃楸组之间(图 3)。这一结果从核基因片段方面进一步验证了现代分布的美国白核桃可能是从古老的胡桃楸组长期演化到黑胡桃组中的结论(图 3)。
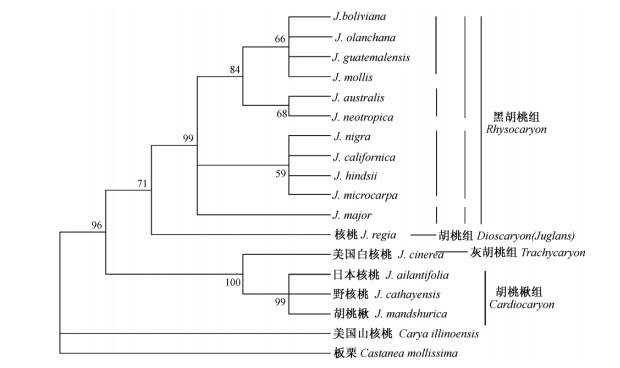 |
图 3 利用核糖体内转录间隔区(ITS)对胡桃属植物的系统发育树分析(最大简约法) Fig. 3 Phylogenetic analyses of internal transcribed spacer(ITS)data(maximum parsimony method) |
近20年来,植物生物地理学发展非常迅速,其中取得重要研究进展的一个领域就是第四纪冰期剧烈变化引起的全球气候变化对植物地理分布、遗传结构和进化历史的影响(Avise,2000; Hewitt,2000; 2004; Yang et al.,2008; Albert et al.,2010; Wang et al.,2012)。这些变化影响着不同纬度地区动植物的分布和遗传结构(Hewitt,2000; Avise,2000; Hua et al.,2013; Hadid et al.,2014)。化石记录、孢粉学、古气候数据解读植物分布区变化留存下的遗传信息也成为推测过去气候变迁物种迁移路线的有效手段(Avise,2000; Jaramillo-Correa et al.,2004; Schönswetter et al.,2005; Alsos et al.,2012),利用当前植物种群的遗传变异分布式样来研究物种的生物地理历史(Comes et al.,1998)。分子钟和化石证据广泛用于估算东亚和北美地区物种分化时间(Azuma et al.,2001; Smith et al.,2008)。胡桃属植物可能作为第三纪孑遗植物,对研究古植物区系的变迁和古地理及第三纪气候波动和环境变迁有着重要的科学价值。根据分子钟估算和化石证据研究表明,胡桃科可能起源于0.55~0.65亿年前第三纪始新世时期,气候地理分布范围为从北美到欧洲的亚热带和温带地区(Rogers,2004); 而最近研究表明胡桃属植物的起源地可能为欧亚大陆,东亚才是该属植物的避难所(Jørgensen et al.,2012); 且核桃的栽培历史已有6 800多年(Beer et al.,2008; Joly et al.,2009)。胡桃属植物和枫杨属植物分化时间大约在0.54亿年前的古新世/晚新世时期,而胡桃属植物的2个组灰胡桃组和黑胡桃组大约分化于0.5亿年前的中始新世(Fjellstrom et al.,1995)。黑胡桃组和灰胡桃组植物化石(美洲)约为0.4~0.5亿年前,J. megacinerea植物化石(亚洲)约为0.25亿年前,J. tephrodes植物化石(欧洲)约为0.3亿年前(Manchester,1987)。现有分子地理学研究表明,许多分布于欧亚大陆的温带植物起源于青藏高原及其周边区域(钱前等,2013; Jia et al.,2012; Gao et al.,2012)。
从生物地理学和地质历史分析表明,从中生代(约7亿年前)到晚中新世或早上新世(约1亿年前),亚洲和美洲之间曾经通过白令海峡路桥(或者可能为阿留申路桥)连接(Fujita,1978; McKenna,1983; Denk et al.,2011; Smith et al.,2006),欧洲-美洲路桥也有存在(McKenna,1983)。因此,可以推测胡桃属植物可能通过路桥在亚洲、欧洲和美洲等地区之间进行迁移。在进化历史中,胡桃属植物是由黑胡桃组和胡桃楸组的共同祖先分化成现在的这些物种,胡桃组植物现代地理分布格局可能是由于欧亚大陆隔离分化或者中亚地层活动造成的。亚洲-美洲和欧洲-美洲路桥消失后,由于长期地理隔离、基因流中断、遗传漂变和选择适应等因素使得该属植物分化成两大组,即北美地区和欧亚地区2部分(Stanford et al.,2000)。灰胡桃组(美国白核桃)从古老的胡桃楸组中分化到美国黑胡桃组中,时间大约在更新世冰期,而化石证据表明美国白核桃在渐新世和新近纪后期广泛分布于新世界(美洲大陆及邻近群岛)和旧世界(欧洲、亚洲和非洲)(Aradhya et al.,2007)。后因青藏高原隆升、秦岭太白山与东北长白山等地质事件,使得胡桃属物种因地理隔离分化成现在的4个组(Jia et al.,2012; Gao et al.,2012)。美国白核桃也可能是于渐新世中期气候变暖时由北美迁移到欧亚大陆,过去气候变化和洲际大陆动荡是造成现代物种的生物地理格局的主要原因(Stanford et al.,2000; Wang et al.,2013)。另外,更新世冰期,胡桃楸组有较强的基因渐渗到黑胡桃组植物,而胡桃组可能是黑胡桃组的祖先种。利用基因片段(matK)分析胡桃属植物生物地理历史的结果说明化石证据和叶绿体基因片段分析胡桃属植物分化时间不一致(Aradhya et al.,2007)。胡桃楸的谱系地理历史研究结果表明,胡桃楸在第四纪冰期有2个独立的避难所,且有着不对称的基因流存在于2个避难所之间(Bai et al.,2010)。这说明,第三纪时期物种广泛分布在东亚和北美地区,第四纪冰期胡桃属植物向南退缩至避难所,冰期后迅速重建扩张分化成美国黑核桃和美国白核桃2大组,而经过几次收缩和扩张、南迁和北移,美国白核桃最终形成现代地理分布格局。这样可以推测胡桃楸组现代分布的物种是否是由祖先种美国白核桃分化而来?如果能对美国白核桃的地理洲际间断分布格局进化历史和生物地理学进行系统研究和解释,这一结果将会为生物地理学的研究提供一个非常经典的理论参考依据(Gemma et al.,2012; Viruel et al.,2013; Liu et al.,2013)。
3 小结与展望随着分子生物学和测序技术的发展,胡桃属植物在系统发育和生物地理学方面的研究也有着长足的进步,已从形态学、分子生物学、孢粉学和化石等角度进行了比较详细的研究(路安民,1982; Manchester,1987; Fjellstrom et al.,1995; Stanford et al.,2000; Aradhya et al.,2007)。目前,普遍被接受用于系统发育和生物地理学研究的植物DNA分子标记主要有以下几种,即叶绿体基因片段rbcL、matK、ITS和psbA-trnH(Bai et al.,2010; Li et al.,2011a; 袁明等,2011; Viruel et al.,2013)。利用线粒体基因片段对该领域的研究较少(Jaramillo-Correa et al.,2004),但是上述这些DNA分子标记对于确定植物大类群(尤其是包括大量近缘物种的类群)内部系统发育关系的实际应用能力尚不清楚(钱前等,2013)。最初人们利用1~2套单亲遗传的叶绿体基因片段对植物系统发育与生物地理学进行研究(Baldwin,1992; Stanford et al.,2000; Taberlet et al.,2007)。为了发现更多的基因序列变异,采用多套cpDNA片段进行分析,然而结果表明,在部分植物类群中仍不能很好解决分支之间和分支内部的系统发育关系问题(Aradhya et al.,2007; Zeng et al.,2010)。核糖体DNA内转录间隔区(internal transcribed spacer,ITS)序列是核糖体DNA上的一个非编码区域,在系统发育学中应用广泛(Chen et al.,2001; Potter et al.,2002a; Li et al.,2011b)。结合核基因ITS与叶绿体DNA片段测序分析植物系统发育与生物地理学可以将2种遗传标记的优点发挥出来,但在部分植物类群中对组间与种间的亲缘关系用这2个标记仍难以区分清楚(Li et al.,2010)。在菊科(Asteraceae)中研究植物系统发育关系最好的DNA分子标记为ITS的部分序列ITS2(Gao et al.,2010),中国科学院昆明植物研究所李德铢研究组联合全国19个科研院所和高校的62名研究人员组成的“中国植物条形码研究团队”也将ITS/ITS2纳入种子植物核心条形码的范围(Li et al.,2011b)。前人对胡桃属植物系统发育研究是基于RFLP,RAPD,ITS,cpDNA等分子标记技术(Fjellstrom et al.,1995; Stanford et al.,2000; Aradhya et al.,2007),其中,既有单亲遗传的叶绿体基因片段,又有双亲遗传的核基因片段,同时具有显性和共显性遗传标记。但根据最新研究进展显示: 低(单)拷贝核基因不仅可以用来重建被子植物系统发育关系,且还得到了更加可靠的研究结论,也可以用来估算各大类群的起源时间。所以,今后低(单)拷贝核基因方面的证据对于被子植物的系统发育重建是必不可少的(Zhang et al.,2012; Mao et al.,2012; Huang et al.,2012)。
到目前为止,由于没有对胡桃属属内所有植物进行全面系统分析,所以属内不同物种之间的系统发育关系研究仍然需要更多的证据来证明其假设和结论的可靠性。关于组内物种的系统地位还需理清。如,基于形态学描述的胡桃属4组植物共包含16个种,不包含以下5个没有分组的物种: J. sorayensis,J. steyermarkii,J. peruviana,J. jamaicensis,J venezuelensis(Dandekar et al.,2005; 图 2); Aradhya等(2007)在胡桃属植物分布图中也没有列出J. guatemalensis(图 1); 麻核桃的起源和系统地位; 野核桃是否归并于胡桃楸等科学问题还有待于深入研究。因此,对于胡桃属植物进化历史、洲际间断分布的生物地理学和不同物种之间杂交渐渗也需要进一步阐明(Mao et al.,2012)。胡桃属植物系统发育关系和生物地理问题仍然需要更多的形态学、孢粉学、分子生物学、遗传学和化石等证据来进一步研究证实(Manos et al.,2007)。
生物地理学的研究通常采用叶绿体DNA变异来检测群体遗传结构式样,原因是绿色植物重要的细胞器叶绿体在植物光合中起着非常重要的作用,叶绿体基因组在进化上相对比较保守,重复少,在大部分植物中为单亲遗传,且突变率非常低(Xiang et al.,2011; Li et al.,2011a)。因此,研究种子植物从属内和种内水平到亚科和种群水平(Bai et al.,2010)的进化历史过程、冰期避难所、遗传分化、群体扩张和迁移等生物地理事件,叶绿体基因片段(cpDNA)显然是很好的分子手段(Aradhya et al.,2007; Taberlet et al.,2007; Bai et al.,2010; Gemma et al.,2012)。然而,由于叶绿体基因为单亲遗传分子标记,通过叶绿体基因片段(cpDNA)无法同时检测种子和花粉传播的生物地理学事件。另外,一套叶绿体仅能反应某个物种一个基因片段的谱系地理历史(Kikuchi et al.,2013)。微卫星分子遗传标记(microsatellites,or simple sequence repeats,SSR)具有高度多态性、共显性和重复性(Dib et al.,1996; Woeste et al.,2002; Li et al.,2002),该遗传标记结合贝叶斯方法进行分析后,可以很好地检测种内遗传结构和阐明物种的谱系地理历史(Bai et al.,2010; Hoban et al.,2010)。因此,各种分子标记仍然是研究胡桃属植物的一个非常重要的工具(邹喻萍等,2001; Ahmed et al.,2012; Hoban et al.,2009; Broders et al.,2011; 赵鹏等,2013)。针对胡桃属植物的研究中,只发现了少数几个物种的特异性标记(Germain et al.,1993; Potter et al.,2002b; Woeste et al.,2009; McCleary et al.,2009),后续从大量测序结果中找出胡桃属种间和种内的特异分子标记也迫在眉睫(Zhao et al.,2012)。随着二代测序技术的发展和测序成本的降低,植物转录组测序、基因组测序和功能基因测序将会非常普遍,这些可能会对非模式生物的研究提供非常有利的工具(Ciarmiello et al.,2011; Xu et al.,2012)。二代测序技术对研究胡桃属植物分子系统发育和生物地理将会产生深远影响,可以利用该技术开发大量的SSR、SNP、低拷贝核基因等遗传标记用于该领域,不仅可进行植物大类群的研究,同时可以进行科、属、种各个分类单元的分子系统发育与生物地理学研究,有助于更加全面地了解植物基因组的遗传变异特征、基因组大小变异的规律和机制。海量的数据分析将会使得人们对非模式生物研究也进入一个新的阶段,而这些序列数据将会很快被广泛应用到胡桃属植物的系统发育和生物地理学研究中(Ciarmiello et al.,2011; Zhou et al.,2011),同时生物信息学方面的研究也将成为非模式胡桃属植物和整个生物领域的热点。
| [1] |
成锁占,杨文衡.1987.根据同工酶对核桃属十个种分类学的研究.园艺学报,14(2): 90-96.( 1) 1)
|
| [2] |
胡先骕.1934.静生汇报.5:305( 1) 1)
|
| [3] |
匡可任,路安民.1979.中国植物志:第21卷.北京:科学出版社,30-35.( 3) 3)
|
| [4] |
路安民,张志耘.1990.胡桃木的分化、进化和系统关系.植物分类学报,28(2): 96-102.( 1) 1)
|
| [5] |
路安民.1982.论胡桃科植物的地理分布.植物分类学报,20(3): 257-271.( 1) 1)
|
| [6] |
穆英林,郗荣庭,吕增仁.1990.核桃属部分种的小孢子发生及核型研究.武汉植物研究,8(4): 301-309.( 1) 1)
|
| [7] |
齐建勋,郝艳宾,朱艳,等.2011.核桃属种质资源的EST-SSR标记研究.园艺学报,38(3): 441-448.( 1) 1)
|
| [8] |
钱前,瞿礼嘉,袁明,等.2013.2012年中国植物科学若干领域重要研究进展.植物学报,48(3): 231-287.( 2) 2)
|
| [9] |
曲泽洲.1990.北京果树志.北京:北京出版社,489-490.( 1) 1)
|
| [10] |
王滑,郝俊民,王宝庆,等.2007.中国核桃8个天然居群遗传多样性分析.林业科学,43(7): 120-124.( 1) 1)
|
| [11] |
吴燕民,裴东,奚声珂.2000.运用RAPD对核桃属种间亲缘关系的研究.园艺学报,27(1): 17-22.( 1) 1)
|
| [12] |
郗荣庭,张毅萍.1996.中国果树志:核桃卷.北京:中国林业出版社,56-78.( 1) 1)
|
| [13] |
奚声珂.1987.我国胡桃属(Juglans L.)种质资源与核桃(J.regia L.)育种.林业科学,23(3): 342-349.( 2) 2)
|
| [14] |
杨自湘,奚声珂.1989.胡桃属十种植物的过氧化物同工酶分析.植物分类学报,27(1): 53-57.( 1) 1)
|
| [15] |
袁明,王小菁,钱前,等.2011.2010年中国植物科学若干领域重要研究进展.植物学报,46(3): 233-275.( 1) 1)
|
| [16] |
赵鹏.2011.核桃属植物种质资源分子鉴定与遗传分析.杨凌:西北农林科技大学博士学位论文.( 1) 1)
|
| [17] |
赵鹏,Keith E.Woeste,程飞,等.2013.美国黑核桃SSR反应体系优化.植物研究,32(2):213-221.( 1) 1)
|
| [18] |
邹喻萍,葛颂,王晓东.2001.系统与进化植物学中的分子标记.北京:科学出版社,68- 107.( 1) 1)
|
| [19] |
Ahmed N,Mir J I,Mir R R,et al. 2012.SSR and RAPD analysis of genetic diversity in walnut(Juglans regia L.)genotypes from Jammu and Kashmir,India.Physiology and Molecular Biology of Plants,18(2): 149-160.( 1) 1)
|
| [20] |
Albert J S,Crampton W G R.2010.The geography and ecology of diversification in Neotropical freshwaters.Nature Education,3(10): 13.( 1) 1)
|
| [21] |
Alsos I G,Ehrich D,Thuiller W,et al. 2012.Genetic consequences of climate change for northern plants.Proceedings of the Royal Society of London Series B-Biological Sciences,279(1735): 2042-2051.( 1) 1)
|
| [22] |
Angiosperm Phylogeny Group.2003.An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG Ⅱ.Botanical Journal of the Linnean Society,141(4): 399-436.( 1) 1)
|
| [23] |
Angiosperm Phylogeny Group.2009.An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants: APG Ⅲ.Botanical Journal of the Linnean Society,161(2): 105-121.( 1) 1)
|
| [24] |
Aradhya M K,Potter D,Gao F,et al. 2007.Molecular phylogeny of Juglans (Juglandaceae): a biogeographic perspective.Tree Genetics & Genomes,3(4): 363-378.( 19) 19)
|
| [25] |
Avise J C.2000.Phylogeography: The history and formation of species.Cambridge,MA: Harvard University Press.( 3) 3)
|
| [26] |
Azuma R,Baillot Y,Behringer R,et al. 2001.Recent advances in augmented reality.Computer Graphics and Applications,IEEE,21(6): 34-47.( 1) 1)
|
| [27] |
Bai W N,Liao W J,Zhang D Y.2010.Nuclear and chloroplast DNA phylogeography reveal two refuge areas with asymmetrical gene flow in a temperate walnut tree from East Asia.New Phytologist,188: 892-901.( 5) 5)
|
| [28] |
Bai W N,Zeng Y F,Zhang D Y.2007.Mating patterns and pollen dispersal in a heterodichogamous tree,Juglans mandshurica(Juglandaceae).New Phytologist,176: 699-707.( 1) 1)
|
| [29] |
Baldwin B G.1992.Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: An example from the compositae.Molecular Phylogenetics Evolution,1(1): 3-16.( 2) 2)
|
| [30] |
Beer R,Kaiser F,Schmidt K,et al. 2008.Vegetation history of the walnut forests in Kyrgyzstan(Central Asia): natural or anthropogenic origin? Quaternary Science Review,27: 621-632.( 1) 1)
|
| [31] |
Broders K D,Woeste K E,SanMiguel P J,et al. 2011.Discovery of single-nucleotide polymorphisms(SNPs)in the uncharacterized genome of the ascomycete Ophiognomonia clavigignenti-juglandacearum from 454 sequence data.Molecular Ecology Resources,11(4): 693-702.( 1) 1)
|
| [32] |
Chen Y C,Eisner J D,Kattar M M,et al. 2001.Polymorphic internal transcribed spacer region 1 DNA sequences identify medically important yeasts.Journal of Clinical Microbiology,39(11): 4042-4051.( 1) 1)
|
| [33] |
Ciarmiello L F,Piccirillo P,Pontecorvo G,et al. 2011.A PCR based SNPs marker for specific characterization of English walnut(Juglans regia L.)cultivars.Molecular Biology Reports,38(2): 1237-1249.( 1) 1)
|
| [34] |
Comes P H,Kadereit J W.1998.The effects of quaternary climatic changes on plant distribution and evolution.Trends Plant Science,3(11):432-438.( 1) 1)
|
| [35] |
Cronquist A.1988.The evolution and classification of flowering plants.Bronx,NY: New York Botanical Garden,555.( 1) 1)
|
| [36] |
Damasceno N R T,Pérez-Heras A,Serra M,et al. 2011.Crossover study of diets enriched with virgin olive oil,walnuts or almonds.Nutrition Metabolism and Cardiovascular Diseases,21:14-20.( 1) 1)
|
| [37] |
Dandekar A,Leslie C,McGranahan G.2005.Juglans regia walnut//Litz R E.Biotechnology of fruit and nut crops.Wallingford,UK: CABI International Publishing,307-323.( 2) 2)
|
| [38] |
Denk T,Grímsson F,Zetter R,et al. 2011.The biogeographic history of Iceland-the North Atlantic land bridge revisited.Late Cainozoic Floras of Iceland.Springer Netherlands,647-668.( 1) 1)
|
| [39] |
Dib C,FauréS,Fizames C,et al. 1996.A comprehensive genetic map of the human genome based on 5,264 microsatellites.Nature,380(6570): 152-154.( 1) 1)
|
| [40] |
Dode L A.1909a.Contribution to the study of the genus Juglans(English translation by Cuendett R E).Bull Soc Dendrol France,11: 22-90.( 3) 3)
|
| [41] |
Dode L A.1909b.Contribution to the study of the genus Juglans(English translation by Cuendett R E).Bull Soc Dendrol France,12:165-215.( 3) 3)
|
| [42] |
Fjellstrom R G,Parfitt D E.1995.Phylogenetic analysis and evolution of the genus Juglans(Juglandaceae)as determined from nuclear genome RFLPs.Plant Systematics and Evolution,197(1/4): 19-32.( 10) 10)
|
| [43] |
Fujita K.1978.Pre-Cenozoic evolution of northeast Siberia.Journal of Geology,86: 159-172.( 1) 1)
|
| [44] |
Gao J,Wang B S,Mao I F,et al. 2012.Demography and speciation history of the homoploid hybrid pine Pinus densata on the Tibetan Plateau.Molecular Ecology,21: 4811-4827.( 2) 2)
|
| [45] |
Gao T,Yao H,Song J Y,et al. 2010.Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family.BMC Evolutionary Biology,10: 324.( 1) 1)
|
| [46] |
Gemma E B,Provan J.2012.Post-glacial dispersal,rather than in situ glacial survival,best explains the disjunct distribution of the Lusitanian plant species Daboecia cantabrica(Ericaceae).Journal of Biogeography,40(2): 335-344.( 2) 2)
|
| [47] |
Germain E,Hanquier I,Monet R.1993.Identification of eight Juglans spp.and their interspecific hybrids by isoenzymatic electrophoresis.Acta Hort,311:73-81( 1) 1)
|
| [48] |
Gunn B F,Aradhya M,Salick J M,et al. 2010.Genetic variation in walnuts(Juglans regia and J.sigillata;Juglandaceae): Species distinctions,human impacts,and the conservation of agrobiodiversity in Yunan,China.American Journal of Botany,97(4): 660-671( 1) 1)
|
| [49] |
Hadid Y,PavlíDček T,Beiles A,et al. 2014.Sympatric incipient speciation of spiny mice Acomys at "Evolution Canyon",Israel.Proceedings of the National Academy of Sciences,111(3): 1043-1048.( 1) 1)
|
| [50] |
Hewitt G.2000.The genetic legacy of the Quaternary ice ages.Nature,405(6789): 907-913.( 2) 2)
|
| [51] |
Hewitt G M.2004.Genetic consequences of climatic oscillations in the Quaternary.Philosophical Transactions of the Royal Society of London Series B: Biological Sciences,359(1442): 183-195.( 1) 1)
|
| [52] |
Hoban S M,Borkowski D S,Brosi S L,et al. 2010.Range-wide distribution of genetic diversity in the North American tree Juglans cinerea: a product of range shifts,not ecological marginality or recent population decline.Molecular Ecology,19: 4876-4891.( 2) 2)
|
| [53] |
Hoban S M,McCleary T S,Schlarbaum S E,et al. 2009.Geographically extensive hybridization between the forest trees American butternut and Japanese walnut.Biology Letter,5(3):324-327.( 1) 1)
|
| [54] |
Hua X,Wiens J J.2013.How does climate influence speciation.American Naturalist,182: 1-12.( 1) 1)
|
| [55] |
Huang X H,Kurata N,Wei X H,et al. 2012.A map of rice genome variation reveals the origin of cultivated rice.Nature,490: 497-501.( 1) 1)
|
| [56] |
Jaramillo-Correa J P,Beaulieu J,Bousquet J.2004.Variation in mitochondrial DNA reveals multiple distant glacial refugia in black spruce(Picea mariana),a transcontinental North American conifer.Molecular Ecology,13(9): 2735-2747.( 2) 2)
|
| [57] |
Jia D R,Abbott R J,Liu T L,et al. 2012.Out of the Qinghai-Tibet Plateau: evidence for the origin and dispersal of Eurasian temperate plants from a phylogeographic study of Hippophae rhamnoides(Elaeagnaceae).New Phytologist,194: 1123-1133.( 2) 2)
|
| [58] |
Joly C,Visset L.2009.Evolution of vegetation landscapes since the Late Mesolithic on the French West Atlantic coast.Review of Palaeobotany and Palynology,154: 124-179.( 1) 1)
|
| [59] |
Jørgensen T,Haile J,Mller P,et al. 2012.A comparative study of ancient sedimentary DNA,pollen and macrofossils from permafrost sediments of northern Siberia reveals long-term vegetational stability.Molecular Ecology,21(8):1989-2003.( 1) 1)
|
| [60] |
Kikuchi S,Bédard J,Hirano M,et al. 2013.Uncovering the protein translocon at the chloroplast inner envelope membrane.Science,339(6119): 571-574.( 1) 1)
|
| [61] |
Li J H,Liu Z J,Salazar G A,et al. 2011a.Molecular phylogeny of Cypripedium (Orchidaceae: Cypripedioideae)inferred from multiple nuclear and chloroplast regions.Molecular Phylogenetics Evolution, 61: 308-320.( 2) 2)
|
| [62] |
Li D Z,Gao L M,Li H T,et al. 2011b.Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS)should be incorporated into the core barcode for seed plants.Proceedings of the National Academy of Sciences of the United States of America,108: 19641-19646.( 2) 2)
|
| [63] |
Li L F,Häkkinen M,Yuan Y M,et al. 2010.Molecular phylogeny and systematics of the banana family(Musaceae)inferred from multiple nuclear and chloroplast DNA fragments,with a special reference to the genus Musa. Molecular Phylogenetics and Evolution,57(1): 1-10.( 1) 1)
|
| [64] |
Li Y C,Korol A B,Fahima T,et al. 2002.Microsatellites: genomic distribution,putative functions and mutational mechanisms: a review.Molecular Ecology,11(12): 2453-2465.( 1) 1)
|
| [65] |
Liu X L,Wen J,Nie Z L,et al.2013.Polyphyly of the Padus group of Prunus(Rosaceae)and the evolution of biogeographic disjunctions between eastern Asia and eastern North America.Journal of Plant Research,126(3): 351-361.( 1) 1)
|
| [66] |
Lu A M,Stone D E,Grauk L J.1999.Flora of China: Vol.4.Beijing,China: Science Press and Missouri Botanical Garden Press,277-285.( 3) 3)
|
| [67] |
Malvolti M E,Fineschi S,Pigliucci M.1994.Morphological integration and genetic variability in Juglans regia L.Journal of Heredity,85: 389-394.( 1) 1)
|
| [68] |
Manchester S R.1987.The fossil history of the Juglandaceae.Monographs in Systematic Botany,Missouri Botany Garden,21: 1-37.( 8) 8)
|
| [69] |
Manos P S,Soltis P S,Soltis D E,et al. 2007.Phylogeny of extant and fossil Juglandaceae inferred from the integration of molecular and morphological data sets.Systematic Biology,56(3): 412-430.( 1) 1)
|
| [70] |
Manos P S,Stone D E.2001.Evolution,phylogeny,and systematics of the Juglandaceae.Annals of the Missouri Botanical Garden,88(2): 231-269.( 1) 1)
|
| [71] |
Manning W E.1978.The classification within the Juglandaceae.Annals of the Missouri Botanical Garden,65(4):1058-1087.( 8) 8)
|
| [72] |
Mao K,Milne R I,Zhang L,et al. 2012.Distribution of living Cupressaceae reflects the breakup of Pangea.Proceedings of the National Academy of Sciences of the United States of America,109: 7793-7798.( 2) 2)
|
| [73] |
McCleary T S,Robichaud R L,Nuanes S,et al. 2009.Four cleaved amplified polymorphic sequence(CAPS)markers for the detection of the Juglans ailantifolia chloroplast in putatively native J.cinerea populations.Molecular Ecology Resoures,9(2): 525-527.( 1) 1)
|
| [74] |
McKenna M C.1983.Holarctic landmass rearrangement,cosmic events,and Cenozoic terrestrial organisms.Annals of the Missouri Botanical Garden,70(3): 459-489.( 1) 1)
|
| [75] |
Mohsenipoor S,Vahdati K,Amiri R,et al. 2009.Study of the genetic structure and gene flow in Persian walnut(Juglans regia L.)using SSR markers.Acta Hortic,861(To be published).( 1) 1)
|
| [76] |
Orel G,Marchant A,McLeod J,et al. 2003.Characterization of 11 Juglandaceae genotypes based on morphology,cpDNA,and RAPD.HortScience,38(6): 1178-1183.( 1) 1)
|
| [77] |
Polechko M A,Clarkson R B.1986.A serological study of the systematics of the Juglandaceae.Biochemical Systematics and Ecology,14(1): 33-39.( 1) 1)
|
| [78] |
Pollegioni P,Woeste K,Major A,et al. 2009.Characterization of Juglans nigra(L.),Juglans regia(L.)and Juglans ×intermedia(Carr.)by SSR markers: a case study in Italy.Silvae Genetica,58(1/2): 68-78.( 1) 1)
|
| [79] |
Potter D,Gao F,Baggett S,et al. 2002a.Defining the source of pardox: DNA sequecce markers for North American walnut(Juglans L.)species and hybrids.Science Horticultural,94(1/2): 157-170.( 2) 2)
|
| [80] |
Potter D,Gao F,Aiello G,et al. 2002b.Intersimple sequence repeat markers for fingerprinting and determining genetic relationships of walnut(Juglans regia)cultivars.Journal of American Society Horticultural Science,127(1): 75-81.( 1) 1)
|
| [81] |
Rehder A.1940.Manual of cultivated trees and shrubs hardy in North America.2nd revised edition.New York,London: Collier Macmillan Ltd.( 1) 1)
|
| [82] |
Rogers R.2004.Temperate Ecosystems,Juglandaceae.Amsterdam,Holland: Elsevier Ltd,1427-1430.( 1) 1)
|
| [83] |
Ros E,Mataix J.2006.Fatty acid composition of nuts-implications for cardiovascular health.British Journal of Nutrition,96(S2): S29-S35.( 1) 1)
|
| [84] |
Ross-Davis A,Huang Z,Mckenna J,et al. 2008.Morphological and molecular methods to identify butternut(Juglans cinerea)and butternut hybrids: relevance to butternut conservation.Tree Physiology,28:1127-1133.( 1) 1)
|
| [85] |
Schönswetter P,Stehlik I,Holderegger R,et al. 2005.Molecular evidence for glacial refugia of mountain plants in the European Alps.Molecular Ecology,14(11): 3547-3555.( 1) 1)
|
| [86] |
Smith S A,Donoghue M J.2008.Rates of molecular evolution are linked to life history in flowering plants.Science,322(5898): 86-89.( 1) 1)
|
| [87] |
Smith T,Rose K D,Gingerich P D.2006.Rapid Asia-Europe-North America geographic dispersal of earliest Eocene primate Teilhardina during the Paleocene-Eocene thermal maximum.Proceedings of the National Academy of Sciences,103(30): 11223-11227.( 1) 1)
|
| [88] |
Stanford A,Harden M,Parks C R.2000.Phylogeny and biogeography of Juglans (Juglandaceae)based on matK and ITS sequence data.American Journal of Botany,87: 872-882( 12) 12)
|
| [89] |
Taberlet P,Coissac E,Pompanon F,et al. 2007.Power and limitations of the chloroplast trnL(UAA)intron for plant DNA barcoding.Nucleic Acids Research,35(3): e14.( 2) 2)
|
| [90] |
Vinson J A,Cai Y.2012.Nuts,especially walnuts,have both antioxidant quantity and efficacy and exhibit significant potential health benefits.Food Function,(3): 134-140.( 1) 1)
|
| [91] |
Viruel J,Catalán P,Segarra-Moragues G.2013.Disrupted phylogeographical microsatellite and chloroplast DNA patterns indicate a vicariance rather than long-distance dispersal origin for the disjunct distribution of the Chilean endemic Dioscorea biloba(Dioscoreaceae)around the Atacama Desert.Journal of Biogeography,39(6): 1073-1085.( 2) 2)
|
| [92] |
Wang H,Pei D,Gu R,et al. 2008.Genetic diversity and structure of walnut populations in central and southwestern China revealed by microsatellite markers.Journal of American Society Horticultural Science,133(2):197-203.( 2) 2)
|
| [93] |
Wang J F,Gong X,Chiang Y C,et al. 2013.Phylogenetic patterns and disjuct distribution in Liggularia hodgsonii Hook.(Asteraceae).Journal of Biogeography,40: 1741-1754.( 2) 2)
|
| [94] |
Wang W,Ortiz R C,Jacques F M,et al. 2012.Menispermaceae and the diversification of tropical rainforests near the Cretaceous-Paleogene boundary.New Phytologist, 195: 470-478.( 1) 1)
|
| [95] |
Wen J.1999.Evolution of eastern Asian and eastern North American disjunct distributions in flowering plants.Annual Review of Ecology and Systematics,30: 421-455.( 1) 1)
|
| [96] |
Williams J G,Kubelik A R,Livak K J,et al. 1990.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers.Nucleic Acids Research, 18(22): 6531-6535.( 1) 1)
|
| [97] |
Woeste K,Burns R,Rhodes O,et al. 2002.Thirty polymorphic nuclear microsatellite loci from black walnut.Journal of Heredity,93(1): 58-60.( 2) 2)
|
| [98] |
Woeste K,Farlee L,Ostry M,et al. 2009.A forest manager's guide to butternut.North Journal of American Forestry,26(1):9-14.( 1) 1)
|
| [99] |
Wu Z Y.1983.On the significance of Pacific intercontinental discontinuity.Annals of the Missouri Botanical Garden,70: 577-590.( 1) 1)
|
| [100] |
Xiang Q Y,Thomas D T,Xiang Q P.2011.Resolving and dating the phylogeny of Cornales-Effects of taxon sampling,data partitions,and fossil calibrations.Molecular Phylogenetic Evolution, 59: 123-138.( 1) 1)
|
| [101] |
Xu X,Liu X,Ge S,et al. 2012.Resequencing 50 accessions of cultivated and wild rice yields markers for identifying agronomically important genes.Nature Biotechnology, 30: 105-111.( 1) 1)
|
| [102] |
Xu X,Walters C,Antolin M F,et al. 2010.Phylogeny and biogeography of the eastern Asian-North American disjunct wild-rice genus(Zizania L.,Poaceae).Molecular Phylogenetic Evolution,55: 1008-1017( 1) 1)
|
| [103] |
Yang F S,Li Y F,Ding X,et al. 2008.Extensive population expansion of Pedicularis longiflora(Orobanchaceae)on the Qinghai-Tibetan Plateau and its correlation with the Quaternary climate change.Molecular Ecology,17: 5135-5145.( 1) 1)
|
| [104] |
Zeng C X,Zhang Y X,Triplett J K,et al. 2010.Large multi-locus plastid phylogeny of the tribe Arundinarieae(Poaceae: Bambusoideae)reveals ten major lineages and low rate of molecular divergence.Molecular Phylogenetic Evolution, 56(2): 821-839.( 1) 1)
|
| [105] |
Zhang N,Zeng L,Shan H,et al. 2012.Highly conserved low-copy nuclear genes as effective markers for phylogenetic analyses in angiosperms.New Phytologist,195: 923-937.( 1) 1)
|
| [106] |
Zhao P,Woeste K.2011.DNA markers identify hybrids between butternut(Juglans cinerea L.)and Japanese walnut(Juglans ailantifolia Carr.).Tree Genetics & Genomes,7(3): 511-533.( 2) 2)
|
| [107] |
Zhao P,Zhang S X,Woeste K.2012.Molecular identification and genetic analysis of Juglans resources.Saarbrücken,Germany: Lambert Aacdemic Publishing.( 1) 1)
|
| [108] |
Zhao P,Zhao G F,Zhang S X,et al. 2014.RAPD derived markers for separating Manchurian walnut (Juglans mandshurica)and Japanese walnut(J.ailantifolia)from close congeners.Journal of Systematics and Evolution,52(1): 101-111.( 1) 1)
|
| [109] |
Zhou R,Ling S,Zhao W,et al. 2011.Population genetics in nonmodel organisms: Ⅱ.natural selection in marginal habitats revealed by deep sequencing on dual platforms.Molecular Biology and Evolution,28(10): 2833-2842.( 1) 1)
|
 2014, Vol. 50
2014, Vol. 50

