文章信息
- 杨钤, 谢映平, 樊金华, 邵生富, 吴俊, 王彦士, 赵常胜, 张英伟
- Yang Qian, Xie Yingping, Fan Jinhua, Shao Shengfu, Wu Jun, Wang Yanshi, Zhao Changsheng, Zhang Yingwei
- 日本松干蚧3个地理种群的遗传分化
- Genetic Differentiation of Matsucoccus matsumurae from Three Geographic Populations in China
- 林业科学, 2013, 49(12): 88-96
- Scientia Silvae Sinicae, 2013, 49(12): 88-96.
- DOI: 10.11707/j.1001-7488.20131213
-
文章历史
- 收稿日期:2013-01-03
- 修回日期:2013-03-22
-
作者相关文章
2. 浙江省金华市林业局 金华 321000:
3. 山东省青岛市林业局 青岛 266061:
4. 吉林省伊通满族自治县林业局 伊通 130700:
5. 辽宁省抚顺市林业局 抚顺 113006
2. Jinhua Forestry Bureau, Zhejiang Province Jinhua 321000;
3. Qingdao Forestry Bureau, Shandong Province Qingdao 266061;
4. Yitong Forestry Bureau, Jilin Province Yitong 130700;
5. Fushun Forestry Bureau, Liaoning Province Fushun 113006
日本松干蚧(Matsucoccus matsumurae)是我国一种重要的外来毁灭性害虫,在北方主要危害赤松(Pinus densiflora)、油松(P. tabulaeformis)和黑松(P. thunbergii),在南方主要危害马尾松(P.massoniana)。在分类上属于半翅目(Hemiptera)松干蚧科(Matsucoccidae)松干蚧属(Matsucoccus),1903年由Kuwana在日本东京的黑松上首次发现,并作为新种发表,是该属的模式种(Kuwana,1907)。松干蚧自20世纪40年代在我国辽宁旅顺和山东烟台首先发现,之后沿我国东部地区扩散蔓延,并先后在辽宁全境,吉林的伊通县,山东的烟台、青岛,上海,江苏的南京,浙江的杭州、金华等地大面积暴发,对松林造成危害。日本松干蚧在我国1年发生2代,雌虫产卵量大,种群增长快,而且虫体表面有多种泌蜡腺体,能分泌蜡质,对虫体起保护作用,因而防治十分困难。除普遍进行化学防治外,许多地方还大量砍伐染虫林木,以阻止蚧虫的扩散。该虫已被长期列为森林植物检疫对象(林业部野生动物和森林植物保护司,1990)。
长期以来,昆虫分类都以传统的形态分类法为主,由于近缘种的形态结构十分相似,该方法对近缘种种间关系的鉴定有局限性。蚧虫属于小型、变态特殊的昆虫,雌雄异形表现得非常明显,因此蚧虫的分类都是以雌成虫的形态特征作为主要分类依据,这些表型特征主要是虫体表面的泌蜡腺孔的种类、数量、分布位置,身体上的刚毛和刺的形状、数量、分布特点,这使形态结构十分相似的蚧虫近缘种的区分和鉴定更加困难,“张冠李戴”的现象时有发生。
近年来,随着分子生物学的快速发展,利用分子信息进行昆虫分类研究越来越受到重视,其中随机扩增多态性DNA技术(random amplified polymorphicDNA,RAPD)(Welsh et al.,1990;Williams et al.,1990)具有操作简便、灵敏以及对模板DNA浓度、纯度要求不高等特点,已经在昆虫分类和系统学研究中得到运用(鲁亮等,1995)。松干蚧雌成虫大小为2~3 mm,基因组DNA含量较少,并且不同地理种群的基因型差异不大,因此,选用RAPD-PCR方法解决上述问题具有较大优势。
本文采用RAPD方法,对在辽宁省、山东省、浙江省采集的松干蚧标本进行研究,明确了3个地区松干蚧种群内的遗传变异程度和种群间的遗传多样性,可为解决日本松干蚧虫种问题和害虫的管理提供依据。
1 材料与方法 1.1 昆虫根据各地方虫害的发生规律,分别于2011年3—4月、 5—6月、 7—8月对浙江金华(ZJJH)、山东青岛(SDQD)、辽宁抚顺(LNFS)3个地点的松干蚧雌成虫进行采集。ZJJH的标本采集于马尾松上,SDQD和LNFS的标本均采集于油松上。在各地点采集带有显露期松干蚧的松树枝条带回实验室内收集松干蚧雌成虫,将蚧虫饥饿48 h,使其消化道内的物质充分消化,然后浸泡于无水乙醇中(张迎春等,2002),-20 ℃条件下保存,备用。
1.2 DNA提取方法采用优化的酚/氯仿法(张迎春等,2002)提取基因组DNA。每个采集地点各取15头雌成虫,将每头成虫置于蒸馏水中浸泡24 h,烘干,各管中加入300 μL预冷的提取液(Tris 1 mol·L-1,EDTA0.25 mol·L-1,NaCl 1.5 mol·L-1,pH 8.0)。经微量研磨棒研碎后,各加入10 μL的10% SDS以及4μL的RNase A[生工生物工程(上海)有限公司],37 ℃水浴1 h。然后在各离心管中加入6 μL的Proteinase K[生工生物工程(上海)有限公司],37℃水浴2 h。分别加入等体积的Tris平衡酚,抽提20 min,离心10 min(5 000 g)。取上清液,加入等体积的酚:氯仿:异戊醇(25 : 24 : 1),抽提20 min,离心10 min(5 000 g)。取上清液,经2倍体积的无水乙醇沉淀2 h及2倍体积的75%乙醇洗涤、干燥后,加TE缓冲液(10 mmol·L-1 Tris-HCl,1mmol·L-1 Na2EDTA H2O)稀释至50 ng·μL-1,-20 ℃保存,备用。
1.3 RAPD扩增反应条件的优化通过参阅蚧虫分子生物学相关文献(刘全超,2012;Frey et al.,2008;高宝嘉等,2007),并经过预试验筛选获得4条10 bp随机引物用于本研究,引物序号及序列见表 1。引物由生工生物工程(上海)有限公司和北京全式金生物技术有限公司合成。
|
|
RAPD-PCR扩增反应体系中含有: 10×buffer(含Mg2+)2.5 μL,dNTPs(2.5 mmol·L-1)2.0 μL,随机引物(10 μmol· L-1)S500、11N010145和11NO10146为1.5 μL,S1415为1.0 μL,模板DNA(50 ng·μL-1)0.4 μL(S500和11NO10146)、 0.8 μL(11N010145)和1.0 μL(S1415),Taq DNA聚合酶1.0 U(北京全式金生物技术有限公司),补加ddH2 O至25 μL。扩增反应程序为: 94 ℃预变性10 min;94 ℃变性30 s,32 ℃退火30 s,72 ℃延伸1 min,45个循环;72 ℃终延伸7 min。在确定上述反应条件之前,首先依据表 2对影响RAPD-PCR结果较大的3种因素即随机引物浓度、模板DNA浓度以及退火温度进行单因素试验设计,即每次只改变1个因素水平,保持其他因素水平不变,筛选出最优的反应体系组合。
|
|
用经优化的扩增反应条件组合对3个地理种群共45头松干蚧雌成虫的基因组DNA进行RAPDPCR扩增,试验设计3次重复。扩增产物用1.2%琼脂糖凝胶分离,电泳缓冲液为0.5倍TBE,取样品DNA 8 μL加入2 μL上样缓冲液,混匀后点入胶孔。标准分子质量为100 bp DNA Ladder,120 V恒压电泳40 min,电泳结束后,0.5 mg·L-1的溴化乙锭(EB)中染色15 min,凝胶成像系统拍照记录。
1.5 数据分析对于凝胶成像系统记录的RAPD图谱,根据DNA Marker计算不同扩增片段的分子质量大小,同一引物的扩增产物在琼脂糖凝胶电泳中迁移率相同的条带被认为是同源的,属于同一位点的条带,按清晰可见的强带或反复出现的弱带记为1、无记为0,形成二元数据。应用Popgene 32软件计算Shannon's信息指数及群体间遗传距离等。使用NTSYS2.10e计算种群间的遗传相似系数,并根据其相似性系数用UPGMA法进行聚类分析。
2 结果与分析 2.1 基因组DNA提取结果用本研究优化所得方法提取松干蚧雌成虫的基因组DNA,提取的样品经琼脂糖凝胶电泳检测,结果表明(图 1):基因组DNA分子质量约为9 416bp,DNA带型整齐,无扩散或弥散现象,无明显的RNA污染,同时也未见因漩涡操作而留下机械切割痕迹。说明该方法提取效果较好,提取出来的DNA可满足RAPD研究的需要。
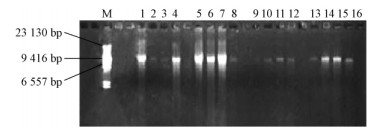 |
图 1 酚/氯仿法提取的基因组DNA的电泳检测
Fig. 1 Electrophoretogram of genomic DNA
extracted with phenol / chloroform method
M: λ-Hind Ⅲ digest DNA marker;
1 - 16: 16 头山东青岛松干蚧雌成虫 Individuals of
pine bast scale female adults from SDQD. |
选用松干蚧雌成虫基因组DNA和4条随机引物为优化材料,根据表 2因素水平设计,分别对随机引物浓度、模板DNA浓度以及退火温度进行优化。图 2为退火温度为32 ℃时4条随机引物的RAPDPCR扩增条带,图 3为退火温度为36 ℃时的扩增条带,退火温度为34 ℃时4条随机引物均未检测出扩增条带。比较图 2和图 3可知,当退火温度为32 ℃时,RAPD-PCR扩增条带更清晰、更稳定。因此,选择松干蚧雌成虫RAPD反应的退火温度为32 ℃。比较图 2中4条随机引物产生的扩增条带,综合考虑结果与成本,对于随机引物S500,S1415,11N010145和11N010146,随机引物和模板DNA的量分别选择0.4和1.5 μL、 1.0和1.0 μL、 0.8和1.5 μL以及0.4和1.5 μL。
 |
图 2 退火温度为32 ℃时4条随机引物扩增效果检测
Fig. 2 RAPD patterns amplified with the 4 random primers in annealing temperature 32 ℃
M: 100 bp DNA ladder;1—5:随机引物为1.0 μL,模板DNA分别为0.2,0.4,0.6,0.8和1.0 μL;6—10:随机引物为1.5 μL,模板DNA分别为0.2,0.4,0.6,0.8和1.0 μL;11—15:随机引物为2.0 μL,模板DNA分别为0.2,0.4,0.6,0.8和1.0 μL。下同。 1-5: 1.0 μL random primers and the volume of genomic DNA are 0.2,0.4,0.6,0.8 and 1.0 μL,respectively;6-10: 1.5 μLrandom primers and the volume of genomic DNA are 0.2,0.4,0.6,0.8 and 1.0 μL,respectively;11-15: 2.0 μL random primersand the volume of genomic DNA are 0.2,0.4,0.6,0.8 and 1.0 μL,respectively. The same below. |
 |
图 3 退火温度为36 ℃时4条随机引物扩增效果检测
Fig. 3 RAPD patterns amplified with 4 random primers in annealing temperature 36 ℃
|
采用4条随机引物分别对ZJJH、 SDQD、 LNFS松干蚧3个地理种群各15头雌成虫的基因组DNA进行RAPD扩增,试验设计3次重复,扩增条带的分子质量均集中在200~2 000 bp(部分扩增图谱见图 4~6)。带型表明松干蚧雌成虫3个地理种群间及种群内不同个体间多态性普遍存在。重复1共检位点比率为100%。
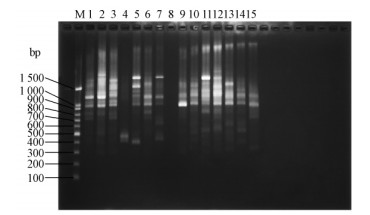 |
图 4 重复1中随机引物11N010145对LNFS15头松干蚧雌成虫基因组DNA的扩增
Fig. 4 RAPD patterns of genomic DNA of 15 individuals of pine bast
scale female adults from LNFS amplified with primer
11N010145 in replicate 1
M: 100 bp DNA ladder; 1—15: LNFS15 头松干蚧雌成虫
15 individuals of pine bast scale female adults from LNFS. |
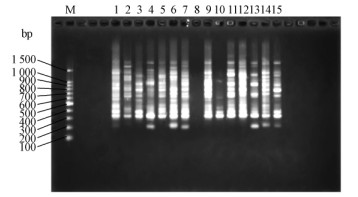 |
图 5 重复2中随机引物S500对ZJJH15头松干蚧雌成虫基因组DNA的扩增
Fig. 5 RAPD patterns of genomic DNA of 15 individuals
of pine bast scale female adults from ZJJH amplified
with primer S500 in replicate 2
M: 100 bp DNA ladder; 1—15: ZJJH15 头松干蚧雌成虫
15 individuals of pine bast scale female adults from ZJJH. |
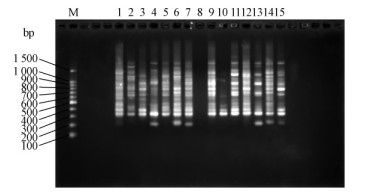 |
图 6 重复3中随机引物S500对ZJJH15头松干蚧雌成虫基因组DNA的扩增
Fig. 6 RAPD patterns of genomic DNA of 15 individuals
of pine bast scale female adults from ZJJH amplified
with primer s500 in replicate 3
M: 100 bp DNA ladder 1 - 15: 浙江金华 15 头松干蚧雌成虫
15 individuals of pine bast scale female adults from ZJJH. |
3次重复的Shannon's信息指数和Nei's基因多样性指数(表 3)显示,群体水平上,重复1、重复2和重复3的有效等位基因数、 Shannon's信息指数和Nei's基因多样性指数遗传多样性最高的为LNFS松干蚧种群,最低的为SDQD松干蚧种群。物种水平上,3次重复的遗传分化系数(Gst)分别为0.207 8,0.191 9和0.207 5,表明总的遗传变异中分别有20.78%,19.19%和20.75%的变异存在于种群间,79.22%,80.81%和79.25%的变异存在于种群内(表 4),此结果与Nei's遗传分化一致。此外,3次重复计算所得的基因流分别为1.906 4,2.105 3和1.909 6(表 4),表明种群之间有一定的基因交流。
|
|
|
|
由3次重复试验的Nei's遗传一致度(表 5上三角值)和遗传距离(表 5下三角值)可以看出:重复1中ZJJH与SDQD的遗传距离为0.040 7,ZJJH与LNFS的遗传距离为0.047 4,SDQD与LNFS的遗传距离为0.065 0;重复2中ZJJH与SDQD的遗传距离为0.035 5,ZJJH与LNFS的遗传距离为0.040 2,SDQD与LNFS的遗传距离为0.056 2;重复3中ZJJH与SDQD的遗传距离为0.044 9,ZJJH与LNFS的遗传距离为0.051 4,SDQD与LNFS的遗传距离为0.067 3。上述结果表明松干蚧3个地理种群间存在微小差异,并且ZJJH种群与SDQD种群有着很近的亲缘关系。
|
|
通过NTSYS2.10e计算个体间遗传相似系数,并根据该系数建立UPGMA聚类分析图(图 7)。3次重复的总体趋势均显示ZJJH与SDQD亲缘关系接近。
 |
图 7 3个地理种群45头雌成虫的UPGMA聚类分析图
Fig. 7 UPGMA dendrograms of 45 individuals of female adults from 3 populations
|
从基于Nei's遗传距离建立的UPGMA聚类分析图(图 8)可以看出: 3次重复所得结果一致,均为ZJJH与SDQD先聚为一簇。这符合遗传距离的趋势,并且与图 7反映的趋势一致。
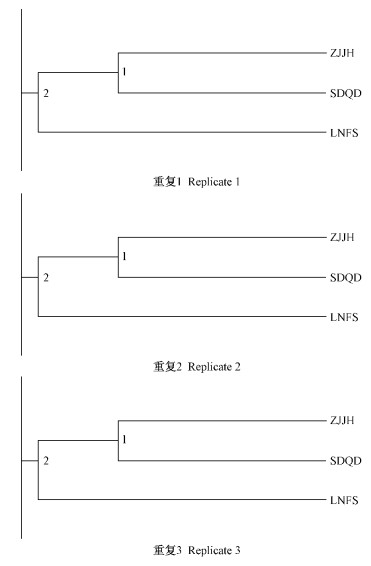 |
图 8 3个地理种群的UPGMA聚类分析
Fig. 8 UPGMA dendrograms of three geographical populations
|
根据化石昆虫资料的研究证明,松干蚧是松树上一类比较古老的昆虫,它们与松树有协同进化的关系,对生态环境的适应性很强,从欧洲、亚洲到美洲均有分布,其中某些种类是造成松林毁灭的害虫(Koteja,1987;1998;2008)。松干蚧在过去的蚧虫分类系统中一直属于古蚧类(Archeococcoidea)的珠蚧科(Margarodidae),但Koteja(1987)根据对蚧虫口器结构的研究已经将其从珠蚧科分离出来,独立为松干蚧科,本科只有一个属,即松干蚧属,日本松干蚧是其模式种,这一结果目前已经被世界蚧虫学界普遍接受(Foldi,2004;Hodgson et al,2006)。在20世纪中叶,随着松干蚧在美国以及欧洲和地中海地区的一些国家发生的加剧,对松干蚧的研究大量增加。我国的松干蚧也是在此期间进入大流行阶段,并被列为森林害虫检疫对象和重点防治害虫;尽管在防治上投入了大量人力、物力和财力,但目前每年仍有大面积的虫害发生。
笔者曾观察记录ZJJH、 SDQD、 LNFS 3个地理种群各30头日本松干蚧雌成虫的形态特征。通过对比发现: 3个地理种群日本松干蚧雌成虫的腹节均为8节,触角均为9节;腺孔均在3 000个以上,且背面分布多于腹面,越靠近腹部末端分布越多,在生殖孔附近密集分布;雌成虫触角上的刚毛数量在ZJJH种群约65根,SDQD种群约64根,LNFS种群约66根;对于雌成虫足部跗节、胫节、腿节、转节和基节上的刚毛数,ZJJH种群依次为10~11,17~24,9 ~21,5~6,8~13根,SDQD种群分别为10,20~24,16~21,5~6,10~13根,LNFS种群分别为7~8,14~15,13~28,10~11,15~20根;雌成虫的背疤都分布于第2~8腹节,并且在背面排列成横行,背疤数量在ZJJH种群约148个,SDQD种群约151个,LNFS种群约153个;雌成虫的爪冠毛都是2根,在ZJJH、 SDQD种群其长度不超过爪端,在LNFS种群爪冠毛稍长于爪。由此可见,ZJJH和SDQD 2个种群雌成虫的形态特征更接近,这与本试验分子生物学研究的结果一致。
杨平澜等(1976)认为当时我国发生的松干蚧是日本松干蚧。此后,汤祊德(1978)把辽宁油松树上的松干蚧定为一个新种即辽宁松干蚧,其主要依据是:辽宁松干蚧成虫“远小”于日本松干蚧;日本松干蚧雌成虫胸足跗冠毛长过爪端,辽宁松干蚧新种的则短,长不达爪端;第1龄若虫的尾毛和体长之比、胸足胫节和跗节长之比不同;雄成虫胸足跗节数;1年发生代数表现为辽宁松干蚧1年2代,日本松干蚧1年1代。随后,杨平澜(1979)对上述5点形态特征的描述进行了反驳,仍主张分布于我国辽、鲁、江、浙的松干蚧均为日本松干蚧。近年来,我国尚无相关方面确定性的报道,这对害虫的科学管理造成困惑;因为利用松干蚧性信息素和其他化学信息等手段进行害虫的监测、预报和防治,都需要准确的物种信息。
近年来,随着分子生物学的发展,为昆虫分类提供了新的技术(De Leon et al.,2010;Hille et al.,2005;Hoy et al.,2000),Booth等(2006)根据分子生物学证据,结合形态学特征,重新评估松干蚧属的北美种和亚洲种,认为分布于美国东部的松干蚧(M. resinosae)和分布于韩国的松干蚧(M. thunbergianae)都是日本松干蚧的同物异名,即它们都是同一物种。这为解决我国松干蚧的分布和物种问题提供了借鉴。目前,应用于昆虫系统演化及分类的分子标记技术主要有限制性片段长度多态性(RFLP)、数目可变串联重复多态性(VNTR)、随机扩增多态性DNA(RAPD)。考虑到日本松干蚧虫体小、基因组DNA含量少、遗传背景不清楚的特点,本研究选择了对模板DNA要求不高的RAPD技术,研究LNFS、 SDQD和ZJJH 3个代表性的地理种群的遗传多样性,可为我国的松干蚧物种问题提供分子生物学证据。
笔者的研究结果表明,3个地理种群的Nei's遗传距离均小于0.1,因此3个地理种群中的松干蚧属于同一虫种,分布于LNFS的松干蚧也应该是日本松干蚧。根据聚类分析,SDQD种群与ZJJH种群在2次重复中都先聚集在一起,说明2种群之间的亲缘关系较近,由此推测ZJJH的日本松干蚧极有可能是从山东青岛扩散蔓延过去的,这一结果与日本松干蚧在20世纪侵入我国初期,同时暴发于山东烟台地区和辽宁旅顺地区的情况相吻合。
松干蚧属共有20多种,除了日本松干蚧之外,还有分布在英国、法国、德国等17个国家的英国松干蚧(M. pini),分布在德国的德国松干蚧(M. mugo),分布在以色列、约旦、地中海东部的克里特岛和塞浦路斯等6个国家和地区的以色列松干蚧(M. josephi),分布在俄罗斯的苏联松干蚧(M. boratynskii),分布在保加利亚、阿尔及利亚、西班牙等9个国家的地中海松干蚧(M. feytaudi)等种类。近年来欧洲一些国家正在研究以色列松干蚧、地中海松干蚧和日本松干蚧的性信息素化学成分,利用性激素对雄成虫进行引诱活性和林间诱捕试验。同时,他们的研究还发现,一些捕食性天敌如花蝽和草蛉等昆虫可以利用松干蚧的性激素作为信号物质,定位和搜寻松干蚧,这在松干蚧的生物防治中具有非常重要的意义(Branco et al.,2004;2006)。而我国在这些方面尚未开展研究,因此,本文的研究结果为我国开展松干蚧生物防治提供了非常重要的基础。
以前关于日本松干蚧DNA提取和遗传多样性研究的报道很少,本研究中酚/氯仿法对提取DNA进行改进,成功地提取了适合RAPD-PCR扩增的日本松干蚧雌成虫基因组DNA。与传统方法相比,该方法直接用微量研磨棒在1.5 mL EP管中对单头雌成虫进行研磨,减少了基因组DNA的损耗。同时,在离心的过程中降低了离心转速,缩短了离心时间,减少了机械力对基因组DNA的破坏。由图 1可看出,由该方法提取的基因组DNA带型整齐,无拖尾、无明显的扩散或弥散现象。此外,有报道指出RAPD技术重复性差,但本次研究中都设有重复,而且3次重复所得结果一致,表明结果具有可信性。
| [1] |
高宝嘉,李梦,王春和,等.2007.皱大球蚧(Eulecanium kuwana)和瘤坚大球蚧(Eulecanium gigantean)地理种群的RAPD遗传分化.生态学报,27(3): 918-923.( 1) 1)
|
| [2] |
林业部野生动物和森林植物保护司.1990.森林植物检疫对象和检疫技术.长春:吉林科学技术出版社,14-18.( 1) 1)
|
| [3] |
刘全超.2012.草履蚧不同寄主和地理种群分化及遗传多样性研究.保定:河北农业大学硕士学位论文.( 1) 1)
|
| [4] |
鲁亮,归鸿.1995.RAPD技术的特点及其在昆虫分类中的应用.昆虫学报,38(1): 117-122.( 1) 1)
|
| [5] |
汤祊德.1978.关于松干蚧的讨论及一新种描记.昆虫学报,21(2): 164-169.( 1) 1)
|
| [6] |
杨平澜.1979.关于我国的日本松干蚧鉴定问题.昆虫学报,22(1): 108-114.( 1) 1)
|
| [7] |
杨平澜,胡金林,仁遵义.1976.中国的松干蚧.昆虫学报,19(2): 199-204.( 1) 1)
|
| [8] |
张迎春,刘波,郑哲民,等.2002.不同保藏处理的昆虫标本DNA提取及其随机扩增多态DNA反应.昆虫学报,45(5): 693-695.( 1) 1)
|
| [9] |
Booth J M,Gullan P J.2006.Synonymy of three pestiferous Matsucoccus scale insects(Hemiptera: Coccoidea: Matsucoccidae)based on morphological and molecular evidence.Proceedings of the Entomological Society of Washington,108(4): 749-760.( 1) 1)
|
| [10] |
Branco M,Jactel H,Silva E B,et al.2004.Effect of trap design,trap size and pheromone dose on male capture of two pine bast scales species(Hemiptera: Matsucoccidae): implications for monitoring and mass-trapping.Agricultural and Forest Entomology,6(3): 233-239.( 1) 1)
|
| [11] |
Branco M,Franco J C,Dunkelblum E,et al.2006.A common mode of attraction of larvae and adults of insect predators to the sex pheromone of their prey(Hemiptera: Matsucoccidae).Bulletin of Entomological Research,96(2): 179-185.( 1) 1)
|
| [12] |
De Leon J H,Setamou M.2010.Molecular evidence suggests that populations of the Asian citrus psyllid parasitoid Tamarixia radiata(Hymenoptera: Eulophidae)from Texas,Florida,and Mexico represent a single species.Annals of the Entomological Society of America,103(1): 100-110.( 1) 1)
|
| [13] |
Foldi I.2004.The Matsucoccidae in the Mediterranean basin,with a world list of species(Hemiptera: Sternorrhyncha: Coccoidea).Annales de la SociétéEntomologique de France(N.S.),40: 145-168.( 1) 1)
|
| [14] |
Frey J E,Frey B.2008.Molecular identification of six species of scale insects(Quadraspidiotus sp.)by RAPD-PCR: assessing the field-specificity of pheromone traps.Molecular Ecology,4(6): 777-780.( 1) 1)
|
| [15] |
Hille A,Miller M A,Erlacher S.2005.DNA sequence variation at the mitochondrial cytochrome oxidase I subunit among pheromotypes of the sibling taxa Diachrysia chrysitis and D.tutti(Lepidoptera: Noctuidae).Zoologica Scripta,34(1): 49-56.( 1) 1)
|
| [16] |
Hodgson C J,Foldi I.2006.A review of the Margarodidae sensu Morrison(Hemiptera: Coccoidea)and some related taxa based on the morphology of adult males.Zootaxa,1263: 1-250.( 1) 1)
|
| [17] |
Hoy M A,Jeyaprakash A,Morakote R,et al.2000.Genomic analyses of two populations of Ageniaspis citricola(Hymenoptera: Encyrtidae)suggest that a cryptic species may exist.Biological Control,17(1): 1-10.( 1) 1)
|
| [18] |
Koteja J.1987.Matsucoccidae(Homoptera,Coccinea),living fossils.Bollettino del Laboratorio di Entomologia Agraria,43: 41-44.( 1) 1)
|
| [19] |
Koteja J.1998.Fossil coccids do exist.Inclusion Wrostek,28: 21.( 1) 1)
|
| [20] |
Koteja J,Azar D.2008.Scale insects from lower cretaceous amber of Lebanon(Hemiptera: Sternorrhyncha: Coccinea).Alavesia,2: 133-167.( 1) 1)
|
| [21] |
Kuwana S I.1907.A new Xylococcus in Japan.Bull Imp Agric Exp Sta,1(2): 209-212.( 1) 1)
|
| [22] |
Welsh J,Mcclelland M.1990.Fingerprinting genomes using PCR with arbitrary primers.Nucleic Acids Research,18(24): 7213-7218.( 1) 1)
|
| [23] |
Williams J G K,Kubelik A R,Livak K J,et al.1990.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers.Nucleic Acids Research,18(22): 6531-6535.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

