文章信息
- 曾艳玲, 谭晓风, 蒋瑶, 刘敏, 王建勇, 周俊琴
- Zeng Yanling, Tan Xiaofeng, Jiang Yao, Liu Min, Wang Jianyong, Zhou Junqin
- 油茶果糖-1, 6-二磷酸醛缩酶基因(CoFBA4)的分子特征与表达分析
- Molecular Characterization and Expression Analysis of Fructose-1, 6-Diphosphate Aldolase Gene (CoFBA4) from Camellia oleifera
- 林业科学, 2013, 49(11): 164-170
- Scientia Silvae Sinicae, 2013, 49(11): 164-170.
- DOI: 10.11707/j.1001-7488.20131123
-
文章历史
- 收稿日期:2013-04-03
- 修回日期:2013-06-29
-
作者相关文章
果糖-1,6-二磷酸醛缩酶(fructose-1,6-diphosphate aldolase,FBA,EC4. 1. 2. 13),简称醛缩酶,是糖酵解代谢途径中第4步关键酶,催化果糖-1,6-二磷酸(Fru-1,6-BP)可逆地裂解为磷酸二羟丙酮(DHAP)和3-磷酸甘油醛(G-3-P)(Rutter,1964)。其中,DHAP经甘油磷酸脱氢酶的催化,还原生成3-磷酸甘油,然后在脂酰转移酶的作用下生成磷脂酸,再经磷脂酸磷酸酶水解,形成油脂合成必需的酯化甘油骨架(Vigeolas et al.,2007);G-3-P则经过多步反应最终形成丙酮酸,产生乙酰辅酶A,经乙酰辅酶A羧化酶作用形成脂肪酸合成的底物丙二酸单酰辅酶A,经过一系列聚合反应形成油脂合成的另一必需元素脂肪酸(Thelen et al.,2002)。
油茶(Camellia oleifera)是我国大力推广的优良食用油料树种,但产油率低是制约油茶产业快速发展的主要原因。为了从根本上提高油茶产油率,近年来关于油茶油脂合成代谢途径调控基因的研究较多(谭晓风等,2008a;2008b;张党权等,2008),但是大部分都集中在脂肪酸方面,对于调控甘油三磷酸的基因研究甚少。油茶果糖-1,6-二磷酸醛缩酶不仅影响脂肪酸的合成,而且为油脂骨架的构建提供原料。因此,本文根据本实验室构建的油茶种仁转录组数字化文库分析,从油茶种仁中分离克隆了CoFBA4的全长cDNA,采用生物信息学方法对其进行了较为系统的分析,并在此基础上开展了亚细胞定位研究及表达差异检测。
1 材料与方法 1.1 材料油茶优良品种‘华硕’(‘HS’)(谭晓风等,2011)采自湖南省长沙市望城县东城镇中南林业科技大学油茶基地,‘华鑫’(‘HX’)、‘华金’(‘HJ’)、‘衡东65’(‘HD65’)、‘衡东17 ’(‘HD17’)、‘茶陵78’(‘CL78’)、‘茶陵二仙268’(‘CLEX268’)、‘茶陵二仙343’(‘CLEX343’)等品种采自株洲马家河油茶基地,采样后迅速存于-80 ℃超低温冰箱备用。本生烟(Nicotiana benthamiana)、载体pEGAD和农杆菌(Agrobacterium tumefaciens)菌株GV3103为湖南大学生物学院生物能源与材料研究中心惠赠,大肠杆菌(Escherichia coli)菌株DH5 本实验室保藏。
本实验室保藏。
TransStartTM FastPfu DNA Polymerase和pEASY-Blunt Simple Cloning Kit购自北京全式金生物技术有限公司,2×Taq PCR MasterMix、质粒提取试剂盒和DNA凝胶回收试剂盒购自天根生化科技(北京)有限公司,RNA提取和反转录试剂盒购自Invitrogen公司,QuantiFast SYBR Green PCR Kit购自QIAGEN公司。
1.3 方法1)总RNA提取与cDNA合成 取油茶近成熟种仁,根据RNA提取和反转录试剂盒说明书提供的方法提取总RNA,电泳检测之后,逆转录成cDNA第1链,用RNaseH消化cDNA产物。
2)全长cDNA克隆 根据本实验室构建的‘华硕’油茶转录组数据库得到的CoFBA4基因序列设计特异引物,分别为CoFBA4F: GTCCTCCTCCTGCCGATTCGCCACT;CoFBA4R: AATCGGCAGGAGGAGGACGATCCAG,由北京华大生物技术有限公司合成。PCR反应体系(50 μL)为5’×Trans Start FastPfu Buffer(Mg2+plus)10.0 μL,2.5 mmol·L-1dNTPs 5.0 μL,cDNA 1.0 μL,Trans Start Fast PfuDNA Polymerase 1.0 μL,CoFBA4F(10 μmol·L-1)1 μL,CoFBA4R(10 μmol·L-1)1 μL,超纯水31.0μL。PCR扩增条件为95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸8 min;4 ℃保温。采用1.2%的琼脂糖凝胶电泳,根据试剂盒说明进行回收连接,转化大肠杆菌DH 5 株,抗生素筛选后提取阳性克隆质粒,委托上海博尚生物技术有限公司测序。
株,抗生素筛选后提取阳性克隆质粒,委托上海博尚生物技术有限公司测序。
3)序列生物信息学分析 利用NCBI的BLAST功能(http:∥www.ncbi.nlm.nih.gov/BLAST)搜索同源的核苷酸序列及氨基酸序列;利用MEGA 4.0(http:∥www.bio-soft.net)程序的UPGMA算法构建FBA蛋白的系统进化树;利用软件AnthePro 5.0(http:∥www.bio-soft.net)进行蛋白跨膜结构预测,http:∥cello.life.nctu.edu.tw在线进行亚细胞定位;http:∥expasy.org/tools/protscale.html在线分析蛋白亲疏水性;https:∥www.predictprotein.org在线分析蛋白二级结构。
4)不同无性系特异表达与出油率检测 取不同无性系油茶种仁提取RNA,逆转录为cDNA,采用实时荧光PCR技术确定CoFBA4基因的相对表达量,每样做3个平行重复检测。PCR引物分别为qCoFBA4F: GAGATTCTTCTTGATGGGGAT;qCoFBA4R: CATAGTGAGCGTGTATTTGGC。PCR反应体系为2×QuantiFast SYBR Green PCR Master Mix12.5 μL,qCoFBA4F(10 μmol·L-1)0.5 μL,qCoFBA4R(10 μmol·L-1)0.5 μL,cDNA 1.0 μL,超纯水9.5 μL。PCR扩增条件为95 ℃预变性5 min;95 ℃变性10 s,55 ℃退火30 s,共40个循环。油脂提取采用索氏提取法(谢鹏等,2010),出油率=(提取油茶种仁的油脂质量/油茶种仁干粉质量)×100%。利用软件SPSS Statistics 17.0(http:∥www.stathome.cn)进行统计学分析。
5)表达载体构建 根据载体pEGAD多克隆位点设计5'端添加EcoRⅠ酶切位点的上游引物CoFBA4ZF: 5'-CCGGAATTCATGGCGTGTTCGAG TT-3';5'端添加Sam Ⅰ酶切位点的下游引物CoFBA4ZR: 5'-TCCCCCGGGTTAGTAGGTATA GCC-3'。以阳性质粒为模板PCR扩增,回收连接至pEASY-Blunt载体上,经测序正确后采用EcoRⅠ和SamⅠ双酶切连接至载体pEGAD多克隆位点,构建包含CoFBA4和eGFP融合植物表达载体。
6)亚细胞定位观察 将融合植物载体转化农杆菌,挑取阳性克隆单菌落接种于3 mL含有利福平(Rif,50 μg·mL-1)和卡那霉素(Kan,100 μg·mL-1)的LB液体培养基中,28 ℃,180 r·min-1培养16~24 h。取1 mL活化后菌液于50 mL同样的LB培养基中,28 ℃,200 r·min-1培养至OD600为0.8~1.0。3 000 r·min-1,离心20 min,收集菌体细胞,用终浓度为10 mmol·L-1 MgCl2、10 mmol·L-1MES(调节pH至5.7)、200 μmol·L-1乙酰丁香酮的无菌水溶液重悬至OD600为1.0。室温放置3 h后,用去掉针头的无菌1 mL针筒浸润生长1个月左右烟草植株中部完全伸长的3个叶片。接种后的烟草植株置于28 ℃、光/暗周期(16 h/8 h)培养36 h,将浸润部位剪成0.5~1.0 cm2小块,叶背面朝上,在激光共聚焦显微镜下观察eGFP的荧光信号,分析CoFBA4蛋白的亚细胞定位。同时,采用基因枪法将重组质粒转化洋葱(Allium cepa)表皮细胞,观察eGFP的荧光信号(Hiei et al.,1994)。
2 结果与分析 2.1 CoFBA4基因全长cDNA克隆根据油茶种子转录组数据设计特异引物,结合RACE技术,获得1 643 bp左右的目的片段,测序结果表明,其中包含193 bp的5’端非编码区、265 bp的3’非编码区和1个1 185 bp的完整开放阅读框,编码394个氨基酸,分子质量为42 740.4 Da,等电点理论值为7.55,即生理中性下该蛋白为碱性。带负电荷残基(Asp+Glu)数为40,带正电荷残基(Arg+Lys)数41,分子式为C1884H2988N524O582S14。估算的半寿期(half-life)分别为: 30 h(哺乳动物网状细胞,体外)、>20 h(酵母,体内)、>10 h(大肠杆菌,体内);稳定指数(Ⅱ)为42.28,属于不稳定蛋白。含有果糖-1,6-二磷酸醛缩酶中的底物特异结合位点: R91,K181,K263(图 1)。将该油茶果糖-1,6-二磷酸醛缩酶基因命名为CoFBA4,GenBank登录号为JX914590。
 |
图 1 CoFBA4基因编码区核酸序列及推导氨基酸序列
Fig. 1 Nucleotide and predicted amino acid sequence of CoFBA4
方框区为底物特异结合位点,下划线部分为果糖二磷酸醛缩酶活性位点。 Specificity binding sites in box,fructose-1,6-diphosphate aldolase activity site with underline. |
自然界中存在2种机制的果糖-1,6-二磷酸醛缩酶: Ⅰ型和Ⅱ型。高等真核生物中主要存在的是Ⅰ型果糖-1,6-二磷酸醛缩酶,特别是动物和高等植物(Schnarrenberger et al.,1990)。根据进化树结果(图 2),来源不同的20条FBA蛋白家族可分为A,B 2个亚族。A亚族支系庞大,既包含藻类也含有草本和林木类FBA,一方面说明该亚族分化较早,另一方面本文所选2种藻类[盐藻(Dunaliellasalina)和衣藻(Chlamydomonas reinhardtii)]均属于A亚族原始分支,说明藻类FBA进化较慢,且A亚族植物FBA可能较B亚族的植物FBA构型或酶活性等方面更接近Ⅱ型醛缩酶;B亚族较A亚族分化晚些,且主要包含植物类FBA,说明该分支可能均为Ⅰ型醛缩酶,与A亚族醛缩酶行使的功能有较大差异。CoFBA4归为A亚族,与拟南芥(Arabidopsis thaliana)遗传距离最近为0.043,与葡萄遗传距离最远为0.677,这说明CoFBA4可能是油茶果糖-1,6-二磷酸醛缩酶基因家族中分化早期的成员之一。
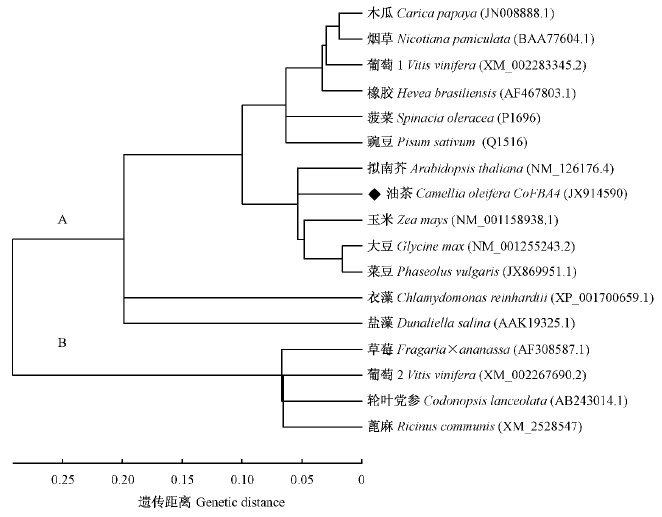 |
图 2 CoFBA4 基因编码蛋白的系统进化树分析
Fig. 2 Phylogenetic tree analysis of CoFBA4 protein
|
采用生物信息学方法分析,CoFBA4推测5—10,38,42—44,54—56,61—69,78—83,85,97,100—102,107—116,139—141,144—151,161—163,166,183—201,204—208,213,216—224,228,235,239—240,242—243,245—248,253—254,257—266,285—287,297—305(最大疏水值位于第301个残基,赋值1.800),315—317,330,332—333,338,340,356—359,361—364,367,369,371—372,390等区段的计119个残基具疏水性,占全部残基总数的30.20%,由此可以推测油茶CoFBA4为亲水性的蛋白。
蛋白质二级结构预测结果显示,CoFBA4蛋白的二级结构中□-螺旋(用H表示)占43.65%,β-折叠(用E表示)占12.18%,无规则卷曲(用L表示)占44.16%。包含了1个果糖二磷酸醛缩酶活性位点,位于第225—265位氨基酸外,还包括N-糖基化位点3个(10—13,72—75,155—158),cAMP和cGMP依赖的蛋白激酶磷酸化位点1个(37—40),蛋白激酶磷酸化位点3个(31—33,36—38,54—56),酪蛋白激酶磷酸化位点5个(46—49,121—124,153—156,309—312,375—378),酪氨酸激酶磷酸化位点1个(241—249),N-豆蔻酰化位点5个(27—32,83—88,176—181,347—352,372—377),酰胺化位点2个(75—78,124—127)。经分析,CoFBA4蛋白不含跨膜区(图 3)。
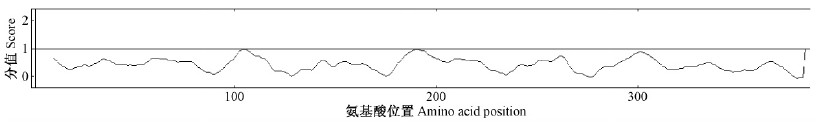 |
图 3 CoFBA4 基因编码蛋白的跨膜结构预测
Fig. 3 Transmembrane domains prediction of CoFBA4 protein
分值大于1.0时说明该区域为跨膜结构,分值小于1.0时说明该区域无跨膜结构。 Region with transmembrane structure if scores>1.0,region without transmembrane structure if scores<1.0. |
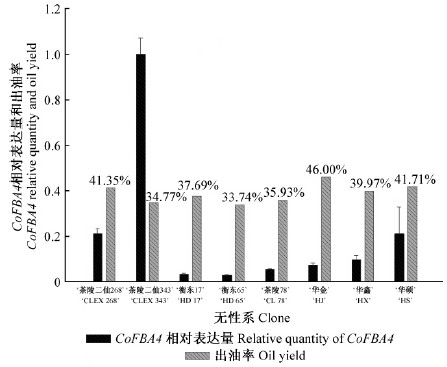
基因表达差异以油茶GAPDH基因作为内参(王保明,2012),采用实时荧光定量PCR的方法对油茶不同无性系10月18日采收的种仁中CoFBA4基因进行相对表达丰度分析。结果(图 4)显示,花期不同的无性系之间比较,存在表达量低出油率高、表达量高出油率低的现象,采用双变量相关性统计分析,结果显示显著性为0.488,说明相关性不强。但是,根据采摘时花期分为3组:花后约365天采摘、花后约350天采摘以及花后约335天采摘。取组员最多的进行统计学分析(表 1),结果显示Pearson相关性为0.967,显著性为0.033,不仅说明二者显著相关,而且表现为花期接近的无性系种仁CoFBA4基因表达丰度与出油率基本呈正相关关系。
|
|
将CoFBA4推导氨基酸序列在线进行亚细胞定位预测,结果显示排在前3位的为外膜、细胞外和周质,可靠性分别为2.100,1.488和0.685。采用基因枪法将带有绿色荧光蛋白基因(eGFP)的目的融合表达载体转化至洋葱表皮细胞,同时通过注射烟草法将融合表达载体转化烟草叶片,获得高效瞬时表达;在激光共聚焦显微镜下观察eGFP荧光信号,从烟草亚细胞定位观察看,CoFBA4可能定位于细胞膜或细胞膜与细胞壁之间,而结合洋葱表皮亚细胞定位,发现CoFBA4融合蛋白主要在细胞壁与细胞膜之间的位置表达,说明CoFBA4可能定位于细胞外(图 5),这与CoFBA4亚细胞定位预测结果基本一致。
 |
图 5 CoFBA4 基因亚细胞定位
Fig. 5 Subcellular localization of CoFBA4 gene
A: 洋葱表皮细胞; B: 烟草。Ⅰ: 紫外激发的荧光信号; Ⅱ: 可见光的信号; Ⅲ: Ⅰ和Ⅱ叠加。 A: Epidermal cells of onion; B: Tobacco. Ⅰ: The fluorescence signal ultraviolet excitation; Ⅱ: Signal light; Ⅲ:Ⅰadd Ⅱ. |
油茶常规育种周期长,且常规杂交育种可能伴有遗传累赘。采用基因工程新方法改良油茶,可实现单一性状或多个性状的定向改良,大大缩短育种周期,而且不会带有遗传累赘。20世纪90年代,我国开始启动油茶分子生物学方面的研究,近年来更是研究的热点,构建了油茶近成熟种子EST文库(谭晓风等,2006)、油茶种仁转录组和表达谱数据库(林萍等,2011;陈英等,2011),挖掘出大量与油茶油脂合成相关基因。糖酵解途径是油脂合成代谢途径的上游代谢途径,该途径各调控酶的作用不仅影响糖酵解的速率和产物而且影响油脂合成的效率,譬如,Focks等(1998)曾得到一个丙酮酸激酶基因缺失的皱叶拟南芥突变体,研究发现其80%的种子表现出随糖酵解活性降低而含油量下降的特性。果糖-1,6-二磷酸醛缩酶是糖酵解代谢途径中枢调控酶,其酶促产物为油脂合成所需脂肪酸和甘油同时提供不可或缺的原料。在植物细胞中,果糖-1,6-二磷酸醛缩酶至少存在2种异构形式,它们有部分同源的基因编码,但结构不同且分别定位于细胞质或叶绿体(Tsutsumi et al.,1994)。本研究亚细胞定位结果显示CoFBA4定位于细胞外,且系统进化分析将其归为支系庞大的A亚族,这说明植物醛缩酶随着生物进化、基因家族成员的增加,各自表达部位和行使功能也出现了更多分化。通常情况下,人们认为分泌蛋白需要借助信号肽的疏水核心穿过内质网(ER)膜,但Hamman等依据多肽转运过程处于亲水环境的现象提出了亲水转位子孔道的转运模式,这种模式中由结合蛋白BiP调控ER膜的选择通透性(刘丹如等,2006),N-糖基化修饰可以使蛋白质打上不同标记,改变多肽构象,在高尔基体上还可形成蛋白聚糖被分泌到细胞外形成细胞外基质或黏液层(周蕾等,2011),这些可能是促成没有跨膜结构的CoFBA4定位于细胞外的原因,但具体是否如此还需进一步证明。
虽然目前人们对植物果糖-1,6-二磷酸醛缩酶基因家族的功能研究主要集中在激素调控、抗逆性等方面(吴耀荣等,2006;Rajjou et al.,2006;Konishi et al.,2004;Osakabe et al.,2005;Jiang et al.,2007),但有研究表明,在烟草体内过量表达拟南芥叶绿体醛缩酶,总生物量明显增加,尤其是在高CO2浓度(1 375 mg·m-3)时,转基因植株生物量达到野生型植株的2.2倍,这说明过量表达醛缩酶能促进CO2的固定(Uematsu et al.,2012),从而促进糖类代谢和脂类代谢。本研究结果也显示,相近花期的油茶种仁CoFBA4基因表达丰度与出油率基本呈显著正相关关系,理论上初步证明通过分子设计育种方法改良油茶果糖-1,6-二磷酸醛缩酶的表达可以提高油茶产量或单位面积出油率。
| [1] |
陈 英,江香梅,张 露,等. 2011. 基于油茶组59 万条EST 序列的转录组学初步分析. 林业科学,47(2): 161-163.( 1) 1)
|
| [2] |
林 萍,曹永庆,姚小华,等. 2011. 普通油茶种子4 个发育时期的转录组分析. 分子植物育种,9(4): 498-505.( 1) 1)
|
| [3] |
刘丹如,宋长征,张更林. 2006. 植物表达分泌蛋白的运输及定位. 生物技术通报, 17(3): 425-428.( 1) 1)
|
| [4] |
谭晓风,胡芳名,谢禄山,等. 2006. 油茶种子EST 文库构建及主要表达基因的分析.林业科学,42(1): 43-48.( 1) 1)
|
| [5] |
谭晓风,陈鸿鹏,张党权,等. 2008a. 油茶FAD2基因全长cDNA的克隆和序列分析. 林业科学,44(3): 70-75.( 1) 1)
|
| [6] |
谭晓风,王威浩,刘卓明,等. 2008b. 油茶ACP基因的全长cDNA克隆及序列分析. 中南林业科技大学学报, 28(4): 8-14.( 1) 1)
|
| [7] |
谭晓风,袁德义,袁 军,等. 2011. 大果油茶良种'华硕'.林业科学,47(12): 184.( 1) 1)
|
| [8] |
王保明. 2012. 油茶ACCase基因的克隆及功能研究. 长沙: 中南林业科技大学博士学位论文.( 1) 1)
|
| [9] |
吴耀荣,谢 旗. 2006. ABA与植物胁迫抗性. 植物学通报, 23(5): 511-518.( 1) 1)
|
| [10] |
谢 鹏, 谭晓风, 王 渊,等. 2010. DTOPSIS法在油茶优良无性系综合评价中的应用研究. 广东农业科学,(6): 10-11.( 2) 2)
|
| [11] |
张党权,谭晓风,陈鸿鹏,等. 2008. 油茶SAD 基因的全长cDNA 克隆及生物信息学分析等. 林业科学,44 (2): 155-159.( 1) 1)
|
| [12] |
周 蕾,顾 建. 2011. N-糖基化位点鉴定方法和非经典N-糖基化序列. 生命科学, 23 (6): 605-611.( 1) 1)
|
| [13] |
Focks N,Benning C. 1998. Low-seed-oil mutant of Arabidopsis with a deficiency in the seed-specific regulation of carbohydrate metabolism. Plant Physiol, 118(1): 91-101.( 1) 1)
|
| [14] |
Hiei Y,Ohta S,Komari T,et al. 1994. Efficient transformation of rice (Oryza sativa L.) mediated by Agrobacterium and sequence analysis of the boundaries of the T-DNA. Plant Journal,6(2): 271-282.( 1) 1)
|
| [15] |
Jiang Y,Yang B,Harris N S,et al. 2007. Comparative proteomic analysis of NaCl stress-responsive proteins in Arabidopsis roots. Journal of Experimental Botany,58(17): 3591-3607.( 1) 1)
|
| [16] |
Konishi H,Yamane H,Maeshima M,et al. 2004. Characterization of fructose-bisphosphate aldolase regulated by gibberellin in roots of rice seedling. Plant Molecular Reporter,56(6): 839-848.( 1) 1)
|
| [17] |
Osakabe Y,Maruyama K,Seki M,et al. 2005. Leucine-rich repeat receptor-like kinase1 is a key membrane-bound regulator of abscisic acid early signaling in Arabidopsis. Plant Cell, 17(4): 1105-1119.( 1) 1)
|
| [18] |
Rajjou L,Belghazi M,Huguet R,et al. 2006. Proteomic investigation of the effect of salicylic acid on Arabidopsis seed germination and establishment of early defense mechanisms. Plant Physiol, 141(3): 910-923.( 1) 1)
|
| [19] |
Rutter W J. 1964. Evolution of aldolase. Federation Proceedings, 23: 1248-1257.( 1) 1)
|
| [20] |
Schnarrenberger C,Jacobshagen S,Muller B,et al. 1990. Evolution of isozymes of sugar phosphate metabolism in green algae. Progress in Clinical and Biological Research,344: 743-764.( 1) 1)
|
| [21] |
Thelen J J,Ohlrogge J B. 2002. Metabolic engineering of fatty acid biosynthsis in plants. Metabolic Engineering,4(1): 12-21.( 1) 1)
|
| [22] |
Tsutsumi K,Kagaya Y,Hidaka S,et al. 1994. Structural analysis of the chloroplastic and cytoplasmic aldolase-encoding genes implicated the occurrence of multiple loci in rice. Gene, 141(2): 215-220.( 1) 1)
|
| [23] |
Uematsu K,Suzuki N,Iwamae T,et al. 2012. Increased fructose 1, 6-bisphosphate aldolase in plastids enhances growth and photosynthesis of tobacco plants. Journal of Experimental Botany,63 (8): 3001-3009.( 1) 1)
|
| [24] |
Vigeolas H,Waldeck P,Zank T, et al. 2007. Increasing seed oil content in oil-seed rape (Brassica napus L.) by over-expression of a yeast glycerol-3-phosphate dehydrogenase under the control of a seed-specific promoter. Plant Biotechnology Journal,5(3): 431-441.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49