文章信息
- 牛苏燕, 范国强, 赵振利, 邓敏捷, 董焱鹏
- Niu Suyan, Fan Guoqiang, Zhao Zhenli, Deng Minjie, Dong Yanpeng
- 泡桐的microRNAs及其功能预测
- MicroRNAs of Paulownia Plants and Their Functional Prediction
- 林业科学, 2013, 49(11): 77-82
- Scientia Silvae Sinicae, 2013, 49(11): 77-82.
- DOI: 10.11707/j.1001-7488.20131110
-
文章历史
- 收稿日期:2013-05-13
- 修回日期:2013-09-12
-
作者相关文章
MicroRNA(miRNA)是一类在植物中广泛存在、长度约21 nt的非编码的单链小 RNAs,在植物的生长发育和逆境胁迫中发挥着重要作用。自miRNA 在秀丽隐杆线虫(Caenorhabditis elegans)(Lee et al.,1993)中被发现以来,拟南芥(Arabidopsis thaliana)(Sunkar et al.,2004)、水稻(Oryza sativa)(Sunkar et al.,2005)、小麦(Triticum aestivum)(Yao et al.,2007)、玉米(Zea may)(Erica et al.,2006)、地黄(Rehmannia glutinosa)(Yang et al.,2011)、杨树(Populus)(Chen et al.,2012)、鹅掌楸(Liriodendron chinense)(Wang et al.,2012)和杉木(Cunninghamia lanceolata)(Wan et al.,2012)等多种植物的 miRNA 也相继公布于世。目前,在传统克隆法基础上发展起来的小分子 RNA(sRNA)深度测序技术,不仅可以一次性获得大量的 sRNA 序列,而且还能够快速、全面地发掘该物种的 miRNA(Morin et al.,2008 ; Cummins et al.,2006 ; He et al.,2004 ; Xue et al.,2008),为研究 miRNA 功能提供了强有力的工具,故在植物 miRNA 研究中得到广泛应用(Glazov et al.,2008 ; Moxon et al.,2008 ; Wei et al.,2009)。泡桐(Paulownia)为玄参科(Scrophulariaceae)多年生落叶乔木,是中国重要的速生用材和庭院绿化树种之一。由于其具有速生、材质优良和用途广泛等特性,深受广大农民的喜爱。近年来,科技工作者虽然开展了泡桐分子生物学研究(范国强等,2001 ; 2011 ; 2012; 黎明等,2008),但至目前国内外还未见有关泡桐 miRNAs 的文献报道。本研究在构建泡桐 sRNA 文库和高通量测序的基础上,结合生物信息学方法,进行泡桐 miRNA 分析及其靶基因预测工作,以期为阐明 miRNA 在泡桐生长发育过程中的生物学功能和开展泡桐分子育种研究奠定基础。
1 材料与方法 1.1 试验材料以河南农业大学泡桐研究所林木生物技术实验室培育的由体细胞胚胎发生途径培养的白花泡桐(Paulownia fortunei)、豫杂一号泡桐(P. tomentosa ×P. fortunei)、毛泡桐(P. tomentosa)、南方泡桐(P.australis)二倍体及其对应四倍体培养30天的组培苗为试验材料。组培苗培养温度为(25 ± 2)℃,光照强度为130 μmol·m-2s-1,光照时间为每天16 h。收取等量上述泡桐顶芽,用液氮速冻后置于 -80 ℃冰箱内保存备用。
1.2 泡桐 RNA 的提取将收集到的泡桐顶芽混合均匀,参照 Trizol 试剂(Invitrogen)说明书提取总 RNA,通过 Agilent2001 BioAnalyzer 检测 RNA 的完整性,RIN≥8 。28 S和18 S 的 RNA 的比值≥1.5 :1,用 Qubit RNA AssayKit 对起始的总 RNA 进行准确定量,并检测 RNA 的纯度,A260 /280 ≥1.8 。
1.3 sRNA 文库的构建与测序首先取总 RNA 4 μg 连接3 '和5 ' RNA 接头,再进行逆转录反应,将带有接头的 sRNA 片段反转录成 cDNA,然后进行12个循环的 PCR 扩增。扩增产物 PAGE 胶电泳后,回收片段长度为145~160 bp 条带构建 pair-end 文库,最后利用 Illumina 技术进行 sRNA 测序。
1.4 测序数据分析Illumina 测序所得的原始 reads,去除接头、低质量和污染序列以及小于18 nt 的 reads 后,得到高质other ncRNA,不含 miRNA)进行比对,去除完全比对到该数据库上的 unique reads,剩余 reads 用于预测新 miRNA。
1.5 新 miRNA 靶基因预测为更好了解泡桐新 miRNAs 的功能,用 SangermiRbase(Griffiths-Jones et al.,2008)预测靶基因方法,将用 Patscan(Dsouza et al.,1997)鉴定的 miRNA序列与泡桐 unigenes 序列(邓敏捷等,2013)进行比对(错配碱基数小于3个、0缺失、0插入),筛选新miRNA 的靶基因,并分析其功能。
2 结果与分析 2.1 泡桐 sRNAs 的深度测序用 Illumina 测序技术对构建的泡桐 sRNA 文库进行测序,共获得108 054 879条原始 reads。去除接头,低质量、污染以及片段长度小于18 nt 的序列后,共得到87 066 707条长度大于18 nt 的高质量 reads(表 1)。其中,与泡桐转录组完全匹配的有41 399 208条,占高质量序列的47.55 % 。泡桐 sRNA 序列长度分布比例差异很大,主要集中在20~25 nt 之间。其中,长度24 nt 的数量最多、高达23 199 407条,占高质量 reads 的26.65 %,这与多数被子植物的研究结果一致(Lin et al.,2013 ; Zhao et al.,2010 ; Song et al.,2010); 其余依次为21 nt(12 439 402 ; 14.29 %)、 23 nt(8 910 902 ;10.23 %)和22 nt(6 584 867 ; 7.56 %)(图 1)。种类数最多的是24 nt(6 834 125 ; 47.76 %); 其次是23 nt(2 304 920 ; 16.11 %),25 nt(951 335 ;6.65 %)和21 nt(929 676 ; 6.50 %)分别位于第3和第4位。植物中存在不同长度 sRNAs 的原因可能是其功能与靶基因调控方式具有对应关系(Wu et al.,2010)。
|
|
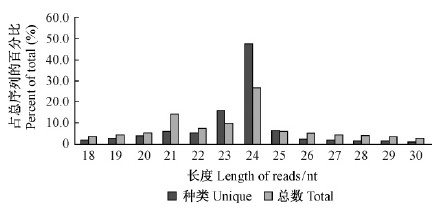 |
图 1 高质量 reads 长度分布
Fig. 1 Length distribution of high-quality reads
|
由 sRNAs 类别和注释结果(表 2 、图 2)看出,用Patscan 软件将获得的特异 sRNA 比对到已知miRNAs 上的序列(保守 miRNAs)只有641种、6 061 631条,非编码的特异 sRNA(rRNA,snRNA,snoRNA,tRNA和other ncRNA)共有34 022种(占总种类的0.24 %)、9 920 372条(占总数量的11.99 %),而功能未被注释的特异 sRNA 序列有14 276 076种(占总种类的99.76 %)、70 566 280条(占总数量的81.05 %)。将 sRNAs 库中完全匹配到泡桐转录组的序列与 miRBase 数据库(Release19.0,August 2012)成熟的 miRNAs 序列同源性比对,鉴定出来自14个 miRNAs 家族的44个保守miRNAs。其中,miR858家族仅有1个 miRNA 成员,其余 miRNAs 家族都有多个 miRNA 成员,如miR396家族有4个成员,miR156家族有6个成员。此外,保守 miRNA 在泡桐中的表达丰度也存在差异,不仅不同家族的 miRNA 表达丰度不同,而且即使同一家族中不同 miRNA 成员的表达丰度也存在差异。如 miR166家族的表达丰度高达2 745 892,而 miR167家族的表达丰度只有282 ; 在 miR398家族中,不同成员的表达丰度为45~183 245 。miR167家族不同成员的表达丰度为3~248 。其他miRNA 家族中也有类似情况出现。
|
|
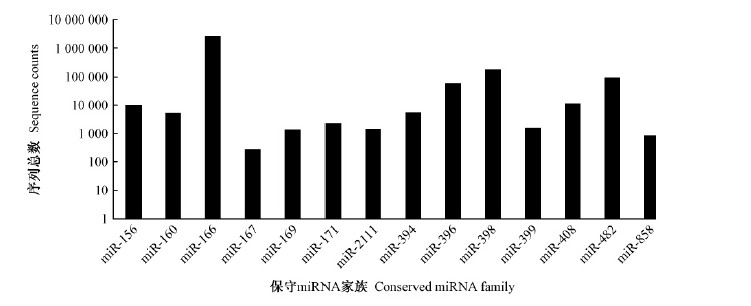 |
图 2 泡桐保守 miRNA 的表达丰度
Fig. 2 Abundance of conserved miRNAs in Paulownia plants
|
利用 Patscan 软件对泡桐 sRNA 文库中未注释序列进行比对后,发现27个候选的新 miRNAs(用BLASTN 在 miRBase 19.0数据库中则未找到)(表 3),这些新 miRNA 长度分布在18~24 nt 之间,但以21 nt 为主。 Mfold 软件分析结果显示,候选新miRNA 前体的最小折叠自由能在 -153.13~ -679.90 kJ·mol-1之间,平均为 -308.15 kJ ·mol-1,比拟南芥和小麦等植物 miRNA 前体的平均自由能低(Yao et al.,2007)。此外,这些不同新miRNA 表达丰度也存在着差异。其中,pau-m0001表达丰度最高,而 pau-m00012 * 表达丰度最低。
|
|
因植物 miRNAs 与其靶基因具有高度互补配对的特性(Sunkar et al.,2005 ; Jong-Rhoades et al.,2004),本研究利用泡桐转录组数据库和生物信息学分析方法预测到泡桐27个新 miRNAs 的156个靶基因。其中,大部分 miRNAs 预测到了靶基因,并且部分 miRNAs 有多个靶基因(但有些靶基因功能未知),而只有少数 miRNA 没有预测到靶基因。这些属于不同基因家族的靶基因主要编码与泡桐生长发育、胁迫响应、信号转导、转录调节、跨膜运输和DNA 加工等相关的蛋白质(编码功能未知蛋白质的靶基因未列出)(表 4)。此外,miRNAs 调控靶基因的数量或靶基因活性受制于 miRNA 的数量也不尽相同。例如,多酚氧化酶基因受到多个miRNA 家族(pau-m0001,pau-m0003,pau-m0004)的调控,而 pau-m0004又可同时调控多酚氧化酶Ⅱ和质膜型钙调转运蛋白酶等多个基因。
|
|
利用生物信息学开展 miRNA 及其靶基因功能预测工作的基础是不同物种间 miRNA 序列的高度保守性。正是由于 miRNAs 具有高度的保守性、时序性和组织特异性,故即使同一物种的不同器官或组织、不同生长条件皆会对其miRNA 的表达产生显著的影响。为了更全面地获得泡桐的 miRNA 并进行靶基因功能预测,本文利用8种(品种)泡桐叶片混合样品为材料提取 RNA,构建了泡桐小 RNAs 文库,获得的 sRNAs 总数在10 805万以上,共得到片段长度大于18 nt 的高质量 reads 达到87 066 707条,与泡桐转录组完全匹配的有41 399 208条,占高质量 reads 的47.55 %,说明本次泡桐 sRNA 的测序质量和组装效果是值得信赖的。并且,构建的sRNA 文库准确性高、覆盖度广、信息全面,这必将为以后研究miRNAs 在泡桐生长发育过程中的作用提供数据支撑。虽然利用生物信息学方法得到泡桐44个保守 miRNAs和27个新 miRNAs 中的大部分功能在现有数据库中没有注释,但这也意味着泡桐生长发育过程中存在大量功能未知的新 miRNA 需要挖掘和研究,从而使泡桐基因组数据库的信息得到丰富,为深入开展泡桐分子生物学研究奠定坚实基础。此外,本试验中鉴定出的大部分miRNA 在泡桐中的表达丰度较低,仅有 miR166家族和miR398家族等少数 miRNA 表达量较高,这种情况在 Song等(2010)和Qiu等(2009)结果中也出现过; 而在小麦和水稻等植物中低表达的 miR156家族(Yao et al.,2007)中的 miRNAs 却在泡桐中大量表达,该结果与柑橘(Citrus trifoliata)(Song et al.,2010)、红豆杉(Taxus chinensis)(Qiu et al.,2009)和毛白杨(Populus tomentosa)(Chen et al.,2012)等研究结果一致。造成该结果的原因,一方面可能与木本植物和草本植物间的遗传差异有关,另一方面也可能与 miR156家族、miR166家族和miR398家族的 miRNAs 在不同类植物中的调控机制存在差异有一定联系。
在 miRNA 靶基因功能预测方面,本试验从27个新 miRNAs 中预测到156个靶基因。其中,pau-m0007靶定多个靶基因,如 F-box 蛋白 CPR30 、E3泛素蛋白连接酶 BRE1 -like 1和生长素诱导蛋白5 NG4 。研究表明,F-box 蛋白质是 SCF 复合体的一个亚基,通过参与 SCF 复合体的形成介导了泛素化蛋白底物的特异性识别,在其降解过程中发挥关键作用(杨娜等,2008)。拟南芥和金鱼草(Antirrhinum majus)中的 F-box 蛋白质,参与了生长素信号转导、开花、叶片衰老和逆境胁迫响应等多种生理过程(秘彩莉等,2006 ; 许振华等,2010)。本试验中,泡桐的 F-box 蛋白 CPR30和E3泛素蛋白连接酶 BRE1 -like 1很可能也参与了蛋白质的降解,提高了泡桐的抗逆能力。此外,生长素诱导蛋白5 NG4可能参与了泡桐根系形成,扩大了泡桐的分布区域(蒋建平,1990),并在泡桐生长发育中发挥重要作用(Busov et al.,2004)。 Pau-m0008的靶基因β连环蛋白样蛋白参与细胞增殖,可能与泡桐速生特性有一定关联。 Pau-m0001的靶基因丝氨酸 /苏氨酸蛋白激酶是一类蛋白质可逆磷酸化过程的关键酶,参与调节防御反应机制信号识别与信号的传导途径,其功能可能与泡桐对逆境的响应程度有关(罗茂,2011)。总之,pau-m0001,pau-m0007和pau-m0008通过参与一系列生理生化代谢活动调控泡桐的生长发育进程,至于相关 miRNA 靶基因鉴定及其功能验证等工作将在泡桐基因组测序结束后作进一步研究。
| [1] |
邓敏捷,董焱鹏,赵振利,等. 2013. 基于Illumina高通量测序的泡桐转录组研究. 林业科学,49(6): 30-36.( 1) 1)
|
| [2] |
范国强,彭海风,翟晓巧,等. 2001. 泡桐叶片蛋白质多态性及其聚类分析. 植物学通报, 18(6):739-743.( 1) 1)
|
| [3] |
范国强,赵改丽,翟晓巧,等. 2011. 硫酸二甲酯处理豫杂一号泡桐丛枝病幼苗形态变化及其SSR分析. 东北林业大学学报,9(5):30-33.( 1) 1)
|
| [4] |
范国强,赵改丽,翟晓巧,等. 2012. 硫酸二甲酯对毛泡桐丛枝病幼苗植原体及SSR扩增位点的影响. 南京林业大学学报: 自然科学版, 36(3):5-8.( 1) 1)
|
| [5] |
蒋建平. 1990. 泡桐栽培学. 北京: 中国林业出版社.( 1) 1)
|
| [6] |
黎明,翟晓巧,范国强,等. 2008. 土霉素对豫杂一号泡桐丛枝病幼苗形态和DNA甲基化水平的影响. 林业科学,44(9):152-156.( 1) 1)
|
| [7] |
罗茂. 2011. 玉米抗纹枯病相关miRNA鉴定及功能分析. 雅安: 四川农业大学博士学位论文.( 1) 1)
|
| [8] |
秘彩莉,刘旭,张学勇,等. 2006. F-box蛋白质在植物生长发育中的功能. 遗传, 28(10): 1337-1342.( 1) 1)
|
| [9] |
许振华,谢传晓. 2010. 植物microRNA与逆境响应研究进展. 遗传, 32(10): 1018-1030.( 1) 1)
|
| [10] |
杨娜,侯巧明,南洁,等. 2008. 泛素连接酶的结构与功能进展. 生物化学与生物物理进展, 35(1): 14-20.( 1) 1)
|
| [11] |
Busov V B, Johannes E, Whetten R W, et al. 2004. An auxin-inducible gene from loblolly pine (Pinus taeda L.) is differentially expressed in mature and juvenile-phase shoots and encodes a putative transmembrane protein. Planta, 218(6): 916-927.( 1) 1)
|
| [12] |
Chen L, Ren Y Y, Zhang Y Y, et al. 2012. Genome-wide identification and expression analysis of heat-responsive and novel microRNAs in Populus tomentosa. Gene, 504:160-165.( 2) 2)
|
| [13] |
Cummins J M, He Y, Leafy R J, et al. 2006. The colorectal microRNAome. Proceedings of the National Academy of Sciences of the United States of America, 103 (10): 3687-3692. ( 1) 1)
|
| [14] |
Dsouza M, Larsen N, Overbeek R. 1997. Searching for patterns in genomic data. Trends in Genetics: TIG, 13(12): 497-498.( 1) 1)
|
| [15] |
Erica M, Luca G, Mario E P. 2006. Characterization of five microRNA families in maize. J Exp Bot, 57(11): 2601-2612.( 1) 1)
|
| [16] |
Glazov E A, Cotte P A, Barris W C, et al. 2008. A microRNA catalog of the developing chicken embryo identified by a deep sequencing approach. Genome Research, 18(6): 957-964.( 1) 1)
|
| [17] |
Griffiths-Jones S, Saini H K, van Dongen S, et al. 2008. miRBase: tools for microRNA genomics. Nucleic Acids Research, 36(suppl 1): D154-D158. ( 1) 1)
|
| [18] |
He L, Harmon G J. 2004. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics, 5(7):522-531.( 1) 1)
|
| [19] |
Jong-Rhoades M W, Bartel P. 2004. Computational identification of plant microRNAs and their targets, including a stress induced miRNA. Mol Cell, 14(6): 787-799.( 1) 1)
|
| [20] |
Lee R C, Feinbaum R L, Ambros V. 1993. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell, 75(5): 843-854.( 1) 1)
|
| [21] | Li R, Li Y, Kristiansen K, et al. 2008. SOAP: short oligonucleotide alignment program. Bioinformatics, 24(5): 713-714. |
| [22] |
Lin Y L, Lai Z X. 2013.Comparative analysis reveals dynamic changes in miRNAs and their targets and expression during somatic embryogenesis in longan (Dimocarpus longan Lour.). PloS one, 8(4): e60337.( 1) 1)
|
| [23] |
Morin R D, Connor M D, Griflith M, et al. 2008. Application of massively parallel sequencing to microRNA profiling and discovery in human embryonic stem cells. Genome Research, 18(4):610-621.( 1) 1)
|
| [24] |
Moxon S, Jing R, Szittya G, et al. 2008. Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening. Genome Research, 18(10):1602-1609.( 1) 1)
|
| [25] |
Qiu D, Pan X, Wilson I W, et al. 2009. High throughput sequencing technology reveals that the taxoid elicitor methyljasmonate regulates microRNA expression in Chinese yew (Taxus chinensis). Gene, 436(1): 37-44.( 2) 2)
|
| [26] |
Song C N, Wang C, Zhang C Q, et al. 2010. Deep sequencing discovery of novel and conserved microRNAs in trifoliate orange (Citrus trifoliata). BMC Genomies, 11(1):431.( 3) 3)
|
| [27] |
Sunkar R, Zhu J K. 2004. Novel and stress-regulated microRNAs and other small RNAs from Arabidopsis. Science Signaling, 16(8): 2001-2019.( 1) 1)
|
| [28] |
Sunkar R, Girke T, Jain P K, et al. 2005. Cloning and characterization of microRNAs from rice. Plant Cell, 17(5):1397-1411.( 2) 2)
|
| [29] |
Wan L C, Wang F, Guo X Q, et al. 2012. Identification and characterization of small non-coding RNAs from Chinese fir by high throughput sequencing. BMC Plant Biology, 12(1):146.( 1) 1)
|
| [30] |
Wang K, Li M, Gao F, et al. 2012. Identification of Conserved and Novel microRNAs from Liriodendron chinense floral tissues. PLoS one, 7(9):e44696. ( 1) 1)
|
| [31] |
Wei B, Cai T, Zhang R Z, et al. 2009. Novel microRNAs uncovered by deep sequencing of small RNA transcriptomes in bread wheat (Triticum aestivum L.) and Brachypodium distachyon (L.) Beauv. Funct Integr Genomics, 9(4): 499-511.( 1) 1)
|
| [32] |
Wu L, Zhou H Y, Zhang Q Q, et al. 2010. DNA methylation mediated by a microRNA pathway. Molecular Cell, 38(3): 465-475.( 1) 1)
|
| [33] |
Xue X, Sun J, Zhang Q, et al. 2008. Identification and characterization of novel microRNAs from Schistosoma japonicum. PLoS one, 3(12):e4034.( 1) 1)
|
| [34] |
Yang Y H, Chen X J, Chen J Y, et al. 2011. Identification of novel and conserved microRNAs in Rehmannia glutinosa L. by solexa sequencing. Plant Mol Biol Rep, 29(4):986-996.( 1) 1)
|
| [35] |
Yao Y Y, Guo G G, Ni Z F, et al. 2007. Cloning and characterization of microRNAs from wheat (Triticum aestivum L.). Genome Biol, 8(6): R96.( 3) 3)
|
| [36] |
Zhao C Z, Xia H, Fazier T P, et al. 2010. Deep sequencing identifies novel and conserved microRNAs in peanuts (Arachis hypogaea L.). BMC Plant Biology, 10 (1):3.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

