文章信息
- 张圣, 黄华宏, 林二培, 童再康
- Zhang Sheng, Huang Huahong, Lin Erpei, Tong Zaikang
- 杉木与台湾杉EST-SSR标记的开发与应用
- Development and Application of EST-SSR Markers for Cunninghamia lanceolata and Taiwania cryptomerioides
- 林业科学, 2013, 49(10): 173-180
- Scientia Silvae Sinicae, 2013, 49(10): 173-180.
- DOI: 10.11707/j.1001-7488.20131027
-
文章历史
- 收稿日期:2013-01-07
- 修回日期:2013-05-11
-
作者相关文章
简单序列重复(Simple Sequence Repeats,SSR)标记是基于SSR中核苷酸单位串联重复次数不同而开发的一种分子标记技术,具有多态性丰富、呈共显性遗传、易于PCR检测,在基因组上分布均匀等特点(Powell et al.,1996)。直接利用现有的EST(Expressed Sequence Tag)序列开发SSR标记就是目前采用较多的一种策略。
EST-SSR标记反映的是基因编码部分,可以直接获得基因表达的信息,为功能基因提供“绝对”的标记,这有可能对决定重要表型性状的等位基因进行直接鉴定(Chen et al.,2001)。EST-SSR标记应用广泛,目前已在多种植物上得到应用,如甘蔗(Saccharum officinarum)(Cordeiro et al.,2001)、小麦(Triticum aestivum)(Eujayl et al.,2001;2002)、大麦(Hordeum vulgare)(Kota et al.,2001;Thiel et al.,2003)、玉米(Zea mays)、高粱属(Sorghum)、水稻(Oryza sativa)(Kantety et al.,2002)、葡萄(Vitisvinifera)(Scott et al.,2000)、大豆(Glycine max)(Song et al.,2004)、火炬松(Pinus taeda)(Liewlaksaneeyanawin et al.,2004)、云杉(Piceaasperata)(Rungis et al.,2004)、白桦(Betulaplatyphylla)(王艳敏等,2008)、核桃属(Juglans)(朱艳等,2009;齐建勋等,2011)、茶树(Camelliasinensis)(周炎花等,2011)、荔枝(Litchi chinensis)(孙清明等,2011)、棉花属(Gossypium)(王为等,2012)和木油桐(Vernicia montana)(Xu et al.,2012)等。
杉木(Cunninghamia lanceolata)为我国重要针叶用材树种,也是我国林木育种研究最为系统的树种之一,研究者围绕杉木育种资源遗传多样性的评价开展了许多卓有成效的工作。施季森等(1993)利用生化标记研究了杉木遗传多态性与多基因位点遗传结构,得出大多数群体中明显存在高阶配子不平衡现象,配子不平衡与地理、气候因子之间相关呈随机分布,奠基者效应、群体的再分化、分布区内环境多样性以及2 000多年人工栽培历史,均是产生和保持杉木群体遗传多样性及结构的重要因素。尤勇等(1998)利用RAPD标记研究了杉木7个种源的遗传变异,结果表明7个种源彼此间的遗传多态性水平较高,多态性位点所占比例为79.8%,遗传距离在0.225~0.440 7之间。李梅等(2001a;2001b)利用RAPD标记分别进行了杉木优树分子遗传变异的研究和杉木杂交亲本分子遗传变异与子代生长相关的研究,得出30株优树的多态性谱带比例达66.90%,具较高水平的遗传变异,杉木亲本的分子遗传变异与子代生长的相关关系较为复杂,它对亲本选配有一定的参考价值。然而,这些研究多采用同功酶,以及RAPD、ISSR、AFLP等标记技术,尚未涉及EST-SSR标记。近年来GenBank中积累的杉木及其同科的台湾杉EST序列日益增多,可为EST-SSR标记的开发提供重要资源。为此,本文拟对公共数据库中的杉木及台湾杉(Taiwaniacryptomerioides)EST序列开展SSR特征分析,开发相关SSR引物,并应用于30个杉木无性系的遗传多样性分析,以期为杉木育种资源系统评价和分子辅助育种提供重要依据。
1 材料与方法 1.1 植物材料参试材料为30个杉木无性系,皆采自浙江省开化县林场,具体来源见表 1。
|
|
基因组DNA的提取采用改良的CTAB法,具体步骤参照Doyle等(1990)的报道。
1.3 EST序列的获得及EST-SSR引物的设计从NCBI(http://www.ncbi.nlm.nih.gov)数据库中获取杉木及台湾杉的EST序列,数据截止2011年12月17日。用软件CAP3(http://seq.cs.iastate.edu/download.html)将EST序列组装成单基因后,利用在线软件Batchprimer3(http://probes.pw.usda.gov/batchprimer3/)搜寻EST序列中的SSR位点并设计对应引物。SSR位点搜寻标准:二核苷酸至少重复6次,三核苷酸至少重复4次,四、五、六核苷酸至少重复3次。引物设计参数标准为: PCR产物大小为100~350 bp,最佳选择为150 bp;引物长度18 ~23 bp,最佳选择为20 bp;退火温度为50~70 ℃,最佳温度为59 ℃;引物(G+C)含量为40%~60%,最佳为50%;其他为系统默认值。设计的EST-SSR引物由南京金斯瑞生物科技有限公司合成。
1.4 引物的筛选用8个不同来源的杉木无性系DNA为模板进行引物筛选。PCR反应采用10 μL反应体系,其中2×ComTaq PCR SuperMix(购自杭州西格玛生物技术有限公司)5 μL,上下游引物各0.25 μL,50 ng DNA模板1 μL,用水补足10 μL。PCR反应程序: 94 ℃预变性3 min;94 ℃变性30 s,退火温度54 ℃ 30 s,72 ℃延伸40 s,共32个循环;最后72 ℃延伸5 min。扩增产物经8%非变性聚丙烯酰胺凝胶电泳后进行银染检测。
1.5 SSR位点验证为了验证扩增片段中含有SSR位点,对扩增获得的片段进行克隆测序。具体操作如下:从PAGE胶中割取目的片段,放入1.5 mL离心管中,加20.0 μL TE缓冲液,4 ℃下浸提过夜。离心后,取2.0 μL上清作为模板,进行PCR扩增。扩增产物用3%的琼脂糖凝胶电泳检测,并从胶上割取目的片段,利用AxyPrep DNA凝胶回收试剂盒(Axygen公司)进行回收。回收后进行连接转化,交由南京金斯瑞公司测序。
1.6 EST-SSR多态性检测及数据统计记录电泳图目的片段范围内重复性好且清晰的条带,根据分子量大小,从大到小依次记作A,B,C,D,……,1条为纯合,2条为杂合,分别记录为aa,ab,bb,ac,ad,cd等,根据等位基因的位置获得原始数据,采用POPGENE version 1.31软件计算群体的等位基因数(observed number of alleles,na)、有效等位基因数(effective number of alleles,ne)和Shannon指数(Shannon’s information index,Ⅰ)。利用多态信息含量计算程序PIC _ Calc0.6计算多态性引物的PIC值。利用软件NTSYS-pc分析30个无性系的遗传关系,选用SM相似系数、UPGMA法进行聚类分析。
2 结果与分析 2.1 EST序列所含微卫星的分布、频率和特点408条杉木EST序列经CAP3拼接后获得Contigs 66条,Singlets 245条。通过Batchprimer3搜寻,共发现28个SSR位点分布在26条序列中,发生频率为8.36%,其中有2条含有2个SSR位点。2 643条台湾杉EST序列经CAP3拼接后获得Contigs 115条,Singlets 269条。含SSR位点的序列共27条,发生频率约为7.03%。27条序列共含SSR位点32个,其中2条各含有3个SSR位点,1条含有2个SSR位点。
由表 2可知,28个杉木SSR位点共有二、三、四、五、六核苷酸5种重复类型,其中以三核苷酸为基序的SSR类型最多,约占60.71%,以四核苷酸为基序的SSR类型次之,约占25%,而以二、五、六核苷酸为基序的SSR类型相对较少,分别为3.57%,7.14%和3.57%。32个台湾杉SSR位点的分布特征与杉木类似,也表现为二、三、四、五、六核苷酸5种重复类型,其中以三核苷酸为基序的SSR类型最多,约占53.13%,其次是四核苷酸为基序的SSR类型,约占25%。另外,从具体基序看,无论是杉木还是台湾杉,三碱基重复类型中所包含基序种类是最多的,均为11种,而且杉木SSR位点中是AGA/TCT,而台湾杉SSR位点中是GAT。四碱基类型基序次之,杉木和台湾杉分别有6种和7种;杉木中(TTTC)3出现频率最高,为28.57%,其他几种重复类型的出现频率均为14.29%;台湾杉中出现频率较高的是(AAAT)3/(ATTT)3和(AGGC)3/(AGGG)3,都为25%,其他几种类型的出现频率均为12.5%。其余核苷酸类型重复基序均只出现1次。
|
|
为检测所开发ESTSSR标记的可用性,分别设计22对杉木和28对台湾杉EST-SSR引物,并以8个不同来源的杉木无性系DNA为模板,进行引物检测与筛选。结果表明20对杉木EST-SSR引物和9对台湾杉EST-SSR引物能扩增出理想的PCR产物(表 3),引物有效扩增率分别为90.9%和32.1%。
|
|
为进一步验证所开发ESTSSR标记的真实性,根据条带是否与预测大小较为一致,或是否具有多态性的原则,从12对引物的PAGE电泳凝胶上回收了19个片段,并进行了测序分析(表 4)。由表 4可以看出仅有1条片段不含有SSR位点,即测序的扩增片段中SSR位点的含有率为95%。由片段9,10,11和12可以看出,片段差异是由SSR的重复次数引起(序列比对结果见图 1)。由片段6,7,8可以得出,当扩增片段远大于预测片段大小时,片段的差异性就不一定是由SSR的重复次数引起,有可能是片段中含有内含子造成。对于这种条带,在后续的读带时不进行统计。
|
|
 |
图 1 片段9,10,11,12和引物设计序列0的序列比对结果
Fig. 1 The alignment results of sequence 9,10,11,12 and 0
片段大小差异由TG 重复次数不同引起。 The differences of the sequences are caused by the repeat numbers of TG. |
利用筛选出的20对杉木EST-SSR引物和9对台湾杉EST-SSR引物,对30个杉木无性系DNA进行PCR扩增。结果有9对杉木EST-SSR引物扩增出多态性,占可扩增出目的片段引物的31%,呈现出较高的多态性(图 2);9对台湾杉EST-SSR引物中仅1对具多态性。10对引物中扩增获得的等位基因数最多为5个,最少为2个,平均等位基因数为2.9,幅度为2~5个(表 5)。
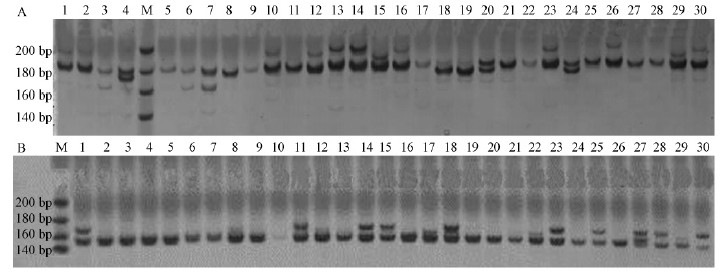 |
图 2 2 对引物在30 个杉木无性系间的多态性
Fig. 2 Polymorphisms showed in 30 different Chinese Fir clones by two pairs of primers
A.引物CL-12 Primer CL-12; B.引物CL-17 Primer CL-17. |
|
|
10个微卫星标记的多态性信息含量(PIC)为0.109 7~0.585 5,平均为0.375 7。根据Botstein等(1980)的理论,有2个标记为低度多态位点(0<PIC<0.25),6个标记为中度多态位点(0.25<PIC <0.5),剩余2个为高度多态位点(PIC>0.5),CL-20位点PIC最高(0.585 5),CL-5位点PIC最低(0.109 7)(表 5)。进一步采用POPGENE对这30个无性系的遗传多样性进行评价。结果显示这些无性系的平均等位基因数为1.666 7、有效等位基因平均数为1.666 7、Shannon多样性指数为0.462 1。而利用软件NTSYS-pc 进行聚类表明,以相似系数0.80为界限,可以把30个无性系分成6大类(图 3)。
 |
图 3 基于EST-SSR标记对30份材料的UPGMA聚类
Fig. 3 Dendrogram of 30 different materials based on EST-SSR markers
|
本文对408条杉木EST序列和2 643条台湾杉EST序列进行SSR特征分析,分别搜寻到28个杉木SSR位点和32个台湾杉SSR位点。在此基础上共设计SSR引物50对,其中10对引物的扩增条带具有多态性。扩增产物测序结果表明,绝大部分扩增片段均含有相应的SSR位点。采用多态性引物对30个杉木无性系遗传多样性进行分析,结果表明平均等位基因数为1.666 7、有效等位基因平均数为1.666 7、Shannon多样性指数为0.462 1。10个微卫星标记的多态性信息含量(PIC)为0.109 7 ~0.585 5,平均为0.375 7。以相似系数0.80为界限,可以把30个无性系分成6大类。这些结果说明开发和筛选出的EST-SSR引物可用于杉木育种资源的遗传多样性评价。
3.2 讨论1)杉木与台湾杉EST-SSR标记特点已有的研究表明,不同物种EST序列中SSR位点的发生频率不同。本研究分析了杉木EST序列中SSR位点的特征,结果发现杉木EST序列中SSR位点的发生频率约为8.36%,台湾杉EST序列中SSR位点的发生频率约为7.03%,其频率比其他物种的ESTSSR要高。如杨秀艳等(2011)发现1 620条日本落叶松(Larix kaempferi)EST序列中SSR位点的发生频率仅为3.85%,宋跃朋等(2010)发现5 359条与杨树(Populus)木质部形成相关的ESTs序列中SSR位点的频率为5.2%,也比杉木和台湾杉略低。产生这种情况的原因可能是物种差异造成的,也可能是本试验中用于分析的EST序列过少,存在一定的偏向性造成的。随着数据库中杉木EST序列的增加,将来的分析结果可能更加可靠。
EST-SSR标记目前已在多种植物的遗传多样性评价、图谱构建等方面得到广泛应用,但在杉木中依然没有相关的报道。为了充分利用公共数据库中积累的EST序列资源,丰富可用于杉木的分子标记,为杉木育种资源系统评价和分子辅助育种提供重要依据,本文利用杉木和台湾杉的EST序列开发出10对多态性引物,其中10对多态性引物仅有1对来自台湾杉。这说明杉木和台湾杉之间具有一定的亲缘关系,且这些具有多态性的位点可能是种间较保守的基因,但若要开发杉木EST-SSR标记,可能仍要依赖来自杉木本身的EST序列。
2)EST-SSR标记在杉木育种群体中的应用遗传多样性一般代表种内的遗传差异水平,可以反映物种适应环境的能力和被改造的潜力。本试验中,10对多态性引物对30个不同来源杉木无性系的遗传多样性分析结果显示,该群体的Shannon多样性指数平均值为0.462 1、平均等位基因数为1.666 7、有效等位基因平均数为1.666 7,比杨玉玲等(2009)利用ISSR分子标记对24个不同地理种源杉木的分析结果略高,表明所研究杉木育种群体的遗传多样性处于较高的水平。
但基于EST-SSR标记分析,本文也发现以相似系数0.800为界限,这30个无性系可分成6个亚群,但亚群内个体来源并无明显规律可循。这说明本研究开发的EST-SSR分子标记可用于杉木遗传多样性分析,但由于数量有限,对杉木个体间亲缘关系的揭示不是十分清晰。因此,今后有必要采用高通量测序等手段获得更多的杉木EST序列,以开发更多有用的EST-SSR分子标记,并进行性状与标记的关联分析,开发有效的标记,为杉木的分子辅助育种研究提供依据。
3)EST-SSR分子标记存在的问题及解决措施EST-SSR作为一种新型的分子标记,在比较作图、遗传图谱构建、种质资源遗传多样性评价与保护等研究中具有很大优势,但同时它也存在一定的缺陷:一方面,功能基因长期在选择的压力下存在着较高的保守性,而EST-SSR恰好来源于这些相对保守的功能基因序列,因此与Genomic SSR标记相比较,EST-SSR标记的多态性较低(Eujayl et al.,2001);另一方面,SSR分子标记多态性的原理是扩增片段中不同的微卫星重复数目产生不同的片段长度,但是对于长度相同而由不同碱基重复序列组成的SSR或者长度相近的SSR不能有效加以区分;除此之外,EST-SSR来自基因的编码部分,并不包含内含子等相关信息,在进行引物设计时可能会跨越内含子,有可能对扩增结果造成干扰。但相对于Genomic-SSR,EST-SSR的开发更简单方便,同时成本更低,因此随着公共数据库中序列信息的增加,EST-SSR被越来越多的研究者采用。
针对以上EST-SSR存在的不足,研究者们可以利用公共数据库中不断增加的EST序列开发新的EST-SSR标记,而现在高通量测序技术的出现,也使得获得EST序列的成本越来越低,有助于增加多态性SSR引物的数量,从而弥补EST-SSR标记本身多态性较低带来的问题;同时,结合其他分子标记如AFLP,CAPS,SNP等进行比较研究,可以提高试验的可靠性和准确性。另一方面,在对EST-SSR引物的电泳检测中,可以适当增加PAGE胶的浓度,提高凝胶的分辨率;同时,针对与预测片段差异较大的片段,可以通过测序等手段,验证其真实性,避免内含子对读带造成的干扰。
| [1] |
李 梅, 施季森, 何祯祥, 等. 2001a. 杉木优树分子遗传变异的研究. 林业科学, 37(4): 137-141.( 1) 1)
|
| [2] |
李 梅, 施季森, 甘四明, 等. 2001b. 杉木杂交亲本分子遗传变异与子代生长相关的研究. 林业科学研究, 14(1): 35-40.( 1) 1)
|
| [3] |
齐建勋, 郝艳宾, 朱 艳, 等. 2011. 核桃属种质资源的EST-SSR标记研究. 果树学报, 38(3): 441-448.( 1) 1)
|
| [4] |
施季森, 叶志宏, 洪菊生. 1993. 杉木遗传多态性与多基因位点遗传结构. 南京林业大学学报, 17(3): 9-15.( 1) 1)
|
| [5] |
宋跃朋, 江锡兵, 张 曼, 等. 2010. 基于ESTs序列的杨树木质部形成相关EST-SSRs标记的开发与应用. 分子植物育种, 8(5): 963-970.( 1) 1)
|
| [6] |
孙清明, 马文朝, 马帅鹏, 等. 2011. 荔枝 EST 资源的 SSR 信息分析及 EST-SSR 标记开发. 中国农业科学, 44(19):4037-4049.( 1) 1)
|
| [7] |
王 为, 王长彪, 刘 方, 等. 2012. 棉花非冗余性 EST-SSR 新标记的开发及其评价. 作物学报, 8(8):1443-1451.( 1) 1)
|
| [8] |
王艳敏, 魏志刚, 杨传平. 2008. 白桦EST-SSR信息分析与标记的开发. 林业科学, 44(2): 78-84.( 1) 1)
|
| [9] |
杨秀艳, 孙晓梅, 张守攻, 等. 2011. 日本落叶松EST-SSR标记开发及二代优树遗传多样性分析. 林业科学, 47(11): 52-58.( 1) 1)
|
| [10] |
杨玉玲, 马祥庆, 张木清. 2009. 不同地理种源杉木的分子多态性分析. 热带亚热带植物学报, 17(2): 183-189.( 1) 1)
|
| [11] |
尤 勇, 洪菊生. 1998. RAPD 标记在杉木种源遗传变异上的应用. 林业科学, 34 (4): 32-37.( 1) 1)
|
| [12] |
周炎花, 乔小燕, 马春雷, 等. 2011. 广西茶树地方品种遗传多样性和遗传结构的 EST-SSR 分析. 林业科学, 47(3): 59-67.( 1) 1)
|
| [13] |
朱 艳, 郝艳宾, 王克建, 等. 2009. 核桃 EST-SSR 信息分析与标记的初步建立. 果树学报, 26(3):394-398.( 1) 1)
|
| [14] |
Botstein D, White R L, Skolnick M, et al.1980. Construction of a genetic linkage maps in man using restriction fragment length polymorphisms. The American Journal of Human Genetics, 32(3):314-331.( 1) 1)
|
| [15] |
Chen X, Salamini F, Gebhardt C. 2001. A potato molecular function map for carbohydrate metabolism and transport. Theoretical and Applied Genetics, 102(2): 284-295.( 1) 1)
|
| [16] |
Cordeiro G M, Casu R, McIntyre C L, et al. 2001. Microsatellite markers from sugarcane (Saccharum spp.) ESTs cross transferable to helianthus and sorghum. Plant Science, 160(6): 1115-1123.( 1) 1)
|
| [17] |
Doyle J J, Doyle J L. 1990. Isolation of plant DNA from fresh tissue. Focus, 12: 13-15.( 1) 1)
|
| [18] |
Eujayl I, Sorrells M, Baum M, et al. 2001. Assessment of genotypic variation among cultivated durum wheat based on EST-SSRs and genomic SSRs. Euphytica, 119(12): 39-43.( 2) 2)
|
| [19] |
Eujayl I, Sorrells M E, Baum M, et al. 2002. Isolation of EST derived microsatellite markers for genotyping the A and B genomes of wheat. Theoretical and Applied Genetics, 104(23): 399-407.( 1) 1)
|
| [20] |
Kantety R V, LaRota M, Matthews D E, et al. 2002. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat. Plant Molecular Biology, 48(5): 501-510.( 1) 1)
|
| [21] |
Kota R, Varshney R K, Thiel T, et al. 2001. Generation comparison of EST-derived SSRs and SNPs in barley (Hordeum vulgare L.). Hereditas, 135(2/3): 145-151.( 1) 1)
|
| [22] |
Liewlaksaneeyanawin C, Ritland C E, El-Kassaby Y A, et al. 2004. Single-copy, species-transferable microsatellite markers developed from loblolly pine ESTs. Theoretical and Applied Genetics, 109(2): 361-369.( 1) 1)
|
| [23] |
Powell W, Machray G C, Provan J. 1996. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1(7): 215-222.( 1) 1)
|
| [24] |
Rungis D, Berube Y, Zhang J, et al. 2004. Robust simple sequence repeat markers for spruce (Picea spp.) from expressed sequence tags. Theoretical and Applied Genetics, 109(6): 1283-1294.( 1) 1)
|
| [25] |
Scott K D, Eggler P, Seaton G, et al. 2000. Analysis of SSRs derived from grape ESTs. Theoretical and Applied Genetics, 100(5): 723-726.( 1) 1)
|
| [26] |
Song Q J, Marek L F, Shoemaker R C, et al. 2004. A new integrated genetic linkage map of the soybean. Theoretical and Applied Genetics, 109(1): 122-128.( 1) 1)
|
| [27] |
Thiel T, Miehalek W, Varshney R K, et al. 2003. Exploiting EST database for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.). Theoretical and Applied Genetics, 106(3): 411-422.( 1) 1)
|
| [28] |
Xu Wei, Yang Qing, Huai Huyin, et al. 2012. Development of EST-SSR markers and investigation of genetic relatedness in tung tree. Tree Genetics & Genomes, 8(4): 933-940.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

