文章信息
- 张晓申, 范国强, 赵振利, 曹喜兵, 赵改丽, 邓敏捷, 董焱鹏
- Zhang Xiaoshen, Fan Guoqiang, Zhao Zhenli, Cao Xibing, Zhao Gaili, Deng Minjie, Dong Yanpeng
- 豫杂一号泡桐二倍体及其同源四倍体的AFLP和MSAP分析
- Analysis of Diploid and Its Autotetraploid Paulownia tomentosa×P. fortunei with AFLP and MSAP
- 林业科学, 2013, 49(10): 167-172
- Scientia Silvae Sinicae, 2013, 49(10): 167-172.
- DOI: 10.11707/j.1001-7488.20131026
-
文章历史
- 收稿日期:2013-05-13
- 修回日期:2013-06-27
-
作者相关文章
植物多倍化既是对自然环境适应的结果,也是推动其进化和物种形成的重要因素。自然界大约70%的被子植物在进化史中经历过一次或多次多倍化过程(Masterson,1994;Wendel,2000)。多倍体植物具有器官和生物量增大的特征及较强适应生物和非生物胁迫的能力(Hilu,1993;Liu et al.,2002)。植物多倍化过程中,在染色体结构(Swapna et al.,2010;Song et al.,1995;Pires et al.,2004;Pontes et al.,2004)和基因表达模式等发生变化(Lu et al.,2006;Adams et al.,2003;Kashkush et al.,2002;Adams et al.,2004)的同时,也发生了DNA甲基化变化(Levy et al.,2004;Liu et al.,2003;Wang et al.,2004)。研究表明,拟南芥(Arabidopsis thaliana)(Madlung et al.,2002)、小麦(Triticum aestivum)(Shaked et al.,2001;Han et al.,2003)、水稻(Oryzasativa)(Zhang et al.,2006)、陆地棉(Gossypiumhirsutum)(Keyte et al.,2006)和黄瓜(Cucumissativus)(Chen et al.,2008)多倍化后,DNA甲基化水平和甲基化模式都发生了变化。然而,有关DNA甲基化与多倍化关系研究多集中在草本植物(Ochogavía et al.,2009;Rodriguez et al.,2012),而有关木本植物的相关研究则较少(胡宝全等,2011;Bao et al.,2011)。泡桐(Paulownia)是中国重要的速生用材和绿化树种之一,因其独特的生物学特性,能与农作物间作形成独特的生态复合系统,为农作物高产稳产提供了重要生态保障。但因其种质资源匮乏、遗传背景窄等,目前利用现有的二倍体种质资源很难培育出符合人们意愿的泡桐新品种。近年来,不同四倍体泡桐新种质的成功诱导(范国强等,2006;2007a;2007b;2009;2010;赵振利等,2011),一方面扩大了泡桐的遗传背景,另一方面也为筛选泡桐新品种提供了材料支撑。过去,虽然开展过四倍体泡桐叶片结构、生长和木材特性等方面研究工作(张晓申等,2012;翟晓巧等,2012),但至目前国内外还未见四倍体及其二倍体泡桐遗传关系相关文章的报道。为了解二倍体及其同源四倍体泡桐遗传差异及DNA甲基化的作用,本文利用AFLP和MSAP分子标记技术,研究豫杂一号泡桐(Paulownia tomentosa×Paulownia fortunei)二倍体及其同源四倍体DNA序列及甲基化变化,以期为阐明泡桐四倍体优良特性的分子机制奠定基础。
1 材料与方法 1.1 试验材料试验材料为河南农业大学泡桐研究所经体胚发生途径获得、培养30天的豫杂一号泡桐二倍体及其同源四倍体组织培养苗。剪取适量生长状况良好、大小一致幼苗长约0.5 cm的顶芽,用液氮速冻后放于-86 ℃冰箱内备用。组培苗培养条件为:温度(25±2)℃,光照强度130 μmol·m-2s-1,光照时间每天16 h。
1.2 试验方法1)DNA提取 泡桐DNA提取参照张延召等(2009)方法。
2)AFLP和MSAP扩增 上述豫杂一号泡桐二倍体及其四倍体DNA的AFLP和MSAP扩增所需96对引物组合的碱基序列和扩增程序及其产物的电泳分别参照曹喜兵等(2010;2012)的方法。
3)电泳凝胶的谱带分析 电泳结束后,对用硝酸银染色的胶板谱带进行统计分析。统计分析中,H和M分别为EcoRⅠ/HpaⅡ和EcoRⅠ/MspⅠ双酶切产物电泳谱带。每一条谱带代表一个酶切位点。将谱带有或无分别记作1或0。每个DNA样品的H和M扩增谱带可划分为4种:种类I(H,M=1,1),无甲基化发生;种类Ⅱ(H,M=1,0),单链DNA外甲基化;种类Ⅲ(H,M=0,1),双链DNA内甲基化;种类Ⅳ(H,M=0,0),双链DNA外甲基化。DNA甲基化类型分为多态性和单态性。DNA多态性类型包括A(甲基化)型、B(去甲基化)型和C(不定)型。其中,A型中的A1和A2代表DNA重新甲基化(对照样H和M泳道均有带,而处理样仅H或M泳道有带),A3和A4代表DNA超甲基化(对照样仅H或M有1条带,而处理样H和M泳道都没带);B型(Bl,B2,B3和B4)代表DNA去甲基化,DNA甲基化谱带与A型相反;C型代表DNA甲基化的不确定性(对照样与处理样DNA甲基化差异谱带无法确定)。单态性类型为D型(D1,D2和D3)(对照样与处理样间DNA谱带相同)。同时,计算样品的总DNA甲基化水平[(种类Ⅱ+种类Ⅲ)/(种类I+种类Ⅱ+种类Ⅲ)×100%]和总DNA甲基化多态性[(A+B+C)/(A+B+C+D)×100%]及DNA单态性[D/(A+B+C+D)×100%]。
2 结果与分析 2.1 豫杂一号泡桐二倍体及其四倍体DNA碱基序列变化由豫杂一号泡桐二倍体及其四倍体DNA的AFLP扩增结果(图 1)可以看出,豫杂一号泡桐二倍体及其四倍体DNA利用同一引物均可在相同位置扩增出谱带,即豫杂一号泡桐二倍体与其四倍体DNA的AFLP酶切位点没有发生变化,每对引物平均可扩增出50~70条谱带,并且扩增出片段长度均小于500 bp。虽然豫杂一号泡桐二倍体及其四倍体DNA经不同引物扩增后产生的片段大小和数量存在一定的差异,但四倍体及其二倍体泡桐DNA经同一引物扩增后产生了数量和长度相同的DNA片段。该结果说明,豫杂一号泡桐二倍体及其四倍体DNA碱基序列在AFLP水平上相同。
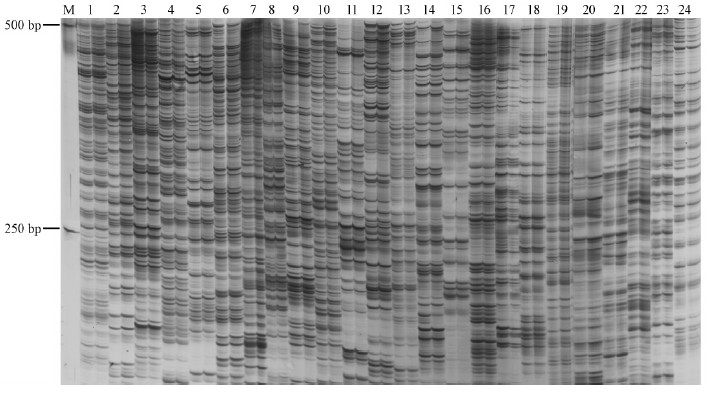 |
图 1 豫杂一号泡桐二倍体及其四倍体DNA的AFLP扩增产物部分SDS-PAGE
Fig. 1 SDS-PAGE of partial AFLP amplification products of the diploid and autotetraploid plants
M: Marker;1,2,3,…,23,24:引物编号Primer code. |
1)豫杂一号泡桐二倍体及其四倍体总DNA甲基化水平变化豫杂一号泡桐二倍体及其四倍体的MSAP电泳谱带统计结果(表 1)表明,二倍体总DNA甲基化水平低于四倍体泡桐。豫杂一号泡桐二倍体有MSAP扩增位点2 093个,其中,DNA全甲基化位点269个(占总扩增位点的12.85%),DNA半甲基化位点493个(占总扩增位点的23.55%),DNA总甲基化位点762个(占总扩增位点的36.41%)。豫杂一号泡桐四倍体MSAP扩增位点总数为2 217个,其中,DNA全甲基化位点332个(占总扩增位点的14.98%),DNA半甲基化位点550个(占总扩增位点的24.81%),DNA总甲基化位点882个(占总扩增位点的39.78%)。也就是说,豫杂一号泡桐四倍体总DNA甲基化水平高于其二倍体的DNA甲基化水平。此外,豫杂一号泡桐二倍体及其四倍体DNA甲基化(CCGG位点)均以双链甲基化为主,并且豫杂一号泡桐四倍体总DNA全甲基化和半甲基化水平皆高于其二倍体。该结果说明,豫杂一号泡桐二倍体染色体加倍形成的四倍体植株的DNA发生了甲基化修饰。
|
|
2)豫杂一号泡桐二倍体及其四倍体DNA的甲基化模式变化豫杂一号泡桐二倍体及其四倍体DNA分别经HapⅡ/EcoRⅠ和MspⅠ/EcoRⅠ酶切后,MSAP扩增产物的电泳结果(图 2、表 2和表 3)表明,豫杂一号泡桐二倍体及其四倍体DNA甲基化模式存在一定差异。四倍体DNA甲基化(A型)位点数为204,占总甲基化多态性位点数的9.97%,去甲基化(B型)位点数为235,占总甲基化多态性位点数的11.48%。与二倍体相比,四倍体DNA甲基化多态性为21.89%,DNA甲基化单态性的为78.11%。也就是说,豫杂一号泡桐二倍体DNA甲基化模式发生变化频率高于其同源四倍体。
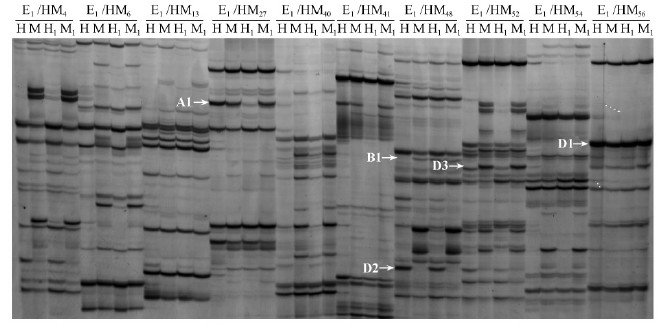 |
图 2 豫杂一号泡桐二倍体及其四倍体的DNA甲基化模式
Fig. 2 2 DNA methylation patterns of the diploid and autotetraploid plants
H、H1分别表示二倍体(2×)、四倍体(4×)豫杂一号泡桐基因组DNA经HapⅡ/EcoRⅠ酶切所产生的扩增结果;M、M1分别表示二倍体(2×)、四倍体(4×)豫杂一号泡桐基因组DNA经MspⅠ/EcoRⅠ酶切所扩增结果;箭头指示不同的DNA甲基变化类型(与表 2中的相对应);E1/HM(4,…,56)指示引物组合。 H and H1,respectively,indicated that total DNA of diploid(2×)and autotetraploid(4×)was digested by HapⅡ/EcoRⅠ;M and M1,respectively,indicated that total DNA of diploid(2×)and autotetraploid(4×)was digested by MspⅠ/EcoRⅠ;arrows indicated thedifferent changes of DNA methylation patterns in Tab. 2;E1/HM(4,…,56)indicated primer combination. |
|
|
|
|
同源四倍体植株通常是由其二倍体经化学诱变染色体加倍形成的,具有器官和生物量增大的明显特征及较强适应生物和非生物胁迫的能力(Hilu,1993;Liu et al.,2002),这些变化必然与染色体加倍后植物DNA的变化有一定的联系。植物染色体加倍前后,DNA碱基序列及其甲基化变化情况,不同研究者得出的结果不完全相同。刘文革等(2004)利用AFLP分析发现,西瓜(Citrullus lanatus)同源四倍体较其二倍体分别有特异DNA片段的增加和消失;王卓伟等(2002)用AFLP比较桑树(Morus alba)二倍体与其同源四倍体的结果表明,二者的DNA碱基序列也发生了一定程度的改变。黄瓜二倍体及其同源四倍体的研究结果也是如此(张晓青等,2006)。但焦锋等(2001)用RAPD分析了桑树二倍体及其同源四倍体的DNA指纹后没有发现差异条带;林强等(2011)利用RAPD分析后认为农桑8号(Morus‘Nongsang 8’)和湖桑199号(Morus‘Husang 199’)同源四倍体与其二倍体DNA指纹图谱相同;聂丽娟等(2009)利用AFLP没有检测到西瓜二倍体及其同源四倍体DNA碱基序列间的变化;马铃薯(Solanum tuberosum)染色体加倍前后DNA碱基序列也没有发生变化(Stupar et al.,2007)。本研究中,利用AFLP分析发现,豫杂一号泡桐二倍体及其染色体加倍后的DNA碱基序列没有发生变化,这可能是由于豫杂一号泡桐四倍体诱导过程中秋水仙碱用量较小,没有引起染色体重叠、不等交换及突变等的缘故。不同研究者试验结果间出现差异的原因可能有以下二方面:第一,不同研究者在诱导同源四倍体过程中使用诱变剂的剂量存在一定差异。如果诱变剂浓度过大,必然引起DNA结构的破坏,导致同源四倍体染色体的重组、不等交换及突变等情况的发生,从而出现DNA谱带的变化(Stupar et al.,2007);第二,不同研究者使用的材料在诱变后的时间跨度上不同。植物染色体加倍后,造成植株细胞内出现了核质不平衡等问题,但植株又要适应外部环境条件,这就往往迫使植物作出适当的形态和遗传变化(Finnegan et al.,2000;Martelotto et al.,2005;2007),最后表现在DNA结构和表观遗传学的变化。同源四倍体诱导后的时间越短,DNA碱基序列的一致性越高,用AFLP等分子标记技术检测出差异性的可能性越小,但由于染色体加倍造成的DNA甲基化变化始终存在,故出现MSAP片段多态性是必然的(王春国等,2009);同源四倍体诱导后的时间跨度越大,DNA碱基序列的变化几率越大,故较容易用AFLP和MSAP技术检测出植物染色体加倍前后DNA碱基序列及其甲基化的变化(胡宝全等,2011;Ochogavía et al.,2009;Bao et al.,2011;Rodriguez et al.,2012)。本研究中,虽然同源四倍体泡桐DNA碱基序列没有发生改变,但与其二倍体泡桐相比,DNA甲基化水平及其甲基化模式都发生了变化,并且株型及抗性也发生了改变(张晓申等,2012;翟晓巧等,2012)。据此认为同源四倍体泡桐DNA甲基化可能是其形态等性状未呈现二倍体倍增效应的原因之一。此外,尽管目前AFLP分子标记技术较能准确反映DNA碱基序列的变化,但毕竟检测的仍是DNA片段。因此,要证明二倍体及其同源四倍体间DNA碱基序列是否发生变化还需在单碱基(SNP)水平上开展研究工作。
| [1] |
曹喜兵,何 佳,翟晓巧,等.2010.泡桐AFLP反应体系的建立及引物筛选.河南农业大学学报,44(2):145-150.( 1) 1)
|
| [2] |
曹喜兵,赵改丽,范国强.2012.泡桐MSAP反应体系的建立.河南农业大学学报,46(5):536-541.( 1) 1)
|
| [3] |
范国强,杨志清,曹艳春,等.2006.秋水仙素诱导兰考泡桐同源四倍体.核农学报,20(6):473-476.( 1) 1)
|
| [4] |
范国强,曹艳春,赵振利,等.2007a.白花泡桐同源四倍体的诱导.林业科学,43(4):30-36.( 1) 1)
|
| [5] |
范国强,杨志清,曹艳春,等.2007b.毛泡桐同源四倍体的诱导.植物生理学通讯,43(1):109-111.( 1) 1)
|
| [6] |
范国强,魏真真,杨志清.2009.南方泡桐同源四倍体的诱导及其体外植株再生研究.西北农林科技大学学报:自然科学版,37(10):83-90.( 1) 1)
|
| [7] |
范国强,翟晓巧,魏真真,等.2010.豫杂一号泡桐体细胞同源四倍体诱导及其体外植株再生.东北林业大学学报,38(12):22-26.( 1) 1)
|
| [8] |
胡宝全,王春国,方成泉,等.2011.不同倍性(2×、3×、4×)鸭梨基因组DNA甲基化水平与模式分析.南开大学学报: 自然科学版,44(2):32-37.( 1) 1)
|
| [9] |
焦 锋,楼程富,张有做,等.2001.桑树变异株系基因组DNA 扩增多态性RAPD 研究.蚕业科学,27(3):165-169.( 1) 1)
|
| [10] |
林 强,邱长玉,朱方容,等.2011.桑树二倍体及其人工诱导同源四倍体遗传差异的RAPD分析.南方农业学报,42(1):11-15.( 1) 1)
|
| [11] |
刘文革,王 鸣,阎志红.2004.西瓜二倍体及同源多倍体遗传差异的AFLP分析.果树学报,21(1):46-49.( 1) 1)
|
| [12] |
聂丽娟,王子成,王一帆,等.2009.二倍体和四倍体西瓜的DNA甲基化差异分析.核农学报,23(1):80-84.( 1) 1)
|
| [13] |
王春国,古 瑜,陈成彬,等.2009.不同倍性西瓜基因组DNA甲基化水平与模式的MSAP.分子细胞生物学报,42(2):118-125.( 1) 1)
|
| [14] |
王卓伟,余茂德,鲁 成.2002.桑树二倍体及人工诱导的同源四倍体遗传差异的AFLP 分析.植物学通报,19(2):194-200.( 1) 1)
|
| [15] |
翟晓巧,张晓申,赵振利,等.2012.四倍体白花泡桐木材的物理特性研究.河南农业大学学报,46(6):651-657.( 1) 1)
|
| [16] |
张延召,曹喜兵,翟晓巧,等.2009.适用于AFLP分析的泡桐DNA 提取方法研究.河南农业大学学报,43(6):610-614.( 1) 1)
|
| [17] |
张晓青,陈劲枫,雷 春,等.2006.不同倍性黄瓜遗传差异的AFLP分析.西北植物学报,26(11):2265-2269.( 1) 1)
|
| [18] |
张晓申,翟晓巧,范国强,等.2012.四倍体泡桐叶片显微结构观察及其抗逆性分析.河南农业大学学报,46(6):646-650.( 1) 1)
|
| [19] |
赵振利,何 佳,赵晓改,等.2011.泡桐9501体外植株再生体系的建立及体细胞同源四倍体诱导.河南农业大学学报,45(1):59-65.( 1) 1)
|
| [20] |
Adams K L, Cronn R, Percifield R, et al. 2003. Genes duplicated by polyploidy show unequal contributions to the transcriptome and organ specific reciprocal silencing. Proc Natl Acad Sci,100(8):4649-4654.( 1) 1)
|
| [21] |
Adams K L, Percifield R, Wendel J F. 2004.Organ-specific silencing of duplicated genes in a newly synthesized cotton allotetraploid. Genetics,168(4):2217-2226.( 1) 1)
|
| [22] |
Bao A L, Hu Q, Xue Z Y, et al. 2011.DNA methylation in genomes of several annual herbaceous and woody perennial plants of varying ploidy as detected by MSAP. Plant Mol Biol Rep, 29(4):784-793.( 1) 1)
|
| [23] |
Chen L, Chen J. 2008.Changes of cytosine methylation induced by wide hybridization and allopolyploidy in Cucumis. Genome, 51(10):789-799.( 1) 1)
|
| [24] |
Finnegan E J, Peacock W J, Dennis E S. 2000. DNA methylation, a key regulation of plant development and other processes. Curr Opion Genet Develop, 10(2):217-223.( 1) 1)
|
| [25] |
Han F P, Fedak G, Ouellet T, et al. 2003.Rapid genomic changes in interspecific and intergenetic hybrids and allopolyploids of Triticeae. Genome, 46(4): 716-723.( 1) 1)
|
| [26] |
Hilu K W.1993.Polyploidy and the evolution of domesticated plants. Am J Bot,80(12):1494-1499.( 1) 1)
|
| [27] |
Kashkush K, Feldman M, Levy A A. 2002.Gene loss, silencing and activation in a newly synthesized wheat allotetraploid. Genetics, 160(4): 1651-1659.( 1) 1)
|
| [28] |
Keyte A L, Percifield R, Liu B, et al. 2006. Intraspecific DNA methylation polymorphism in cotton (Gossypium hirsutum L.). J Hered, 97(5):444-450.( 1) 1)
|
| [29] |
Levy A A, Feldman M. 2004.Genetic and epigenetic reprogramming of the wheat genome upon allopolyploidization. Biol J Linn Soc Lond, 82(4): 607-613.( 1) 1)
|
| [30] |
Liu B, Wendel J F. 2002.Non-mendelian phenomena in allopolyploid genome evolution.Curr Genomics,3(6):489-505.( 1) 1)
|
| [31] |
Liu B, Wendel J F. 2003.Epigenetic phenomena and the evolution of plant allopolyploids. Mol Phylogenet Evol,29(3):365-379.( 1) 1)
|
| [32] |
Lu B B, Pan X Z, Zhang L, et al. 2006.A genome-wide comparison of genes responsive to autopolyploidy in Isatis indigotica using Arabidopsis thaliana Affymetrix genechips. Plant Mol Biol Rep, 24(2):197-204.( 1) 1)
|
| [33] |
Madlung A, Masuelli R W, Watson B, et al. 2002.Remodeling of DNA methylation and phenotypic and transcriptional changes in synthetic Arabidopsis allotetraploids. Plant Physiol,129(2):733-746.( 1) 1)
|
| [34] |
Martelotto L G, Ortiz J P A, Stein J, et al. 2005.A comprehensive analysis of gene expression alterations in a newly synthesized Paspalum notatum autotetraploid. Plant Sci,169(1):211-220.( 1) 1)
|
| [35] |
Martelotto L G, Ortiz J P A, Stein J, et al. 2007.Genome rearrangements derived from autopolyploidization in Paspalum sp. Plant Sci,172(5): 970-977.( 1) 1)
|
| [36] |
Masterson J. 1994.Stomatal size in fossil plants:evidence for polyploidy in majority of angiosperms.Science,264(5157): 421-424.( 1) 1)
|
| [37] |
Ochogavía A, Cervigni G, Selva J P, et al. 2009.Variation in cytosine methylation patterns during ploidy level conversions in Eragrostis curvula. Plant Mol Biol,70(1-2):17-29.( 1) 1)
|
| [38] |
Pires J C, Zhao J W, Schranz M E, et al. 2004.Flowering time divergence and genomic rearrangements in resynthesized polyploids (Brassica). Biol J Linn Soc Lond, 82(4):675-688.( 1) 1)
|
| [39] |
Pontes O, Neves N, Silva M, et al. 2004.Chromosomal locus rearrangements are a rapid response to formation of the allotetraploid Arabidopsis suecica genome. Proc Natl Acad Sci,101(52):18240-18245.( 1) 1)
|
| [40] |
Rodriguez M P, Cervigni G D L, Quarin C L, et al. 2012.Frequencies and variation in cytosine methylation patterns in diploid and tetraploid cytotypes of Paspalum notatum. Biologia Plantarum,56(2):276-282.( 1) 1)
|
| [41] |
Shaked H, Kashkush K, Ozkan H, et al. 2001.Sequence elimination and cytosine methylation are rapid and reproducible responses of the genome to wide hybridization and allopolyploidy in wheat. Plant Cell,13(8):1749-1759.( 1) 1)
|
| [42] |
Song K, Lu P, Tang K, et al. 1995. Rapid genome change in synthetic polyploids of Brassica and its implications for polyploid evolution. Proc Natl Acad Sci, 92(17):7719-7723.( 1) 1)
|
| [43] |
Stupar R M, Bhaskar P B, Yandell B S, et al. 2007.Phenotypic and transcriptomic changes associated with potato autopolyploidization. Genetics,176(4):2055-2067.( 1) 1)
|
| [44] |
Swapna M, Sivaraju K, Sharma R K, et al. 2010.Single-strand conformational polymorphism of EST-SSRs: a potential tool for diversity analysis and varietal identification in sugarcane. Plant Mol Biol Rep,29(3):505-513.( 1) 1)
|
| [45] |
Wang J, Tian L, Madlung A, et al. 2004.Stochastic and epigenetic changes of gene expession in Arabidopsis polyploids. Genetics, 167(4): 1961-1973.( 1) 1)
|
| [46] |
Wendel J F. 2000.Genome evolution in polyploids. Plant Mol Biol,42(1):225-249.( 1) 1)
|
| [47] |
Zhang H Y, Peng H, Li Y, et al. 2006.Patterns of DNA cytosine methylation between haploid and corresponding diploid rice. Chin Sci Bull, 51(14):1721-1728.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

