文章信息
- 李长江, 孙帆, 张通, 张凌云
- Li Changjiang, Sun Fan, Zhang Tong, Zhang Lingyun
- 青杄PwPSAF基因的克隆与组织表达分析
- Cloning and Tissue Expression Analysis of PwPSAF in Picea wilsonii
- 林业科学, 2013, 49(10): 40-47
- Scientia Silvae Sinicae, 2013, 49(10): 40-47.
- DOI: 10.11707/j.1001-7488.20131007
-
文章历史
- 收稿日期:2013-02-01
- 修回日期:2013-08-04
-
作者相关文章
PSAF(photosystem I reaction center F),又称光系统Ⅰ亚基Ⅲ(subunit Ⅲ of photosystem Ⅰ),是绿色植物光系统Ⅰ(PSⅠ)反应中心蛋白F亚基,为PSⅠ复合体的重要组成部分(Farkas et al.,2011a)。PSAF蛋白由核基因编码,在受到光信号或细胞分裂素的诱导后表达,同时也受到Ca2+、细胞质激酶与磷酸酶的诱导(Chandok et al.,2001;Wöstemeyer et al.,2003)。大量研究表明,PSAF在藻类光合作用中,特别在PSⅠ的电子传递过程中起到重要作用。Chitnis等(1991)首先在蓝藻(Cyanobacterium synechocystis)中鉴定了PSAF基因。之后,在莱茵衣藻(Chlamydomonas reinhardtii)的研究中发现,PSAF蛋白介导质体蓝素、细胞色素c6与PS Ⅰ反应中心结合(Farah et al.,1995),且发现特异互作位点为PSAF的Lys27与细胞色素c6的Glu69残基、PSAF的Lys23残基与细胞色素c6的Glu69/Glu70残基(Sommer et al.,2006)。进一步研究发现,STT7基因的失活将能保护莱茵衣藻PSAF突变体免受高光下的氧化胁迫作用(Berry et al.,2011),而PSAJ能稳定PSAF的结构与功能,当psaj缺失突变后,质体蓝素与细胞色素c6向反应中心P700的电子转移水平将显著下降(Fischer et al.,1999)。在聚球藻(Synechococcus elongatus)与莱茵衣藻中,也证明了PSAF蛋白介导细胞色素c6与质体蓝素中的电子向PSⅠ反应中心的传递(Hippler et al.,1999),而且在这个过程中PSAF的N端起到重要作用(Hippler et al.,1998)。在聚球藻中发现,当psaf突变后,电子转移线路会发生改变(Van Der Est et al.,2004),而PSAB的分支电子转移将会增加(Agalarov et al.,2008)。
在高等植物中,对于PSAF功能的研究并不多见。Flieger等(1993)在菠菜(Spinacia oleracea)中发现位于启动子区-220 bp与-179 bp之间的一个42 bp区域对于PSAF基因的表达有重要的调控作用。进一步研究表明PSAF与质体蓝素相互作用,并形成了对传递电子有重要作用的二硫桥(Farkas et al.,2011a),通过核磁共振技术证明,Mg2+和Mn2+与菠菜的质体蓝素结合,进而调节其与PSAF的结合(Farkas et al.,2011b)。同时,PSAF蛋白8与63位的半胱氨酸受到硫氧还蛋白的还原作用,而蛋白的活化则需要受到质体蓝素的氧化作用(Farkas et al.,2011c)。相对于在藻类中PSAF功能的研究,植物中该蛋白功能还知之甚少。
青杄(Picea wilsonii)属于松科(Pinaceae)云杉属(Picea)植物,是多年生木本针叶树种,对干旱、阴冷等环境有较强的适应能力,为我国重要的园林绿化树种。本研究基于青杄cDNA文库与PwPSAF的EST序列,利用RACE PCR获取PwPSAF的全长cDNA序列。通过生物信息学工具预测PwPSAF的分子组成、理化性质、二级结构、三级结构等;同时,利用RT-qPCR技术检测其在青杄各组织的特异表达、种子萌发过程的表达水平,以及干旱、盐胁迫处理下PwPSAF在各组织的表达水平。本研究将为林木中PSAF的功能研究奠定基础,以进一步研究其在林木发育及抗逆过程中的功能,也可为林木遗传育种、优质林木基因的筛选等提供理论依据。
1 材料与方法 1.1 试验材料 1.1.1 植物材料青杄花粉和种子采集于中国科学院植物所植物园;青杄混合组织cDNA文库由Invitrogen(英潍捷基)公司利用Gateway技术构建。3年生青杄幼苗的根、茎与针叶用于组织特异表达试验,采集后置于-80 ℃备用。
1.1.2 试验试剂pDONR222为文库载体,由Invitrogen公司提供,pEASY-T1载体购于全式金公司,PCR Taq PCR mix、DNA marker(DL2000)、大肠杆菌(Escherichia coli)感受态DH5α等购于天根公司,反转录试剂盒(RevertAidTM First Str and cDNASynthesis Kit)购于Fermentas公司。荧光定量PCR试剂盒(SYBR FAST qPCR Kit)购于美国KAPA公司。RNA提取试剂盒购于AidLab公司。其他试剂均购买于AMRESCO公司。
以青杄cDNA文库为模板,根据PwPSAF的EST序列与pDONR222载体的序列设计引物,进行RACE PCR试验。5'-RACEPCR引物为5-race-f与5-race-r,3'-RACE PCR引物为3-race-f与3-race-r。经过回收、测序后获得PwPSAF的末端序列,与EST序列拼接后获得PwPSAF的全长cDNA序列。设计引物psaf-forward与psaf-reverse,经过PCR后得到开放阅读框完整的核酸序列,连接到pEASY-T1上,获得PwPSAF单克隆。所用到的引物见表 1。
|
|
运用DNAMAN软件进行核酸序列拼接及翻译,运用ProtParam工具分析蛋白的氨基酸组成及理化特性(http://web.expasy.org/protparam/)。利用TMHMM工具进行蛋白跨膜预测(http://www.cbs.dtu.dk/services/TMHMM-2.0/);利用ProtScale工具进行蛋白疏水性分析(http://web.expasy.org/cgi-bin/protscale/protscale.pl);运用GOR4工具进行蛋白的二级结构分析(http://gor.bb.iastate.edu/);运用SWISS-MODEL工具进行蛋白的三级结构分析(http://swissmodel.expasy.org/)。多序列比对由ClustalX工具完成,系统发育树由MEGA5构建完成,构建系统树计算方法为邻位相连法。
1.2.3 组织特异表达试验用于试验的3年生青杄的根、茎、针叶、种子及花粉等总RNA的提取利用AidLab公司的植物RNA提取试剂盒。琼脂糖凝胶电泳的结果中28 S与18 S比较明显即可进行cDNA第1条链的合成,运用Fermentas公司的反转录试剂盒进行。均一化浓度之后在StepOnePlusReal Time RT-PCR仪器上进行荧光定量PCR来检测基因在不同组织的相对表达量,所用特异引物为psaf-rt-a与psaf-rt-b,内参基因为青杄延伸因子蛋白EF1-α(Yu et al.,2011),引物为EF1-α-F与EF1-α-R(表 1),试验设置3次重复。
1.2.4 种子萌发试验种子萌发试验采用水培法。将吸水滤纸铺在培养皿中,青杄种子分散放入湿润的滤纸上培养,试验过程中保持培养皿底部水分充足。将培养皿置于25 ℃光照培养箱中,光周期为16 h光照/8 h黑暗,培养10天,分别在培养0,2,4,6,8,10天时采样,并置于-80 ℃保存备用。提取各时期萌发幼体总RNA后,经反转录、RT-qPCR等试验检测各个时期PSAF的表达量,各个时期取样及RT-PCR试验均设置3次重复。
1.2.5 干旱与盐胁迫试验干旱胁迫处理参考喻方圆(2002)方法,盐胁迫试验参考朱振贤(2007)研究方法,分别有改动。具体如下:将3年生青杄幼苗栽植于营养土与蛭石(体积比1∶ 1)混合的培养基质中,置于温室中,温度为25 ℃,空气湿度为60%,光周期为16 h光照/8 h黑暗。干旱试验中,对试验组青杄进行控水处理4周(不浇水),对照组每周浇灌500 mL清水。盐胁迫试验中,试验组每周分别浇灌500 mL 2%,4%,6%的NaCl溶液,并每天喷施5mL对应浓度的NaCl溶液于青杄幼苗针叶做加强处理,处理4周,对照组每周浇500 mL清水。干旱与盐胁迫试验中每个处理均设置3个重复,取样后放入-80 ℃备用。提取青杄幼苗各组织(根、茎与针叶)总RNA后进行反转录、RT-qPCR等试验检测各组织PwPSAF的表达量变化,取样及RT-PCR试验均设置3次重复。
2 结果与分析 2.1 青杄PSAF的克隆经过RACE PCR成功获得PwPSAF的末端序列,与EST序列拼接后获得全长cDNA序列。PwPSAF全长cDNA序列由1 139 bp组成,5'-UTR为41 bp,在42 bp处发现起始密码子ATG,在801 bp处发现终止密码子TAG,编码区共759 bp,编码253个氨基酸。3'-UTR共321 bp,并在末端发现Poly(A)15尾巴(图 1)。
 |
图 1 青杄PSAF的全长cDNA序列及蛋白质氨基酸序列
Fig. 1 Full length cDNA sequence and deduced protein amino acid sequence of PwPSAF
|
根据ProtParam工具预测PSAF的分子量为27.04 kDa,理论等电点为9.57。丙氨酸(Ala)为12.3%,赖氨酸(Lys)、丝氨酸(Ser)与亮氨酸(Leu)含量均为9.5%,甘氨酸(Gly)为7.1%,分子式C1210H1953N331O351S9,预测半衰期约为30 h,不稳定指数为50.47,说明蛋白质不稳定。利用Protscale工具预测蛋白质疏水性,结果如图 2A,蛋白质第75位亮氨酸(Leu)分值为2.244,疏水性最大,第58位天冬酰胺(Asn)分值最小,为-3.722。TMHMM工具预测蛋白有2个跨膜区域,分别位于64—86 aa与176—198 aa(图 2B)。
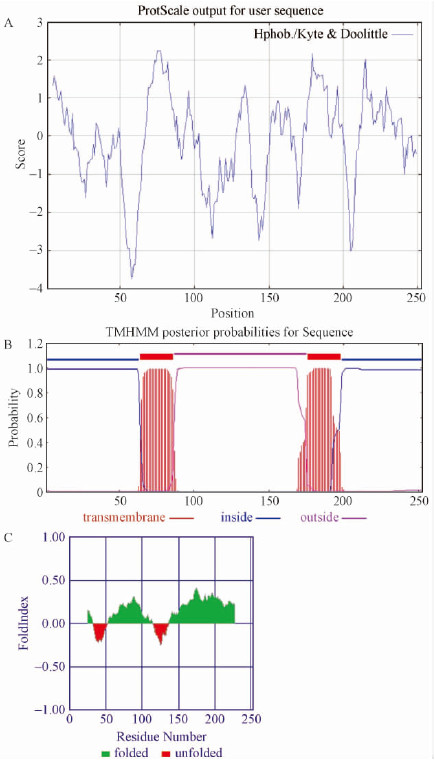 |
图 2 PwPSAF的理化性质分析
Fig. 2 Physical and chemical properties analysis of PwPSAF
A: ProtScale工具预测PwPSAF疏水性结果;B: TMHMM工具预测PwPSAF跨膜分析结果;C: FoldIndex工具预测PwPSAF固有无序化分析结果。 A: Hydrophobic analysis of PwPSAF byProtScale;B: Transmembrane analysis of PwPSAF by TMHMM;C:Intrinsically disordered protein analysis of PwPSAF by FoldIndex. |
利用FoldIndex工具进行蛋白质固有无序化分析,结果表明蛋白质在N端及115—135 aa之间存在无序化特征,预测此蛋白质具有很强的动态结构,在行使其功能时蛋白质的结构容易发生改变(图 2C)。
2.2.2 多序列比对及进化树分析通过NCBI上的Blastp工具可以检测到PwPSAF在其他物种中的同源蛋白质,如北美云杉(Picea sitchensis)、毛果杨(Populus trichocarpa)、葡萄(Vitis vinifera)、马铃薯(Solanum tuberosum)、拟南芥(Arabidopsis thaliana)等PSAF蛋白质(表 2)。运用Clustalx工具进行多序列比对(图 3),结果发现不同植物的PSAF拥有保守的CD Ⅰ(Val-Cys-Ser)与CD Ⅱ(Ser-Ala/Tyr-Ala-Leu-Ala-Leu)结构,以及大部分的C端序列。利用MEGA5进行不同物种中PSAFs的进化树分析发现: PwPSAF与北美云杉的PSAF聚为一簇(图 4),裸子植物PSAF聚在Clade Ⅲ,双子叶植物PSAF聚在Clade Ⅰ,单子叶植物PSAF聚在Clade Ⅱ。
|
|
 |
图 3 不同物种PSAF蛋白多序列比对结果
Fig. 3 Multiple protein sequences alignment of PSAF of various species
Conserved residues in this alignmentare shaded in black(homology level=100%),pink(homology level>75%) and blue(homology level>50%). |
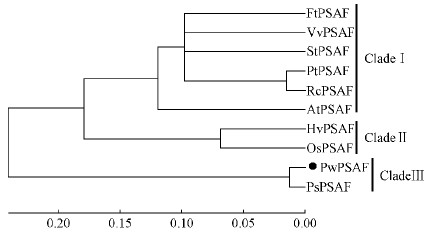 |
图 4 青杄PSAF进化树分析
Fig. 4 Phylogenetic tree analysis of PwPSAF
多序列比对中的保守序列标记为黑色(相似性=100%),粉色(相似性>75%),蓝色(相似性>50%)。 Conserved residues in this alignmentare shaded in black(homology level=100%),pink(homology level>75%) and blue(homology level>50%). |
利用GOR4工具进行蛋白的二级结构预测,发现PwPSAF中α-螺旋(α-helix)为27.27%,无规则卷曲(r and omcoil)占50.20%,延伸链(extended str and )为22.53%,且α-螺旋主要分布在60—74 aa、100—145 aa及N端220 aa处(图 5A)。
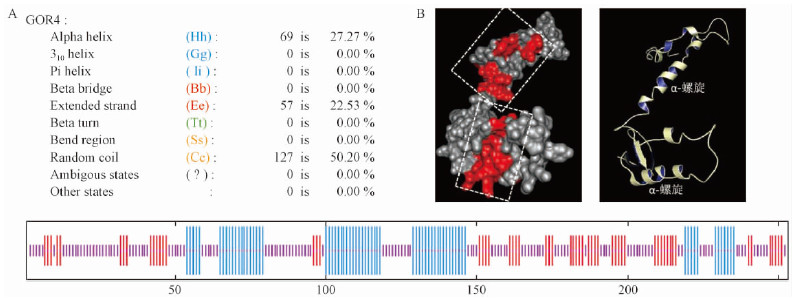 |
图 5 青杄PSAF的二级结构与三级结构
Fig. 5 Predicted secondary and tertiary structure of PwPSAF
A: PSAF二级结构,由GOR4工具进行预测;B: PSAF三级结构,由Swiss-model工具预测,虚线方框内为预测的跨膜区域。 A: Secondary structure ofPwPSAF predicted by GOR4 tool;B: Tertiary structure of PwPSAF predicted by Swiss-model,and dotted boxes indicate the transmembrane domains. |
三级结构由SWISS-MODEL工具预测(图 5B),蛋白含有较多的α-螺旋结构,且在空间上有一定距离,可以分别形成疏水区域或跨膜区域,跨膜区域在N端与C端均有分布。
2.3 组织表达分析 2.3.1 组织特异性表达分析提取青杄各组织的总RNA以及青杄种子在萌发不同时期幼体的总RNA,进行琼脂糖凝胶电泳,显示总RNA的提取质量较好,28 S与18 S条带清晰(图略),可以进行后续试验。
利用RT-qPCR技术检测PwPSAF在青杄各组织的表达量,结果发现PwPSAF在针叶中表达量最高,其次是在茎与种子中(图 6A)。在种子萌发的过程中,PwPSAF在第2天的表达量最高,之后开始急剧下降,随着子叶生长,从第6天开始PwPSAF在萌发幼体中的表达量逐步增高(图 6B)。
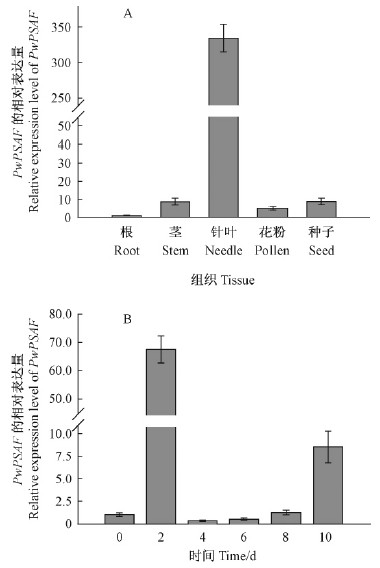 |
图 6 PwPSAF在青杄不同组织(A)及种子萌发不同时间(B)的表达分析
Fig. 6 Expression levels of PwPSAF in different organs(A) and during seed germination(B)of Picea wilsonii
所有结果均由三次重复试验所得。 Values are means±SE from three independent assays. |
提取胁迫处理后的青杄各组织总RNA,进行反转录与浓度均一化后,进行RT-qPCR试验。结果显示,在干旱情况下,PwPSAF基因在根与茎中表达量与对照相比基本无变化,值得注意的是在针叶中PwPSAF的表达量下调10倍左右(图 7A)。在盐胁迫下,PwPSAF的表达量随着盐浓度的增加,在针叶中不断下降,而在6%的NaCl溶液处理下,根与茎中PwPSAF的表达量上调(图 7B)。
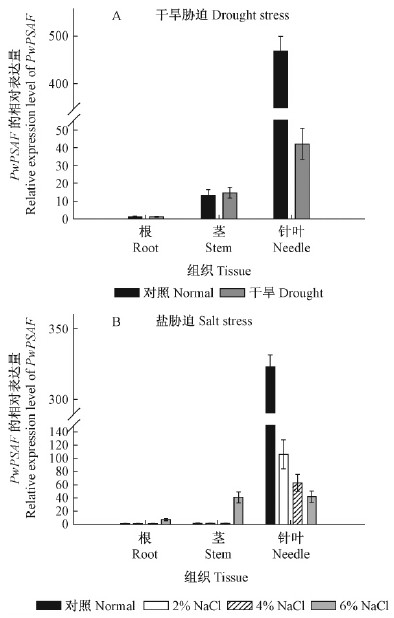 |
图 7 干旱与盐胁迫下PwPSAF在青杄不同组织的表达情况
Fig. 7 Expression level of PwPSAF in different organs of Picea wilsoniiunder drought and salt stresses
所有结果均由3次重复试验所得。 Values are means±SE from three independent assays. |
本研究成功分离了青杄PwPSAF基因,基于其蛋白质氨基酸组成进行了生物信息学分析。运用RT-qPCR技术检测了PwPSAF在青杄不同组织、种子萌发不同时期,以及在干旱与盐胁迫下的表达情况。结果显示PwPSAF是一个由253 aa组成的膜蛋白,含有典型的PSAK结构域,通过blast检索发现其氨基酸序列与北美云杉的EF087687高度同源,二者相似性达到98.02%。进化树分析也表明二者聚为一簇,由于青杄与北美云杉均为松科云杉属植物,推测二者在进化上有共同的祖先。
针叶是青杄进行光合作用的主要场所,相比于其他组织(如根、茎等),PwPSAF在青杄的针叶中表达量最高,暗示了该基因可能参与青杄针叶的光合过程。这与其他物种中PSAF主要参与光合作用的功能一致(Sommer et al.,2006;Farkas et al.,2011a)。同时,这也与作者对青杄PSAK(photosystem reaction center subunit K)的研究结果类似(李长江等,2012),PwPSAK与PwPSAF均为PSⅠ复合体的重要组成部分,且二者基因均在青杄的针叶中高表达。
重要的是,在种子萌发试验中PwPSAF的表达量在第2天急剧升高,这个时期是青杄种子的吸胀期,种子打破休眠,大量基因被诱导激活表达,在第4天PwPSAF的表达量又下调;随着子叶的生长,PwPSAF表达量又逐步升高。PwPSAF在青杄种子萌发过程中表达量的变化说明PwPSAF在种子萌发早期被诱导,参与了种子的萌发过程,但在萌发不同阶段可能起着不同的作用。另一种可能是该基因受到植物自身发育过程及外界环境信号的调控。Chandok等(2001)认为PSAF的表达受到光信号的诱导,因此推断在本试验中,种子萌发6天后,PwPSAF受到光信号的诱导,表达量又开始逐渐增加。然而,PwPSAF的表达量在青杄种子萌发的0~4天内升高又急剧下调的分子机制还有待研究。PwPSAF在青杄种子萌发与针叶中的表达模式说明了其不但参与了青杄的光合作用过程,而且与光形态建成的动态过程有关。
大量研究表明,在干旱及盐胁迫等非生物逆境条件下,植物的光合作用会明显受抑。在玉米(Zeamays)中的研究表明干旱会降低玉米的光合速率,增强叶片对光的敏感性,严重制约光合作用(卜令在对杨树的研究中发现,受到高温与干旱胁迫后,核酮糖1,5-二磷酸羧化酶(Rubisco)的表达量将降低,但PS Ⅰ中PSAD(photosystem reaction centersubunit D)的表达被诱导上调(He et al.,2008)。在本研究中也表明,干旱会在分子水平降低PwPSAF的表达量,进而影响PS Ⅰ的功能,这与Batlang(2010)在水稻(Oryza sativa)中的研究结果相同,即在受到干旱胁迫后,水稻中PSⅠ的F亚基、K亚基与N亚基的表达量均被下调。盐胁迫也严重影响植物的光合作用。有研究表明,在盐胁迫下,光合作用中起到重要作用的Rubisco活性降低,植物的光合效率下降(张娟等,2008);在高光胁迫下,植物体内的活性氧(ROS)大量产生,直接破坏PSⅡ的反应中心,从而抑制光合作用(Murata et al.,2007),而盐胁迫则通过抑制PSBA基因的表达抑制PSⅡ的修复(Allakhverdiev et al.,2002)。关于盐胁迫对PSⅠ的影响报道较少。在本试验中发现,盐胁迫下青杄PSAF在针叶的表达量下调,这说明盐胁迫也对PSⅠ产生不利影响。且在本研究中,随着盐害的增加,PwPSAF下调的程度增大。值得注意的是在受到6% NaCl溶液处理后,茎中PwPSAF的表达量上调,推测植物体针叶中光合系统受到损害,而植物自身为了维系光合产能,调高了茎中的表达量,然而具体的分子机制还有待研究。
| [1] | 卜令铎, 张仁和,常 宇,等. 2010. 苗期玉米叶片光合特性对水分胁迫的响应.生态学报, 30(5): 1184-1191. |
| [2] |
李长江, 曹一博, 张凌云. 2012. 青杄PSAK的克隆及生物信息学分析. 生物技术, 22(3): 4-9.( 1) 1)
|
| [3] |
喻方圆. 2002. 几个针叶树种苗木逆境生理研究. 南京: 南京林业大学博士学位论文.( 1) 1)
|
| [4] |
张 娟, 姜闯道, 平吉成. 2008. 盐胁迫对植物光合作用影响的研究进展. 农业科学研究, 29(3): 74-80.( 1) 1)
|
| [5] |
朱振贤. 2007. 几种主要造林树种盐胁迫响应及耐盐机理研究. 南京: 南京林业大学硕士学位论文.( 1) 1)
|
| [6] |
Agalarov R, Byrdin M, Rappaport F, et al. 2008. Removal of the PsaF polypeptide biases electron transfer in favor of the PsaB branch of cofactors in Triton X-100 photosystem I complexes from Synechococcus sp. PCC 7002. Photochemistry and Photobiology, 84 (6): 1371-1380.( 1) 1)
|
| [7] |
Allakhverdiev S I, Nishiyama Y, Miyairi S, et al. 2002. Salt stress inhibits the repair of photodamaged photosystem Ⅱ by suppressing the transcription and translation of psbA genes in Synechocystis. Plant Physiology, 130 (3): 1443-1453.( 1) 1)
|
| [8] |
Batlang U. 2010. Identification of drought-responsive genes and validation for drought resistance in rice. Virginia Polytechnic Institute and State University. Doctor of Philosophy.( 1) 1)
|
| [9] |
Berry L L, Brzezowski P, Wilson K E. 2011. Inactivation of the STT7 gene protects PsaF-deficient Chlamydomonas reinhardtii cells from oxidative stress under high light. Physiologia Plantarum, 141 (2): 188-196.( 1) 1)
|
| [10] |
Chandok M R, Sopory S K, Oelmuller R. 2001. Cytoplasmic kinase and phosphatase activities can induce PsaF gene expression in the absence of functional plastids: evidence that phosphorylation/dephosphorylation events are involved in interorganellar crosstalk. Molecular & General Genetics: MGG, 264 (6): 819-826.( 2) 2)
|
| [11] |
Chitnis P R, Purvis D, Nelson N. 1991. Molecular cloning and targeted mutagenesis of the gene PsaF encoding subunit Ⅲ of photosystem I from the cyanobacterium Synechocystis sp. PCC 6803. The Journal of Biological Chemistry, 266 (30): 20146-20151.( 1) 1)
|
| [12] |
Farah J, Rappaport F, Choquet Y, et al. 1995. Isolation of a psaF-deficient mutant of Chlamydomonas reinhardtii: efficient interaction of plastocyanin with the photosystem I reaction center is mediated by the PsaF subunit. The EMBO Journal, 14 (20): 4976-4984.( 1) 1)
|
| [13] |
Farkas D, Franzen L G, Hansson O. 2011a. Cloning, expression and purification of the luminal domain of spinach photosystem 1 subunit PsaF functional in binding to plastocyanin and with a disulfide bridge required for folding. Protein Expression and Purification, 78 (2): 156-166.( 3) 3)
|
| [14] |
Farkas D, Hansson O. 2011b. An NMR study elucidating the binding of Mg(Ⅱ) and Mn(Ⅱ) to spinach plastocyanin. Regulation of the binding of plastocyanin to subunit PsaF of photosystem I. Biochimica et Biophysica Acta(BBA)-Bioenergetics, 1807 (12): 1539-1548.( 1) 1)
|
| [15] |
Farkas D, Hansson O. 2011c. Thioredoxin-mediated reduction of the photosystem I subunit PsaF and activation through oxidation by the interaction partner plastocyanin. FEBS Letters, 585 (12): 1753-1758.( 1) 1)
|
| [16] |
Fischer N, Boudreau E, Hippler M, et al. 1999. A large fraction of PsaF is nonfunctional in photosystem I complexes lacking the PsaJ subunit. Biochemistry, 38 (17): 5546-5552.( 1) 1)
|
| [17] |
Flieger K, Tyagi A, Sopory S, et al. 1993. A 42 bp promoter fragment of the gene for subunit Ⅲ of photosystem I (psaF) is crucial for its activity. The Plant Journal, 4 (1): 9-17.( 1) 1)
|
| [18] |
He C Y, Zhang J G, Duan A G, et al. 2008. Proteins responding to drought and high-temperature stress in Populus×euramericana cv. ‘74/76'. Trees-Struct Funct, 22 (6): 803-813.( 1) 1)
|
| [19] |
Hippler M, Drepper F, Haehnel W, et al. 1998. The N-terminal domain of PsaF: precise recognition site for binding and fast electron transfer from cytochrome c6 and plastocyanin to photosystem I of Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America, 95(13): 7339-7344.( 1) 1)
|
| [20] |
Hippler M, Drepper F, Rochaix J D, et al. 1999. Insertion of the N-terminal part of PsaF from Chlamydomonas reinhardtii into photosystem I from Synechococcus elongatus enables efficient binding of algal plastocyanin and cytochrome c6. The Journal of Biological Chemistry, 274 (7): 4180-4188.( 1) 1)
|
| [21] |
Murata N, Takahashi S, Nishiyama Y, et al. 2007. Photoinhibition of photosystem Ⅱ under environmental stress. Biochimica et Biophysica Acta, 1767 (6): 414-421.( 1) 1)
|
| [22] |
Sommer F, Drepper F, Haehnel W, et al. 2006. Identification of precise electrostatic recognition sites between cytochrome c6 and the photosystem I subunit PsaF using mass spectrometry. The Journal of Biological Chemistry, 281 (46): 35097-35103.( 2) 2)
|
| [23] |
Van Der Est A, Valieva A I, Kandrashkin Y E, et al. 2004. Removal of PsaF alters forward electron transfer in photosystem I: evidence for fast reoxidation of QK-A in subunit deletion mutants of Synechococcus sp. PCC 7002. Biochemistry, 43 (5): 1264-1275.( 1) 1)
|
| [24] |
Wöstemeyer A, Oelmüller R. 2003. The promoter of the spinach PsaF gene for the subunit Ⅲ of the photosystem I reaction center directs β-glucuronidase gene expression in transgenic tobacco roots. Implication of the involvement of phospholipases and protein kinase C in PsaF gene expression. Journal of Plant Physiology, 160 (5): 503-508.( 1) 1)
|
| [25] |
Yu Y, Li Y, Huang G, et al. 2011. PwHAP5, a CCAAT-binding transcription factor, interacts with PwFKBP12 and plays a role in pollen tube growth orientation in Picea wilsonii. Journal of Experimental Botany, 62 (14): 4805-4817.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

