文章信息
- 欧阳昆唏, 李俊成, 黄浩, 刘明骞, 陈晓阳
- Ouyang Kunxi, Li Juncheng, Huang Hao, Liu Mingqian, Chen Xiaoyang
- 团花树α扩展蛋白基因的克隆及表达分析
- Molecular Cloning and Expression Analysis of α-Expansin Genes in Anthocephalus chinensis
- 林业科学, 2013, 49(9): 62-71
- Scientia Silvae Sinicae, 2013, 49(9): 62-71.
- DOI: 10.11707/j.1001-7488.20130909
-
文章历史
- 收稿日期:2012-12-24
- 修回日期:2013-04-14
-
作者相关文章
作为一类细胞壁蛋白,扩展蛋白(Expansins,EXP)以独特方式诱导依赖pH的细胞壁的伸展和膨压的松弛(Cosgrove et al.,2002)。尽管扩展蛋白作用于细胞壁的真正机制模式还不清楚,但有许多证据表明这类蛋白是通过打断细胞壁多糖类物质之间的非共价键(氢键),允许细胞壁多聚物在细胞膨压作用下蠕动,导致细胞快速伸展,从而促进植物生长(Cosgrove,2000; Cosgrove et al.,2002; Gray-Mitsumune et al.,2008; 2004; Hutchison et al.,1999; Lee et al.,2002; McQueen-Mason et al.,1994;Wang et al.,2011)。
自从McQueen-Mason等(1992)第1次从黄瓜(Cucumis sativus)下胚轴中发现2个扩展蛋白以来,在各种不同植物中,克隆到越来越多的扩展蛋白基因,如: 拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、毛果杨(Populus trichocarpa)、木瓜(Carica papaya)、玉米(Zea mays)和烟草(Nicotiana tabacum)(http://homes.bio.psu.edu/expansins/genes.htm)。根据系统发育关系,扩展蛋白分为4类,分别是:α-expansin(EXPA),β-expansin(EXPB),expansin-likeA(EXPLA)和expansin-like B(EXPLB)(Kende et al.,2004)。已有的研究主要集中于EXPA和EXPB(Lee et al.,2001; Sampedro et al.,2005),而且表明扩展蛋白参与植物生长的许多过程,包括根和叶片的生长(Pien et al.,2001)、纤维伸长(Ruan et al.,2001)、果实成熟(Sane et al.,2005)、种子萌发(Chen et al.,2000)、组织脱落(Cho et al.,2000)以及柱头的花粉管侵入(Dai et al.,2007)等。
团花树( Anthocephalus chinensis)是茜草科(Rubiaceae)团花属植物,常绿大乔木,广泛分布于南亚和东南亚(罗献瑞等,1999)。由于其生长迅速,在菲律宾被称颂为“宝石树”(Fox,1971),并在1972年的世界林业大会上被称为“奇迹树”。在团花树中,扩展蛋白基因的克隆已有报道(欧阳昆唏等,2010),但对其整个家族的了解知之甚少。本文应用基因组步移,在线基因全长分析及3’RACE技术克隆到扩展蛋白基因的其他成员,并对其在幼嫩的不同组织的表达模式进行分析,为研究扩展蛋白在团花树生长过程中的不同作用奠定基础。
1 材料与方法 1.1 材料团花树生长于华南农业大学科研基地,3~5年生,取处于生长旺盛的幼嫩组织,侧芽、嫩叶及胸径处的形成层,液氮速冻,-80 ℃保存备用。形成层组织取样按照Gray-Mitsumune等(2004)的方法。而幼嫩的根采自温室中培养至1 m高的实生苗。Taq DNA聚合酶、LA Taq 聚合酶、dNTP、PMD19-T 载体、3’RACE 试剂盒、第1条链cDNA 合成试剂盒、限制性内切酶和SYBR Premix Ex Taq Ⅱ购自 TaKaRa 公司; T4 DNA Ligase与DNaseⅠ购自NEB 公司; 植物DNA、总RNA 提取试剂购自Promega; DNA凝胶回收试剂盒、感受态大肠杆菌菌株DH5α购于天根生物技术有限公司; 用Primer5设计的所有引物(表 1)由上海生工生物公司合成; 测序由深圳华大生物技术有限公司完成。
|
|
以团花树形成层为材料,提取基因组DNA。以生长旺盛的4个组织,侧芽、嫩叶、形成层及嫩根为材料,分别提取总RNA,经DNaseⅠ处理,分别按照PrimeScript® Ⅱ1st str and cDNA Synthesis Kit合成cDNA第1条链,用于基因全长的克隆; 按照PrimeScript® RT Master Mix合成用于荧光定量的cDNA及根据3’RACE试剂盒合成用于3’RACE的cDNA。
1.2.2基因组步移文库的构建用限制性内切酶AluⅠ、DraⅠ、EcoRⅤ、HaeⅢ、RsaⅠ、SspⅠ和StuⅠ对提取的DNA进行酶切、回收,用于构建不同的基因组步移文库。制备文库所用平末端接头的50 μL 反应体系: 5 μL Long Adaptor(100 μmol·L-1),5 μL Short Adaptor(100 μmol·L-1),1.7 μL NaCl(3 mol·L-1),3 8.3 μL ddH2O; 其反应条件为: 95 ℃ 5 min,室温下自然冷却。8 μL步移文库构建的反应体系为: 4.7 μL 酶切产物,2 μL 上述已制备好的平末端接头,0.5 μL T4 DNA Ligase,0.8 μL 10×Ligation Buffer; 其反应条件为: 16 ℃连接过夜,于65℃灭活10 min,加入32 μL ddH2O稀释。
1.2.3 扩展蛋白基因保守区的克隆、上游和下游基因组步移、开放阅读框(ORF)的在线预测及3’RACE扩展蛋白保守区的扩增按照欧阳昆唏等(2010)方法,以DNA为模板,72 ℃延伸由1 min改为1.5 min,其他都一致。根据扩增到的保守区设计向上游和下游步移的巢式PCR所需引物。将扩增到的保守区、上游和下游序列在DNAMAN 6.0软件中进行拼接,利用在线分析工具(http://linux1.softberry.com/berry.phtml?topic=fgenesh&group=programs&subgroup=gfind, http://www.cbs.dtu.dk/services/NetGene2),对拼接到的DNA序列的ORF、内含子与外显子进行预测。因为在不同物种之间,EXPA的相似性极高(Sampedro et al.,2005),所以将预测得到的ORF编码的氨基酸序列在NCBI中进行比对以进一步确认ORF的准确性,并设计引物扩增ORF。用扩增到的ORF序列与基因组进行比较,分析在ORF中的内含子和外显子。在此基础上,按照3’RACE试剂盒的说明,获得各个EXPA基因的3’端非编码区(3’Untranslated Region,3’UTR)。所有扩增所用引物见表 1。
1.2.4基因AcEXPA1-16所编码的氨基酸序列的信号肽预测、比对及系统进化树分析基因AcEXPA1-16所编码的氨基酸序列的信号肽由在线分析工具SignalP软件(http://www.cbs.dtu.dk/services/SignalP/)进行预测。
采用在线工具EMBL-EBI Web server(http://www.ebi.ac.uk/Tools/msa/clustalw2/)的设定参数(Weight Matrix,Gonnet; Gap Opening Penalty,10.0; Gap Extension Penalty,0.2; Residue-specificPenalty,ON; Hydrophilic Penalty,ON)对16个扩展蛋白基因所编码的氨基酸序列进行比对,再用在线工具BOXSHADE(http://www.ch.embnet.org/software/BOX_form.html)观察比对结果。
利用在线工具EMBL-EBI Web server对团花树的16个EXPA基因和其他植物的EXPA基因所编码的氨基酸序列进行多重比较,然后按照Gray-Mitsumune等(2004)的方法,采用MEGA 5软件(Tamura et al.,2011)对比对结果构建系统进化树。
1.2.5基因AcEXPA1-16的RT-qPCR分析以团花树中的环孢素A受体基因AcCyP(GenBank注册号JX902587)为内参,对扩展蛋白基因AcEXPA1-16在侧芽、嫩叶、形成层及嫩根中的表达量进行RT-qPCR分析。RT-qPCR分析在CFX96(BIO-RAD)定量PCR仪上运行。为了确保扩增子的特异性,基因AcEXPA1-16所用的定量引物都设计在3’UTR上,并对扩增子进行测序。定量所用引物见表 1。
2 结果与分析 2.1基因组步移文库的构建由于团花树基因组DNA比较复杂且未见报道,所以本试验采用7种平末端限制性内切酶AluⅠ、DraⅠ、EcoRⅤ、HaeⅢ、RsaⅠ、SspⅠ和StuⅠ进行酶切。根据图 1的电泳分析结果可以看出,在所使用的限制性内切酶中,AluⅠ、HaeⅢ、RsaⅠ和SspⅠ所产生的DNA片段弥散均匀,DraⅠ和EcoRⅤ次之,而StuⅠ最差,而且基因组条带清晰可见。这也说明在团花树的基因组DNA中,StuⅠ酶切位点很少,而前6种酶切位点较多且均匀分布。因此采用DNA凝胶回收试剂盒对前6种酶切产物进行过柱回收并与平末端接头连接构建6种步移文库。
 |
图 1 基因组DNA 的酶切结果
Fig. 1 The results of genomic DNA cleavage
|
以DNA为模板,按照欧阳昆唏等(2010)的方法,PCR产物电泳结果见图 2。对图 2中的3个不同大小的条带分别进行凝胶回收、克隆并转化感受态细胞DH5α,各自筛选30个阳性克隆送去测序。排除重复序列,找到16条不同的保守区序列,包括已知的AcEXPA1(GenBank注册号FJ417847),并经BLAST比对进一步确认。根据获得的15条保守序列,设计向上游和下游步移的引物扩增两端未知序列。拼接这3段序列并在线分析是否已获得基因的ORF,并用引物扩增ORF,再按照3’RACE试剂盒方法获得3’UTR序列。根据Kende等(2004)的命名法则,把这新获得的15个EXPA基因命名为AcEXPA2-16,GenBank的注册号为JF922686-JF922700,其相对应的基因组DNA序列的注册号为JF922701-JF922715。
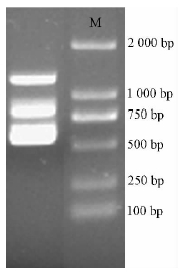 |
图 2 兼并引物的扩增产物
Fig. 2 PCR products amplified by the degenerated primers
|
表 2显示了基因AcEXPA1-16的序列的具体信息,包括cDNA长度、编码的氨基酸长度及所预测的信号肽氨基酸长度。基因AcEXPA1-16所编码的氨基酸长度从240AA到258AA不等,而预测的信号肽的氨基酸长度有19AA到28AA。
|
|
通过基因AcEXPA1-16所编码的氨基酸序列比对,从图 3可以发现编码的16个团花树EXPA蛋白都含有EXPA蛋白的主要保守特征,包括N端的信号肽、8个半胱氨酸(cysteine,C)残基、1个组氨酸域(His-Phe-Asp,HFD)和羧基末端的4个保守色氨酸(tryptophan,W)残基,而在AcEXPA12的第1个W被亮氨酸(leucine,L)所取代。
 |
图 3 AcEXPA1-16预测的蛋白序列比对结果
Fig. 3 Alignment of the predicted protein sequences of AcEXPA1-16
下划线表示N端的信号肽,字母C、W以及HFD各自表示保守的半胱氨酸、色氨酸残基和1个组氨酸( His_Phe_Asp,HFD)域,方框表示成熟蛋白在N端的RIPGV基序及C端的KNFRV基序。 The N-terminal signal peptides are underlined. The conserved Cys, Trp residues and His-Phe-Asp motif residues are indicated by the letter C, W, and HFD respectively. RIPGV and KNFRV motifs at both the N and C end of mature proteins are boxed. |
对AcEXPA1-16基因组序列与cDNA 序列进行比较。图 4显示基因AcEXPA1-16编码序列和内含子/外显子结构模式极其保守,但内含子长度和3’UTR序列存在极大差异。内含子1长度变幅从AcEXPA7的85 bp到AcEXPA8的404 bp; 而内含子2最短的只有98 bp,最长为1 524 bp; 且通过比对发现内含子之间和3’UTR之间的序列各自存在很高的特异性。
 |
图 4 团花树EXPA家族的结构示意
Fig. 4 Schematic representation of the structure of the EXPA family in A. chinensis
黑色和灰色框分别表示外显子和3’UTR; 连接2个框之间的线条表示内含子; 并且在图中标出了起始密码子、终止密码子和各个片段的长度。 Exons and UTRs were indicated as black and grey boxes, respectively, and lines between boxes indicated as introns. The start code, stop code and length of fragment were labeled in the figure. |
根据已有的报道(Cosgrove,1998; Gray-Mitsumune et al.,2004; Link et al.,1998),EXPA蛋白由4个亚家族组成。对AcEXPA1-16与其他植物的24个EXPA蛋白进行系统进化树构建(图 5)。由图可知,在团花树中有4个EXPA蛋白属于A亚家族,分别是AcEXPA1、AcEXPA5、AcEXPA8和AcEXPA9。这类亚家族蛋白拥有独特的保守序列,在成熟蛋白的N端存在RIPGV氨基酸序列,而在C端存在KNFRV氨基酸序列。由相关报道显示,这2个特有的基序是A亚家族的标志序列并且这类亚家族在次生木质部大量存在(高燕,2011; Gray-Mitsumune et al.,2004; Wang et al.,2011)。AcEXPA2、AcEXPA3、AcEXPA4、AcEXPA6与AcEXPA11属于B亚家族,AcEXPA7、AcEXPA10、AcEXPA12、AcEXPA13、AcEXPA14和AcEXPA15属于C亚家族,而属于D亚家族的只有1个AcEXPA16。
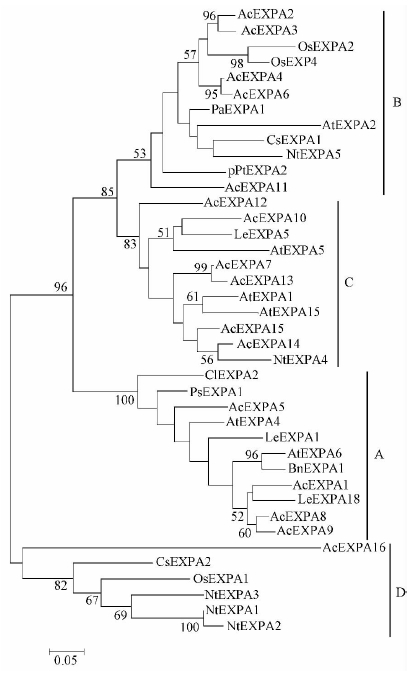 |
图 5 α-expansin 基因的系统进化树分析
Fig. 5 Phylogenetic analysis of α-expansin genes
团花树Anthocephalus chinensis: AcEXPA1(FJ417847),AcEXPA2-16 (JF922686-JF922700); 拟南芥Arabidopsis thaliana: AtEXPA1 (U30476),AtEXPA2 (U30481),AtEXPA4 (129526),AtEXPA5 (U30478),AtEXPA6(U30480),AtEXPA15(126361); 油菜Brassica napus: BnEXPA1 (AJ000885); 杉木Cunninghamia lanceolata: ClEXPA2(EF158813); 黄瓜Cucumis sativus: CsEXPA1 (U30382), CsEXPA2 (U30460); 蕃茄Lycopersicon esculentum: LeEXPA1 (U82123),LeEXPA5 (AF059489),LeEXPA18 (AJ004997); 烟草 Nicotiana tabacum: NtEXPA1 (AF049350),NtEXPA2 (AF049351), NtEXPA3 (AF049352),NtEXPA4 (AF049353),NtEXPA5 (AF049354); 水稻Oryza sativa: OsEXPA1 (Y07782),OsEXPA2 (U30477),OsEXPA4 (U85246); 杏Prunus armeniaca: PaEXPA1 (U93167); 豌豆Pisum sativum: PsEXPA1(X85187); 火炬松Pinus taeda: pPtEXPA2(U64890). |
为筛选到合适的内参引物,从图 6A可以看出,在4个泳道中引物对AcCyP-Q所扩增的产物条带清晰单一亮度相似,无二聚体; 图 6B显示了其溶解峰聚拢且无杂峰;与标准曲线图 6C相比,它的扩增效率为103.7%。
 |
图 6 内参引物对AcCyP-Q的鉴定
Fig. 6 Identification of reference gene pair AcCyP-Q
A. 凝胶电泳检测; B. 溶解曲线; C. 标准曲线。1,2,3,4,CK -分别表示以芽、嫩叶、形成层、嫩根100 ng cDNA 为模板及阴性对照的PCR 产物泳道。 A. Gel electrophoresis; B. The melting curve; C. The standard curve. 1, 2, 3, 4 and CK- represent lane of PCR product amplified with 100 ng bud, tender leaf, cambium region, young root cDNA as template and negative control respectively. |
从图 7可以看出,AcEXPA1-16 在4个不同的组织中的表达存在极大的差异,在形成层中AcEXPA8的相对表达量最高,AcEXPA1和AcEXPA15次之,其余表达很低或不表达。在嫩叶中,AcEXPA4、AcEXPA6、AcEXPA8的相对表达量在20倍以上。在4个组织中,AcEXPA3和AcEXPA12均极少表达,而AcEXPA16仅在嫩根中表达。
 |
图 7 AcEXPA1-16基因在团花树的芽、嫩叶、形成层和嫩根4 个快速生长组织中的表达模式分析
Fig. 7 Analysis of expression pattern of 16 α-expansin
genes in four rapid growth tissues, bud, tender leaf, cambium
region and young root in A. chinensis by real-time PCR
|
进化树分析显示了EXPA被分为的4个分支(图 5)与Link和Cosgrove(1998)报道的结果相似,并且他们也推测在同一个分支中的EXPA在某方面的功能可能相似。在进化树中,AcEXPA1、AcEXPA5、AcEXPA8和AcEXPA9同属于A亚家族,而这个亚家族含有多个在初生和次生维管组织中高表达的EXPA 基因,如AtEXPA4和AtEXPA6(S. Turner,data published online at http://affymetrix.arabidopsis.info/narrays/experimentbrowse.pl),以及LeEXPA18基因(Reinhardt et al.,1998)和ClEXPA2基因(高燕,2011; Wang et al.,2011)。这个亚家族中的所有EXPA蛋白都拥有独特的保守序列,在成熟蛋白的N 端存在RIPGV氨基酸序列,而在C端存在KNFRV氨基酸序列,并且作为A亚家族的标志(图 3)。AcEXPA2、AcEXPA3、AcEXPA4、AcEXPA6和AcEXPA11属于B 亚家族。而在这个亚家族中的其他成员在生长的根和下胚轴中表达量很高,如:火炬松(Pinus taeda)在响应吲哚丁酸处理时,pPtEXPA2在不定根形成的过程中和下胚轴的基部存在表达(Hutchison et al.,1999); 黄瓜CsEXPA1在下胚轴存在表达(Shcherban et al.,1995)。AcEXPA7、AcEXPA10、AcEXPA12、AcEXPA13、AcEXPA14和AcEXPA15则属于C亚家族,拟南芥的AtEXPA1与AtEXPA5以及蕃茄(Lycopersicon esculentum)的LeEXPA5同属于这一分支。在拟南芥中,AtEXPA1基因表达并加速了气孔的开放(Zhang et al.,2011),而AtEXPA5主要在拟南芥的地上表达(Park et al.,2010)。已知蕃茄的LeEXPA5基因在果实膨大期有所表达,并且果实在尺寸最大
的绿色期表达量最高,而在此后的果实成熟早期,其表达量又下降(Brummell et al.,1999)。与AcEXPA16同属于D亚家族的OsEXPA1基因,在水稻根的生长区存在很高的表达(Cho et al.,1997),而在本文中,AcEXPA16仅在根中特异表达(图 7)。由于团花树缺乏遗传信息,因此在本试验中,曾选择与其近源物种的多个常用看家基因为内参,如: Actin(DQ531565)、Ubiquitin(AF297089)、GAPDH(GW453007)、Tubulin(EU007434)和EF1-alpha(AF120093),但均未找到合适的引物。而环孢素A受体基因Cyclophilin的引物对AcCyP-Q符合荧光定量的要求,在缺乏遗传信息的团花树中可作为内参基因用于分析基因在不同组织的相对表达(图 6)。
EXP基因具有独特表达模式以满足各种不同的生理活性。如,在咖啡(Coffea arabica)的不同发育阶段,3个EXPA基因在外胚乳、胚乳和果皮的3个不同组织中的表达模式各不相同(Budzinski et al.,2011); 在番茄种子的发育和萌发阶段,2个EXPA基因在不同的胚胎组织中存在不同的表达和定位(Chen et al.,2001); 在水稻的叶片、茎秆和根中4个EXPA基因的表达模式也不同(Cho et al.,1997)。此外,EXP基因独特的表达模式受到启动子的调控(Cosgrove,1998)。在拟南芥中,根据报告基因分析,许多EXP基因的启动子表现出各种不同的组织特异性表达(Cho et al.,2000; Cosgrove et al.,1998; Sun et al.,2011),并且在拟南芥根毛细胞中特异表达的AtEXPA7和AtEXPA18基因的启动子区含有根毛特异的cis元件(RHEs)(Kim et al.,2006)。在本文中,AcEXPA8在形成层中的表达量极高,存在明显的组织特异性(图 7)。表明,AcEXPA8拥有一个表达特异的启动子,能在形成层的特定环境下调控基因表达,包括生长发育信号和激素。
新形成的叶片需要经历细胞的分裂和增大,从而长成完全大小的叶片。已有记录显示,EXP基因的表达模式和活性与叶片增大相关。Keller和Cosgrove(1995)从蕃茄生长叶片中提取的扩展蛋白能够诱导黄瓜幼苗细胞壁的伸展。在拟南芥生长的叶片中,AtEXPA10优先表达,而在Cho和Cosgrove(2000)用AtEXPA10构建正义和反义载体转化拟南芥的试验中发现,与对照植株相比,正义转基因植株拥有较大的叶片,而反义植株的叶片较小。Muller等(2007)也从玉米生长的叶片中鉴定出与叶片生长相关的19个EXP基因,而它们又被分为与细胞分裂、叶片伸展及细胞壁分化相关的3组,并认为它们在叶片的增宽和伸长中扮演着不同的角色。在本文中,AcEXPA4、AcEXPA6、AcEXPA15、AcEXPA14和AcEXPA10在嫩叶中都有大量表达(图 7),这些表明这些AcEXPA基因可能通过协同作用在团花树叶片的生长中起着重要的作用。
EXPA蛋白A亚家族中其他物种的EXPA基因多在初生和次生维管组织中高表达,与这些基因相似,AcEXPA8基因在形成层中也存在高表达(图 7)。这些表明AcEXPA8基因在团花树木质部发生的过程中可能起到重要的作用。并且,已有许多研究证实了EXPA与纤维素的新陈代谢及纤维素含量的增加有关(Gray-Mitsumune et al.,2008; Wang et al.,2011; Zenoni et al.,2004)。这为在团花树中研究α-expansin基因功能与木质部的生长和材质的关系提供了基础,并最终为林木分子育种提供潜在的候选基因。
| [1] |
高燕. 2011. 杉木木材形成过程扩展蛋白基因的克隆与表达分析. 林业科学,47(11):44-51.( 2) 2)
|
| [2] |
罗献瑞,高蕴璋,陈伟球,等. 1999. 茜草科//罗献瑞. 中国植物志. 北京: 科学出版社, 260-262.( 1) 1)
|
| [3] |
欧阳昆唏,李娜,张东方,等. 2010. 团花树形成层扩展蛋白基因cDNA的克隆和序列分析. 热带亚热带植物学报, 18(2):151-158.( 3) 3)
|
| [4] |
Brummell D A,Harpster M H,Dunsmuir P. 1999. Differential expression of expansin gene family members during growth and ripening of tomato fruit. Plant Mol Biol,39(1):161-169.( 1) 1)
|
| [5] |
Budzinski I G,Santos T B,Sera T,et al. 2011. Expression patterns of three alpha-expansin isoforms in Coffea arabica during fruit development. Plant Biology, 13(3):462-471.( 1) 1)
|
| [6] |
Chen F,Dahal P,Bradford K J. 2001. Two tomato expansin genes show divergent expression and localization in embryos during seed development and germination. Plant Physiol, 127(3):928-936.( 1) 1)
|
| [7] |
Chen F,Bradford K J. 2000. Expression of an expansin is associated with endosperm weakening during tomato seed germination. Plant Physiol, 124(3):1265-1274.( 1) 1)
|
| [8] |
Cho H T,Cosgrove D J. 2000. Altered expression of expansin modulates leaf growth and pedicel abscission in Arabidopsis thaliana. Proc Natl Acad Sci Usa,97(17):9783-9788.( 3) 3)
|
| [9] |
Cho H T,Kende H. 1997. Expression of expansin genes is correlated with growth in deepwater rice. Plant Cell,9(9):1661-1671.( 2) 2)
|
| [10] |
Cosgrove D J. 1998. Cell wall loosening by expansins. Plant Physiol, 118(2):333-339.( 2) 2)
|
| [11] |
Cosgrove D J. 2000. Loosening of plant cell walls by expansins. Nature,407(6802):321-326.( 1) 1)
|
| [12] |
Cosgrove D J,Li L C,Cho H T,et al. 2002. The growing world of expansins. Plant Cell Physiol,43(12):1436-1444.( 2) 2)
|
| [13] |
Cosgrove D J,Tatyana S,Durachko D M,et al. 1998. Highly specific and distinct expression patterns for two alpha-expansin genes in Arabidopsis. 9th International Conference on Arabidopsis Research, Madison, Wisconsin, USA.( 1) 1)
|
| [14] |
Dai S,Chen T,Chong K,et al. 2007. Proteomics identification of differentially expressed proteins associated with pollen germination and tube growth reveals characteristics of germinated Oryza sativa pollen. Molecular & Cellular Proteomics,6(2):207-230.( 1) 1)
|
| [15] |
Fox J E D. 1971. Anthocephalus chinensis, the laran tree of sabah. Econ Bot, 25(3):221-233.( 1) 1)
|
| [16] |
Gray-Mitsumune M,Blomquist K,Mcqueen-Mason S,et al. 2008. Ectopic expression of a wood-abundant expansin pttexpa1 promotes cell expansion in primary and secondary tissues in aspen. Plant Biotechnology Journal,6(1):62-72.( 2) 2)
|
| [17] |
Gray-Mitsumune M,Mellerowicz E J,Abe H,et al. 2004. Expansins abundant in secondary xylem belong to subgroup A of the α-expansin gene family. Plant Physiol, 135(3):1552-1564.( 5) 5)
|
| [18] |
Hutchison K W,Singer P B,Mcinnis S,et al. 1999. Expansins are conserved in conifers and expressed in hypocotyls in response to exogenous auxin. Plant Physiol, 120(3):827-832.( 2) 2)
|
| [19] |
Keller E,Cosgrove D J. 1995. Expansins in growing tomato leaves. Plant J,8(6):795-802.( 1) 1)
|
| [20] |
Kende H,Bradford K,Brummell D,et al. 2004. Nomenclature for members of the expansin superfamily of genes and proteins. Plant Mol Biol,55(3):311-314.( 2) 2)
|
| [21] |
Kim D W,Lee S H,Choi S B,et al. 2006. Functional conservation of a root hair cell-specific cis-element in angiosperms with different root hair distribution patterns. Plant Cell, 18(11):2958-2970.( 1) 1)
|
| [22] |
Lee Y,Choi D,Kende H. 2001. Expansins: ever-expanding numbers and functions. Curr Opin Plant Biol,4(6):527-532.( 1) 1)
|
| [23] |
Lee Y,Kende H. 2002. Expression of α-expansin and expansin-like genes in deepwater rice. Plant Physiol, 130(3):1396-1405.( 1) 1)
|
| [24] |
Link B M,Cosgrove D J. 1998. Acid-growth response and α-expansins in suspension cultures of bright yellow 2 tobacco. Plant Physiol, 118(3):907-916.( 2) 2)
|
| [25] |
McQueen-Mason S,Durachko D M,Cosgrove D J. 1992. Two endogenous proteins that induce cell wall extension in plants. Plant Cell,4(11):1425-1433.( 1) 1)
|
| [26] |
McQueen-Mason S,Cosgrove D J. 1994. Disruption of hydrogen bonding between plant cell wall polymers by proteins that induce wall extension. Proc Natl Acad Sci USA,91(14):6574-6578.( 1) 1)
|
| [27] |
Muller B,Bourdais G,Reidy B,et al. 2007. Association of specific expansins with growth in maize leaves is maintained under environmental, genetic, and developmental sources of variation. Plant Physiol, 143(1):278-290.( 1) 1)
|
| [28] |
Park C H,Kim T W,Son S H,et al. 2010. Brassinosteroids control AtEXPA 5 gene expression in Arabidopsis thaliana. Phytochemistry,71(4):380-387.( 1) 1)
|
| [29] |
Pien S,Wyrzykowska J,Mcqueen-Mason S,et al. 2001. Local expression of expansin induces the entire process of leaf development and modifies leaf shape. Proc Natl Acad Sci USA,98(20):11812-11817.( 1) 1)
|
| [30] |
Reinhardt D,Wittwer F,Mandel T,et al. 1998. Localized upregulation of a new expansin gene predicts the site of leaf formation in the tomato meristem. Plant Cell, 10(9):1427-1437.( 1) 1)
|
| [31] |
Ruan Y L,Llewellyn D J,Furbank R T. 2001. The control of single-celled cotton fiber elongation by developmentally reversible gating of plasmodesmata and coordinated expression of sucrose and K+ transporters and expansin. Plant Cell, 13(1):47-60.( 1) 1)
|
| [32] |
Sampedro J,Cosgrove D J. 2005. The expansin superfamily. Genome Biology,6(12):242.( 2) 2)
|
| [33] |
Sane V A,Chourasia A,Nath P. 2005. Softening in mango (Mangifera indica cv. Dashehari) is correlated with the expression of an early ethylene responsive, ripening related expansin gene, MiExpA 1. Postharvest Biol Tec,38(3):223-230.( 1) 1)
|
| [34] |
Shcherban T Y,Shi J,Durachko D M,et al. 1995. Molecular cloning and sequence analysis of expansins—a highly conserved, multigene family of proteins that mediate cell wall extension in plants. Proc Natl Acad Sci USA,92(20):9245-9249.( 1) 1)
|
| [35] |
Sun T,Zhang Y,Chai T. 2011. Cloning, characterization, and expression of the BjEXPA 1 gene and its promoter region from Brassica junceal. Plant Growth Regul,64(1):39-51.( 1) 1)
|
| [36] |
Tamura K,Peterson D,Peterson N,et al. 2011. Mega5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 28(10):2731-2739.( 1) 1)
|
| [37] |
Wang G,Gao Y,Wang J,et al. 2011. Overexpression of two cambium-abundant chinese fir (Cunninghamia lanceolata) α-expansin genes ClEXPA 1 and ClEXPA 2 affect growth and development in transgenic tobacco and increase the amount of cellulose in stem cell walls. Plant Biotechnology Journal,9(4):486-502.( 4) 4)
|
| [38] |
Zenoni S,Reale L,Tornielli G B,et al. 2004. Downregulation of the Petunia hybrida α-expansin gene PhEXP 1 reduces the amount of crystalline cellulose in cell walls and leads to phenotypic changes in petal limbs. Plant Cell, 16(2):295-308.( 1) 1)
|
| [39] |
Zhang X Q, Wei P C,Xiong Y M,et al. 2011. Overexpression of the arabidopsis α-expansin gene AtEXPA 1 accelerates stomatal opening by decreasing the volumetric elastic modulus. Plant Cell Rep,30(1):27-36.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

