文章信息
- 黄海燕, 杜红岩, 乌云塔娜, 刘攀峰
- Huang Haiyan, Du Hongyan, Wuyun Tana, Liu Panfeng
- 基于杜仲转录组序列的SSR分子标记的开发
- Development of SSR Molecular Markers Based on Transcriptome Sequencing of Eucommia ulmoides
- 林业科学, 2013, 49(5): 176-181
- Scientia Silvae Sinicae, 2013, 49(5): 176-181.
- DOI: 10.11707/j.1001-7488.20130523
-
文章历史
- 收稿日期:2012-09-13
- 修回日期:2012-11-07
-
作者相关文章
简单重复序列(simple sequence repeat,SSR),又称微卫星,是广泛存在于真核和原核生物基因组中的1~6个核苷酸串联重复单元,研究发现基因组中平均每50 kb 就有1个SSR(Morgante et al.,1993;Kalia et al.,2011),SSR标记主要包括基因组SSR和表达序列标签SSR(EST-SSR)。SSR标记具有多态性高、重复性高、共显性、易检测、操作简单、无放射、所用时间较短、覆盖面广等优点,在DNA指纹图谱的构建、遗传多样性分析、基因定位、分子标记辅助育种等方面得到广泛应用(Morgante et al.,1993;Milee et al.,2008;罗冉等,2010)。SSR分子标记引物的开发是进行SSR分子标记研究的前提,从基因组开发SSR标记的引物需要cDNA文库构建、SSR克隆筛选、测序等步骤,其步骤复杂、工作量大、开发成本高等,从而限制了gSSR标记的发展(程小毛等,2011)。表达序列标签(ExpressedSequence Tag,EST)来源于基因的转录区,直接反映基因的表达信息,虽然近年来在公共数据库中EST的数量剧增,但与转录组数据相比EST 数据还是相当缺乏。随着高通量测序技术的快速发展和测序成本的降低,利用新一代高通量测序技术对植物全基因组范围内进行测序,并产生丰富的转录组数据,其中包含了大量的EST 序列(Simon et al.,2009)。利用转录组序列开发SSR标记既有EST-SSR标记的优点,同时其海量的数据为SSR标记的开发提供了比EST-SSR更全面的信息,提高了遗传多样性和分子标记辅助育种研究的准确性。
杜仲(Eucommia ulmoides)为第四纪冰川侵袭后残留下的古老树种,雌雄异株。中国为现存杜仲的原产地,杜仲自然分布在我国的中亚热带到暖温带地区。杜仲(2n=34)是我国特有的优质天然橡胶和中药资源,杜仲胶作为战略资源具有十分广阔的应用前景(杜红岩等,2000;2009;杜红岩,2010;李芳东等,2001)。我国拥有世界99%的杜仲资源,具有独特的资源优势。但目前杜仲的遗传背景并不清晰,这给杜仲杂交育种、种质资源保存、新种质挖掘等带来了很大困难,RAPD,AFLP,ISSR等现代分子生物学手段为杜仲遗传多样性、种质创新和品种改良等研究带来很大便利(张檀等,1993;王瑷琦等,2006;邓建云等,2006;Wu et al.,2011;Kalia et al.,2011)。目前关于杜仲SSR标记,只有Deng等(2006)利用FIASCO 法开发了19对具有多态性的SSR引物,但杜仲为单科单种物种,经作者验证这些引物还远远难以满足研究需要。在GenBank 中公布的杜仲EST 序列只有29条、Nucleotide 只有232条(截止2012年7月),这在很大程度上限制了杜仲SSR标记的开发和利用。为此,中国林业科学研究院经济林研究开发中心利用高通量测序技术成功获得杜仲叶和果实的转录组数据,本文对这些数据进行了系统分析,最终找到了适用于杜仲SSR标记研究的引物,为进行杜仲遗传多样性分析、雌雄株鉴定、分子标记辅助育种等奠定了基础。
1 材料与方法 1.1 材料转录组测序样本为采自中国林业科学研究院经济林研究开发中心院内‘华仲6 号’(Eucommia ulmoides‘Huazhong No.6’)的叶子和幼果,液氮速冻后送华大基因公司(北京)进行转录组测序,共包含49 610条Unigenes。
SSR扩增所用的8份杜仲样品于2012年5月取自中国林业科学研究院经济林研究开发中心杜仲基因库(表 1)。样品取健壮、无病虫害的当年生杜仲嫩叶,液氮速冻后保存于-80 ℃冰箱中备用。
|
|
1)DNA的提取采用天根生物公司(北京)新型植物基因组提取试剂盒提取杜仲基因组DNA,用1%的琼脂糖凝胶电泳检测DNA的质量,-20 ℃保存备用。
2)杜仲转录组SSR位点的鉴别及SSR引物设计对杜仲叶和果实的49 610条Unigenes 利用在线微卫星位点扫描工具SSRIT(simple sequencerepeat identification tool)(http://www.gramene.org/db/searches/ssrtool)查找SSR位点,查找标准:二、三、四、五、六核苷酸的最小重复次数分别为9、7、5、5、5 次,并且SSR位点侧翼序列长度≥50 bp。
用Primer5和Oligo7软件进行引物设计和评价。设计引物时设置的主要参数为:GC 含量40%~70%,退火温度58~63 ℃,引物长18~25bp,预期扩增产物长度100~500 bp,且无二级结构和二聚体。引物命名为EU 加序号,如EU-1。
3)SSR引物筛选挑选85对引物由金斯瑞生物技术公司(江苏南京)合成以用于扩增,2×Taq PCRMasterMix 购自天根生物公司(北京)。PCR反应体系为20 μL:DNA模板25 ng,2×Taq PCRMasterMix9 μL,上下游引物浓度0.4 μmol·L-1,去离子水补足20 μL。PCR反应在Mastercycler gradient(eppendorf)上进行扩增:94 ℃预变性4min;再进行35个循环,每个循环包括94 ℃变性30 s,60 ℃复性30 s,72 ℃延伸1min;最后72 ℃延伸10 min。
PCR扩增产物首先用2%的琼脂糖凝胶电泳检测有无特异性条带,有特异性条带的再用6%的非变性聚丙烯酰胺凝胶在垂直板电泳仪(美国Bio-Rad)中进行电泳分离,200 V 电压下电泳200~240min,电泳后用Gelred 荧光染料(美国)染色30~60min,用复日FR-980H 生物电泳图像分析系统(上海复日)进行观察、拍照。
2 结果与分析 2.1 杜仲转录组中SSR位点的分布特点在杜仲转录组的49 610条Unigene 序列中发现1 442个SSR位点,分布在1 334条Unigene 中,发生频率(含有SSR的Unigene 数量与总Unigene 数量之比)为2.69%。其中有1 238条Unigene 序列只含1个SSR位点,含2个及2个以上SSR位点的Unigene序列有96条,SSR的分布频率(SSR的个数与总Unigene 的数量比)为2.90%,杜仲转录组序列中平均26.13 kb 就能发现1个SSR位点(表 2)。
|
|
杜仲转录组中SSR的主要重复类型是二核苷酸重复,占SSR总数的69.90%;其次是三核苷酸重复,占SSR总数的22.82%;四、五、六核苷酸重复类型的数量很少,总计7.28%(图 1)。
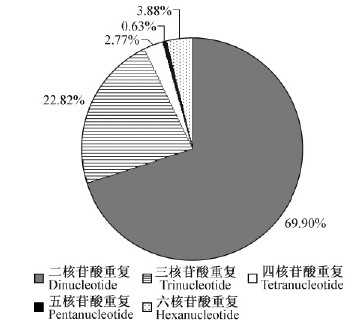 |
图 1 SSRs不同重复单元
Fig. 1 Types of SSR motifs
|
杜仲转录组SSR位点的序列长度分布从18~92个碱基不等,平均长度23 bp,二、三、四、五、六核苷酸重复的平均长度分别为23、24、22、28、32 bp(表 2)。重复序列长度18~24 bp的最多,占72.95%;其次是长度25~39 bp的序列重复,占24.89%;大于40 bp的重复序列仅2.15%(图 2)。
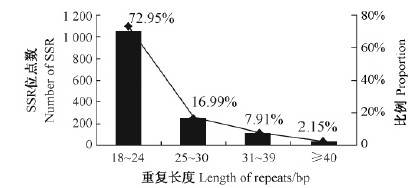 |
图 2 SSR重复长度分布
Fig. 2 Distribution of the length of repeats
|
杜仲转录组SSR重复单元的重复次数分布在5~46次之间。其中5~10 次重复的有946个SSR,占65.60%;11~15 次重复的有395个SSR,占27.39%;15~20 次重复的有82个SSR,占5.69%;大于20 次重复的仅有19个SSR,占1.32%(图 3)。二核苷酸单元最多重复46 次,三核苷酸单元最多重复17 次,四、五核苷酸单元最多重复都为8 次,六核苷酸单元最多重复7 次。
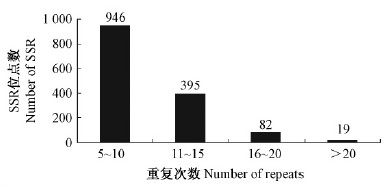 |
图 3 SSR重复次数分布
Fig. 3 Distribution of the number of repeats
|
1 442个SSR位点共包含150种重复单元,二、三、四、五、六核苷酸重复各有11、48、29、9、53种。
出现频率最高的2个重复类型是AG/TC,CT/GA,分别占总SSRs的32.73%,26.07%,共计占58.80%;其次是AC/TG和AGA/TCT,分别占总SSRs的4.79%,3.74%。在二核苷酸重复中,AG/TC,CT/GA 出现的次数最多,共占二核苷酸SSRs的84.13%;同时在二核苷酸重复中还发现了少量的CG 重复,占二核苷酸SSRs的0.10%(图 4)。
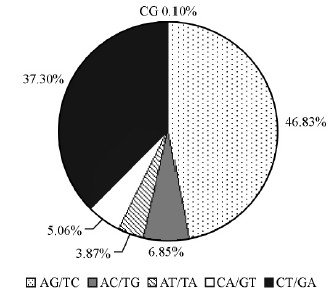 |
图 4 二核苷酸重复中不同重复单元的比例
Fig. 4 Percentage of different motifs in dinucleotide repeats
|
杜仲转录组SSRs出现的频率较低,重复单元的类型较丰富。从杜仲转录组49 610条Unigenes 中筛选出1 442个SSRs,分布于1 334条Unigenes 中,平均分布距离是26.13 kb,出现频率为2.9%,与已公布木本植物的SSR分布频率相比明显偏低。设定的SSR长度不同,SSRs的分布频率将有所变化,如,李小白等(2007)研究发现把油菜(Brassica napus)EST-SSR最小SSR长度标准由12 bp增加到20 bp,EST-SSR的频率从15.58%降低到11.61%,但仍高于杜仲转录组SSRs的出现频率。当把杜仲转录组SSR查找的标准设定为二核苷酸重复次数≥7,三核苷酸重复次数≥6,四、五、六核苷酸重复次数≥5,并且SSR位点侧翼序列长度≥50 bp时,SSR出现的频率为6.54%,平均分布距离为11.61 kb,高于杨树(Populus)14.0 kb(Cardle et al.,2000)、柑橘(Citrus)5.7 kb(Chen et al.,2006)、银杏(Ginkgobiloba)12.02 kb(樊洪泓等,2009),低于茶树(Camellia sinensis)3.68 kb(杨华等,2011)、北美鹅掌楸(Liriodendron tulipifera)8.5 kb(Xu et al.,2010)、橡胶树(Hevea brasiliensis)3.93 kb(安泽伟等,2009)等木本植物(图 5)。
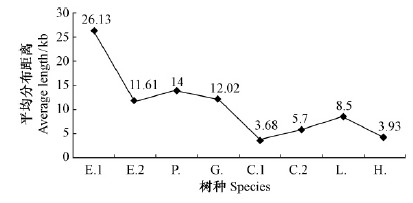 |
图 5 杜仲与其他树种SSR平均分布距离对比
Fig. 5 Comparison of the SSR average distance between Eucommia ulmoides and other tree species
|
合成85对SSR引物,SSR位点包括2~6核苷酸重复单元。选用8份差异较大的杜仲资源的核DNA为模板对85对引物进行扩增检测。85对引物中有50对引物扩增出特异性条带,扩增效率59%;其中有5对引物扩增片段与预期产物片段大小不符,剩余45对引物中有20对引物的扩增产物具有多态性(表 3),占设计引物总数23%。引物EU-12,33,69,108 在8个优良无性系之间的扩增的PAGE 效果图见图 6,4对引物的目的片段长度分别在150~200 bp,180~230 bp,100~150 bp,100~150 bp之间。
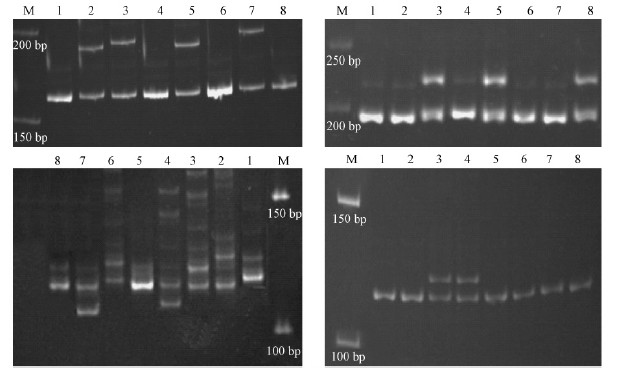 |
图 6 引物EU-12,33,69,108 在8个优良无性系间的多态性
Fig. 6 Polymorphism of primer EU-12,33,69,108 showed in different excellent clones of Eucommia ulmoides
|
|
|
杜仲转录组中微卫星的种类较为丰富,2~6核苷酸重复类型都有出现,主要重复类型为二、三核苷酸重复,占SSR总数的92.72%,四、五、六核苷酸重复类型的数量很少,总计7.28%。大多数植物的SSR主要以二、三核苷酸重复为主,但优势的重复单元有差别。杜仲转录组中最常见的SSR重复类型是二核苷酸重复,这一结果与拟南芥(Arabidopsisthaliana)、花生(Arachis hypogaea)、甜菜(Betavulgaris)、梨(Pyrus)和葡萄(Vitis vinifera)等一致(Wei et al.,2011)。在二核苷酸重复单元中AG/TC(占总SSRs的32.73%)占主导地位,这与报道的多数植物情况相同,同时也发现了一次CG 重复,表现出了明显的偏倚性。杜仲转录组SSRs出现的频率较低,这一方面由物种的特异性引起,另一方面也与搜索条件有很大的关系。
杜仲转录组SSRs出现的频率较低,但是重复单元的类型较丰富,平均分布距离为26.13 kb,出现频率为2.9%。当降低SSR查找标准,发现杜仲的出现频率有所提高,但仍低于很多木本植物的SSR出现频率。因此在进行物种间SSRs频率对比时应注意查找参数的设定,不同的查找参数其结果相差也很大,应根据需要合理设置查找参数。
在合成的85对引物中有3对引物扩增片段与预期产物片段大小不符,大于预期产物片段长度,这可能与扩增片段中插入内含子有关,属于无效扩增(Saha et al.,2004)。有多态性的20对引物的片段大小在100~400 bp,其中长度在100~250 bp之间的引物有14对,占70%。因此,建议在设计SSR标记引物时要注意引物长度控制在100~250 bp之间,多态性较高,扩增效果也较好。这20对具有多态性的引物重复单元为二、三核苷酸重复,因此在设计SSR引物时要侧重于含低级单元的序列,同时也验证了低级单元SSR的多态性普遍比高级单元的高(Dreisigacker et al.,2004)这一推断。
本研究表明利用杜仲转录组数据开发SSR标记是可行的,本研究开发的引物将为杜仲遗传多样性分析、分子标记辅助育种、育种群体的建立等奠定基础。
| [1] |
安泽伟,赵彦宏,程汉,等. 2009. 橡胶树EST-SSR 标记的开发与应用. 遗传,31(3): 311-319.( 1) 1)
|
| [2] |
程小毛,黄晓霞. 2011. SSR 标记开发及其在植物中的应用. 中国农学通报,27(5): 304-307.( 1) 1)
|
| [3] |
邓建云,李建强,黄宏文. 2006. 一株具有特异AFLP 指纹图谱的杜仲古树. 武汉植物学研究,24(6): 509-513.( 2) 2)
|
| [4] |
杜红岩,李钦,李福海. 2009. 杜仲种仁桃叶珊瑚苷含量的测定及积累规律. 林业科学研究,22(5): 744-746.( 1) 1)
|
| [5] |
杜红岩. 2010. 我国的杜仲胶资源及其开发潜力与产业发展思路. 经济林研究,28(3): 1-6.( 1) 1)
|
| [6] |
杜红岩,赵戈,卢绪奎. 2000. 论我国杜仲产业化与培育技术的发展. 林业科学研究,13(5): 554-561.( 1) 1)
|
| [7] |
樊洪泓,李廷春,李正鹏,等. 2009. 银杏EST 序列中微卫星的分布特征. 基因组学与应用生物学,28 (5): 869-873.( 1) 1)
|
| [8] |
李芳东,杜红岩. 2001. 杜仲. 北京: 中国中医药出版社.( 1) 1)
|
| [9] |
李小白,张明龙,崔海瑞. 2007. 油菜EST 资源的SSR 信息分析. 中国油料作物学报,29(1): 20-25.( 1) 1)
|
| [10] |
罗冉,吴委林,张旸,等. 2010. SSR 分子标记在作物遗传育种中的应用. 基因组学与应用生物学,29(1): 137-143.( 1) 1)
|
| [11] |
王瑷琦,黄璐琦,邵爱娟,等. 2006. 孑遗植物杜仲的遗传多样性RAPD 分析和保护策略研究. 中国中药杂志,31 (19): 1583-1586.( 1) 1)
|
| [12] |
杨华,陈琪,韦朝领,等. 2011. 茶树转录组中SSR 位点的信息分析. 安徽农业大学学报,38(6): 882-886.( 1) 1)
|
| [13] |
张檀,张康健,杨吉安,等. 1993. 杜仲脂酶同工酶地理递变及生长预测的研究. 西北林学院学报,8(4): 20-23.( 1) 1)
|
| [14] |
Cardle L,Ramsay L,Milbourne D,et al. 2000. Computational and experimental characterization of physically clustered simple sequence repeats in plants. Genetics,156(2): 847-854.( 1) 1)
|
| [15] |
Chen Chunxian,Zhou Ping,Choi Y A,et al. 2006. Mining and characterizing microsatellites from citrus ESTs. Theor Appl Genet,112(7): 1248-1257.( 1) 1)
|
| [16] |
Deng Jianyun,Liu Yifei,Huang Hongwen. 2006. Development and characterization of microsatellite markers in Eucommia ulmoides Oliver (Eucommiaceae). Molecular Ecology Notes,6 (2): 496-498.( 1) 1)
|
| [17] |
Dreisigacker S,Zhang P,Warburton M L,et al. 2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments. Crop Science,44(2): 381-388.( 1) 1)
|
| [18] |
Kalia R,Rai M,Kalia S,et al. 2011. Microsatellite markers: an overview of the recent progress in plants. Euphytica,177 (3): 309-334.( 2) 2)
|
| [19] |
Milee A,Neeta S,Harish P. 2008. Advances in molecular marker techniques and their applications in plant sciences. Plant Cell Rep,27(4): 617-631.( 1) 1)
|
| [20] |
Morgante M, Olivieri A M. 1993. PCR-amplified microsatellites as markers in plant genetics. Plant J,13(1): 175-182.( 2) 2)
|
| [21] |
Wu Mingqian,Chen Shuanglin,Wang Meixia,et al. 2011. An analysis of the genetic diversity and genetic structure of Eucommia ulmoides using inter-simple sequence repeat(ISSR)markers. African Journal of Biotechnology,10(84): 19505-19513.( 1) 1)
|
| [22] |
Xu Meng,Sun Yaguang,Li Huogen. 2010. EST-SSRs development and paternity analysis for Liriodendron spp. New Forests,40(3): 361-382.( 1) 1)
|
| [23] |
Wei Wenliang, Qi Xiaoqiong, Wang Linhai, et al. 2011. Characterization of the sesame (Sesamum indicum L.)global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers. BMC Genomics,12(1): 451.( 1) 1)
|
| [24] |
Simon S A,Zhai Jixian,Nandety R S,et al. 2009. Short-read sequencing technologies for transcriptional analyses. Annu Rev Plant Bio,60: 305-333.( 1) 1)
|
| [25] |
Saha M C,Mian M A R,Eujayl I,et al. 2004. Tall fescue EST-SSR markers with transferability across several grass species. Theor Appl Genet,109(4): 783-791.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

