文章信息
- 胡佳续, 宋传生, 林彩丽, 耿显胜, 田国忠
- Hu Jiaxu, Song Chuansheng, Lin Caili, Geng Xiansheng, Tian Guozhong
- 4种植物病害植原体病原质粒全序列测定及分子特征
- Sequencing the Full-Length DNA and the Molecular Characterization of Four Plasmids from Plant Pathogens of Phytoplasma Disease
- 林业科学, 2013, 49(4): 90-97
- Scientia Silvae Sinicae, 2013, 49(4): 90-97.
- DOI: 10.11707/j.1001-7488.20130413
-
文章历史
- 收稿日期:2012-03-31
- 修回日期:2012-10-31
-
作者相关文章
植原体(Phytoplasma)是日本学者土居养二在1967年发现的(Doi et al.,1967),是一类无细胞壁、不能人工培养的原核微生物,寄生于植物韧皮部筛管及刺吸式介体昆虫的肠道、淋巴及唾腺等组织内,由叶蝉(Cicadella viridius)和蝽蟓(Halyomorpha mista)等刺吸式昆虫传播,也可通过嫁接和菟丝子(Cuscuta chinensis)等传播(廖晓兰等,2002; 车海彦等,2006)。植原体能够引起植物丛枝、黄化、簇生、矮化、顶枯等症状。目前世界上报道的植原体病害已有1000多种,给农林业生产带来了严重的经济损失(Welliver,1999)。
泡桐(Paulownia sp.)丛枝病(Paulownia witches’-broom disease)是一种由泡桐丛枝植原体(Paulownia witches’-broom phytoplasma)引起的侵染性病害,症状主要表现为腋芽和不定芽大量萌发至簇生成团,外观似鸟巢,有些病树上会出现花变叶症状,常常不能正常开花。林彩丽(2009)从泡桐丛枝植原体南阳株系中克隆并测定了2种质粒pPaWBNy-1和pPaWBNy-2。长春花(Catharanthus roseus)为夹竹桃科观赏性草本植物,已报道的自然侵染长春花的植原体株系在目前分类体系中分别属于不同的16Sr组或亚组(蔡红等,2003)。宋传生(2009)测定了我国海南长春花绿变植原体1种质粒的部分序列。陈泠伶(2010)从台湾长春花叶黄化植原体株系中测定出5种质粒,分别为p05PLY-1、p05PLY-2、p08PLY-1、p09PLY-1和p09PLY-2。桑树(Morus alba)萎缩病(Mulberry dwarf disease)是以植原体为病原的桑树重要病害,在我国各大蚕区均有分布。目前由于没有有效的治疗药物,所以该病又称为桑树的癌症(Ji et al.,2009; 张合禹等,2010)。至今未见有桑萎缩植原体质粒的研究报道。由植原体引起的苦楝(Melia azedarach)丛枝病表现为枝条节间缩短,侧枝丛生,叶片变小且黄化,生长缓慢,常导致苦楝大量死亡,严重影响林业经济的发展(陈作义等,1984; 金开璇等,1982; 徐梅卿等,2008)。宋传生等(2011)测定了苦楝丛枝植原体福清株系的一个4 446 bp的质粒pCWBFq。
质粒是染色质外的遗传物质,植物病原菌的质粒通常编码与致病性和昆虫介体传播等相关的基因(Vivian et al.,2001)。质粒可以通过获得或丢失功能基因来适应变化的环境,因此寄主菌质粒DNA进化速度比染色体DNA更快(Eberhard,1990;Sundin,2007)。目前,已被测序和发表的植原体质粒有29个,它们大部分为环状,线状质粒很少。在这些质粒的基因中,Rep、dnaG、ssb对质粒的自我复制起作用,cop影响质粒的拷贝数,在某些植原体中,致病能力与质粒的拷贝数可能有关联(Hisashi et al.,2001)。除了复制相关基因外,植原体质粒编码的绝大多数蛋白为膜蛋白与分泌蛋白,它们可能影响植原体与寄主的互作以及介体昆虫的传播(Yoshiko et al.,2009)。根据植原体质粒编码的Rep蛋白将植原体质粒分为2类,一类质粒编码与双生病毒(geminiviruses)类似的Rep,另一类质粒编码与pLS1家族相似的复制相关蛋白(Oshima et al.,2001)。
国内外关于植原体质粒相关研究较少,特别是质粒对于植原体繁殖、致病性以及与寄主互作的影响都有待深入研究(Lin et al.,2010)。目前分别从16Sr Ⅰ、16Sr Ⅱ、16Sr Ⅵ、16Sr Ⅺ及16Sr Ⅻ组植原体中测定了20,3,2,1和3种质粒,其余组植原体未见有质粒的报道。
本研究测定比较了苦楝丛枝、长春花绿变、桑萎缩及泡桐丛枝植原体上各一个质粒的完整DNA序列,利用信息学软件预测并分析了所编码蛋白的结构与功能,进一步明确该植原体分类地位,为植原体快速检测和鉴定、质粒基因结构和功能以及致病机制等研究奠定基础。
1 材料与方法 1.1 材料长春花绿变海南株系和泡桐丛枝植原体山东株系保存在本实验室组培苗上,分别编号为PeVHn和PaWBG33D(田国忠等,2005); 苦楝丛枝病材料采自福建省福清市,编号CWBFq; 桑树萎缩病材料采自河南濮阳市,编号MDPy。
1.2 方法 1.2.1 总DNA的提取参照天根公司所制的植物基因组DNA提取试剂盒所附说明书(DP305-02)。
1.2.2 质粒扩增和序列测定根据已发表植原体质粒pAYWB-Ⅳ(CP000065)、EcOYNIM(AB097150)、pPaWBNy-1(EF426472)序列比对的结果设计引物pF1fwd/pF1rev(表 1)。以感病的苦楝、桑树、泡桐和长春花总DNA为模板,进行PCR扩增,分别获得质粒片段pCWBFq-2 F1、pMDPy F1、pPaWBG33D F1和pPeVHn F1,然后根据所得到片段的序列设计引物pF2-1fwd、pF2-2fwd、pF2-3rev和pF2-4rev,利用引物对pF2-1fwd/pF2-3rev 从罹病的苦楝和桑树中分别扩增到质粒片段pCWBFq-2 F2和pMDPy F2,利用引物对pF2-2fwd/pF2-3rev和pF2-2fwd/pF2-4rev分别从患病的泡桐和长春花中扩增到质粒片段pPaWBG33D F2和pPeVHn F2。
|
|
PCR 扩增及测序方法如下。25 μL总体积:DNA 1 μL,2xTaq MIX 12.5 μL,dd H2O 1 μL,10mmol·L-1正反向引物各0.5 μL。扩增步骤为: 预变性,94 ℃,4 min; 变性,94 ℃,30 s,退火和延伸温度见表 1,共35次循环; 最后延伸,72 ℃,10 min。PCR产物进行琼脂糖凝胶电泳检测,阳性PCR产物送博迈德基因公司测序。
1.2.3 序列比对和蛋白特征预测利用DNAMAN6.0软件进行序列装配,采用BLAST(Basic Local Alignment Search Tools)工具(http://www.ncbi.nlm.nih.gov/balst/)分析序列同源性,采用标准遗传密码的NCBI ORF寻找器(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)预测可读框,使用DNAMAN进行序列比对,进化树分析采用MEGA4.1(MolecμLar Evolutionary Genetics Analysis,version 4.1),启动子预测采用在线启动子预测软件SoftBerry(http://linux1.softberry.com/berry.phtml)。
2 结果与分析 2.1 质粒片段的PCR扩增与序列测定在患病的植物总DNA中,扩增到长度为3 675bp的片段pF1CWBFq-2 F1,3 608 bp的pMDPyF1,3 598 bp的pPaWBG33D F1,3 700 bp的pPeVHnF1,484 bp的pCWBFq-2 F2,485 bp的pMDPy F2,596 bp 的pPaWBG33D F2和603 bp的pPeVHn F2。在健康植物总DNA中均没有扩增到特异性片段(图 1)。
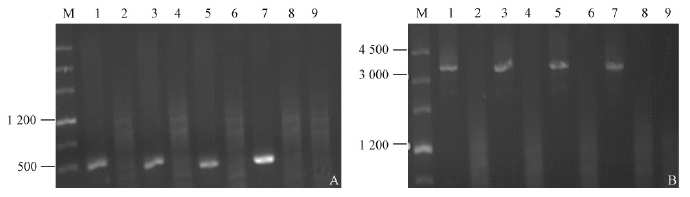 |
图 1 4种植原体的质粒片段的PCR产物琼脂糖凝胶电泳
Fig. 1 PCR amplification of the plasmid fragments of four phytoplasmas and agarose gel electrophoresis
A. M: Marker Ⅲ; 1, 3, 5, 7: pPaWBG33D F2, pMDPy F2, pCWBFq-2 F2, pPeVHn F2;
Lane 2, 4, 6, 8: 健康对照Healthy control; Lane 9: 空白对照None template control.
B. M: Marker Ⅲ; 1, 3, 5, 7: pPaWBG33D F1, pMDPy F1, pCWBFq-2 F1, pPeVHn F1;
Lane 2, 4, 6, 8: 健康对照Healthy control; Lane 9: 空白对照None template control. |
利用DNAMAN软件分别对不同植原体的测定片段进行拼接装配,得到4种环状质粒,分别是3 833 bp的桑树萎缩植原体濮阳株系质粒,命名为pMDPy; 长度为3 913 bp的苦楝丛枝植原体福清株系质粒,命名为pCWBFq-2; 长度为3 843 bp的泡桐丛枝植原体山东株系质粒,命名为pPaWBG33D; 长度为3 943 bp的长春花绿变植原体海南株系质粒,命名为pPeVHn(图 2)。序列分析表明,质粒pMDPy、pPaWBG33D、pCWBFq-2及pPEVHn的A+T含量分别为72.56%,74.12%,72.3%和75.78%。预测此4种质粒序列在同一条链的同一方向均有5个编码蛋白,且都大于100个氨基酸。将每个质粒ORF1上第1个核苷酸的位置定位为1,ORF5编码单链结合蛋白(single-str and ed DNA binding protein,SSB),ORF1编码复制相关蛋白(replication associated protein,RepA)。
 |
图 2 质粒pCWBFq-2、pMDPy、pPaWBG33D及pPeVHn示意
Fig. 2 Schematic diagrams of the plasmids pCWBFq-2, pMDPy,pPaWBG33D and pPeVHn
|
从4种质粒的ORF组织结构看,pMDPy、pPaWBG33D和pCWBFq-2更加相似,这3种质粒ORF1和ORF2之间的非编码区的序列最长,大小在816~821 bp之间。而质粒pPeVHn的ORF1和ORF2间的非编码区的序列仅为254 bp; pMDPy、pPaWBG33D和pCWBFq-2的ORF5和0RF1之间的非编码区仅有48 bp左右,而pPEVHn中相对应的序列为724 bp。
2.3 4种植原体编码的蛋白质序列同源性比较对此4种不同植原体质粒编码的蛋白与GenBank等数据库进行同源性比对,表 2列出了4种质粒的每一个ORF所编码蛋白质氨基酸序列的比对结果。结果显示,pPaWBG33D与以前测定的泡桐丛枝植原体南阳株系质粒pPaWBNy-2的氨基酸序列同源性最高,其中pPaWBG33D的SSB蛋白与pPaWBNy-2的相似值为100%(Lin et al.,2008)。pCWBFq-2比pCWBFq短544 bp,且少一个ORF,应为苦楝丛枝植原体上新发现的一个质粒。与pCWBFq-2的编码蛋白RepA、P2、P4及SSB相似值最高的分别为pCWBFq的RepA、P3、P5及SBB,可能这2种质粒间存在共同的进化起源; 而pCWBFq-2的P2与洋葱(Omanense sp.)黄化野生型的质粒pOYW编码的蛋白同源性最高,序列一致性为47.21%。从桑树萎缩植原体中首次测定的质粒pMDPy,其编码的5个基因均与pCWBFq相对应的基因有最高的相似性,其中ORF4与pCWBFq对应的序列同源性达到95.24%。前述3种植原体质粒之间显示较高的序列相似性,而pPeVHn明显与这3种质粒的关系要远,而与pOYW和pOYM及台湾报道的长春花黄化植原体质粒p05PLY-2关系更近。
|
|
4个质粒的RepA蛋白和SSB蛋白与其他质粒的相似度较高,且都在88%以上。pCWBFq-2、pMDPy、pPeVHn的P3蛋白与其他质粒编码的蛋白相似度较低,其中pCWBFq-2的P3蛋白与pOYW编码的蛋白相似值仅为47.21%。
对质粒编码的5个蛋白分别进行比对,结果显示pPaWBG33D RepA蛋白与pPaWBNY-2 RepA相似性最高达99.7%,与pPaWBNY-1 RepA的相似性仅为72.3%; pMDPy RepA蛋白与pCWBFq-2RepA的相似性达97%,与宋传生等(2011)测定的pCWBFq RepA相似性为91.3%; pCWBFq-2 RepA与pCWBFq RepA相似性为92.4%。4种质粒的RepA蛋白之间的序列相似值变化很大,在11.6%~97%之间,其中pPeVHn与台湾报道的长春花黄化植原体质粒p05PLY的同源性达81.2%,并与pAYWB-Ⅱ(70.8%)、pAYWB-Ⅳ(75.1%)及pCPa(60.3%)有较高的一致性,而与质粒pCWBFq-2、pMDPy和pPaWBG33D的相似值仅为11.6%~12.2%之间,与其他质粒的RepA的相似值也在10.2%~12.7%之间。质粒pCWBFq-2、pMDPy和pPaWBG33D之间的Rep相似性在88%~97%之间。
17个不同植原体质粒上的SSB蛋白之间表现出相对较高的序列相似性,相似值最低为74%,而最高的达100%。其特点是除pAYWB-Ⅲ SSB序列长度为120个氨基酸外(末端多出16个氨基酸,KGVIEKEPKSFDDWV),其余质粒的SSB长度皆为104个氨基酸。16Sr Ⅰ组植原体质粒起始端16个氨基酸完全相同,而所测定的4种质粒的起始端37个氨基酸完全一致; 如,洋葱黄化植原体质粒pOYM SSB与pMDPy、pCWBFq及p05PLY2的SSB同源性都为74%,所测定的4种质粒pPaWBG33D、pCWBFq-2、pPeVHn及pMDPy的SSB蛋白之间的序列同源性在87.5%~93.3%之间,可能反映了它们关系较近但却是不同种类的植原体。
质粒编码蛋白P3(ORF3)氨基酸序列虽然存在多个明显的保守区域或结构域,但不同植原体不同质粒间的序列的变异却很大,所比对的15个16Sr Ⅰ组植原体质粒P3序列的相似性在28.1%~99.5%之间,pPeVHn、pCWBFq-2、pMDPy和pPaWBG33D间的相似性在34.1%~67.1%之间。序列的长度从144至200个氨基酸不等。不同质粒的该编码蛋白末端序列无论是长度还是氨基酸种类皆表现出高度的变异性; pPaWBG33D与pPAWBNy-2的氨基酸序列相似性最高,为99.5%,仅有一个氨基酸的差异(50位异亮氨酸Ⅰ/苏氨酸T),与pAYWB-Ⅰ为99.48%,有2个氨基酸差异。pAYWB-Ⅱ与pAYWB-Ⅳ为98.7%(2个是氨基酸差异),pOYW与EcAYWB为95.2%(7个位点,9个氨基酸变异),pOYNIM与pEcAYWB为95.1%(5个差异)。最低的pOYW与pAYWB-Ⅱ为28.1%,pAYWB-Ⅱ与EcAYWB为28.8%。
2.4 质粒DNA 全长序列和单基因系统进化分析构建质粒DNA全序列的系统发育树,结果显示(图 3),这4种质粒与其他16Sr Ⅰ组的质粒聚为一个大的分支。pPaWBG33D与pPaWBNy-2最近,归为16Sr Ⅰ组D亚组。pMDPy和pCWBFq-2与pCWBFq最近,pPeVHn与台湾报道的长春花黄化植原体的5种质粒及pOYW关系最近。在亚组水平上,基于16Sr DNA 序列的同源性已把CWB、MD划分为16Sr Ⅰ组B亚组,但质粒的进化分析结果显示CWB和MD更接近于D 亚组的PaWB。
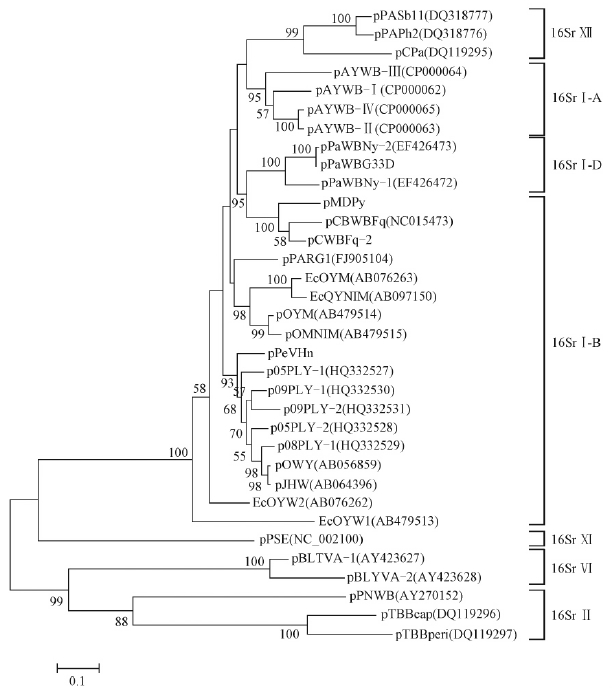 |
图 3 基于质粒全长序列所建立的系统发育树
Fig. 3 Bootstrapped phylogenetic analysis for phytoplasmal plasmids based on the full-length sequences of all given plasmids
|
对这4种质粒编码的每个氨基酸序列分别进行进化分析,结果与用完整DNA序列分析的结果基本一致。同时进一步肯定了pPaWBG33D与pPaWBNy-2最近,表明pPaWBG33D与pPaWBNy-2可能是泡桐丛枝植原体不同地理株系的相同种类的质粒。pMDPy与pCWBFq关系更近,而与pPaWB较远。由于苦楝丛枝植原体福州株系的pCWBFq-2与该株系上已测定的质粒pCWBFq(4 446 bp)关系最近,但大小比pCWBFq短了544bp,且少一个ORF,所以判断pCWBFq-2为苦楝丛枝植原体中新发现的第2种质粒。与用完整DNA序列分析的结果类似,各预测蛋白氨基酸列进化分析结果也进一步显示了在16Sr Ⅰ组植原体的质粒中,海南长春花绿变植原体与另外3种亚热带或温带植物上发生的植原体进化关系更远。
2.5 质粒非编码区序列分析在质粒pMDPy、pCWBFq-2和pPaWBG33D序列中,ORF1与ORF2之间的非编码区序列最长,相似度较高,在71%到89%之间。在pPeVHn序列中,2段最长的非编码序列分别与质粒pMDPy、pCWBFq-2和pPaWBG33D最长非编码区的部分序列有较高的相似性。与pMDPy、pCWBFq-2和pPaWBG33D相比,pPeVHn中5个基因的顺序没有变化,但个别基因可能会在质粒进化过程中出现位移或重组。
采用在线启动子预测发现,pMDPyORF1-ORF2间隔区的序列长度为816 bp,共预测到3个启动子序列和1个核糖体结合位点(AAAGGAGTA);pCWBFq-2的ORF1-ORF2间隔区的序列长度为821 bp,预测到2个启动子序列; pPeVHn中ORF1-ORF2间隔区的序列长度为254 bp,预测到1个可能的启动子,ORF5-ORF1间隔区的序列长度为727 bp,预测到2个启动子和2个可能的核糖体结合位点(AGAGG和AAAGGAGTA); 在pPaWBG33D中,ORF1-ORF2间隔区的序列长度为869 bp,共预测到3个可能的启动子。
3 讨论本研究测定及分析了采自不同地区的4种16Sr Ⅰ组植原体中各1种质粒的完整DNA序列。其中桑树萎缩植原体的1种质粒为该植原体上首次测定。pCWBFq-2苦楝丛枝植原体福清株系中与pCWBFq不同的质粒。虽然之前已有关于我国长春花绿变植原体海南株系质粒部分序列的测定结果,但本研究完成了一个环状质粒分子的序列测定。本研究所测定的泡桐丛枝植原体质粒pPaWBG33D来源于保存在中度抗病泡桐无性系MB33组培苗上的植原体山东株系,而之前Lin等(2009)2种所测定的两个质粒pPaWBNy-1和pPaWBNy-2来源于保存在感病泡桐无性系中林3号组培苗中的河南南阳株系,pPaWBG33D与pPaWBNy-2可能是相同的质粒,序列的差异可能与2菌株因不同地理和繁殖寄主作用下的遗传变异有关(田国忠等,2005)。
本研究在扩增植原体质粒片段时,所设计的引物序列位于质粒上最保守的ssb区域,因而可以同时扩增和检测这4种属于16Sr Ⅰ组的不同亚组的植原体质粒。该研究采用1对通用引物(Plasmid2F/Plasmid2R)先扩增到了这4种质粒约94%的核苷酸序列,测序后根据末端序列设计4种质粒的特异性引物,扩增出约200 bp的小片段。这种方法的优点是采用此通用引物可以对含有ssb的质粒的大部分序列进行快速准确的测序。从大量植原体株系中鉴定特异质粒并寻找突变菌株,从而更好地研究植原体的致病机制、寄主专化性和质粒基因转移与重组规律; 也有助于高效快速地获得完整质粒并转化到其他菌株细胞内,以深入研究其致病相关性。也可以为植原体的多指标、多基因分类提供新的理论依据。由于ssb基因仅在16Sr Ⅰ组植原体质粒中被发现(宋传生等,2011),所以该方法可能不适用于其他组植原体的质粒研究。由于16Sr Ⅰ组植原体是众多植物病害的病原,用此法对该组植原体质粒进行的快速鉴定研究会有较大的应用前景(Lee et al.,2004)。
以质粒全序列构建的进化树和16S rDNA序列系统发育树基本一致,验证了基于质粒基因的系统进化分析与染色体16S rDNA的分类结果在一定范围内存在一致性。在亚组水平上,根据16S rDNA序列,已把CWB、MD划为16Sr Ⅰ组B亚组,但质粒全序列分析结果显示,其更接近于D亚组的PaWB。由于长春花可感染多种植原体且表现类似的症状,因而不同地区的所鉴定的植原体可能属于不同的16Sr组或16Sr Ⅰ组内的不同亚组。在目前发现的24个植原体质粒中,序列变异非常明显,相似性在11%~99%之间(图 6),这种变异的产生可能与植原体对不同寄主植物和介体昆虫的选择以及对不同地理生态环境的适应有关系(Liefting et al.,2006)。
已知植原体质粒编码的Rep蛋白分为质粒编码与双生病毒(geminiviruses)类似的Rep(Gibbs et al.,2006)和与pLS1家族相似的复制相关蛋白(Hisashi et al.,2001)。因而,根据本研究对4种质粒的rep编码蛋白氨基酸序列的分析比对结果分析推断,pMDPy、pPaWBG33D和pCWBFq-2应与pAYWB-Ⅰ和Ⅲ、EcOYW1和2一样,属于质粒编码与双生病毒(geminiviruses)类似的REP,而pPEVHn则与pOYW和pAYWⅡ和Ⅳ一样,所编码的蛋白质与pLS1家族相似(Saccardo et al.,2011; Shigetou et al.,2001)。
质粒是细菌细胞内一种自我复制的环状双链DNA分子,能稳定地独立存在于染色体外,具有自主复制、传给子代、丢失及在细菌间转移等特性,与细菌的遗传变异有关,一般不整合到宿主染色体上。质粒编码非细菌生命所必须的某些生物学性状,如性菌毛、细菌素、毒素和耐药性等,通常不携带宿主菌在正常条件下生长的必须基因(Barbara et al.,2009)。在植原体中,质粒可能参与了植原体存活、致病以及与寄主的互作,而且对致病性和介体昆虫传播有影响。质粒易于通过获得或丢失功能基因以适应环境的变化,因此进化速度快于比宿主染色体DNA。在某些植原体中质粒的拷贝数也有可能影响植原体的致病能力,这对质粒的研究是非常重要的。本研究测定的每个质粒的5个ORF中,只有ORF1和ORF5为已知功能基因,其余3个功能未知。Lin等(2009)和宋传生等(2011)的研究都预测了这3个蛋白皆为分泌型蛋白,具有明显的跨膜区域,已通过原核细胞表达了改蛋白,并通过Western blot技术证明pPaWBNy-1的ORF4(与本研究中的ORF3同源)基因能够在发病的泡桐植株中表达。P3的同源蛋白在洋葱黄化植原体中有较多的研究,并被证实与介体昆虫的传播特性有关。Shigetou(2001)发现质粒pOYM ORF3编码蛋的白是具有2个跨膜螺旋的膜蛋白,推测包括ORF3在内的膜蛋白可能在与介体昆虫的互作中发挥功能。Ishii等(2009)通过免疫组织化学分析证明OY-NIM植原体的EcOYNIM的ORF3不能够被翻译成蛋白是由于启动子的缺失和突变造成的,这导致了植原体OYNIM不能被介体昆虫传播。从以上3个ORF编码蛋白氨基酸序列的变异程度可以推断,这些基因可能与植原体的致病性、寄主和介体专化性及对环境变化的适应性有更密切的关系。
| [1] |
蔡红, 陈惠, 李凡. 2003. 长春花黄化植原体核糖体蛋白基因片段序列分析. 微生物学报, 30(1) :34-37.( 1) 1)
|
| [2] |
车海彦, 罗大全. 2006. 植原体病害的检测方法研究进展. 华南热带农业大学学报, 03:69-73.( 1) 1)
|
| [3] |
陈泠伶. 2010. 日日春叶片黄化病植物菌质体之质体选殖与分析. 台湾大学植物病理与微生物学研究所硕士学位论文.( 1) 1)
|
| [4] |
陈作义, 沈菊英, 郑冠标. 1984. 海南岛苦楝丛枝病的初步调查研究. 热带作物学报, (5) :121-123.( 1) 1)
|
| [5] |
金开璇, 蔡希灼, 阿斯诺来. 1982. 苦楝丛枝病类细菌、类菌原体的电镜观察. 林业科学, 22(3) :422-424.( 1) 1)
|
| [6] |
廖晓兰, 罗宽, 朱水芳. 2002. 植原体的分类及分子生物学研究进展. 植物检疫, 03:167-172.( 1) 1)
|
| [7] |
林彩丽. 2009. 泡桐丛枝病植原体质粒分子特征及其编码蛋白功能研究. 中国农业大学博士学位论文.( 1) 1)
|
| [8] |
宋传生, 林彩丽, 田国忠, 等. 2011. 苦楝丛枝植原体质粒的测定与分子特征. 微生物学报, 51(9) :1158-1167.( 4) 4)
|
| [9] |
宋传生. 2009. 木本植物黄化类植原体的检测鉴定及苦楝丛植植原体质粒的测定, 中国林业科学研究院硕士学位论文.( 1) 1)
|
| [10] |
田国忠, 温秀军, 李永, 等. 2005. 枣疯病和泡桐丛枝病植原体不同分离物组织培养保藏和嫁接传病试验比较. 林业科学研究, 18 (1) :1-9.( 2) 2)
|
| [11] |
徐梅卿, 何平勋. 2008. 中国木本植物病原总汇. 哈尔滨: 东北林业大学出版社, 816-817.( 1) 1)
|
| [12] | 张和禹, 汪泰初, 鲍先巡. 2010. 桑树萎缩病的研究进展. 中国蚕业, 31(1) :9-12. |
| [13] |
Barbara E, Funnell, Gregory J, et al. 2009. 质粒生物学. 陈惠鹏, 张惟材, 等译. 北京: 化学工业出版社.( 1) 1)
|
| [14] |
Doi Y, Teranaka M, Yora K, et al. 1967. Mycoplasma or PLT group-like micoorganisms found in the phloem elements of plants infected with mulberry dwarf, potato witches' broom, aster yellows, or paulownia witches’broom. Annals of the Phytopathological Society of Japan. 33:259-266.( 1) 1)
|
| [15] |
Eberhard W G. 1990. Evolutionin bacterial plasmids and levels of selection. The Quarterly Review of Biology, 65(1) : 3-22.( 1) 1)
|
| [16] |
Gibbs M J, Smeianov V V, Steele J L, et al. 2006. Two families of replike genes that probably originated by interspecies recombination are represented in viral, plasmid, bacterial and parasitic protozoan genomes. Molecular Biology and Evolution, 23:1097-1100.( 1) 1)
|
| [17] |
Hisashi N, Shin-Ichi M, Kenro O, et al. 2001. In planta expression of a protein encoded by the extrachromosomal DNA of a phytoplasma and related to geminivirus replication ptoteins. Microbiology, 147:507-513.( 2) 2)
|
| [18] |
Ishii Y, Kakizawa S, Hoshi A, et al. 2009. In the non-insecttransmissible line of onion yellows phytoplasma ( OYNIM), the plasmid-encoded transmembrane protein ORF3 lacks the major promoter region. Microbiology, 155:2058-2067.( 1) 1)
|
| [19] |
Ji X L, Gai Y P, Mu Z M. 2009. Comparative proteomic analysis provides new insights into mulberry dwarf responses in mulberry (Morus alba L. ) . Proteomics, 9:5328-5339.( 1) 1)
|
| [20] |
Lee I M, Gundersen-Rindal D E, Davis R E, et al. 2004. ‘Candidatus Phytoplasma asteris’, a novel phytoplasma taxon associated with aster yellows and related diseases. International Journal of Systematic and Evolutionary Microbiology, 54:1037-1048.( 1) 1)
|
| [21] |
Liefting L W, Andersen M T, Lough T J, et al. 2006. Comparative analysis of the plasmids from two isolates of ‘ Candidatus Phytoplasma australiense’. Plasmid, 56:138-144.( 1) 1)
|
| [22] |
Lin C L, Zhou T, Li H F, et al. 2009. Molecular characterization of two plasmids from paulownia witches'-broom phytoplasma and and detection of a plasmid-encoded protein in infected plants. European Journal of Plant Pathology, 123:321-330.( 2) 2)
|
| [23] |
Lin C L, Piao C G, Li Y, et al. 2010. Characterization of P33 protein encoded by plasmid from paulownia witches’- broom phytoplasma. The 18 th IOM Congress. Chiancciano Terme, Italy, 146.( 1) 1)
|
| [24] |
Oshima K, Kakizawa S, Nishigawa H, et al. 2001. A plasmid of phytoplasma encodes a unique replication protein having both plasmid- and virus-like domains: clue to viral ancestry or result of virus/plasmid recombination? Virology, 285(2) :270-277.( 1) 1)
|
| [25] |
Saccardo F, Cettul E, Palmano S, et al. 2011. On the alleged origin of geminivurses from extrachromosomal DNAs of phytoplasmas. BMC Evolustionary Biology, 11:185-197.( 1) 1)
|
| [26] |
Shigetou N. 2001. In planta expression of a protein encoded by the extrachromosomal DNA of a phytoplasma and related to geminivirus replication ptoteins. Microbiology, 147:507-513.( 2) 2)
|
| [27] |
Sundin G W. 2007. Genomic insights into the contribution of phytopathogenic bacterial plasmids to the evolutionary history of their hosts. Annual Review of Phytopathology, 45:129-151.( 1) 1)
|
| [28] |
Vivian A, MurilloJ, Jackson R W. 2001. The roles of plasmids in phytopathogenic bacteria: Mobile arsenals? Microbiology, 147:763-780.( 1) 1)
|
| [29] |
Welliver R. 1999. Diseases caused by phytoplasmas. Plant Pathology Circular, 25(1) :17-22.( 1) 1)
|
| [30] |
Yoshiko I, Shigeyuki K, Ayaka H, et al. 2009. In the non-insecttransmissible line of onion yellows phytoplasma ( OY-NIM), the plasmid-encoded transmembrane protein ORF3 lacks the major promoter region. Microbiology, 155: 2058-2067.( 1) 1)
|
 2013, Vol. 49
2013, Vol. 49

