文章信息
- 梁艳, 徐刚标, 张合平, 吴雪琴, 申响保, 王爱云
- Liang Yan, Xu Gangbiao, Zhang Heping, Wu Xueqin, Shen Xiangbao, Wang Aiyun
- 南岭山地伯乐树天然种群和人工种群遗传多样性比较
- Genetic Diversity of Natural and Planted Populations of Bretschneidera sinensis from Nanling Region
- 林业科学, 2012, 48(12): 45-52.
- Scientia Silvae Sinicae, 2012, 48(12): 45-52.
-
文章历史
- 收稿日期:2012-06-28
- 修回日期:2012-10-23
-
作者相关文章
种内遗传变异是物种适应性进化的基础(Gillies et al., 1999),研究种群遗传结构与组成,获取遗传多样性水平及分布的信息,是开展物种遗传多样性保护及其遗传改良的前提(Zeng et al., 2003; Feres et al., 2009)。自20世纪70年代以来,等位酶、DNA分子标记技术已广泛应用于珍稀濒危植物遗传多样性及种群遗传结构的研究(Hamrick et al., 1991;Nybom, 2004)。珍稀濒危植物迁地保护是生物多样性保护的重要组成部分,也是回归引种及野生居群恢复重建的基因和原始材料的保障,但迁地保护种群是否维持了天然种群的遗传完整性,一直受到人们普遍关注(Burgarella et al., 2007)。有研究表明,在缺乏基本种群遗传信息情况下营建的大多数人工林,遗传多样性降低,适应性潜力下降(Pandey et al., 2004;Finkeldey et al., 2007)。因此,植物迁地保护种群的遗传多样性保护效果的评价已日益受到重视(Leiva et al., 2009; Ferreira et al., 2012)。
伯乐树(Bretschneidera sinensis)又名钟萼木,为伯乐树科(Bretschneideraceae)伯乐树属(Bretschneidera)植物,是我国特有的单种科植物,高大落叶乔木,总状花序顶生,两性花,木质蒴果,种子大(张宏达等,2004),属第三纪古热带植物区系的孑遗种,被列为国家一级重点保护植物(傅立国,1992;汪松等,2004),分布于云南、广西、广东、湖南、湖北、江西、福建、浙江等地海拔500~1 500 m的湿润沟谷坡地或溪旁的常绿阔叶林或针阔混交林中。虽然伯乐树分布区较广,但天然资源稀少,呈零星、片断化分布(吴征镒等,2003;王美娜等,2011),多数株间相距1 000 m以上(彭莎莎等,2011),偶见株间相距不足200 m的2~5株成年树木形成的小群落,属偶见种(Gillies et al., 1999)。由于伯乐树物种的古老性和稀有性,在研究被子植物的系统发育及古地理、古气候等方面具有重要的科学价值,但其研究基础十分薄弱(乔琦等,2011a; 2011b);近年才有学者开展伯乐树遗传多样性的初步研究(彭莎莎等,2011;王美娜等,2011),并在国际植物园保护联盟(Botanic Gardens Conservation International,BGCI)中国项目等项目资助下,在南昆山省级自然保护区、车八岭国家级自然保护区营建了小规模的迁地保存林,但这些迁地保护工作是在没有开展保护遗传学研究背景下进行的。
地处贵州、湖南、广西、广东、江西五省边境的南岭山地(中华人民共和国林业部林业区划办公室,1978),地质历史较为古老,自然环境复杂多样,孕育了丰富的植物资源,保留着大量的孑遗植物(庞雄飞,1993;陈涛等,1994),是我国具有国际意义的14个陆地生物多样性关键地区之一(陈灵芝,1993)。该地区建立的自然保护区多,是伯乐树资源分布相对集中的地区,也是开展伯乐树遗传多样性保护的重点地区。
简单重复间隔序列(inter-simple sequence repeats, ISSR)标记多态性高,比RAPD分子标记重复性好,比AFLP和SSR标记更易获得且费用低,已成功应用于许多植物的种群遗传多样性研究(Nybom, 2004)。由于ISSR是显性标记,需要假定种群处于Hardy-Weiberg平衡状态才能估算等位基因频率,这会使估算的种群遗传多样性参数产生偏差,因此许多学者往往采用表型数据(多态百分比、Shannon表型多样性指数)及分子方差分析(analysis of molecular variance, AMOVA)方法进行遗传多样性分析。本研究采用ISSR标记分析、比较南岭山地伯乐树天然种群与人工种群遗传多样性及种群遗传结构,评价现有的迁地保护人工种群(基因库)的遗传完整性,旨在为制定南岭山地伯乐树遗传多样性迁地保护策略提供理论依据。
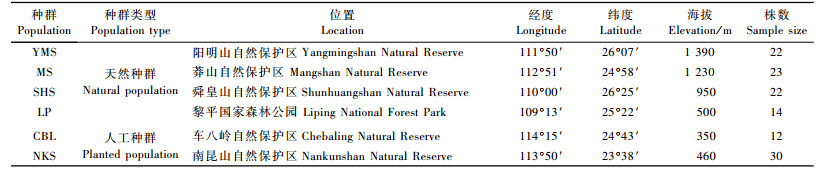
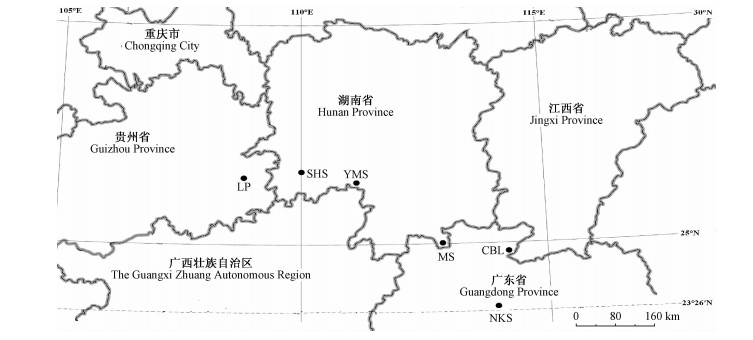
1 材料与方法 1.1 材料于2011年5月至8月采样。尽管计划在南岭山地范围内按网格式等距离设计采样地点,由于伯乐树为偶见种,天然种群内个体数很少,最终在株数相对较多(10株以上)的湖南省阳明山、莽山、舜皇山和贵州黎平国家森林公园的天然种群中采集成年树木,株间距200 m以上。南昆山、车八岭人工迁地保护种群均来源于本地。南昆山人工种群于2009年造林,株行距2 m×3 m,造林成活率达95%以上,其母种群中仅有12株不同龄级树木,且集中在800 m2左右的范围内(王美娜等,2011),个体间存在着亲子代亲缘关系;车八岭人工种群的具体造林年份不清(由于2008年冰害,母种群现仅存3株大树)。野外采集的新鲜嫩叶立即放入装有硅胶的密封袋中,并记录各种群的地理位置及个体数(表 1;图 1)。叶片带回实验室后,放入-70 ℃冰箱中保存备用。
|
|
|
图 1 伯乐树种群采样点的地理位置 Fig.1 Geographic locations of B. sinensis populations sampled |
采取改良的CTAB法(张玉梅等,2012)提取伯乐树基因组DNA,保存于-20 ℃下备用。0.8%的琼脂糖凝胶电泳, 紫外分光光度计检测其浓度。
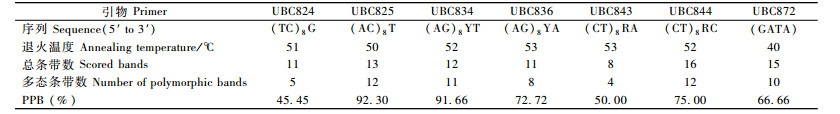
1.2.2 ISSR反应体系建立与优化通过正交试验确定ISSR优化体系为:20 μL反应体系,Mg2+ 2.0 mmol·L-1,dNTP 0.25 mmol·L-1,引物0.40 μmol·L-1,DNA 70 ng,Taq酶1.75 U。所有分子试剂购自天根生物有限公司。PCR扩增在Veriti 96 well Thermal Cycler PCR扩增仪上进行,扩增程序为:94 ℃预变性4 min;94 ℃变性40 s,退火40 s(退火温度随引物而定,见表 2),72 ℃延伸90 s,40个循环;72 ℃延伸7 min;4 ℃保存。
|
|
采用加拿大哥伦比亚大学(UBC)2006年公布的序列,由上海英俊生物技术有限公司合成的ISSR引物进行扩增筛选。从100条引物中筛选出扩增条带稳定、清晰的7条引物,对全部样品进行扩增。扩增产物用1.8%琼脂糖凝胶电泳检测,缓冲液1×TAE,电压为5 V·cm-1,每组试验(扩增与电泳)重复3次以上,G-BOX紫外凝胶成像系统观察,拍照记录。
1.3 数据处理与分析采用人工计带法判读电泳图谱。相同迁移位置上条带有无分别计为“1”和“0”,构成“0/1”型矩阵。采用POPGENE version 1.31软件(Yeh et al., 1999)估算物种及种群多态条带百分率(PPB);计算Shannon表型多样性指数(H)和多样性组分,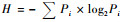
利用ARLEQUIN3.1(http://cmpg.unibe.ch/software/arlequin3)软件包中AMOVA计算种群内、种群间及种群类型间的遗传方差分量σw2,σb2,σa2,2个类型种群遗传分化系数ΦST(Excoffier et al., 1992)以及所有成对种群遗传分化系数ΦST。采用NTSYS-pc软件(Rohlf, 1998)对种群间遗传分化系数ΦST进行UPGMA(unweighted pair-group method with arithmetic-means)聚类分析,绘制树状聚类图。
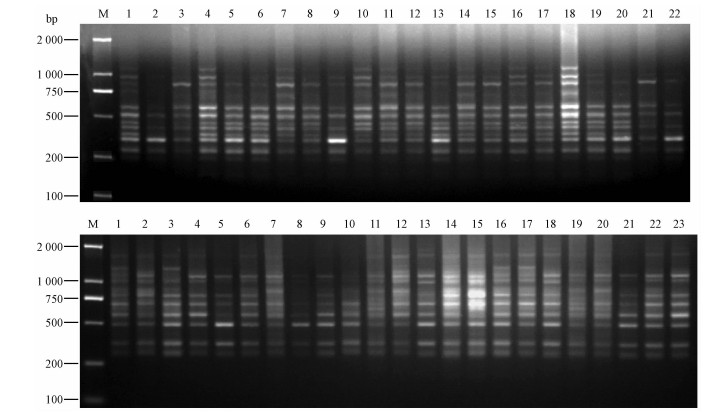
2 结果与分析 2.1 种群遗传多样性分析7条ISSR引物对来自6个伯乐树种群的123个DNA样本进行PCR扩增,共扩增出86个条带(表 2),扩增片段大小在200~1 800 bp之间(部分扩增结果见图 2)。其中,多态条带62条,多态条带百分率(PPB)为72.09%。每条ISSR引物扩增出8~16个条带,平均为12个条带;多态条带百分率为45.45%~92.30%,其平均值为70.54%。
|
图 2 引物UBC834对YMS和UBC844对MS样品扩增结果 Fig.2 The amplification of ISSR marker 836 for YMS and 844 for MS |
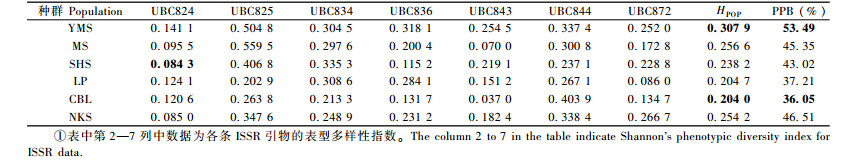
由伯乐树各种群的ISSR多态性参数(表 3)可知,伯乐树各种群多态条带百分率(PPB)和Shannon表型多样性指数(HPOP)变化范围分别为36.05%~53.49%,0.204 0~0.307 9。各种群的多态条带百分率(数据未列出)和Shannon表型多样性指数明显因引物而异,如,引物UBC824检测到舜皇山种群(SHS)的遗传多样性最低(HPOP=0.084 3),但引物UBC834检测的结果正好相反。总体而言,湖南阳明山种群(YMS)遗传多样性最高,广东车八岭(CBL)种群遗传多样性最低。
|
|
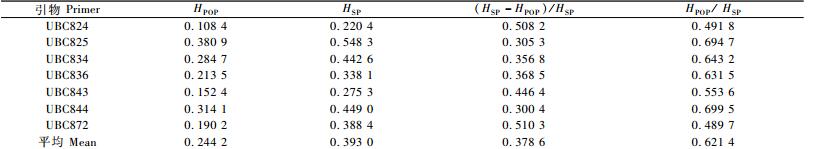
基于Shannon表型多样性指数分析结果(表 4)表明,各种群的Shannon表型多样性指数平均值为0.244 2,而物种水平的Shannon表型多样性指数为0.393 0。这表明,62.14%遗传变异存在种群内,37.86%遗传变异存在种群间。
|
|
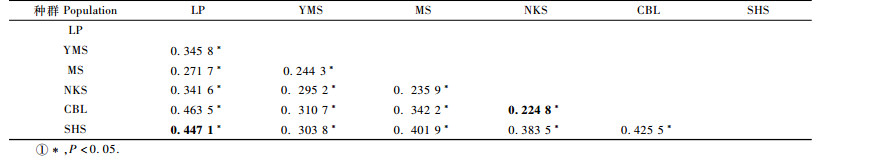
AMOVA分析结果(表 5)表明,天然与人工2类种群类型间变异占总变异3.04%,种群类型间差异不显著。种群间和种群内遗传变异分别占总变异的31.06%和65.90%,种群间遗传分化极显著(ΦST=0.341 0,P < 0.001)。
|
|
由伯乐树2类种群的遗传多样性参数比较结果(表 6)可知,伯乐树天然种群类型和人工种群类型多态条带百分率分别为70.93%和53.49%,种群平均表型Shannon多样性指数分别为0.251 8,0.229 1,种群的总体表型Shannon多样性指数分别为0.388 2,0.294 1,两者均说明天然种群遗传多样性高于人工种群。基于表型多样性指数方法估算的天然种群类型中种群间变异占总变异35.14%,而人工迁地种群类型中种群间变异占总变异22.10%;基于AVOMA分析,天然种群遗传分化系数为ΦST=0.340 7,人工种群遗传分化系数为ΦST=0.224 8。这些种群遗传参数表明,伯乐树天然种群间遗传分化较人工迁地保护种群明显。
|
|
利用AMOVA计算的成对种群间遗传分化系数ΦST值在0.224 8 ~ 0.477 1之间(表 7)。其中,贵州黎平种群(LP)与湖南舜皇山(SHS)种群间遗传距离最大(ΦST=0.477 1),广东车八岭(CBL)与南昆山(NKS)种群间遗传距离最小(ΦST=0.224 8),各种群间遗传分化达到显著水平(P < 0.05)。
|
|
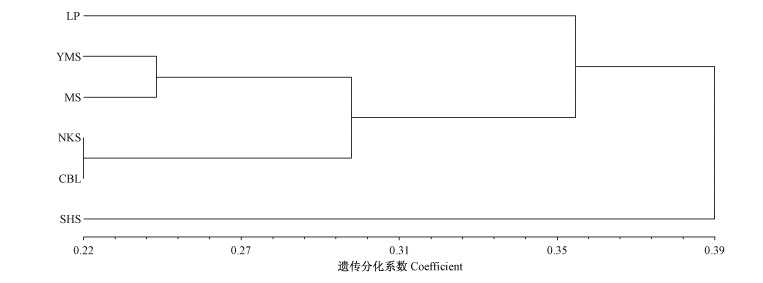
基于ISSR标记检测的伯乐树成对种群间ΦST值,用NTSYS-pc软件对6个种群进行UPGMA聚类(图 3)。结果表明,6个种群可分为3组:贵州黎平(LP)和湖南舜皇山(SHS)种群各为一组,湖南莽山(MS)、湖南阳明山(YMS)、南昆山(NKS)与车八岭(CBL)种群为一组。
|
图 3 伯乐树种群间ΦST的UPGMA聚类 Fig.3 Dendrogram of UPGMA based on ΦST of different populations of B. sinensis |
从ISSR扩增结果来看,来自南岭山地的4个伯乐树天然种群的多态条带百分率和Shannon表型多样性指数分别为70.93%和0.388 2,略高于彭莎莎等(2011)对浙江、江西5个天然种群的23株个体的初步研究结果(PPB=63.19%,HSP=0.340 5)。伯乐树间断性零星分布于云南、贵州、广西、湖北、湖南、广东、福建、浙江,分布区内环境复杂多样。本研究材料来源于伯乐树分布相对集中的南岭山地种群,因此,伯乐树实际的遗传多样性可能会更高,这表明伯乐树在物种水平维持相对较高的遗传多样性。在朝鲜连翘(Abeliophyllum distichum )(Kang et al., 2000)、大花溲疏(Deutzia grandiflora) (Helenurm, 2001)、贝叶石南属Leucopogon obtectus (Xue et al., 2004)、报春花属Primula apennina (Crema et al., 2009)、巴伦西亚雪片莲(Leucojum valentinum )(Jordán-Pla et al., 2009)、矢车菊属Centaurea wiedemanniana (Sozen et al., 2010)等珍稀、特有植物种中也发现同样情况。在缺乏有关伯乐树生物学资料及种群进化历史资料的前提下,初步认为,伯乐树曾经可能是连续的广布种且具有很高的遗传多样性,尽管近期人为活动加剧了生境片断化导致天然资源稀少,但种内仍存在着丰富的遗传变异。
本研究的主要目的是比较南岭地区伯乐树天然种群与人工种群的遗传多样性,评价人工迁地保护种群的遗传完整性。研究结果表明,南岭地区伯乐树天然种群多样性高于人工迁地保护种群(表 6),这在欧洲云杉(Picea abies) (Rajora, 1999)、小干松(Pinus contorta) (Thomas et al., 1999)、银杏(Ginkgo biloba) (葛永奇等,2003)、水杉(Metasequoia glyptostroboides)(Li et al., 2005)、白云杉与恩格曼氏云杉的杂交种(Picea glauca × engelmanni) (Stoehr et al., 1997)、圣栎(Quercus ilex)(Burgarella et al., 2007)、萌芽白骨壤(Avicennia germinans)(Leiva et al., 2009)、云杉(Picea asperata) (Wang et al., 2010)、水松(Glyptostrobus pensilis)(吴则焰等,2011)、南方红豆杉(Taxus chinensis var. mairei)(李乃伟等,2011)等树种研究中也有类似报道。普遍认为,人工种群遗传多样性丢失的原因是营建人工林的母树资源有限或非随机采样,即种子可能仅采集于同一种群或少数植株(Rajora, 1999;Li et al., 2005;Burgarella et al., 2007;吴则焰等,2011)。但在窄叶南洋杉(Araucaria angustifolia)(Ferreira et al., 2012)、西藏长叶松(Pinus roxburghii)(Gauli et al., 2009)的天然与人工种群的遗传多样性比较研究中发现,大多数窄叶南洋杉人工种群的遗传多样性并没有降低,而西藏长叶松人工与天然种群的遗传多样性基本一致。这可能是这2个树种分别为当地的主要造林树种,年造林面积大,用种量多,人工林建立的种子材料来源于多个不同种群中的不同个体。伯乐树人工迁地保护种群遗传多样性降低是由于种质材料仅来自单个种群,不能充分反映南岭山地天然种群遗传组成,换言之,当前伯乐树迁地保护的规模尚未达到遗传多样性保护的要求。
3.2 伯乐树天然种群与人工种群遗传结构比较本研究表明,南岭山地伯乐树天然种群遗传分化较大(ΦST=0.340 7),接近于Nybom(2004)统计的植物种群RAPD分子标记遗传分化系数平均数(ΦST=0.35),远高于王美娜等(2011)对2个伯乐树种群的研究结果(GST=0.167),这可能与取样的种群不同有关,王美娜等(2011)研究的2个种群相距很近,实为一个复合种群,也可能与研究的标记类型不同有关。
植物种群遗传结构与其物种的生活史、分布区、交配系统、种子传播方式及群落演替阶段有关(Hamrick et al., 1991;Nybom, 2004)。南岭山地伯乐树天然种群遗传分化与广布种(ΦST=0.34)相接近,但高于长寿命林木(ΦST=0.25)及特有种(ΦST=0.26) (Nybom, 2004)。伯乐树天然种群片断化分布,南岭山地起伏,走向多变,地形复杂,形成丰富的局部小气候(陈涛等,1994),种群间为高山阻隔,势必促进种群遗传分化(Weller et al., 1996)。20世纪森林过度开发、开荒毁林,导致伯乐树种群数量及种群内个体数目减少,也可能加剧种群遗传分化(Stefenon et al., 2008)。
Nybom (2004)根据104种植物的ISSR标记研究数据总结,认为异交、自交及混合交配植物种群遗传分化系数ΦST平均值分别为0.27,0.65和0.40,伯乐树种群ΦST接近于混合交配植物。遗憾的是,迄今为止尚未开展伯乐树生殖生物学特性研究。
根据在一些公路、沟旁天然生长的伯乐树幼树周围2 km范围内没有发现成年大树的事实认为,鸟类、动物及水流可能是其种子传播的一条重要途径。ISSR标记表明,伯乐树天然种群遗传分化系数高于种子靠动物(ΦST=0.27)和水(或风)传播(ΦST=0.25)的植物,低于种子靠重力传播的植物(ΦST=0.45)。有关伯乐树种子传播方式有待于进一步研究。
伯乐树天然种群遗传分化(ΦST=0.340 7)高于人工迁地保护种群(ΦST=0.224 8),可能是伯乐树天然种群所处的生境(南岭山地中部及西部余脉,见图 1)条件差异大,而建立的人工迁地保护种群的种子采集地生境条件差异不大(大庚岭以南,见图 1),也可能是人工种群来源于母种群的部分个体而产生的偏差。
3.3 伯乐树遗传资源保护物种遗传多样性水平决定着物种对自然或人工选择反应的能力,是制定物种遗传多样性保护与利用策略的必需信息资料(Moreira Reis et al., 2004)。本研究表明,湖南省阳明山、莽山种群遗传多样性较其他种群高,建议南岭山地应优先保护这2个种群。伯乐树幼年期需半阴湿环境,成年树要求“少阴大阳”生境,天然更新困难(乔琦等,2011b),目前各级自然保护区开展的封禁措施虽然有利于伯乐树成年母树的保护,但不利于天然种群更新。建议应加强伯乐树天然种群中幼苗抚育管理,适度砍伐林下灌木丛,修剪林冠层的枝叶,逐步加强幼林光照强度,以满足不同年龄阶段对光照的需求,促进天然种群自然更新能力,扩大其种群内个体数目。
南岭山地伯乐树天然种群存在显著的遗传分化(ΦST=0.340 7),这表明,在制定迁地保护策略过程中应尽可能地从多个种群中收集种质材料,以增加人工迁地保护种群进化潜力(Stefenon et al., 2008)。由于伯乐树分布区内生态环境复杂多样,从多个不同生态地理种群采集种子,一方面可维持迁地保护种群的遗传完整性及适应性进化潜力,防止近交衰退和遗传漂移,另一方面也可能引起来源于生态地理条件差异大的种群材料远交衰退的危险(Ferreira et al., 2012)。因此,在目前缺乏伯乐树种群生殖生物学特性的详细信息条件下,建议分别在云贵高原、武陵山脉、南岭山地、幕阜山脉、武夷山脉等不同生态类型区内的国家级自然保护区,选择适宜伯乐树生长的较湿润的小气候环境,开展伯乐树迁地保护工作,扩大种植规模,增加人工迁地保护种群遗传变异水平,保证其遗传完整性。
| [] | 陈灵芝. 1993. 中国的生物多样性--现状及其保护对策. 北京, 科学出版社: 31-70. |
| [] | 陈涛, 张宏达. 1994. 南岭植物区系地理学研究: Ⅰ植物区系的组成和特点. 热带亚热带植物学报, 2(1): 10–23. |
| [] | 傅立国. 1992. 中国植物红皮书. 北京, 科学出版社: 194-195. |
| [] | 葛永奇, 邱英雄, 丁炳扬, 等. 2003. 孑遗植物银杏群体遗传多样性的ISSR分析. 生物多样性, 11(4): 276–287. |
| [] | 李乃伟, 贺善安, 束晓春, 等. 2011. 基于ISSR标记的南方红豆杉野生种群和迁地保护种群的遗传多样性和遗传结构分析. 植物资源与环境学报, 20(1): 25–30. |
| [] | 庞雄飞. 1993. 南岭山地生物群落简史. 生态学报, 12(1): 21–33. |
| [] | 彭莎莎, 黄华宏, 童再康. 2011. 濒危植物伯乐树遗传多样性的初步研究. 植物遗传资源学报, 12(3): 362–367. |
| [] | 乔琦, 秦新生, 邢福武, 等. 2011a. 珍稀植物伯乐树一年生更新幼苗的死亡原因和保育策略. 生态学报, 31(16): 4709–4716. |
| [] | 乔琦, 邢福武, 陈红峰, 等. 2011b. 中国特有珍稀植物伯乐树的研究进展和科研方向. 中国野生植物资源, 30(3): 4–8. |
| [] | 汪松, 解焱. 2004. 中国物种红色名录. 北京, 中国高等教育出版社: 336. |
| [] | 王美娜, 乔琪, 张荣京, 等. 2011. 广东南昆山与大岭山孑遗植物伯乐树群落特征比较与谱系地理学研究. 广西植物, 31(6): 789–794. |
| [] | 吴则焰, 刘金福, 洪伟, 等. 2011. 水松自然种群和人工种群遗传多样性比较. 应用生态学报, 22(4): 873–879. |
| [] | 吴征镒, 路安民, 汤彦承. 2003. 中国被子植物科属综论. 北京, 科学出版社: 702. |
| [] | 张宏达, 黄云峰, 缪汝槐, 等. 2004. 种子植物系统学. 北京:科学出版社: 319–320. |
| [] | 张玉梅, 徐刚标, 申响保, 等. 2012. 珙桐天然种群遗传多样性的ISSR标记分析. 林业科学, 48(8): 62–67. DOI:10.11707/j.1001-7488.20120810 |
| [] | 中华人国共和国林业部林业区划办公室. 1978. 中国林业区划. 北京, 中国林业出版社: 220-225. |
| [] | Burgarella C, Navascués M, Soto Á, et al. 2007. Narrow genetic base in forest restoration with holm oak (Quercus ilex L.) in Sicily. Ann For Sci, 64(7): 757–763. DOI:10.1051/forest:2007055 |
| [] | Crema A, Cristofolini G, Rossi M, et al. 2009. High genetic diversity detected in the endemic Primula apennina Widmer(Primulaceae) using ISSR fingerprinting. Plant Syst Evol, 280(1/2): 29–36. |
| [] | Excoffier L, Smouse P E, Quattro J M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitrochondrial DNA restriction data. Genetics, 131(2): 479–491. |
| [] | Feres J M, Guidugli M C, Mestriner M A, et al. 2009. Microsatellite diversity and effective population size in a germplasm bank of Hymenaea courbaril var. stilbocarpa (Leguminosae), an endangered tropical tree: recommendation for conservation. Genet Resour Crop Evol, 56(6): 797–807. DOI:10.1007/s10722-008-9402-2 |
| [] | Ferreira D K, Nazareno A G, Mantovani A, et al. 2012. Genetic analysis of 50-year old Brazilian pine (Araucaria angustifolia) plantation: implications for conservation planning. Conserv Genet, 13(2): 435–442. DOI:10.1007/s10592-011-0296-8 |
| [] | Finkeldey R, Hattemer H H. 2007. Tropical forest genetics. Heidelberg, Berlin, Springer: 12-33. |
| [] | Gauli A, Gailing O, Stefenon V M, et al. 2009. Genetic similarity of natural populations and plantations of Pinus roxburghii Sarg. in Nepal. Ann For Sci, 66(7): 702–713. DOI:10.1051/forest/2009063 |
| [] | Gillies A C, Navarro M C, Lowe A J, et al. 1999. Genetic diversity in Mesoamerican populations of mahogany (Swietenia macrophylla), assessed using RAPD. Heredity, 83: 722–732. DOI:10.1046/j.1365-2540.1999.00626.x |
| [] | Hamrick J L, Godt M J W, Murawski D A, et al. 1991. Correlation between species traits and allozyme diversity: implication for conservation biology//Falk D A, Holsinger K E. Genetics and Conservation of Rare Plants. UK: Oxford University Press, 245-253. |
| [] | Helenurm K. 2001. High levels of genetic polymorphism in the insular endemic herb Jepsonia malvifolia. J Hered, 92(5): 427–432. DOI:10.1093/jhered/92.5.427 |
| [] | Jordán-Pla A, Estrelles E, Boscaiu M, et al. 2009. Genetic variability in the endemic Leucojum valentinum. Biologia Plantarum, 53(2): 317–319. DOI:10.1007/s10535-009-0057-4 |
| [] | Kang U, Chang C S, KimY S. 2000. Genetic structure and conservation considerations of rare endemic Abeliophyllum distichum Nakai (Oleaceae) in Korea. J Plant Res, 113(2): 127–138. DOI:10.1007/PL00013923 |
| [] | Leiva D E, Duran V M, Perea N. 2009. Genetic diversity of black mangrove(Avicennia germinans) in natural and reforested areas of Salamanca Island Parkway, Colombian Caribbean. Hydrobiologia, 620(1): 17–24. DOI:10.1007/s10750-008-9611-x |
| [] | Li Y Y, Chen X Y, Zhang X, et al. 2005. Genetic differences between wild and artificial populations of Metasequoia glyptostroboides: implications for species recovery. Conserv Biol, 19(1): 224–231. DOI:10.1111/cbi.2005.19.issue-1 |
| [] | Moreira Reis A M, Grattapaglia D. 2004. RAPD variation in a germplasm collection of Myracrodruon urundeuva (Anacardiaceae), an endangered tropical tree: Recommendations for conservation. Genet Resour Crop Evol, 51(5): 529–538. DOI:10.1023/B:GRES.0000024159.48644.e6 |
| [] | Nybom H. 2004. Comparison of different nuclear DNA markers for estimation intraspecific genetic diversity in plants. Mol Ecol, 13(5): 1143–1156. DOI:10.1111/mec.2004.13.issue-5 |
| [] | Pandey M, Gailing O, Leinemann L. 2004. Molecular markers provide evidence for long-distance planting material transfer during plantation establishment of Dalbergia sissoo Roxh. in Nepal. Ann For Sci, 61(6): 603–606. DOI:10.1051/forest:2004056 |
| [] | Rajora O P. 1999. Genetic biodiversity impacts of silvicultural practices and phenotypic selection in white spruce. Theor Appl Genet, 99(6): 954–961. DOI:10.1007/s001220051402 |
| [] | Rohlf F J. 1998.NTSYS-pc version 2.0. Numerical taxonomy and multivariate analysis system. Exeter Software, Setauket, New York. |
| [] | Sozen E, Oxaydin B. 2010. A study of genetic variation in endemic plant Centaurea wiedemanniana by using RAPD markers. Ekoloji, 77: 1–8. |
| [] | Stefenon V M, Gailing O, Finkeldey R. 2008. Genetic structure of plantations and the conservation of genetic resources of Brazilian pine (Araucaria angustifolia). For Ecol Manag, 255(7): 2718–2725. DOI:10.1016/j.foreco.2008.01.036 |
| [] | Stoehr M U, El-Kassaby Y A. 1997. Levels of genetic diversity at different stages of the domestication cycle of interior spruce in British Columbia. Theor Appl Genet, 94(1): 83–90. DOI:10.1007/s001220050385 |
| [] | Thomas B R, Macdonald S E, Hicks M, et al. 1999. Effects of reforestation methods on genetic diversity of lodgepole pine: an assessment using microsatellite and randomly amplified polymorphic DNA markers. Theor Appl Genet, 98(5): 793–801. DOI:10.1007/s001220051136 |
| [] | Wang Z S, Liu H, Xu W X, et al. 2010. Genetic diversity in young and mature cohorts of cultivated and wild populations of Picea asperata Mast (Pinaceae), a spruce endemic in western China. Eur J Forest Res, 129(4): 719–728. DOI:10.1007/s10342-010-0377-0 |
| [] | Weller S G, Sakai A K, Straub C. 1996. Allozyme diversity and identity in Schiedea and Alsinidendron (Caryophyllaceae: Alsinoideae) in the Hawaiian Islands. Evolution, 50(1): 20–34. |
| [] | Xue D W, Ge X J, Hao G, et al. 2004. High genetic diversity in a rare, narrowly endemic primrose species: Primula interjacens by ISSR analysis. Acta Botanica Sinica, 46(10): 1163–1169. |
| [] | Yeh F C, Yang R C, Boyle T. 1999.POPGENE version 1.31: Microsoft Window-based Freeware for Population Genetic Analysis [CP /CD] Edmonton: University of Alberta. |
| [] | Zeng J, Zou Y P, Bai J Y, et al. 2003. RAPD analysis of genetic variation in natural populations of Betula alnoides from Guangxi, China. Euphytica, 134(1): 33–41. DOI:10.1023/A:1026113506563 |
 2012, Vol. 48
2012, Vol. 48