文章信息
- 张健, 张晓军, 任炳忠
- Zhang Jian, Zhang Xiaojun, Ren Bingzhong
- 基于28S rDNA基因的天牛科部分种类的分子系统发育
- Molecular Phylogeny of Some Longhorned Beetles Based on 28S rDNA Sequences
- 林业科学, 2012, 48(10): 86-94.
- Scientia Silvae Sinicae, 2012, 48(10): 86-94.
-
文章历史
- 收稿日期:2011-12-11
- 修回日期:2012-02-14
-
作者相关文章
2. 吉林省林业生物防治中心站 长春 130033;
3. 东北师范大学生命科学学院 长春 130024
2. Jilin Centre Station of Forestry Biological Control Changchun 130033;
3. School of Life Sciences, Northeast Normal University Changchun 130024
天牛科(Cerambycidae)隶属于鞘翅目(Coleoptera)叶甲总科(Chrysomeloidea), 是鞘翅目中最大的科之一,全世界约有45 000种,是鞘翅目昆虫中形态变异最具多样性的类群之一(Hua et al., 2009)。到2005年为止,中国天牛记录已有3 100余种(Hua et al., 2009)。天牛科昆虫绝大多数取食植物,其中很多种类是森林、果木的重要害虫,有些是毁灭性害虫。
传统分类学主要依据天牛科各亚科的蛹、幼虫及成虫的形态等可观察到的特征作为分类的重要依据(Linsley et al., 1961;1972;Gressit et al., 1970;Danilevsky,1985;Lawrence et al., 1995)。但由于天牛科各亚科物种形态和生态特征较为复杂,因而依据不同的分类特征得到的分类系统存在较多争议。目前国际上普遍采用的是Lawrence等在1995年提出的天牛科13亚科的分类系统。尽管目前对于天牛科各物种的研究较多,但是天牛科各亚科之间的系统发育关系尚缺乏系统研究。分子系统学研究为揭示物种间关系的本质带来希望,使生物系统学成为可验证的学科。天牛科作为一类重要的经济昆虫,国内外目前都未系统开展其分子系统学的研究,有必要从分子水平补充完善天牛科的系统分类研究理论及方法体系。
28S rDNA是真核生物编码核糖体大亚基的基因,由于其在进化过程中比较保守,同时在保守序列中也含有12个高变区,是研究生物高级阶元系统发育较好的分子标记(刘殿锋等,2005)。近年来,核基因序列广泛应用于鞘翅目昆虫高级分类阶元研究。Sagemami-Oba等(2007)采用核基因28S rDNA部分序列探讨了叩甲科(Elateridae)9亚科的系统发育关系,解释了叩甲科昆虫生物发光特性的演化。Marvaldi等(2009)采用18S rDNA(V4-V5区与V7-V9区)和28S rDNA(D2区)序列对鞘翅目扁甲系(Cucujiformia)共计104种甲虫进行系统发育重建。结果证明了多食亚目(Phytophaga)的单系性,且象甲总科(Curculionoidea)和叶甲总科(Chrysomeloidea)为姐妹群。本研究通过对部分天牛科昆虫28S rDNA基因片段的序列测定,结合GenBank数据库中天牛科昆虫28SrDNA基因片段序列,重建系统发育树,以期从分子水平探讨天牛科各亚科间的系统发育关系,为天牛科分类系统修订提供参考。
1 材料与方法 1.1 试验材料标本于2009—2010年6月至9月间采自吉林省东部、中部及西部地区。标本采集后,立即放入无水乙醇中固定,浸泡的第1个月更换无水乙醇3~4次,以后一直用无水乙醇保存于塑料瓶中,置于-80 ℃冰箱保存备用。标本依据陈世骧等(1959)、蒲富基(1980)、蒋书楠等(1985;2001)、王直诚(2003)、孟庆繁等(2008)、Hua(2009)等天牛科分类专著进行鉴定。与DNA提取相对应的干制标本保存在东北师范大学生命科学学院标本馆。供试材料种类、采集地及GenBank序列号如表 1所示。
|
|
采用酚-氯仿-异戊醇法提取总基因组DNA(Sambrook et al., 1989),并有所改进。DNA检测采用2种方法:1)1%琼脂糖凝胶电泳法;2)紫外分光光度计法,记录在260 nm下的OD值及260 nm与280 nm OD值之比以备参考。
1.3 PCR扩增及测序用于扩增28S rDNA基因的引物为Kim等(2000)设计的通用引物。引物序列为:上游引物GACTACCCCCTGAATTTAAGCAT(23 bp)和下游引物GACTCCTTGGTCCGTGTTTCAAG(23 bp)。扩增反应体系体积为50 μL,包括5 μL pH为8.3 10×PCR缓冲液,4 μL 2.5 mmol·L-1 MgCl2,1.5 U的Taq DNA聚合酶,1 μL 10 mmol·L-1的dNTP(C,G,A,T),以上试剂均购自Takara生物公司(大连),2 μL 10 μmol·L-1引物(上海生工合成),2 μL DNA模板和33.7 μL H2O。扩增产物采用1%琼脂糖凝胶电泳检测,方法与总DNA样品的检测相同,不同之处在于要用DNA marker(DL2000)标记扩增片段的大小。
PCR产物委托上海生工生物技术公司进行纯化、测序,测序仪为ABI 3730型,测序方法为四色荧光标记双脱氧链终止法,采用正反双向测序,以提高序列的精确度。
1.4 DNA序列数据的处理采用Clustal 1.83软件(Thompson et al., 1997)进行序列比对。比对后的结果采用MEGA 4.0软件(Tamura et al., 2007)进行序列组成分析,计算各分类单元之间的遗传距离及标准误差,各数据组的碱基组成(nucleotide composition)、保守位点(conserved sites)、变异位点(variable sites)及简约信息位点(parsimony information sites)等。
1.5 系统发育树的建立本文采用2种方法,利用自测的32种天牛的28S rDNA序列和GenBank数据库已有的5亚科6种天牛的28S rDNA序列,构建天牛科7亚科38种天牛的系统发育树。从GenBank数据库下载的5亚科6种天牛的序列信息见表 1。外群采用的是象甲科(Curculionidae)的Araucarius minor(序列号AF308351)和步甲科(Carabidae)的Calathus mollis(序列号FJ173071)。
系统发育树的重建使用PAUP 4.0b10(Swofford,2002)和MrBayes 3.1.2(Huelsenbeck et al., 2001a)软件进行,采用最大简约法(maximum parsimony method,MP)和贝叶斯法(Bayesian inference method,BI)分别对所研究的天牛种类进行系统发育重建,并与传统分类方法进行比较。系统树各分支的置信度由Bootstrap 1 000次重抽样检验,DNA序列变异中的颠换和转换赋予相同的加权值。
最大简约分析中,采用均等加权,启发式搜索和TBR分支交换法,随机添加序列(random-addition sequences)项设为10次。利用非参数重复抽样法(Bootstrap)计算节点间的可靠性,进行1 000次重复检验。贝叶斯法分析运算中马尔科夫链的蒙特卡罗方法(Markov chain Monte Carlo process)设置为4条链同时运行,3条热链1条冷链;根据Modeltest 3.7(Posada et al., 1998)对序列进行相应模型比较分析后确认其最佳分析模型(general time reversible model)(Yang et al., 1997;Huelsenbeck et al., 2001b)。以随机树为起始树,运行1 500 000代,每100代抽样1次;在舍弃老化样本(burn-in samples)(占25%)后,根据剩余的样本构建一致树并计算参数贝叶斯验后概率(Bayesian posterior probabilities)(Pbay)。
2 结果与分析 2.1 28S rDNA基因序列组成及变异分析将序列比对结果导入MEGA4.0软件进行序列组成分析。测得的碱基平均含量为:A,19.8%;T,19.3%;G,33.2%;C,27.6%;A+T,39.1%;G+C,60.8%。G+C含量明显高于A+T,碱基组成偏向G和C。系统发育分析的772个位点中,其中保守位点521个,占全部位点的67.5%。简约信息位点136个,占全部位点的17.6%。变异位点216个,占全部位点的28.0%。转换(transition)/颠换(transversion)平均值(R值)为1. 80,转换稍高于颠换。其碱基组成特点与28S rDNA完全相符。
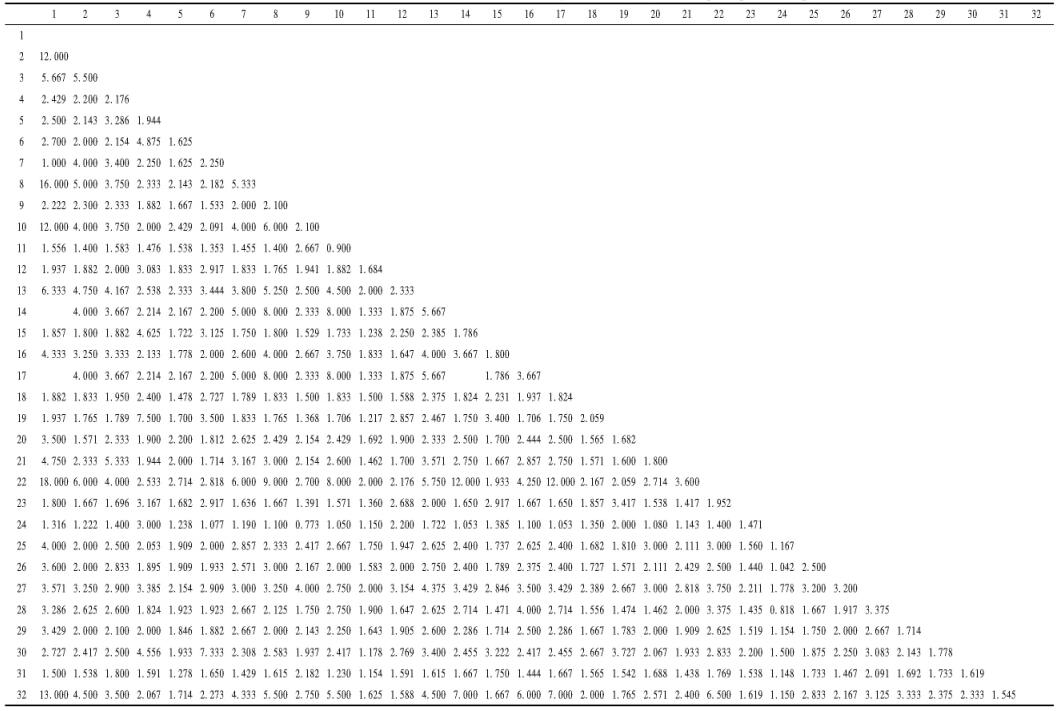
2.2 遗传距离分析采用MEGA4.0软件计算32种天牛之间的非校正P距离,结果见表 2。R值(TS/TV)见表 3。
|
|
|
|
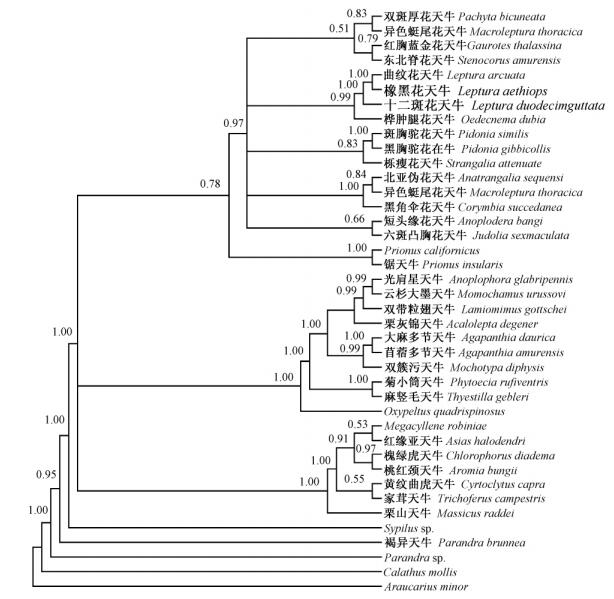
2种方法得到的天牛科昆虫分子系统树表明:所有天牛科种类都聚在一起,与外群明显地分开,证明了天牛科的单系性。最大简约树(图 1)和贝叶斯树(图 2)的结构基本一致,将7亚科38种天牛主要分为4个聚类簇,它们来源于2个支系,支系1包括锯天牛亚科和花天牛亚科,位于系统发育树的顶部;支系2包括沟胫天牛亚科、天牛亚科及盾天牛亚科,位于系统发育树的中部;裸天牛亚科和异天牛亚科位于系统发育树的最基部。

|
图 1 基于28S rDNA基因部分序列构建的最大简约树 Fig.1 Most-parsimonoius tree of Cerambycidae based on the analysis of the 28S rDNA sequences 树长1 300,一致性指数0.682 7,保留指数0.600 4,树节点上数字为大于50%的自举检验值。 Tree length 1 330, consistency index 0.682 7, retention index 0.600 4. Numbers above branches indicate bootstrap values over 50%. |
|
图 2 基于28S rDNA基因部分序列构建的贝叶斯树 Fig.2 Bayesian tree of Cerambycidae based on the analysis of the 28S rDNA sequences 树节点上数字为大于50%的自举检验值。 Numbers above branches indicate bootstrap values over 50%. |
目前研究表明:从总DNA基因组中用PCR扩增线粒体DNA片段的同时,可能扩增出起源于线粒体的假基因Numts(nuclear mitochondrial like sequences),而不仅是线粒体DNA上的目的序列(Richly et al., 2004)。这种情况下,对PCR产物进行电泳检测时,通常有非特异性条带出现,这样测序结果会出现重叠峰,产生错误的测序结果,最终影响系统发育和群体遗传学的研究结果。Numts现象一直引起许多学者的广泛关注,并不断有新的报道。从真菌到昆虫,直至脊椎动物中均发现存在Numts。对于鞘翅目昆虫假基因的研究较少,仅见于小蠹科(Scolytidae)(Jordal,2007)、拟步甲科(Tenebrionidae)(Mestrovic et al., 2006)和虎甲科(Cicindelidae)的昆虫(Pons et al., 2005)。通常认为:全基因组较小的生物体很少有假基因,而全基因组较大的生物体更可能存在假基因。也有学者认为假基因的丰富度与基因组的大小没有直接关系。Koutroumpa等(2009)发现了樟子松松墨天牛(Monochamus galloprovinvialis)存在假基因,这是天牛科昆虫中首次发现假基因。本研究扩增出的PCR产物经电泳检测均显示清晰的条带,亮度较高,未产生非特异性条带,测序结果在后续序列组成和系统发育重建过程中,未发现组成或进化模式上显著不同的序列,且在系统发育重建中采用不同方法得到的结果有一定的重复性,本研究所得到的序列就是目的序列。
本研究基于28S rDNA基因片段序列,采用自行测定的天牛科4亚科32种天牛,并结合由GenBank数据库下载已有的5亚科6种天牛28S rDNA基因片段序列,并以象甲科和步甲科昆虫的28S rDNA基因片段序列作为外群,采用最大简约法和贝叶斯法构建系统发育树,结果证明了天牛科各亚科的单系性;且异天牛亚科处于树的较为基部位置,为较早分化出来的分支,应该为较为原始的类群。此结果与Linsley(1961)基于天牛科幼虫和成虫特征得出的天牛科各亚科亲缘关系,认为异天牛为较为原始的类群的结果相一致。此外,本文的最大简约树结果与蒋书楠等(2001)采用成虫和幼虫的形态特征和生物学特性对天牛科各亚科进行聚类分析的结果部分一致,得到天牛科4亚科从原始到进化的关系为天牛亚科→沟胫天牛亚科→锯天牛亚科→花天牛亚科。认为天牛亚科和沟胫天牛亚科是较为原始的类群,锯天牛亚科和花天牛亚科是较为进化的类群。此外,本研究最大简约树结果将沟胫天牛亚科和天牛亚科聚在一个分支,这与本课题组基于16S rDNA基因部分序列对9种天牛进行系统发育重建得到的结果一致(张健等,2010)。
核基因相对于线粒体基因进化较慢,进化慢的基因对树的中部和端部解析程度较高,进化快的基因对树的端部解析程度较高,联合数据对树的支持要远高于单一基因。因此,在以后的研究中,应该采用线粒体和核基因联合数据集进行系统发育树的构建,将有助于进一步阐明天牛科的系统发育关系。
| [] | 陈世骧, 谢蕴贞, 邓国藩. 1959. 中国经济昆虫志. 第一册.鞘翅目:天牛科(一). 北京, 科学出版社. |
| [] | 蒋书楠, 陈力. 2001. 中国动物志, 昆虫纲. 第二十一卷.鞘翅目:天牛科:花天牛亚科. 北京, 科学出版社. |
| [] | 蒋书楠, 蒲富基, 华立中. 1985. 中国经济昆虫志. 第三十五册.鞘翅目:天牛科(三). 北京, 科学出版社. |
| [] | 刘殿锋, 蒋国芳. 2005. 核基因序列在昆虫分子系统学上的应用. 动物分类学报, 30(3): 484–492. |
| [] | 孟庆繁, 高文韬. 2008. 长白山访花甲虫. 北京, 中国林业出版社. |
| [] | 蒲富基. 1980. 中国经济昆虫志. 第十九册.鞘翅目:天牛科(二). 北京, 科学出版社. |
| [] | 王直诚. 2003. 东北天牛志. 长春, 吉林科学技术出版社. |
| [] | 张健, 李晓强, 刘斐, 等. 2010. 基于线粒体16S rDNA基因的天牛科部分种类分子系统学研究(鞘翅目:天牛科). 昆虫分类学报, 32(Suppl.): 1–7. |
| [] | Danilevsky M L, Miroshnikov A I. 1985. Timber-Beetles of Caucasus (Coleoptera, Cerambycidae). Moscow, The Key Krasnodar: 419. |
| [] | Gressitt J L, Rondon J A. 1970. Cerambycidae of Laos (Disteniidae, Prioninae, Philinae, Aseminae, Lepturinae, Cerambycinae). Pacific Insects Monograph, 24: 1–314. |
| [] | Hua L Z, Hajime N, Saemulson G A, et al. 2009. Iconography of Chinese Longicorn Beetles (1406 Species) in Color. Guangzhou, Sun Yat-sen University Press: 1-474. |
| [] | Huelsenbeck J P, Ronquist F. 2001a. MRBAYES, Bayesian inference of phylogenetic trees. Bioinformatics, 17(8): 754–755. DOI:10.1093/bioinformatics/17.8.754 |
| [] | Huelsenbeck J P, Ronquist F, Nielsen R, et al. 2001b. Bayesian inference of phylogeny and its impact on evolutionary biology. Science, 294(5550): 2310–2214. DOI:10.1126/science.1065889 |
| [] | Jordal B H. 2007. Reconstructing the phylogeny of Scolytinae and close allies:major obstacles and prospects for a solution. Proc RMRSP-45.USDA For Serv Proc, 45: 3–9. |
| [] | Kim C G, Zhou H Z, Imura Y, et al. 2000. Pattern of morphological diversification in the Leptocarabus ground beetles Coleoptera, Carabidae as deduced from mitochondrial ND5 gene and nuclear 28S rDNA sequences. Molecular Biology and Evolution, 17(1): 137–145. DOI:10.1093/oxfordjournals.molbev.a026226 |
| [] | Koutroumpa F A, Lieutier F, Roux-Morabito G. 2009. Incorporation of mitochondrial fragments in the nuclear genome (Numts) of the longhorned beetle Monochamus galloprovincialis(Coleoptera, Cerambycidae). Journal of Zoological Systematics and Evolutionary Research, 47(2): 141–148. DOI:10.1111/jzs.2009.47.issue-2 |
| [] | Lawrence J F, Newton A F. 1995. Families and subfamilies of Coleoptera (with selected genera, notes, references and data onfamily-group names)// Pakaluk J, Slipinsky S. Biology, Phylogeny, and Classification of Coleoptera: Papers celebrating the 80th Birthday of Roy A. Crowson. Warsaw (Poland): Museum I Instytut Zoologii PAN, 779-1007. |
| [] | Linsley E G. 1961. The Cerambycidae of North America.Part Ⅰ.Introduction.University of California Publications in Entomology. CA-Berkeley, University of California Press: 1-97. |
| [] | Linsley E G, Chemsak J A. 1972. The Cerambycidae of North America.Part Ⅵ, No.1.Taxonomy and Classification of the Subfamily Lepturinae.University of California Publications in Entomology. CA-Berkeley, University of California Press: 1-138. |
| [] | Marvaldi A E, Duckett C N, Kjer K M, et al. 2009. Structural alignment of 18S and 28S rDNA sequences provides insights into phylogeny of Phytophaga (Coleoptera:Curculionoidea and Chrysomeloidea). Zoologica Scripta, 38(1): 63–77. DOI:10.1111/zsc.2009.38.issue-1 |
| [] | McKenna D D, Sequeira A S, Marvaldi A E, et al. 2009. Temporal lags and overlap in the diversification of weevils and flowering plants. Proceedings of the National Academy of Sciences, 106(17): 7083–7088. |
| [] | Mestrovic N, Mravinac B, Plohl M, et al. 2006. Preliminary phylogeny of Tribolium beetles (Coleoptera:Tenebrionidae) resolved by combined analysis of mitochondrial genes. European Journal of Entomology, 103(3): 709–715. DOI:10.14411/eje.2006.094 |
| [] | Pons J, Vogler A P. 2005. Complex pattern of coalescence and fast evolution of a mitochondrial rRNA pseudogene in a recent radiation of tiger beetles. Molecular Biology and Evolution, 22(4): 991–1000. DOI:10.1093/molbev/msi085 |
| [] | Posada D, Crandall K A. 1998. MODELTEST:testing the model of DNA substitution. Bioinformatics, 14(9): 817–818. DOI:10.1093/bioinformatics/14.9.817 |
| [] | Richly E, Leister D. 2004. Numts in sequenced eukaryotic genomes. Molecular Biology and Evolution, 21(6): 1081–1084. DOI:10.1093/molbev/msh110 |
| [] | Ruiz C, Jordal B, Serrano J. 2009. Molecular phylogeny of the tribe Sphodrini (Coleoptera:Carabidae) based on mitochondrial and nuclear markers. Molecular Phylogenetics and Evolution, 50(1): 44–58. DOI:10.1016/j.ympev.2008.09.023 |
| [] | Sagemami-Oba R, Oba Y, Ohira H. 2007. Phylogenetic relationships of click beetles (Coleoptera:Elateridae) inferred from 28S ribosomal DNA:Insights into the evolution of bioluminescence in Elateridae. Molecular Phylogenetics and Evolution, 42(2): 410–421. DOI:10.1016/j.ympev.2006.07.017 |
| [] | Sambrook J, Fritsch E F, Maniatis T. 1989. Molecular cloning:A laboratory manual. New York, Coldspring Harbor Laboratory Press, Cold Spring Harbor. |
| [] | Swofford D L. 2002. PAUP* phylogenetic analysis using parsimony (*and other methods). Version 4.Massachusetts: Sinauer Associates, Sunderland. |
| [] | Tamura K, Dudley J, Nei M, et al. 2007. MEGA4:Molecular evolutionary genetics analysis (MEGA) Software Version 4.0. Molecular Biology and Evolution, 24(8): 1596–1599. DOI:10.1093/molbev/msm092 |
| [] | Thompson J D, Gibson T J, Plewniak F, et al. 1997. The CLUSTAL X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25(24): 4876–4882. DOI:10.1093/nar/25.24.4876 |
| [] | Yang Z, Rannala B. 1997. Bayesian phylogenetic inference using DNA sequences:a Markov chain Monte Carlo method. Journal of Molecular Evolution, 14(7): 717–724. DOI:10.1093/oxfordjournals.molbev.a025811 |
 2012, Vol. 48
2012, Vol. 48