文章信息
- 夏明瑞, 周国娜, 高宝嘉
- Xia Mingrui1, Zhou Guon, Gao Baoji
- 不同林分类型油松毛虫COⅠ基因变异分析
- Sequence Variation of COⅠ Gene of Dendrolimus tabulaeformis in Different Types of Stand
- 林业科学, 2012, 48(9): 171-175.
- Scientia Silvae Sinicae, 2012, 48(9): 171-175.
-
文章历史
- 收稿日期:2011-04-01
- 修回日期:2011-06-17
-
作者相关文章
2. 河北北方学院 张家口 075000
2. Hebei North University Zhangjiakou 075000
昆虫和其他生物一样,在长期的生长发育和进化过程中保留了一些遗传特征,使物种得以延续,但同时也存在一些变异,以适应新的环境。这些遗传变异不仅仅存在于昆虫的种间,也存在于同种昆虫的不同种群之间(蔡青年等,2004; 施立明,1990)。不同的寄主植物、地理隔离及其他环境条件都会对昆虫种群的遗传变异产生一定的影响。顾耘等(2002)利用RAPD技术对辣椒(Chili pepper)、烟草(Nicotianatabacum)和番茄(Solanum lycopersicum)上所寄生的棉铃虫(Helicoverpa armigera)进行分析,发现棉铃虫不同寄主种群已经存在明显的遗传分化; 袁一杨等(2008)利用AFLP技术对不同林分下油松毛虫(Dendrolimus tabulaeformis)的遗传多样性研究发现:林分类型是影响油松毛虫种群遗传多样性的重要因素,不同林分生境对油松毛虫产生不同的选择压力,使油松毛虫种群发生遗传分化; 杨宝山等(2009)对不同地理种群银杏大蚕蛾(Caligula japonica)的种群遗传分化进行了研究,并探讨其与生态环境的关系。
随着分子生物学技术的发展,学者们不仅运用RAPD、AFLP、SSR、ISSR等技术对昆虫在不同环境因素影响下的遗传多样性进行研究,而且更多地运用线粒体DNA分子标记技术对昆虫的种群遗传分化进行研究。线粒体DNA具有进化速率较核DNA快,高度的碱基替换率、严格的母性遗传,无遗传重组、倒位、易位等突变等特点(王备新等,2002),被广泛应用于讨论种群遗传结构,通常能够全面反映种群内和种群间的遗传变异。不同的线粒体DNA进化速率不同,其中线粒体细胞色素C亚基Ⅰ基因(COⅠ)所编码的亚基是电子传递链中最重要的核心酶,在线粒体基因中有较丰富的变异,且在细胞内是多拷贝,更有利于PCR扩增,DNA中没有内含子和高突变区,含有较少的非编码区域,容易得到没有争议的结果,适合于种群遗传分化的研究。
油松毛虫是重要的林业害虫,以往的研究发不同林分类型对其种群遗传结构有一定的影响,混交林对油松毛虫种群的基因流有一定阻隔作用,降低了林内的遗传多样性; 种群间的遗传分化与生境有关(袁一杨等,2008; 张学卫,2011)。本研究采用PCR和测序技术对不同林分类型中油松毛虫线粒体COⅠ的部分基因进行测定和分析,探讨不同林分类型对油松毛虫种群遗传分化的影响,为揭示油松毛虫遗传分化与林分类型的关系提供依据。
1 材料与方法 1.1 试验材料供试油松毛虫4个种群于2010年7月全部采自河北省平泉县黄土梁子,其中油松(Pinus tabulaeformis)纯林2块,油松和华北落叶松(Larix principis-rupprechtii)、油松和山杨(Populus davidiana)混交林各1块,各样地间直线距离约为2 km,4个松毛虫种群间相对独立,采集信息见表 1。表 1中的样品数量为每个种群测定数量,编号分别为C1,C2,H1,H2。
|
|
将采回的油松毛虫蛹连同松枝一起放置在室内的养殖笼里,保持笼内湿度,待其羽化后将雌成虫取出,装袋封口标记后-20 ℃保存。羽化后的雌虫全部用于测定
1.3 基因组DNA的提取应用上海生工SK-1205 UNIQ柱式动物基因组DNA抽提试剂盒进行提取,将单头雌性成虫取出,取中后胸肌肉置于预冷的研钵中,加适量液氮迅速研磨至粉末,迅速转移到离心管中,然后按照试剂盒说明进行提取,所提DNA放于-20 ℃保存备用。
1.4 PCR扩增和序列测定引物采用线粒体细胞色素C扩增通用引物,设计参考Simon等(1994),引物序列为: LYQ3(5'-CCTGGATCTTTAATTGGAGA-3')和LYQ4(5'-GGTAAAATTAAAATATAAACTTC-3')。
PCR采用张学卫(2010)所优化的松毛虫线粒体RCR体系,并对退火温度进行优化。50 μL体系中,50 ngDNA模板溶液模板、引物0.2 μmol·L-1、dNTP 0.3 mmol·L-1、Taq DNA聚合酶2.0 U、Mg2+2.5 mmol·L-1,PCR buffer 5 μL,加ddH2O补足至终体积50 μL。扩增条件为: 94 ℃预变性3 min,35个循环,包括94 ℃变性30 s,55 ℃退火60 s,72 ℃延伸60 s,最后在72 ℃充分延伸5 min,扩增产物用1.5%的琼脂糖凝胶电泳检测。
1.5 PCR产物的回收和测序PCR产物的回收采用上海生工SK-1311UNIQ DNA凝胶回收试剂盒,按说明进行操作,回收的DNA样品委托上海生工公司北京中科希林测序部进行测序,所用测序仪器为ABI PRISM 3730xl。
1.6 序列分析方法用Clustal X程序对测得的COⅠ基因片段进行序列对齐,并辅助人工校对,确定序列后在NCBI中用BLAST进行相似性搜索,确定所得序列为目的基因片段。用Mega 4.1计算不同序列间的可变位点、简约信息位点、转换颠换比值、遗传距离及各碱基含量。采用邻接法(neighborjoining method,NJ)和简约法(maximum parsimonymethod,MP)进行聚类分析。运用DNAsp5.0计算基因分化系数(Fst)和基因流(Nm)。
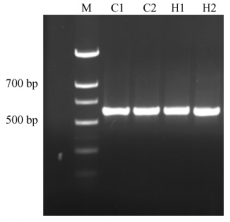
2 结果与分析 2.1 PCR扩增结果利用昆虫线粒体COⅠ基因通用引物扩增,均扩增出1条亮带,且带型一致。大小均在580 bp左右(图 1),且3次重复实验结果一致,符合预定结果。
|
图 1 PCR扩增检测结果 Fig.1 Results of amplification M:标准DNA DNA Marker; C1,C2,H1,H2: PCR扩增产物PRC amplification products. |
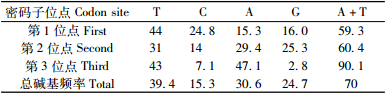
4块林地中油松毛虫扩增的COⅠ基因序列可用于分析的长度为544 bp,测序结果在NCBI中用BLAST进行了相似性检索,结果显示所测序列为所需目的片段。在所测得的544 bp序列中,保守位点480个,变异位点64个,简约信息位点34个,自裔位点30个。序列变异性为11.7%。A,T,C,G的含量分别是30.6%,39.4%,15.3%,14.7%,A+T含量为70%,C+G含量为30%,A+T含量明显高于C+G含量,这与昆虫一般的核苷酸频率相一致。
在密码子不同的位点,碱基含量存在很大差异(表 2)。密码子的第3位点A+T含量为90.1%,远远高于C+G含量,反映出COⅠ基因在密码子的使用上具有明显的偏向性。
|
|
DNA序列的碱基替换类型可分为转换(transition,Ts)和颠换(transversion,Tv),它是衡量序列进化的重要参数。运用MEGA4.1对序列之间的核苷酸转换和颠换数以及R值进行统计分析,核苷酸替换主要发生在密码子第3位点上,为66.7%,第1位点最为保守,为6.7%。第3位点表现出转换高于颠换,转换主要发生在T和C之间,颠换主要发生在A和T之间,转换多于颠换。总体的颠换比为2.17,大于颠换比的临界值2.0。
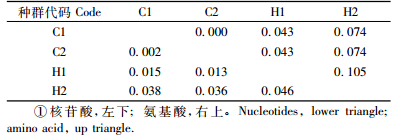
2.3 油松毛虫不同种群间遗传距离及聚类分析采用Mega 4.1软件,基于Kimura 2-paramrters模型计算序列间的遗传距离,基于p-distance模型计算氨基酸的遗传距离(表 3)。从表 3中可以看出:核苷酸遗传距离在0.002~0.046之间,平均遗传距离为0.025,氨基酸遗传距离范围在0.000~0.105之间,平均遗传距离为0.057。2组数据都显示:纯林的C1和C2种群遗传距离差异很小,但与同组的混交都有一定的差异,2种混交林间的差异最大,2混交林种群间出现了遗传分化。
|
|
利用Mega4.1软件,基于Kimura 2参数(2-parameter)距离采用邻近法(NJ)和简约法(MP)进行聚类分析,分别见图 2a和图 2b。可以看出:采用2种不同的方法进行聚类分析,2块纯林中的种群C1和C2先聚为第1支,然后和山杨与油松的混交林H1聚为第2支,最后与落叶松与油松的混交林种群H2聚为第3支。这一结果表明:油松毛虫种群间的遗传分化可能和林分类型有着一定的关系。

|
图 2 基于NJ法(a) MP(b)法构建的不同林分中油松毛虫COⅠ基因的聚类图 Fig.2 MP (a) and NJ(b) dendrograms based on the COⅠ gene sequence of D. tabulaeformis |
种群间的遗传分化程度常用基因分化系数(Fst)和基因流(Nm)等指标来衡量。其中Fst越大表明遗传分化越明显,而Nm则相反,Nm值越大表明群体间基因交流越充分,从而遗传分化越小(李菁等,2010)。利用DNAsp5.0计算种群间的分化程度Fst为在-0.128~0.117之间,而基因流(Nm)都大于4,根据已有的研究可知,C2中种群与H1中种群之间已经出现遗传分化; 而由于种群间基因交流频繁,从而降低了种群间遗传分化程度。
|
|
从mtDNA COⅠ分子水平上对同一区域的不同林分类型中油松毛虫种群的遗传关系进行的初步探讨表明:纯林和混交林中油松毛虫种群间存在一定程度上的遗传分化,但分化程度较低。选择相同地理区域种群,减少了地理区域因素对种群分化的影响。
测定的COⅠ基因全长为544 bp,A,T,C,G的含量分别是30.6%,39.4%,15.3%,14.7%,A+T含量为70%,这种高AT含量的特性是昆虫线粒体序列碱基组成的一种共性(高天翔,2000; 任竹梅等,2002; 顾杰等,2009)。在密码子第3位上A+T含量高达90.1%,和其他昆虫表现相同,如柳蚕(Actias selene)(88.5%)、小稻蝗(Oxya podisma)(90.4%)、线蛱蝶(Limenitis sp.)(92.5%)(濮佳明等,2009; 任竹梅等,2003; 吴东霞,2007),表现出明显的偏好型,这种偏好型是由于第3位点的突变大多为同义突变,受到自然选择压力小,突变后容易固定(周继亮等,2001)。而第2位点较为保守,因为核苷酸的替换,特别是颠换的发生,极大多数是非同义替换,会引起氨基酸改变。
碱基替换是序列进化一个重要参数,Knight等认为,转换颠换之比的值若小于2.0,则此基因序列突变已达到饱和状态(Knight et al., 1993)。本研究中R值为2.17,认为COⅠ基因序列变异并未达到饱和状态,随着遗传差异的增加,转换趋向饱和。碱基的替换主要发生在密码子的第3位点上,占变异总数的66.7%,转换主要发生在T和C之间,颠换主要发生在A和T之间,与戴金霞等(2005)、李爱玲等(2004)发现转换的发生主要以C-T为主的研究结果一致。研究发现:转换/颠换率在不同进化距离的分类阶元之间具有明显差异,通常亲缘关系较近的阶元之间核苷酸替换表现为转换率大于颠换率,亲缘关系远则反之(Simon et al., 1994)。本研究中,油松毛虫线粒体COⅠ基因变异转换大于颠换,验证了上述规律。
本研究表明不同林分类型中的油松毛虫种群在线粒体基因组水平上差异是很小的,平均遗传距离为0.025,这与张学卫等(2011)基于cytb基因对油松毛虫种群分化的研究结果相似,这是由于所采样品都取自同一个地域,有着高度相似的生态环境条件,因而各种群间亲缘关系较近。
通过聚类结果分析,笔者认为各个种群按照不同的林分类型条件而形成一定的遗传分化,首先2块纯林先聚为一支,而后分别与混交林聚为另一支,分析原因可能是由于不同样地中林分结构发生变化,影响到油松体内的营养物质的数量及质量(庞正轰,1990),油松毛虫种群通过自身的改变来适应油松的变化,从而导致油松毛虫的遗传分化。因此,推测林分类型的差异是影响油松毛虫遗传分化的主要因素。
基因分化系数可以反映不同种群之间的基因差异程度。Wright(1978)认为:当Fst≤0.05时,种群几乎无分化,当0.05<Fst≤0.15时,种群中等分化,当0.15<Fst≤0.25,种群发生明显分化,当Fst>0.25时,种群极度分化。本研究中纯林种群与混交林种群间的Fst分别为0.117和0.111,说明纯林和混交林油松毛虫种群间存在的一定的遗传分化,而纯林间遗传分化系数小于0.05,并未出现明显的遗传分化。由此推断,林分类型可能是影响油松毛虫种群遗传分化的因素。
基因流是种群遗传结构均质化的主要因素之一,通常具有较大基因流的物种比具有较小基因流的物种遗传分化小,通常当Nm>4时,种群间交流比较充分,遗传分化较小,当Nm<1时,说明种群可能由于遗传漂变而发生分化(Millar et al., 1991)。本研究中油松毛虫种群间基因流水平均大于4,说明基因交流可能很频繁,导致各种群间遗传分化不明显。油松毛虫有一定的飞行能力,且各样地间地理距离较近,可能是基因交流水平较高的原因之一,从而降低了种群间的遗传差异。
应用COⅠ基因的部分序列对油松毛虫种群遗传分化进行研究,因受自然条件限制,所选定的混交林种类较少,只有山杨与油松混交(针阔混交),落叶松和油松混交(针针混交),但有一定的代表性。在林分选择的地理区域上也有一定的局限性。建议今后采集更多混交类型的林分中的油松毛虫种群、加大每个种群的采集量、选用多种基因序列分析,以便得出更准确的结论。
4 结论本研究基于对mtDNA COⅠ基因部分序列的测定和分析,探讨了不同林分类型中油松毛虫种群间的遗传关系,结果表明同一区域的纯林和混交林油松毛虫种群间存在遗传分化,而同一区域的纯林种群间油松毛虫分化并未分化。
| [] | 蔡青年, 胡熳华, 王宇, 等. 2004. 蚜虫种群遗传多样性的影响因素及分子基础. 昆虫知识, 41(4): 285–290. DOI:10.7679/j.issn.2095-1353.2004.076 |
| [] | 代金霞, 郑哲民. 2005. 基于Cytb基因序列探讨蝽亚科11种昆虫的系统发育关系. 昆虫知识, 42(4): 395–399. DOI:10.7679/j.issn.2095-1353.2005.098 |
| [] | 高天翔, 张秀梅, 渡边精一. 2000. 中华绒螯蟹与日本绒螯蟹线粒体12S rDNA序列比较. 水产学报, 24(5): 413–416. |
| [] | 顾杰, 毛雅琴.王莉萍, 等. 2009. 四纹豆象不同地理种群的遗传分化. 昆虫学报, 52(12): 1349–1355. DOI:10.3321/j.issn:0454-6296.2009.12.009 |
| [] | 顾耘, 张迎春, 王思芳. 2002. 棉铃虫不同寄主种群遗传分化的RAPD分析. 华东昆虫学报, 11(2): 30–34. |
| [] | 李爱玲, 徐安英, 沈兴家, 等. 2004. 家蚕、野桑蚕线粒体Cytb基因片段序列分析及分子进化研究. 蚕业科学, 30(1): 80–84. |
| [] | 李菁, 张颖, 王振营, 等. 2010. 基于线粒体DNA COⅡ基因的亚洲玉米螟中国不同地理种群遗传分化及基因流研究. 昆虫学报, 53(10): 1135–1143. |
| [] | 庞正轰. 1990. 封山林分针叶在生理特性上对赤松毛虫抗虫效应的研究. 北京林业大学学报, 12(2): 6–12. |
| [] | 濮佳明, 武松, 霍锡敏, 等. 2009. 珍稀绢丝昆虫柳蚕的DNA条形编码与系统进化初步分析. 蚕业科学, 35(1): 154–159. |
| [] | 任竹梅, 马恩波, 郭亚平. 2002. 蝗虫总科部分种类Cytb基因序列及系统进化研究. 遗传学报, 29(4): 314–321. |
| [] | 任竹梅, 马恩波, 郭亚平. 2003. 不同地域小稻蝗mtDNA部分序列及其相互关系. 昆虫学报, 46(19): 51–57. |
| [] | 施立明. 1990. 遗传多样性及其保存. 生物科学信息(4): 158–164. |
| [] | 王备新, 杨莲芳. 2002. 线粒体DNA序列特点与昆虫系统学研究. 昆虫知识, 39(2): 88–92. DOI:10.7679/j.issn.2095-1353.2002.026 |
| [] | 吴东霞. 2007. 线蛱蝶亚科部分种类线粒体Cytb及COⅠ基因分子系统学研究. 安徽师范大学博士学位论文. |
| [] | 杨宝山, 侯庆君, 王欢, 等. 2009. 不同地里种群银杏大蚕蛾COⅠ基因序列变异与遗传分化. 昆虫学报, 52(4): 406–412. |
| [] | 袁一杨, 高宝嘉, 李明, 等. 2008. 不同林分类型下油松毛虫(Dendrolinus tabulaeformis Tsai et Liu)种群遗传多样性. 生态学报, 28(5): 2099–2106. |
| [] | 张学卫, 高宝嘉, 周国娜. 2011. 基于Cytb基因序列分析的松毛虫种群遗传结构研究. 生态学报, 31(6): 1–8. |
| [] | 张学卫, 吴丽欣, 高宝嘉. 2010. 松毛虫mtDNA PCR反应体系优化. 河北农业大学学报, 33(4): 77–80. |
| [] | 周继亮, 张亚平, 黄美华, 等. 2001. 蝮亚科蛇线粒体细胞色素b基因序列分析及其系统发育. 动物学报, 47(4): 361–366. |
| [] | Knight A, Mindell D P. 1993. Substitutions bias, weighting of DNA sequence evolution, and the phylogenetic positions of lea's viper. Systematic Biology, 42(1): 18–31. DOI:10.1093/sysbio/42.1.18 |
| [] | Millar C I, Libby W J.1991.Strategies for conserving clinal, ecotypic, and disjunct population diversity in widespread species//Falk D A, Holsinger K E.Genetics and Conservation of Rare Plants.New York: Oxford University Press, 149-170. https://www.sciencedirect.com/science/article/pii/S0006320796001553 |
| [] | Simon C, Frait F, Bechenback A, et al. 1994. Evolution weighting and phylogenetic utility of mitochondrial gene sequence and a compilation of conserved polymerase chain reaction primers. Ann Entamol Soc Am, 87: 651–701. DOI:10.1093/aesa/87.6.651 |
| [] | Wright S. 1978. Variability within and among natural population. Evolution and the Genetics of Populations Vol.4, University of Chicago Press, Chicago, IL: 79–103. |
 2012, Vol. 48
2012, Vol. 48