文章信息
- 张玉梅, 徐刚标, 申响保, 吴雪琴, 覃建庸, 陈娟
- Zhang Yumei, Xu Gangbiao, Shen Xiangbao, Wu Xueqin, Qin Jianyong, Chen Juan
- 珙桐天然种群遗传多样性的ISSR标记分析
- Genetic Diversity of Davidia involucrata Populations Detected by Using ISSR Markers
- 林业科学, 2012, 48(8): 62-67.
- Scientia Silvae Sinicae, 2012, 48(8): 62-67.
-
文章历史
- 收稿日期:2011-12-23
- 修回日期:2012-02-19
-
作者相关文章
2. 西南民族大学 成都 610041
2. Southwest University for Nationalities Chengdu 610041
珙桐(Davidia involucrata)系我国特有的单型属植物,是第三纪古热带植物区系孑遗种,被列为国家一级保护植物,为世界著名的观赏木本植物,在植物系统发育和地史变迁上具有很高的研究价值。珙桐天然种群在102°52′—111°20′E,26°46′—32°20′N范围内呈马蹄形分布,间断性星散和小块状分布于边远山区人迹罕至之处(贺金生等,1995;张清华等,2000),其中,以四川中部、西部和湘鄂西分布最为集中。以往有关珙桐研究主要集中在生理特性、生态特征及繁殖方面(禹玉婷等,2006)。虽然已开展了利用等位酶(李建强等,2000;彭玉兰等,2003)和RAPD标记(宋丛文等,2004;(2005;李雪萍等,2006;徐刚标等,2007)分析种群遗传多样性,但采集的珙桐种群数量较少或种群样本容量过小,代表性不强,研究结果不足以反映整个物种水平的遗传变异情况(Poes et al., 1995)。
基于PCR技术的ISSR(inter simple sequence repeat)标记已成功地应用于种群遗传结构分析,与RAPD标记相比,ISSR比RAPD重复性好(Arcade et al., 2000)。本研究采用ISSR标记对11个代表性珙桐天然种群进行遗传多样性分析,旨在探明珙桐天然种群的遗传变异状况,为珙桐遗传资源保护策略制定提供种群遗传学基本信息。
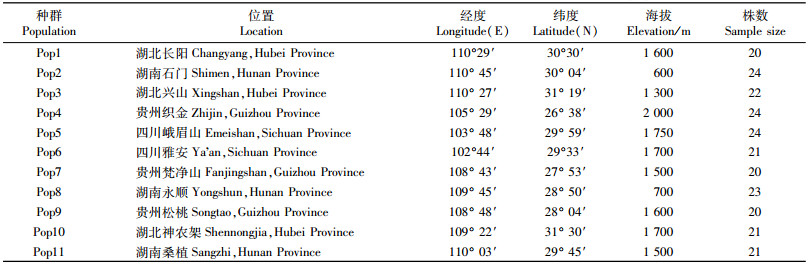
1 材料与方法 1.1 材料收集于2011年5—7月,采集湖南、湖北、四川、贵州4省11个代表性天然珙桐种群(表 1)。每个种群采集20株以上的个体,单株之间距离50 m以上。野外采集的珙桐新鲜嫩叶立即放入装有硅胶的密封袋中,记录各种群的地理位置及个体数。带回实验室后,将叶片放入-70 ℃的冰箱中保存备用,并制作种群凭证标本。标本保存于中南林业科技大学林木遗传育种实验室。
|
|
采用改良的CTAB法(徐刚标等,2007)提取珙桐基因组DNA,并进行纯化,用0.8%琼脂糖凝胶电泳和Eppendorf Biophotometer核酸蛋白分析仪检测DNA的纯度和浓度,将DNA统一稀释为10 ng·μL-1,于-20 ℃下保存备用。
1.2.2 ISSR反应体系建立与优化通过单因素和正交试验确定ISSR优化体系为:25 μL反应体系,Mg2+ 1.8 mmol·L-1,DNA 60 ng,dNTP 0.40 mmol·L-1,引物0.52 μmol·L-1,Taq酶1.25 U,反应程序为94 ℃预变性4 min;94 ℃变性45 s,48~58 ℃退火45 s,72 ℃延伸90 s,36个循环;72 ℃总延伸7 min;4 ℃保存。
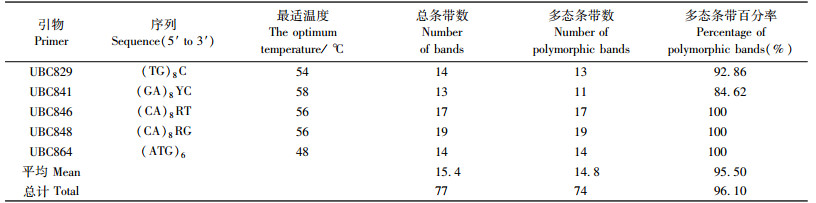
1.2.3 ISSR引物筛选ISSR引物参照加拿大哥伦比亚大学(UBC)2006年公布的序列,由上海英俊生物技术有限公司合成。利用优化的反应体系对100条引物进行筛选,从中筛选出5条引物,其扩增产物电泳条带清晰、稳定且条带多态性高,用Veriti 96 well Thermal Cycler PCR扩增仪对全部样品进行扩增。
1.2.4 电泳及检测用1.6%琼脂糖凝胶电泳检测扩增产物,电泳缓冲液为1×TAE,电压为5 V·cm-1,以200 bp DNA marker作为对照。电泳结束后用G-BOX紫外凝胶成像系统观察,拍照记录。
1.3 数据分析相同迁移位置上条带有无分别计为“1”和“0”,构成“0/1”型矩阵。用POPGENE version 1.31软件(Yeh et al., 1999)估算:1)物种与种群多态条带百分率(PPB);2)种内(HSP)和种群内(HPOP) Shannon多样性指数H(H=-Σpilog2pi,pi为第i条带的频率,不需要假定群体处于Hardy-Weiberg平衡状态)(Fontaine et al., 2004)。
利用ARLEQUIN3.1(http://cmpg.unibe.ch/software/arlequin3)软件包中的分子方差分析(AMOVA)软件计算种群内、种群间变异组分以及ΦST遗传距离。采用NTSYS-pc软件对种群间ΦST遗传距离进行UPGMA(unweighted pair-group method with arithmetic-means)聚类分析,绘制种群间遗传关系图。利用电子地图计算珙桐11个天然种群各采样点的地理距离,采用R软件对种群间ΦST距离与地理距离进行相关性分析。
2 结果与分析 2.1 ISSR-PCR扩增片断多态性从100条ISSR引物中共筛选出5条引物可扩增清晰、可重复且具多态性的条带。用这些引物对11个珙桐天然种群240个体进行PCR扩增,扩增片段大小在100~2 200 bp之间。除引物UBC829和UBC841分别扩增出1条和2条共有条带外,其余引物所扩增的条带均为多态,共扩增出77个条带,其中74个为多态条带,多态条带百分率为96.10%。每条引物扩增出的条带数为13~19条,不同引物多态条带百分率在84.62%~100.00%之间,平均每条引物多态位点百分率为95.50%(表 2)。
|
|
由表 3可知,在物种水平上,珙桐多态条带百分率和Shannon指数分别为96.10%,0.484 9;在种群水平上,各种群多态条带百分率和Shannon指数分别为37.66%~63.44%,0.188 6~0.327 4,其平均值分别为54.07%,0.277 4。其中,四川峨眉山种群遗传多样性最高(PPB=63.64%,HPOP=0.327 4),湖南永顺种群遗传多样性最低(PPB=37.66%,HPOP=0.188 6)。种群间遗传多样性所占比例为:(HSP-HPOP)/HSP=0.427 9。
|
|
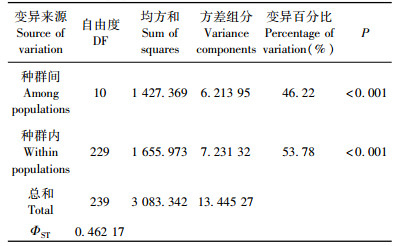
AMOVA分析结果(表 4)表明,珙桐总的遗传变异中46.22%存在于种群间,53.78%存在于种群内,种群遗传分化显著(ΦST=0.462 2,P < 0.001)。
|
|
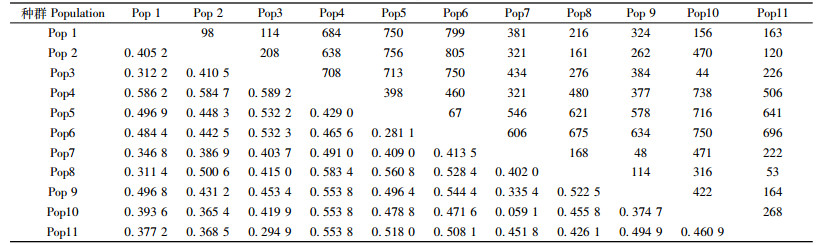
利用AMOVA计算的成对种群间ΦST遗传距离为0.059 1~0.589 2(表 5)。其中,湖北兴山种群与贵州织金种群间遗传距离最大(ΦST=0.589 2),贵州梵净山种群与湖北神农架种群间遗传距离最小(ΦST=0.059 1)。种群间ΦST遗传距离与地理距离呈显著正相关(r=0.546,DF=9,P<0.01)。
|
|
UPGMA聚类分析结果(图2)表明,11个珙桐天然种群可分为3组:湖南桑植、湖北兴山、湖北长阳和湖南永顺种群为一组;湖南石门、贵州梵净山、湖北神农架和贵州松桃种群为一组;四川峨眉山、四川雅安和贵州织金种群为一组。
3 结论与讨论 3.1 珙桐遗传多样性ISSR标记揭示珙桐物种和种群水平多态条带百分比分别为96.10%和54.78%,物种和种群水平Shannon多样性指数分别为0.484 9,0.277 4,与宋丛文等(2004;2005)采用RAPD标记的研究结果基本一致,其结果揭示珙桐物种水平多态条带百分比为97.03%,5个种群多态条带百分比为64.36%~85.15%。这表明,珙桐物种和种群都维持较高的遗传多样性,种群进化过程中没有经历瓶颈效应(Williamson et al., 1999)。
一般认为,物种遗传多样性高低与其地理分布区大小呈显著正相关(Hamrick et al., 1990)。我国特有的濒危植物,如银杉(Cathaya argyrophylla)(Ge et al., 1998)、桫椤(Alsophila spinulosa)(Wang et al., 2004)、苏铁(Cycas guizhouensis)(Xiao et al., 2004)、沙冬青(Ammopiptanthus mongolicus)和新疆沙冬青(Ammopiptanthus nanus)(Ge et al., 2003)、永瓣藤(Monimopetalum chinense)(Xie et al., 2005)及穗花杉(Amentotaxus argotaenia)(Xie et al., 2005)遗传多样性都较低。珙桐系我国特有的珍稀濒危植物,属于拥有较高水平遗传变异的特有种(Smith et al., 1996)。近年来,分子标记揭示了一些珍稀特有植物种维持较高水平的遗传多样性(Ge et al., 1997;Ayres et al., 1999;Kang et al., 2000;Helenurm, 2001;Zawko et al., 2001;Xue et al., 2004;Crema et al., 2009;Jordan-Pla et al., 2009;Sozen et al., 2010)。几种可能的机制被提出:特有种是由许多广域种杂交形成、分布区(或生境)近期发生变化、交配系统、体细胞突变、多次奠基者效应或冰期避难所(Zawko et al., 2001;Xue et al., 2004)。珙桐为第三纪古热带植物区系孑遗种,在受第四纪冰川影响较弱的我国西南地形复杂山地得以残存(张清华等,2000;吴刚等,2000),近年来人类活动加剧,珙桐天然分布面积和种群数量急剧下降并濒于灭绝(吴刚等,2000)。在野外采集样本中发现,珙桐种群遗传多样性高低与现实种群中个体数量多少不相关。据此,初步认为,珙桐现存种群是其冰期避难所,这与前人的研究推测(李建强等,2000;张清华等,2000;彭玉兰等,2003)基本吻合。近期人类活动频繁,生境片断化加剧,天然分布面积和种群数量急剧减少且不连续分布,也可能是其物种与种群水平维持较高遗传变异的原因之一。
珙桐种群内多态条带百分比(37.66%~63.44%)和Shannon多样性指数(0.188 6~0.327 4)变异较大,各种群遗传多样性差异可能与其所处的生境有一定的关联(王凤英等,2005)。
3.2 珙桐种群遗传结构与宋丛文等(2004;2005)采用RAPD标记研究珙桐5个天然种群遗传多样性结果相比,本研究结果中种群遗传分化系数(0.462 2)要大于他们的研究结果(0.125 6),也高于李建强等(2000)采用等位酶标记对6个天然种群遗传多样性研究的结果(0.164 0)。造成结果差异的原因,可能与抽取的种群样本、分子标记不同有关。
对植物种群遗传结构与生活史之间关系研究表明,植物生活史周期、地理分布范围、繁育系统、演替阶段等决定着种群遗传变异大小。寿命长、广域种或地区种、异交或混合交配、重力种子、存在于演替末期群落中的植物种群遗传分化大(Nybom et al., 2000;Nybom, 2004)。与采用RAPD或ISSR标记揭示的植物种群遗传分化系数统计平均数(Nybom, 2004)相比,珙桐种群遗传分化系数(ΦST=0.462 2)接近于借助种子重力扩散的植物(ΦST=0.45),这符合珙桐种子较大、主要靠重力传播(苏瑞军等, 2005)的生物学特性。
繁育系统被认为是引起植物种群遗传分化的主要因素之一(Hamrick et al., 1991;Nybom et al., 2000;Nybom, 2004)。珙桐为雄全同株(andromonoecy)植物(操国兴等,2007),目前还没有充分的繁育系统信息。彭玉兰等(2003)认为珙桐为异交植物,宋丛文等(2005)认为是自交植物,而彭红丽等(2009)认为其为异交,部分自交亲和。Nybom(2004)对植物种群遗传结构作综合分析,混合交配、自交和异交植物的ΦST值分别为0.40,0.65和0.27。从珙桐种群分化系数来看,可能为混合交配植物。但珙桐可根萌无性繁殖并常以数株幼苗围绕母树生长(苏瑞军等, 2005),这可能会影响用种群遗传分化系数对其繁育系统的判断。
珙桐种群遗传分化高于长寿命林木(ΦST=0.25)及特有种(ΦST=0.26),与地区种(ΦST=0.42)相接近(Nybom, 2004),这可能与珙桐种群分布特性有关。珙桐片断化分布,种群分布海拔相差很大(表 1),地形复杂,生境多样化,种群间为高山阻隔,势必促进种群遗传分化(Weller et al., 1996)。
珙桐种群间遗传距离与相应的地理距离呈显著正相关,表明邻近种群可能存在较高的遗传相似性(高丽等,2006)。这种变异模式与UPGMA树大体吻合(图 1)。4个地理距离较近的湖南桑植、湖北兴山、湖北长阳和湖南永顺种群聚为一组;四川峨眉山和四川雅安种群聚为一亚组。对所有样本的基因型数据聚类分析(图未显示)发现,一些个体未能聚合在相应的种群中。在咖啡树(Coffea arabica)(Aga et al., 2005)和春兰(Cymbidium goeringii)(高丽等,2006)种群研究中也发现同类情况。值得一提的是,贵州梵净山与湖北神农架种群遗传相似性高度一致(ΦST=0.059 1),宋丛文等(2004;2005)研究也出现同样的结果,但这2个种群间相距471 km之遥。其原因有待进一步研究。

|
图 1 11个天然珙桐种群的ΦST遗传距离UPGMA聚类图 Fig.1 UPGMA dendrogram based on ΦST genetic distance of 11 natural populations of D. involucrata |
遗传多样性是物种适应外界环境变化的进化潜力基础,遗传多样性丢失将大大降低个体的适应度和物种应对环境变化的适应能力(Frankham et al., 2002)。珙桐物种和种群都维持较高水平遗传多样性,种群间产生较大遗传分化,表明珙桐物种具有较大的进化潜力(Frankham et al., 2002)。Williamson等(1999)认为遗传多样性水平特别高的濒危物种,种群内个体间交配不可能导致近亲衰退(inbreeding depression),其濒危机制是原生境片断化、地理隔离及人类干扰加强的结果。人类活动所导致的森林片断化、生境恶化甚至丧失,可能使原来高水平遗传多样性的大种群隔离为若干个范围狭小的小种群;小种群会导致遗传漂移,可能是珙桐濒危的主要原因之一。这也是其他许多濒危物种的致濒因素(Eriksson et al., 1995;陈俊秋等,2006)。这符合前人研究的结论(吴刚等,2000;张清华等,2000)。据此,建议尽可能地保护珙桐所有的天然种群原生境条件,禁止滥砍滥挖,以保护尽可能多的遗传变异。其中,优先保存四川峨眉山、湖南桑植和贵州梵净山等遗传多样性丰富度高的种群。由于珙桐天然更新困难,应积极开展珙桐人工促进天然更新措施。
珙桐为世界著名的观赏木本植物,种苗具有很高的商业价值,但其对生境要求严格,自行繁殖困难很大(吴刚等,2000),引种人工林中开花结实植株不多(陈迎辉等,2010)。因此,应加强珙桐引种驯化和人工繁殖技术研究。这不仅能满足园林绿化对珙桐苗的需求,缓解由于人为采挖幼苗产生对天然种群生存的威胁,而且可以确保珙桐异地引种人工林正常开花结实,繁衍后代。
| [] | 操国兴, 严娟, 罗畅. 2007. 珍稀濒危植物珙桐繁育系统的初步研究. 西南大学学报:自然科学版, 29(4): 122–126. |
| [] | 陈俊秋, 慈秀芹, 李巧明, 等. 2006. 樟科濒危植物思茅木姜子遗传多样性的ISSR分析. 生物多样性, 14(5): 410–420. |
| [] | 陈迎辉, 彭春良, 李迪友, 等. 2010. 珙桐的生物生态特性和人工引种促进开花结实. 中南林业科技大学学报:自然科学版, 30(5): 64–67. |
| [] | 高丽, 杨波. 2006. 湖北野生春兰资源遗传多样性的ISSR分析. 生物多样性, 14(3): 250–257. |
| [] | 贺金生, 林洁, 陈伟烈. 1995. 我国珍稀特有植物珙桐的现状及其保护. 生物多样性, 3(4): 213–221. |
| [] | 李建强, 张敏华, 黄宏文, 等. 2000. 珙桐的等位酶位点变异分析. 武汉植物学研究, 18(3): 247–249. |
| [] | 李雪萍, 何正权, 陈发菊, 等. 2006. 神农架4个珙桐居群遗传多样性的RAPD分析. 北京林业大学学报, 28(3): 66–70. |
| [] | 彭红丽, 苏智先, 王颖, 等. 2009. 珙桐开花动态及繁育系统的研究. 安徽农业科学, 37(18): 8445–8448. DOI:10.3969/j.issn.0517-6611.2009.18.059 |
| [] | 彭玉兰, 胡远乾, 孙航. 2003. 光叶珙桐的等位酶分析及生物地理学意义. 云南植物研究, 25(1): 55–62. |
| [] | 宋丛文, 包满珠. 2005. 珙桐种质资源保存样本策略的研究. 植物生态学报, 29(3): 422–428. DOI:10.17521/cjpe.2005.0056 |
| [] | 宋丛文, 包满珠. 2004. 天然珙桐群体的RAPD标记遗传多样性研究. 林业科学, 40(4): 75–79. DOI:10.11707/j.1001-7488.20040413 |
| [] | 苏瑞军, 苏智先. 2005. 珙桐种子散布、萌发及其种群龄级分配的关系研究. 林业科学, 41(3): 193–195. |
| [] | 王凤英, 葛学军, 郝刚, 等. 2005. 喜马拉雅-横断山区钟花报春居群遗传多样性及遗传分化. 热带亚热带植物学报, 13(2): 149–153. |
| [] | 吴刚, 肖寒, 李静, 等. 2000. 珍稀濒危植物珙桐的生存与人为活动的关系. 应用生态学报, 11(8): 493–496. |
| [] | 徐刚标, 禹玉婷, 申响保. 2007. 湘鄂西地区珙桐天然群体遗传结构的研究. 中南林业科技大学学报, 27(6): 5–8. |
| [] | 禹玉婷, 徐刚标, 汪晓萍. 2006. 珙桐研究进展. 经济林研究, 24(4): 92–94. |
| [] | 张清华, 郭泉水, 徐德应. 2000. 气候变化对我国珍稀濒危树种——珙桐地理分布的影响研究. 林业科学, 36(2): 47–52. DOI:10.11707/j.1001-7488.20000208 |
| [] | Aga E, Bekele E, Bryngelsson T. 2005. Inter-simlpe sequence repeat(ISSR) variation in forest coffee trees(Coffea arabica L.) populations from Ethiopia. Gent, 124: 213–221. |
| [] | Arcade A F, Anselin P, Faivre Rampant, et al. 2000. Application of AFLP, RAPD and ISSR markers to genetic mapping of Euro-pean and Japanese larch. Theor Appl Genet, 100(2): 299–307. DOI:10.1007/s001220050039 |
| [] | Ayres D R, Ryan F J. 1999. Genetic diversity and structure of the narrow endemic Wyethia reticulata and its congener W.bolanderi (Asteraceae) using RAPD and allozyme techniques. Am J Bot, 86(3): 344–353. DOI:10.2307/2656756 |
| [] | Crema A, Cristofolini G, Rossi M, et al. 2009. High genetic diversity detected in the endemic Primula apennina Widmer(Primulaceae) using ISSR fingerprinting. Plant Syst Evol, 280(1): 29–36. |
| [] | Eriksson G, Namkoong G, Robert J. 1995. Dynamic conservation of forest tree gene resources. Forest Genetic Resources, 23(1): 2–5. |
| [] | Fontaine C, Lovett P N, Sanou H, et al. 2004. Genetic diversity of the shea tree(Bitellaria paradoxa C. F. Gaertn), detected by RAPD and chloroplast microsatellite marker. Heredity, 93: 639–648. DOI:10.1038/sj.hdy.6800591 |
| [] | Frankham R, Ballou J D, Briscoe D A. 2002. Introduction to conservation genetics. Cambridge: Cambridge University Press, 2-9, 96-112. http://www.journals.uchicago.edu/doi/abs/10.1086/658446?journalCode=qrb |
| [] | Ge Song, Hong Deyuan, Wang Haiqun, et al. 1998. Population genetic structure and conservation of an endangered conifer, Cathaya argyrophylla (Pinaceae). Int J Plant Sci, 159(2): 351–357. DOI:10.1086/297556 |
| [] | Ge Song, Zhang Daming, Wang Haiqun, et al. 1997. Allozyme variation in Ophiopogon xylorrhizus, an extreme endemic species of Yunnan, China. Conserv Biol, 11(2): 562–565. DOI:10.1046/j.1523-1739.1997.96011.x |
| [] | Ge Xuejun, Yu Yan, Zhao Nanxian, et al. 2003. Genetic variation in the endangered inner Mongolia endemic shrub Tetraena mongolica Maxim(Zygophyllaceae). Biol Conserv, 111(3): 427–434. DOI:10.1016/S0006-3207(02)00312-9 |
| [] | Hamrick J L, Godt M J. 1990. Allozyme diversity in plant species//Brown A H D, Clegg M T, Kahler A L, et al. Plant Population Genetics, Breeding, and Genetic Resources. Sunderland, Mass: Sinauer, 43-63. |
| [] | Hamrick J L, Godt M J W, Mur awski D A, et al. 1991. Correlation between species traits and allozyme diversity: implication for conservation biology//Falk D A, Holsinger K E. Genetics and Conservation of Rare Plants. Oxford: Oxford University Press, 245-253. |
| [] | Helenurm K. 2001. High levels of genetic polymorphism in the insular endemic herb Jepsonia malvifolia. J Hered, 92(5): 427–432. DOI:10.1093/jhered/92.5.427 |
| [] | Jordán-Pla A, Estrelles E, Boscaiu M, et al. 2009. Genetic variability in the endemic Leucojum valentinum. Biologia Plantarum, 53(2): 317–319. DOI:10.1007/s10535-009-0057-4 |
| [] | Kang U, Chang C S, KimY S. 2000. Genetic structure and conservation considerations of rare endemic Abeliophyllum distichum Nakai(Oleaceae) in Korea. J Plant Res, 113(2): 127–138. DOI:10.1007/PL00013923 |
| [] | Nybom H, Bartish I V. 2000. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants. Perspect Plant Ecol Evol Syst, 3(2): 93–114. DOI:10.1078/1433-8319-00006 |
| [] | Nybom H. 2004. Comparison of different nuclear DNA markers for estimation intraspecific genetic diversity in plants. Mol Ecol, 13(5): 1143–1156. DOI:10.1111/mec.2004.13.issue-5 |
| [] | Poes S, Chaouche K. 1995. Estimation, variance and optimal sampling of gene diversityⅡ: Diploid locus. Theoretical and Applied Genetics, 91(1): 122–130. |
| [] | Smith J F, Pham T V. 1996. Genetic diversity of the narrow endemic Allium aaseae (Alliaceae). Am J Bot, 83(6): 717–726. DOI:10.2307/2445848 |
| [] | Sozen E, Oxaydin B. 2010. A study of genetic variation in endemic plant Centaurea wiedemanniana by using RAPD markers. Ekoloji, 77: 1–8. |
| [] | Wang Ting, Su Yingjuan, Li Xueyan, et al. 2004. Genetic structure and variation in the relict populations of Alsophila spinulosa from southern China based on RAPD markers and cpDNA atpB-rbcL sequence data. Hereditas, 140(1): 8–17. DOI:10.1111/j.1601-5223.2004.01659.x |
| [] | Weller S G, Sakai A K, Straub C. 1996. Allozyme diversity and identity in Schiedea and Alsinidendron(Caryophyllaceae: Alsinoideae) in the Hawaiian Islands. Evolution, 50(1): 23–34. DOI:10.1111/evo.1996.50.issue-1 |
| [] | Williamson P S, Werth C R. 1999. Levels and pattern of genetic variation in the endangered species Abronia macrocarpa(Nyctaginaceae). Am J Bot, 86(2): 293–301. DOI:10.2307/2656946 |
| [] | Xiao Longqian, Ge Xuejun, Gong Xun, et al. 2004. ISSR variation in the endemic and endangered plant Cycas guizhouensis (Cycadaceae). Ann Bot, 94(1): 133–138. DOI:10.1093/aob/mch119 |
| [] | Xie Guowen, Wang Delian, Yuan Yongming, et al. 2005. Population genetic structure of Monimopetalum chinense(Celastraceae), an endangered endemic species of eastern China. Ann Bot, 95(5): 773–777. DOI:10.1093/aob/mci087 |
| [] | Xue Dawei, Ge Xuejun, Hao Gang, et al. 2004. High genetic diversity in a rare, narrowly endemic primrose species: Primula interjacens by ISSR analysis. Acta Botanica Sinica, 46(10): 1163–1169. |
| [] | Yeh F C, Yang R C, Boyle T. 1999. POPGENE version 1.31: Microsoft Window-based Freeware for Population Genetic Analysis. Edmonton: University of Alberta. http://www.scienceopen.com/document?vid=76d06904-138c-442e-8da7-3e9fbd47b3cd |
| [] | Zawko G, Krauss S L, Dixon K W, et al. 2001. Conservation genetics of the rare and endangered Leucopogon obtectus (Ericaceae). Mol Ecol, 10(10): 2389–2396. DOI:10.1046/j.0962-1083.2001.01378.x |
 2012, Vol. 48
2012, Vol. 48