文章信息
- 李金花, 俞宁
- Li Jinhua, Yu Ning
- 利用荧光SSR标记分析中国油橄榄品种遗传多样性
- Genetic Diversity of Olive Cultivars in China Based on Fluorescent SSR Markers
- 林业科学, 2012, 48(6): 47-55.
- Scientia Silvae Sinicae, 2012, 48(6): 47-55.
-
文章历史
- 收稿日期:2011-07-25
- 修回日期:2012-03-20
-
作者相关文章
2. 中国林业科学研究院林业研究所 北京 100091
2. Research Institute of Forestry, Chinese Academy of Forestry Beijing 100091
油橄榄(Olea europaea)是世界著名的优质木本油料树种之一,有4 000多年的栽培历史,现已有40多个国家引种栽植,目前全世界油橄榄种植总面积约为1 060万hm2,总株数超过了10亿株,其中98%集中于地中海沿岸国家(IOC,2011)。油橄榄在长期的引种驯化和栽培历史中形成了许多品种,全世界油橄榄栽培中所使用品种数量已超过2 000个(Bartolini et al., 1998;Bartolini,2008;Ipec et al., 2009)。由于油橄榄主要是异花授粉,导致其具有高度杂合性和DNA多态性,品种间存在多态性和变异性(Muzzalupo et al., 2010;Lopes et al., 2004;Cipriani et al., 2002)。丰富的遗传变异带来了系统分类学上的混乱,而品种的正确鉴别和多样性研究,对于油橄榄种质资源管理、利用、保护和橄榄油贸易都具有重要意义。
我国从20世纪60年代开始引种油橄榄,经过数十年努力,曾一度发展到了2 000万株,但远远不能满足国内橄榄油的消费需求。日益扩大的橄榄油市场供需缺口,以及科技发展和人们健康意识的增强,加快了我国油橄榄产业的迅速发展,但我国的油橄榄种质资源利用、品种选育和更新一直未引起重视,在大力推广油橄榄的种植过程中,普遍存在良种率低、品种混乱的现象(徐纬英,2001;邓明全等,2011)。我国自1964年引种种植油橄榄以来,大量收集和引进了国外油橄榄品种资源(近150余种),经过区域化试验选出了一批适生的品种,如‘佛奥’、‘莱星’、‘皮削利’、‘阿斯’等,并在长期引种驯化的基础上,开展了品种内单株选优、实生选优和杂交育种等选育工作(续九如,1983;贺善安等,1984)。例如,利用前苏联引入的‘柯列’、‘阿斯’和‘尼Ⅱ’品种的种子繁育实生树,对其扦插树进行优良单株选择,选出了‘城固’、‘中山’、‘九峰’、‘鄂植’等一系列品种,这些引种品种和选育品种资源,在今后新品种选育中有很大潜力,而对其进行准确、有效的遗传多样性和遗传变异分析以及品种鉴定,对于正确收集、保存和利用油橄榄种质资源以及品种选育至关重要,是我国油橄榄遗传改良利用的前提。
近十年来,分子标记技术如RAPD(Fabbri et al., 1995;Besnard et al., 2001)、AFLP(Sensi et al., 2003;Owen et al., 2005)和ISSR(Gemas et al., 2004;Martins-Lopes et al., 2007;Gomes et al., 2008)等已被广泛用于国外油橄榄遗传育种研究中。SSR(Simple Sequence Repeat)标记以其多态性高、重复性好、共显性、操作简单等优点,在国外油橄榄遗传变异研究和品种鉴定中得到了越来越广泛的应用(Belaj et al., 2007;Rallo et al., 2000;Sefc et al., 2000;Cipriani et al., 2002),并已经研发出了油橄榄不同SSR引物(Rallo et al., 2000;Cipriani et al., 2002;Carriero et al., 2002),且在油橄榄种质资源遗传多样性分析、品种鉴别和育种工作中的有效性已经得到了证实(Sarri et al., 2006;Sefc et al., 2000;Rallo et al., 2002;Carriero et al., 2002;Cipriani et al., 2002)。在我国,仅有邱源等(2008)对四川收集引种的23个油橄榄品种进行了RAPD分析,马万里等(2006)运用RAPD技术鉴定澳大利亚油橄榄品种,而利用SSR标记开展油橄榄品种遗传多样性、品种分类和鉴定等研究未见报道。
为了进一步探明目前我国甘肃、四川和云南油橄榄三大适生区引种和选育品种间的遗传关系,本研究利用油橄榄SSR标记引物对油橄榄适生区所收集的53个品种107份材料进行遗传多样性分析,结果为揭示油橄榄种质资源遗传多样性、有效保存和利用我国油橄榄种质资源提供理论依据,为进一步引种和选择育种材料以及种质资源的保存利用提供基础资料。
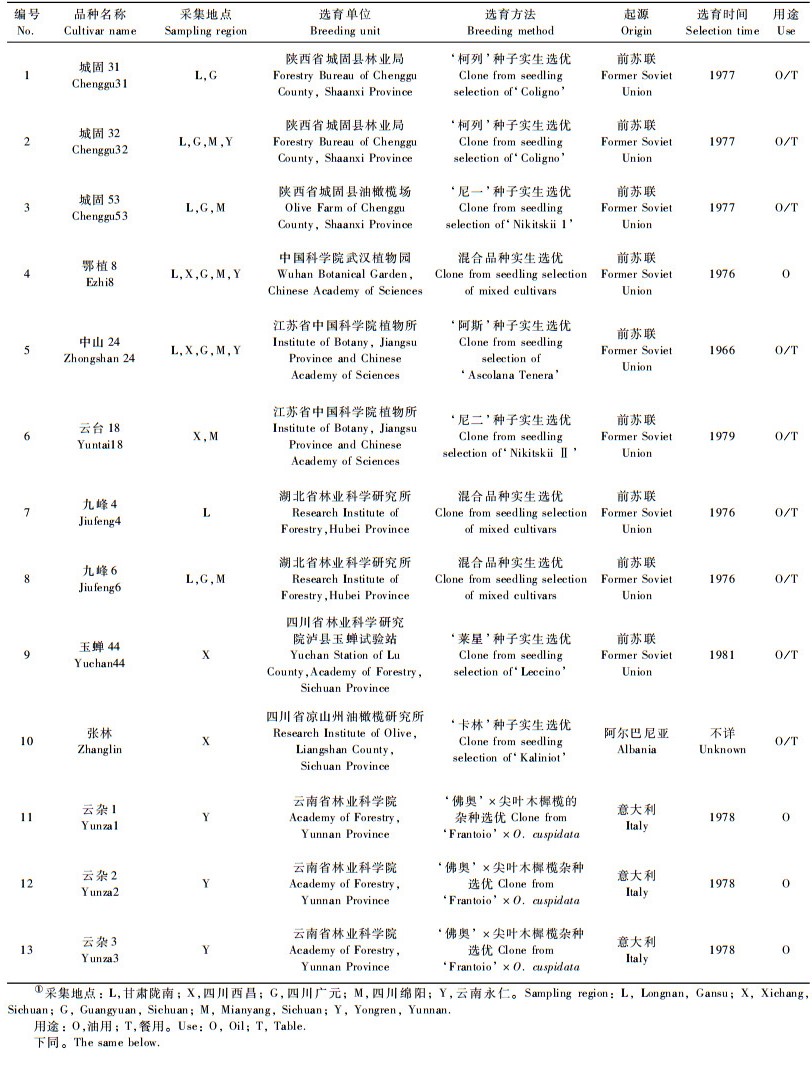
1 材料与方法 1.1 材料 1.1.1 试验材料供试材料采自甘肃、云南和四川油橄榄种质资源收集库和品种园,品种名称、采集地点及其起源如表 1和表 2。
|
|
|
|
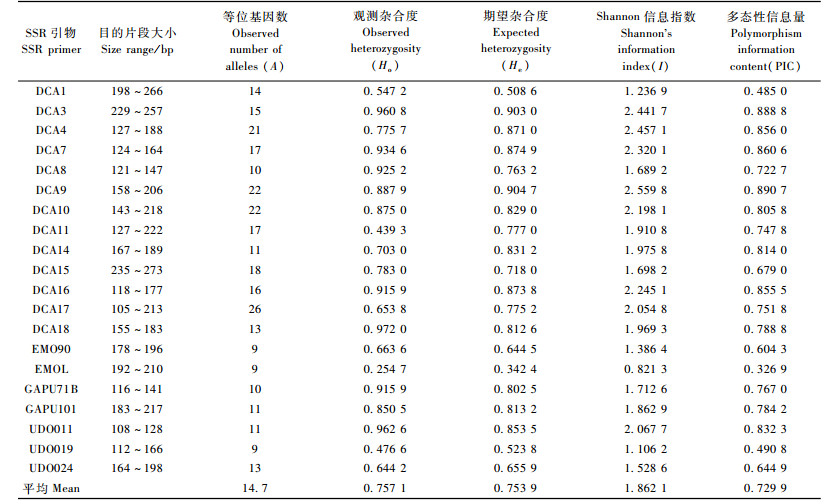
参照文献(Sefc et al., 2000;Cipriani et al., 2002;Carriero et al., 2002;De La Rosa et al., 2002)选取油橄榄SSR引物进行PCR扩增,筛选出多态性高的20对引物(表 3)用于本研究,并用荧光标记物FAM对正向引物进行标记。
|
|
春季采集1年生枝条上幼嫩叶片,利用CTAB法提取总DNA,在0.8%琼脂糖凝胶电泳上测定样品DNA的浓度,以标准λ-DNA为对照,根据测定结果,将所有样品DNA浓度稀释至25 ng·μL-1。
1.2.2 PCR扩增与产物荧光检测PCR反应体系为25 μL:2× Taq mix 12.5 μL,正向引物(10 pmol·L-1)1 μL,反向引物(10 pmol·L-1)1 μL,ddH2O 8.5 μL,DNA 2 μL(50 ng)。PCR反应程序为:95 ℃5 min,35个循环(95 ℃ 30 s,Tm 30 s,72 ℃ 1 min),72 ℃延伸10 min,4 ℃保温,不同引物的Tm值不同。取PCR产物1 μL,加入8.5 μL HI-DI甲酰胺和0.5 μL ROX-500荧光分子量内标混合均匀,95 ℃变性5 min,上样于ABI3730 DNA分析仪上进行毛细管电泳和自动荧光检测,并收集数据。
1.3 数据统计与分析利用GeneMarker V1.95软件分析收集数据,设置Bin和Panel菜单下相应参数,通过人工分析与软件统计得出不同引物对不同参试材料扩增得到的SSR标记片段,以Excel表格输出Allele(基因型分子量数值)和Bin(0,1二元型)数据,并根据统计分析软件的输入要求,利用CONVERT软件(Glaubitz,2004)对数据进行相应的转换。利用Popgene1.31软件(Yeh et al., 1999)计算等位基因数(A)、观测杂合度(Ho)、期望杂合度(He)和Shannon信息指数(I)。利用PowerMarker V3.25软件(Liu et al., 2005)计算多态性信息量(PIC)。利用Freetree 0.9.1.50软件(http://www.natur.cuni.cz/~flegr/programs/freetree.htm)计算Jaccard相似系数和邻接法(NJ)聚类分析,并经1 000次重抽样法对聚类树进行Bootstrap分析,利用TreeView 1.61软件(http://taxonomy.zoology.gla.ac.uk/rod/treeview.html)编辑和拷贝出树状图。对于采集有3份以上材料的品种,利用Arlequin3.5软件(Excoffier et al., 2010)进行分子方差分析(AMOVA)。
2 结果与分析 2.1 SSR标记的多态性53个油橄榄品种107份材料在20个SSR位点检测到的遗传变异汇总至表 3。共检测出294个等位变异,每对引物检测出9~22个等位变异,平均14.7个,其中DCA17位点的等位基因最多,有26个;EMO90,EMOL和UDO019三个位点的等位基因最少,均为9个。对于整个群体,群体的观测杂合度(Ho)平均为0.757 1(0.254 7~0.972 0),期望杂合度(He)平均为0.753 9(0.342 4~0.904 7)。20个位点的Shannon信息指数(I)平均为1.862 1(0.821 3~2.559 8),多态性信息量PIC值平均为0.729 9(0.326 9~0.890 7),其中Shannon信息指数在2以上、PIC值在0.8以上的引物位点有8个,分别是DCA3,DCA4,DCA7,DCA9,DCA10,DCA14,DCA16,UDO011。20个位点的扩增产物片段大约在105~273 bp之间。
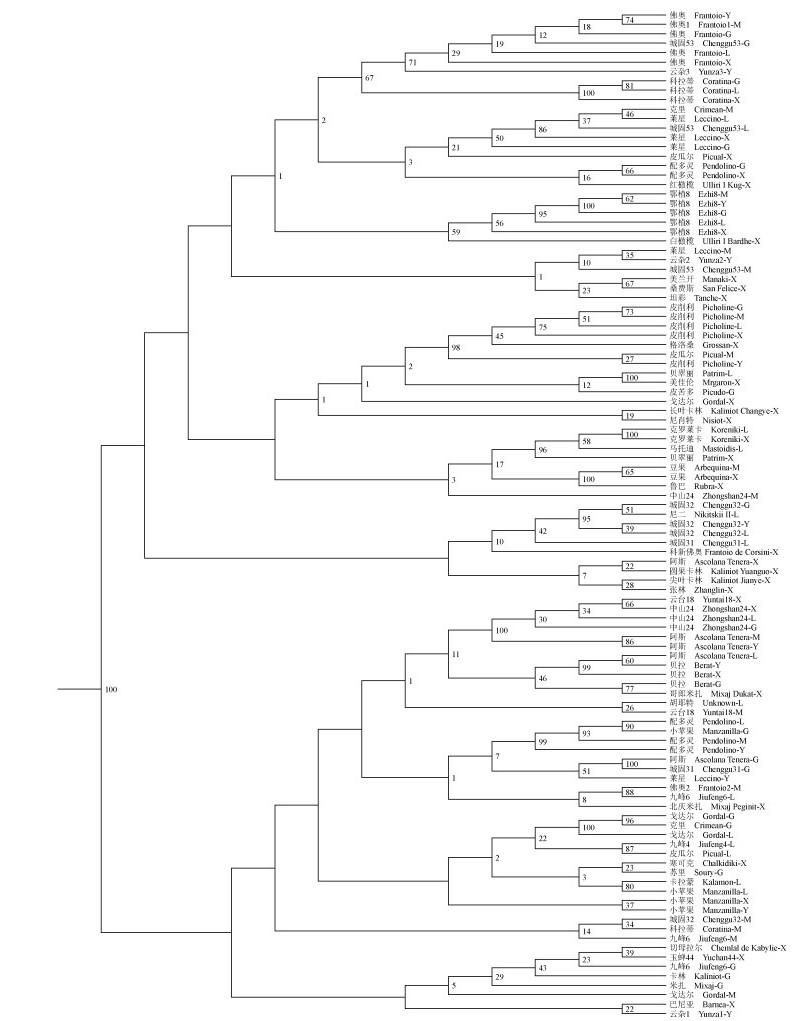
2.2 聚类分析根据53个品种107份参试材料的Jaccard相似系数,利用NJ法进行聚类分析,可将参试材料分为2大组(图 1),我国经引种驯化选育出的品种与从国外直接引种的品种材料交叉分布于不同组中。第1组共有62份材料,包括‘鄂植8’、‘城固32’等选育品种和‘佛奥’、‘科拉蒂’、‘皮削利’、‘克罗莱卡’、‘豆果’等引种品种;第2组共有45份材料,包括‘中山24’、‘九峰6’、‘云台’等选育品种和‘阿斯’、‘贝拉’、‘小苹果’等引种品种。起源于相同国家的品种材料也在不同组中穿插分布、相互渗透,大多数品种并不是根据其地理起源聚类在一组,这是由于选育品种是利用引种品种的种子繁殖产生的实生群体选择优良单株并经无性繁殖建立的品种(徐纬英,2001;邓明全等,2011),因此选育品种和引种品种之间存在较高的遗传相似性。
|
图 1 53个油橄榄品种107份材料基于Jaccard遗传距离基础上的邻近法聚类树状图 Fig.1 Dendrogram of 107 olive accessions obtained by NJ clustering algorithm based on Jaccard coefficients 图中L,X,G,M和Y代表品种采集地点,详见表 1。分枝点处的数字是1 000次抽样bootstrap检验的重复百分比。 In Tab. 1, L, X, G, M, and Y represent the sampling regions of cultivars detailed. Numbers at the nodes are bootstrap percentages out of 1 000. |
由聚类分析的结果可知:大多数情况下,同一个品种的参试材料被分在了同一个大组中,聚类分析图上明确地区分开了所有‘鄂植8’、‘中山24’、‘贝拉’、‘豆果’、‘小苹果’品种材料;但有一些品种材料分在不同大组中,例如,绵阳的‘科拉蒂’(M)被分在第2大组中,其他3个采集地点陇南、西昌和广元的‘科拉蒂’均被分在第1大组的同一个小亚组中,广元和西昌的‘配多灵’(G,X)与陇南、绵阳和云南的‘配多灵’(L,M,Y)分别被分在了第1大组和第2大组的同一个小亚组中。大多数引种驯化选育出的品种材料与其母本品种被分在了同一组中,例如,20世纪70年代江苏省中国科学院植物所利用‘阿斯’种子实生树选育出了‘中山24’,云南省林业科学院利用‘佛奥’与野生尖叶木樨榄(Olea cuspidata)杂交选育出了‘云杂3’,这些品种分别与其母本品种‘阿斯’、‘佛奥’之间存在很近的亲缘关系;但有少数亲缘关系较近的品种并没有聚在同一组中,如‘云杂1’、‘云杂2’和‘云杂3’均是‘佛奥’与尖叶木樨榄人工授粉杂交子代,‘云杂3’与母本品种‘佛奥’被分在第1大组的同一亚组中,而‘云杂1’被分在了第2大组,这些品种有一定的距离和不确定性,需要进一步的试验验证。
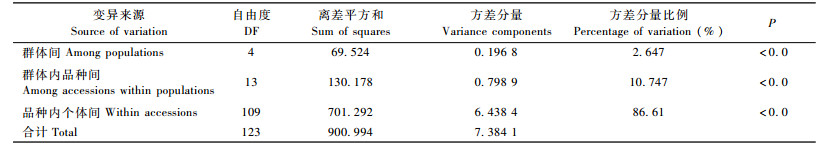
2.3 AMOVA分析采集有3份以上材料的品种共计15个,依照品种的地理起源国家分为前苏联(5个品种)、意大利(5个)、西班牙(3个)、法国(1个)和阿尔巴尼亚(1个)5个群体进行AMOVA分析,结果(表 4)表明,遗传分化系数FST为0.133 94,不同国家的品种群体间遗传变异为2.647 9%,不同国家群体内品种间遗传变异为10.747%,而品种内遗传变异(86.61%)远远超过群体间和群体内不同品种间的遗传变异(13.39%),遗传变异主要分布在品种内,以不同国家品种为群体的群体间遗传分化水平较低。
|
|
对来自我国油橄榄主要种植区三大省份53个油橄榄品种107份材料的SSR遗传多样性分析,结果表明其具有丰富的遗传多样性,证实了油橄榄是一个具有高度变异性树种,反映了品种间农艺性状的多样性,这可能是由于油橄榄种质起源分散,野生和栽培品种间杂交产生新品种(Erre et al., 2010;Bellini et al., 2008;Besnard et al., 2001)。而本文的国内选育品种和国外直接引种品种在聚类分析图上混杂在了一起,这是由于在长期引种过程中经历了品种大规模扩散交换、人工杂交和果实经济性状选择等,这与国外利用SSR标记对伊朗(Omrani-Sabbaghi et al., 2007)、葡萄牙(Gomes et al., 2009)、美国(Koehmstedt et al., 2011)等国油橄榄种质遗传多样性的研究结果和原因一致。另外,本文参试材料来源于5个采集地点,是各地在长期引种试验和选择基础上保存下来的种质,有些品种引种时为播种苗,有些为实生选种产生的品种(品系),有些为播种苗繁育产生的实生变异,是遗传组成差异较大的天然杂种,这些均有可能是造成油橄榄品种具有高水平遗传多样性的原因。
3.2 品种内变异和品种鉴别油橄榄是以异花授粉为主,新品种是在长期的自然演化和种间杂交过程中形成的,对其果实性状进行选择以满足经济需求,并通过无性方式繁育和推广,因而在长期栽培中利用实生变异产生了源于杂合性表型的多无性系(品种群),而不是单无性系品种。许多研究也已经发现和证实了油橄榄的品种内变异(Díaz et al., 2006;Cipriani et al., 2002;Lopes et al., 2004;Muzzalupo et al., 2010;Gomes et al., 2008),Muzzalupo等(2010)利用SSR标记对不同产地的油橄榄品种‘卡罗里’、‘科拉蒂’和‘佛奥’进行品种内变异性分析,认为SSR标记可用于油橄榄品种鉴别和品种内个体鉴别。Omrani-Sabbaghi等(2007)利用SSR标记对伊朗的18个油橄榄品种和30个国外油橄榄品种的87份材料进行遗传多样性分析,结果发现伊朗品种与地中海西部地区品种有不同起源,两者间遗传变异为37.24%,而品种内遗传变异达62.76%,认为可能的原因是长期无性繁育过程中品种异名或混淆。本文中油橄榄品种内个体间的变异非常大(86.61%),远远超过了不同国家群体间和不同国家群体内的品种间的遗传变异。品种内变异可以明确分为2种:一种是SSR位点差异较大的品种,如陇南的‘阿斯’(L)、广元的‘小苹果’(G)、绵阳的‘科拉蒂’(M)和‘中山24’(M)等,绵阳的‘佛奥2’(M),与其他采集点同名品种之间的位点差异较大,聚类在不同大组中,因而可能是引种来源或品种鉴别有误;另一种是SSR位点差异较小的品种,如‘鄂植’(L,X,Y,G,M)、‘中山24’(L,G,X)等,取自不同采集点,聚类在同一大组的同一小组中。此外,还有一些同名的品种被聚类在了2个不同大组中,如广元和西昌的‘配多灵’(G,X)与陇南、绵阳和云南的‘配多灵’(L,M,Y),这是因为西昌的‘配多灵’苗木来自广元,绵阳和云南的‘配多灵’苗木来自陇南,在苗木繁育中出现了品种混淆。这表明在油橄榄大规模引种过程中对品种的起源和来源地等并不完全清楚,相关的信息资料也不准确,以及长期引种栽培和苗木繁育过程中,依靠表型性状的品种鉴别难免出现不同品种之间的混淆、同种异名和同名异种现象,都是造成品种内遗传变异水平高的原因,有必要利用分子标记对这些品种准确鉴别,为这些种质资源的有效利用奠定基础。
3.3 中国油橄榄品种的起源油橄榄在漫长的栽培历史中,大规模的引种使得不同国家油橄榄种质间发生频繁的基因交流,以果实等经济性状而不是地理起源作为品种的选择标准,造成不同国家群体间和不同国家群体内的品种间遗传变异比较小,而品种内的遗传分化水平比较高。我国驯化选育的品种是在我国自然气候条件下引种驯化基础上产生的,各地在油橄榄长期引种试验过程中,利用引种品种开展了品种内单株选优、种子播种实生树选优以及与野生尖叶木樨榄的种间杂交育种工作,这直接说明选育品种与引种品种具有相同的地理起源,聚类分析结果也表明选育品种与引种品种混杂,二者遗传背景一致。
致谢:
本研究在材料收集过程中得到了云南省林业科学院、四川广元市油橄榄研究所、四川凉山州中泽技术开发有限公司、甘肃陇南田园油橄榄科技开发有限公司、四川华欧油橄榄开发公司等单位的协助。
| [] | 邓明全, 俞宁. 2011. 油橄榄引种栽培技术. 北京, 中国农业出版社. |
| [] | 贺善安, 顾姻. 1984. 油橄榄驯化引种. 南京, 江苏科学技术出版社. |
| [] | 马万里, CollinsG. 2006. 运用RAPD技术鉴定澳大利亚油橄榄品种的研究. 内蒙古师范大学学报:自然科学版, 35(3): 337–339. |
| [] | 邱源, 韩华柏, 李俊强, 等. 2008. 23个油橄榄品种的RAPD分析. 林业科学, 44(1): 85–89. DOI:10.11707/j.1001-7488.20080115 |
| [] | 徐纬英. 2001. 中国油橄榄种质资源与利用. 长春, 长春出版社. |
| [] | 续九如. 1983. 汉中地区油橄榄选种和建园的研究. 北京林学院学报, 4(4): 72–86. |
| [] | Bartolini G. 2008. Olive germplasm (Olea europaea L.). http://www.oleadb.eu. |
| [] | Bartolini G, Prevost G, Messeri C, et al. 1998. Olive germplasm: cultivars and world-wide collections. Rome, Italy: FAO. |
| [] | Belaj A, Muñoz-Diez C, Baldoni L, et al. 2007. Genetic diversity and population structure of wild olives from the North-western Mediterranean assessed by SSR markers. Annals of Botany (Lond), 100(3): 449–458. DOI:10.1093/aob/mcm132 |
| [] | Bellini E, Giordani E, Rosati R. 2008. Genetic improvement of olive from clonal selection to cross-breeding programs. Advances in Horticultural Science, 22(2): 73–86. |
| [] | Besnard G, Baradat P, Berville A. 2001. Genetic relationship in the olive (Olea europaea L.) reflects multilocal selection of cultivars. Theoretical and Applied Genetics, 102(2/3): 251–258. |
| [] | Carriero F, Fontanazza G, Cellini F, et al. 2002. Identification of simple sequence repeats (SSRs) in olive (Olea europaea L.). Theoretical and Applied Genetics, 104(2/3): 301–307. |
| [] | Cipriani G, Marrazzo M T, Marconi R, et al. 2002. Microsatellite markers isolated in olive are suitable for individual fingerprinting and reveal polymorphism within ancient cultivars (Olea europaea L.). Theoretical and Applied Genetics, 104(2/3): 223–228. |
| [] | De La Rosa R, James C M, Tobutt K R. 2002. Isolation and characterization of polymorphic microsatellites in olive (Olea europaea L.) and their transferability to other genera in the Oleaceae. Molecular Ecology Notes, 2(3): 265–267. DOI:10.1046/j.1471-8286.2002.00217.x |
| [] | Díaz A, De La Rosa R, Martín A, et al. 2006. Development, characterization and inheritance of new microsatellites in olive (Olea europaea L.) and evaluation of their usefulness in cultivar identification and genetic relationship studies. Tree Genetics & Genomes, 2(3): 165–175. |
| [] | Erre P, Chessa I, Muñoz-Diez C, et al. 2010. Genetic diversity and relationships between wild and cultivated olives (Olea europaea L.) in Sardinia as assessed by SSR markers. Genetic Resources and Crop Evolution, 57(1): 41–54. DOI:10.1007/s10722-009-9449-8 |
| [] | Excoffier L, Lischer H E L. 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 10(3): 564–567. DOI:10.1111/men.2010.10.issue-3 |
| [] | Fabbri A, Hormaza J I, Polito V S. 1995. Random amplified polymorphic DNA analysis of olive (Olea europaea L.) cultivars. Journal American Society for Horticultural Science, 120(3): 538–542. |
| [] | Gemas V J V, Almadanim M C, Tenreiro R, et al. 2004. Genetic diversity in the olive tree (Olea europaea L. subsp. europaea) cultivated in Portugal revealed by RAPD and ISSR markers. Genetic Resources and Crop Evolution, 51(5): 501–511. DOI:10.1023/B:GRES.0000024152.16021.40 |
| [] | Glaubitz J C. 2004. CONVERT: A user-friendly program to reformat diploid genotypic data for commonly used population genetic software packages. Molecular Ecology Notes, 4(2): 309–310. DOI:10.1111/men.2004.4.issue-2 |
| [] | Gomes S, Martins-Lopes P, Lima-Brito J, et al. 2009. Evidence of clonal variation in olive 'Verdeal-Transmontana' cultivar using RAPD, ISSR and SSR markers. Journal of Horticultural Science & Biotechnology, 83(4): 395–400. |
| [] | International Olive Council (IOC). 2011. Olive oil production and consumption in the season 2006/07. http://www.internationaloliveoil.org/downloads/. |
| [] | Ipek A, Barut E, Gulen H, et al. 2009. SSR analysis demonstrates that olive production in the southern Marmara region in Turkey uses a single genotype. Genetic Molecular Research, 8(4): 1264–1272. DOI:10.4238/vol8-4gmr659 |
| [] | Koehmstedt A, Aradhya M K, Soleri D, et al. 2011. Molecular characterization of genetic diversity, structure, and differentiation in the olive (Olea europaea L.) germplasm collection of the United States Department of Agriculture. Genetic Resources and Crop Evolution, 58(4): 519–531. DOI:10.1007/s10722-010-9595-z |
| [] | Liu K J, Muse S V. 2005. PowerMarker: Integrated analysis environment for genetic marker data. Bioinformatics, 21(9): 2121–2129. |
| [] | Lopes M S, Mendonca D, Sefc K M, et al. 2004. Genetic evidence of intra-cultivar variability within Iberian olive cultivars. Hort Science, 39(7): 1562–1565. |
| [] | Martins-Lopes P, Lima-Brito J, Gomes S, et al. 2007. RAPD and ISSR molecular markers in Olea europaea L.: genetic variability and cultivar identification. Genetic Resources and Crop Evolution, 54(1): 117–128. DOI:10.1007/s10722-005-2640-7 |
| [] | Muzzalupo I, Chiappeta A, Benincasa C, et al. 2010. Intra-cultivar variability of three major olive cultivars grown in different areas of central-southern Italy and studied using microsatellite markers. Scientia Horticulturae (Amsterdam), 324(3): 324–329. |
| [] | Omrani-Sabbaghi A, Shahriari M, Falahati-Anbaran M, et al. 2007. Falahati-Anbaran M, et al. 2007. Microsatellite markers based assessment of genetic diversity in Iranian olive (Olea europaea L.) collections. Scientia Horticulturae (Amsterdam), 112(4): 439–447. DOI:10.1016/j.scienta.2006.12.051 |
| [] | Owen C A, Bita E C, Banilas G, et al. 2005. AFLP reveals structural details of genetic diversity within cultivated olive germplasm from the Eastern Mediterranean. Theoretical and Applied Genetics, 110(7): 1169–1176. DOI:10.1007/s00122-004-1861-z |
| [] | Rallo P, Dorado G, Martin A. 2000. Development of simple sequence repeats (SSRs) in olive tree (Olea europaea L.). Theoretical and Applied Genetics, 101(5/6): 984–989. |
| [] | Rallo P, Dorado G, Martin A. 2002. Application of microsatellite markers in olive breeding. Acta Horticluture (ISHS), 586(2): 69–72. |
| [] | Sarri V, Baldoni L, Porceddu A, et al. 2006. Microsatellite markers are powerful tools for discriminating among olive cultivars and assigning them to geographically defined populations. Genome, 49(12): 1606–1615. DOI:10.1139/g06-126 |
| [] | Sefc K M, Lopes M S, Mendonça D, et al. 2000. Identification of microsatellite loci in olive (Olea europaea) and their characterization in Italian and Iberian olive trees. Molecular Ecology, 9(8): 1171–1173. DOI:10.1046/j.1365-294x.2000.00954.x |
| [] | Sensi E, Vignani R, Scali M, et al. 2003. DNA fingerprinting and genetic relatedness among cultivated varieties of Olea europaea L. estimated by AFLP analysis. Sci Hortic (Amsterdam), 97(3/4): 379–388. |
| [] | Yeh F C, Yang R C, Boyle T. 1999. POPGENE (Version 1.31): Microsoft Window-based freeware for population genetic analysis. University of Alberta and the Centre for International Forestry Research. |
 2012, Vol. 48
2012, Vol. 48