文章信息
- 李静, 袁虎威, 李伟
- Li Jing, Yuan Huwei, Li Wei
- 牛枝子抗旱候选基因LpDREB的克隆与表达及单核苷酸多态性分析
- Isolation, Expression and Single Nucleotide Polymorphisms Analysis of Dehydration Responsive Element Binding Gene (LpDREB) in Lespedeza potaninii
- 林业科学, 2012, 48(6): 40-46.
- Scientia Silvae Sinicae, 2012, 48(6): 40-46.
-
文章历史
- 收稿日期:2011-10-08
- 修回日期:2011-10-28
-
作者相关文章
干旱是影响植物生长和发育的重要环境因子之一,它也是一个世界性的社会和经济问题。有关植物耐旱分子机制研究一直是国内外研究的热点领域。许多抗旱基因或抗旱调控因子已被成功克隆(Shinozaki et al., 2007),模式植物中的拟南芥(Arabidopsis thaliana)(Bouchabke et al., 2008)和水稻(Oryza sativa)(http://www.Stress-genomics.org)已经建立了比较完善的抗旱基因调控网络。但是,目前所有关于植物耐旱分子机制的研究都是基于基因水平上的,并没有考虑基因内部的单核苷酸多态性(single nucleotide polymorphism, SNP)所引起的植物个体间、群体间耐旱能力的差异,即同一基因在同一物种的不同个体或群体内存在单核苷酸水平上的差异,这种差异能够影响基因的功能和植物的性状。已有研究结果表明,SNP是决定不同群体或个体之间表型差异的分子基础(Ingvarsson, 2005;Aulchenko et al., 2007)。
牛枝子(Lespedeza potaninii)是胡枝子属(Lespedeza)中最具抗旱能力的一个种,它能在年降水量不足150 mm的干旱地区生长,广泛分布于荒漠、草原及草原化荒漠地带,是新疆、内蒙古、宁夏荒漠草原上的优势灌木种(李新荣, 1997)。与胡枝子属其他种一样,牛枝子还处于自然分布状态,由于天然隔离或生态环境的适应性,种源间、个体间的抗旱能力存在着丰富的变异(骈瑞琪等, 2005)。这种广泛存在的差异为研究基于SNP层次的木本植物个体、群体间的耐旱机制提供了极为重要的材料。
DREB(dehydration responsive element binding)是一类与逆境胁迫相关的转录因子,它能够识别并结合于与逆境胁迫应答有关的基因启动子区域中的DRE顺式作用元件上,启动下游抗逆基因的表达,从而在转录水平上调控多个抗逆基因,在诱导一系列抗非生物胁迫基因和提高植物抗逆性方面起着重要的作用(Agarwal et al., 2006)。目前已经在拟南芥(Haake et al., 2002)、大豆(Glycine max)(Li et al., 2005)、玉米(Zea mays)(Saleh et al., 2006)、水稻(Wang et al., 2008)等植物中克隆得到了DREB基因。已有研究结果表明,DREB基因在植物的抗旱过程中起重要作用,是重要的抗旱转录调控因子。因此,DREB基因是研究基于SNP水平的牛枝子个体间抗旱差异机制的重要候选基因之一。本研究以不同种源的天然牛枝子为材料,克隆了LpDREB基因,分析了该基因的结构和系统进化,研究了LpDREB基因在干旱胁迫下组织和诱导表达规律,并进行了该基因的单核苷酸多态性分析。研究结果为牛枝子抗旱候选基因筛选、抗旱候选基因SNP发掘、基于SNP的牛枝子个体间耐旱机制差异,及以基因为标记的林木分子标记辅助育种研究提供了理论基础。
1 材料与方法 1.1 植物材料试验材料牛枝子种子分别由宁夏银川市、宁夏盐池县、内蒙古呼和浩特市、内蒙古磴口县、甘肃平凉市提供,共5个种源。将饱满种子放于65 ℃热水中浸泡24 h,催芽后在25 ℃人工气候箱中培养。染色体步移文库构建所用DNA由试验材料中的单株牛枝子提取。SNP检测群体由试验材料中300株牛枝子个体组成。
1.2 试验方法 1.2.1 DNA,RNA提取及cDNA合成SNP检测所用模板DNA由300株牛枝子个体采集的叶片等量混合,通过CTAB法(Murray et al., 1980)提取获得。总RNA提取按照北京天恩泽基因有限公司提供的Column Plant RNAout 2.0试剂盒完成,cDNA合成利用promega公司的Reverse Transcription System完成。
1.2.2 染色体步移文库构建选用DraⅠ,EcoRⅤ,Hae Ⅲ,PvuⅡ,RsaⅠ,SspⅠ,StuⅠ,ScaⅠ8种限制性内切酶,对基因组DNA进行酶切。纯化回收后,按照GenomeWalkerTM Kits User Manual连接接头(http://WWW.Clontech.Com/products/listing.asp[DB])。
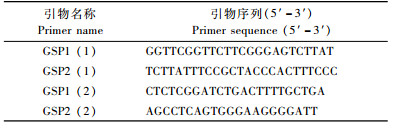
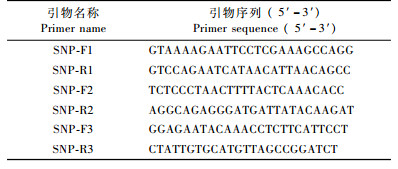
1.2.3 引物设计和PCR反应体系根据NCBI中已注册的DREB氨基酸序列同源区域设计简并引物LpDREBF1:5′-CCTCCRAARCCVACRAARCT-3′;LpDREBR1:5′-GGRAAGTTRAGCCKKGCRAADTC-3′。根据克隆得到的序列片段设计起始PCR和第2次PCR的特异引物(gene specific primer GSP)(表 1),接头引物为AP1:5′-GTAATACGACTCACTATAGGGC-3′;AP2:5′-ACTATAGGGCACGCGTGGT-3′。根据LpDREB基因的核苷酸序列设计用于扩增cDNA序列的引物(LpDREBF2:5′-AGCCTTTTCAGAAACGAGAT-3′;LpDREBR2:5′-AGATCCGGCTAAACAC AATAGA-3′)及用于SNPs检测的特异引物(表 2)。
|
|
|
|
染色体步移PCR中,第1轮PCR反应体系为20 μL,以AP1和GSP1为引物,构建的8个染色体步移文库为模板。第2轮PCR反应体系为50 μL,以AP2和GSP2为引物,以第1轮PCR的反应产物为模板(http://www.clontech.com/products/listing.asp[DB])。用于SNP检测的基因组PCR体系为20 μL,35个循环(李伟等,2007)。
1.2.4 PCR产物测序及结构分析将PCR扩增获得的目的片段纯化回收,连接转化,经菌落PCR后筛选阳性克隆测序。利用DNAman和DNAstar(Lasergene, USA)软件进行序列编辑,应用softberry中FGENESH功能推导出氨基酸序列,进行序列结构分析,利用BioXM2.6软件估算蛋白质的分子质量和等电点,用Phylip和MEGA4.0软件构建LpDREB基因的进化树。
1.2.5 牛枝子干旱胁迫处理根据高琼等(2005)对胡枝子属耐旱性差异的研究,确定干旱胁迫试验体系,取苗龄5个月的牛枝子盆栽苗3株,自然干旱处理(10天)作为试验组。对照组则为相同苗龄的牛枝子盆栽苗3株置于人工气候培养箱中进行正常光照培养。分别采集上述对照及处理后植株的根、茎及叶组织,置于液氮中冻存备用。
1.2.6 LpDREB基因组织及诱导表达根据LpDREB基因序列设计Realtime PCR引物LpDREBRTF:5′-AGACTCCCGAAGAACCGAACCAG-3′;LpDREBRTR:5′-CGGAGTTTGTAAGCGGCTTTG TC-3′, 扩增长度约为95 bp。以肌动蛋白基因Actin作为内参,引物序列ActF:5′-GTGACGTTGATATCA GGAAGGAC-3′;ActR:5′-AAGATAGAGCCACCAA TCCAGAC-3′。RT-PCR反应按照杨帆等(2010)描述的方法进行,利用该基因相对表达量分析其表达模式。
1.2.7 LpDREB基因的SNPs分析利用MEGA4.0和DNAsp软件对LpDREB基因的50个序列进行比对分析,查找LpDREB基因内位于5′非编码区、编码区和3′非编码区的SNPs位点,分析基因序列位于编码区中SNPs位点的同义突变和非同义突变的碱基变异情况及对相应氨基酸的影响。
2 结果与分析 2.1 牛枝子LpDREB基因序列分析以基因组DNA为模板,利用简并引物LpDREBF1和LpDREBR1进行PCR扩增,PCR产物测序后得到1条193 bp的序列。通过Blastp同源性分析,该片段同大豆GmDREB3(序列号DQ055133)相似性达到85%,并且含有DREB基因特有的DREBP/AP2保守区,由此判断该片段属于DREB基因,命名为LpDREB。根据LpDREB基因部分核苷酸序列设计染色体步移引物(表 1),以8个酶切文库为模板,进行巢式PCR,分别获得了长度为737 bp和812 bp的2条序列,利用SegMan软件将3条序列拼接,拼接后序列长度为1 930 bp。通过softberry软件推导出该序列的起始密码子ATG位于286 bp,终止密码子TGA位于1 242 bp。利用引物LpDREBF2和LpDREBR2,以提取的牛枝子总RNA反转录得到的cDNA为模板,PCR扩增得到cDNA序列片段。序列分析结果表明,LpDREB基因cDNA片段全长1 611 bp,包含了285 bp的5′ UTR区,957 bp的ORF区及369 bp的3′ UTR区,可编码长度为318个氨基酸残基的蛋白质(基因注册号为JF900594),AP2/ERF保守区域位于677~856 bp的碱基范围内。cDNA序列同基因组DNA序列进行比对,结果表明该基因内部没有内含子。利用软件BioXM2.6估算蛋白质的分子质量为35.43 ku,等电点为6.87。
2.2 牛枝子DREB蛋白质氨基酸序列同源性及系统进化分析将LpDREB推导的蛋白质氨基酸序列同其他植物DREB蛋白质序列进行同源性比较(图 1)。由图 1可知,该基因编码的氨基酸序列同大豆GmDREB3、柠条锦鸡儿(Caragana korshinskii)CkDREB2、毛果杨(Populus trichocarpa)PtDREB19、玉米ZmDBP2、水稻(Oryza sativa)OsDBF2的蛋白质序列具有较高的同源性,分别为84%,91%,76%,76%,92%。DREB蛋白均包含1个高度保守的DNA结合区域,即APETALA2/乙烯响应因子(AP2/ERF)区域(Riechmann et al., 1998),从图 1可以看出,LpDREB基因的氨基酸序列也含有高度保守的AP2/ERF区域。

|
图 1 DREB蛋白质氨基酸序列比较 Fig.1 Comparison of the deduced amino acid sequence of DREB CkDREB2:柠条锦鸡儿Caragana korshinskii;GmDREB3:大豆Glycine max;OsDBF2:水稻Oryza sativa;PtDREB19:毛果杨Populus trichocarpa;ZmDBP2:玉米Zea mays;LpDREB:牛枝子Lespedeza potaninii。方框内氨基酸序列表示DREB蛋白中AP2/ERF保守区域。Amino acids in the square frame represent the AP2/ERF domain of DREB proteins. |
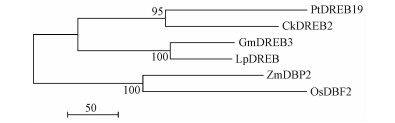
为了分析LpDREB基因同其他植物DREB基因的系统进化关系,构建了LpDREB基因同大豆GmDREB3、柠条锦鸡儿CkDREB2、毛果杨PtDREB19、玉米ZmDBP2、水稻OsDBF2基因的DREB蛋白的系统进化树(图 2)。由图 2可以看出,DREB蛋白由极点向2个方向发生了进化,分别为双子叶植物和单子叶植物。双子叶植物中,牛枝子LpDREB基因和大豆GmDREB3基因的进化关系最近,属于DREB基因亚族A-5组(Chen et al., 2009)。
|
图 2 DREB蛋白的系统进化树 Fig.2 The phylogenetic tree of DREB proteins |
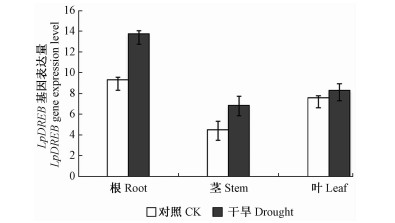
利用荧光定量PCR技术检测了LpDREB基因在牛枝子各组织及干旱胁迫条件下的表达情况。由图 3可知,正常条件下,LpDREB基因在牛枝子的根、茎、叶组织中均有表达,但表达模式不同,根部表达丰度最高,而茎和叶部表达量次之。干旱胁迫处理后,LpDREB基因在不同组织中的转录水平与对照组相比均有不同程度的上调,t检验结果表明根部表达差异最大,达到了显著差异水平(P < 0.05)。LpDREB基因在牛枝子的根部表达量高于茎和叶,且干旱胁迫处理后表达水平差异显著,这与牛枝子生活的地理环境有关,本试验的种子材料来源于干旱的沙漠地区,气候条件及土壤条件使植物的根系朝向更有利于抗胁迫的方向发展。
|
图 3 LpDREB基因的组织及干旱胁迫诱导表达 Fig.3 Expression profile of LpDREB in roots, stems and leaves induced by drought stress |
设计了3对用于SNP检测的特异引物(表 2),模板为300株牛枝子个体混合DNA,PCR产物长度分别为625 bp,545 bp和744 bp,序列之间有80~100 bp重复区域。挑选50个阳性克隆测序,将得到的基因序列进行单核苷酸多态性分析(表 3)。由表 3可知,长度为1 611 bp的序列中共有SNPs位点23个,发生频率为1/70,其中5′非翻译区、外显子和3′非翻译区的SNPs位点分别为4,17,2个,频率为1/71,1/56,1/185。对LpDREB基因中的23个SNPs位点进行变异类型统计(表 4),结果显示,23个SNPs位点中12个属于转换,分别包括3个A⇔G和9个T⇔C,11个颠换分别包括2个A⇔T,5个G⇔C和4个G⇔T,转换与颠换的比值为1.09( < 2.0)。
|
|
|
|
为了检测LpDREB基因编码区内核苷酸的改变对氨基酸序列是否产生了影响,对LpDREB基因编码区内的17个SNPs位点进行了同义突变、非同义突变的分析(表 5)。由表 5可知:17个SNPs位点中9个为同义突变,它们都位于密码子的第3个核苷酸上;非同义突变为8个,其中错义突变8个,无义突变0个;同义突变与非同义突变的比值为1.1(>1)。在8个非同义突变中,有3个位于密码子的第1个核苷酸上,其密码子由原来的CTC,GCT,CTT分别转变为GTC,ACT和GTT,氨基酸由原来的亮氨酸(Leu)、丙氨酸(Gla)和亮氨酸(Leu)分别转变为缬氨酸(Val)、苏氨酸(Thr)和缬氨酸(Val);2个位于密码子的第2个核苷酸上,其密码子由原来的AAT和CTC转变为AGT和CCC,氨基酸由原来的天冬酰胺(Asn)和亮氨酸(Leu)转变为丝氨酸(Ser)和脯氨酸(Pro);3个位于密码子的第3个核苷酸上,其密码子由原来的CAT,TTG和CAC转变为CAA,TTC和CAG,氨基酸由原来的组氨酸(His)、亮氨酸(Leu)和组氨酸(His)转变为谷氨酰胺(Gln)、苯丙氨酸(Phe)和谷氨酰胺(Gln)。结果表明,LpDREB基因编码区内存在着丰富的SNPs,而且近50%的SNPs导致了氨基酸的改变,这一结果为基于SNP水平的牛枝子个体间耐旱机制差异的研究奠定了重要的基础。
|
|
DREB转录因子在植物抗逆反应中扮演着重要的角色(Liu et al., 1998;Agarwal et al., 2006),迄今为止,尚没有关于LpDREB基因调控机制的相关报道。目前关于DREB基因亚族的研究主要集中在A-1和A-2 2个较大的组中(Liu et al., 1998;Nakashima et al., 2006;Shen et al., 2003a),对于其他成员特点及功能的研究却很少。对A-5组成员比如PpDBF1, GmDREB2和GhDBF1(Huang et al., 2006;Liu et al., 2007;Chen et al., 2007)的研究指出,A-5组同A-1组成员都是比较重要的基因资源,这些基因对提高植物抗逆性具有一定的作用。本研究通过染色体步移技术从牛枝子中分离得到LpDREB基因序列全长,进化分析表明LpDREB基因同大豆中的GmDREB3基因(Chen et al., 2009)进化关系最近,同属于A-5组。牛枝子DREB蛋白氨基酸序列同大豆DREB蛋白质氨基酸序列同源性分析结果显示,2个物种的DREBP/AP2保守区域具有高度的相似性,而除保守区域的其他区域片段相似性比较低(图 1),可能是由于牛枝子LpDREB基因同大豆GmDREB基因亲缘关系较远,也可能是由于同一种基因在不同物种中的进化速率不同引起的。在对MYB蛋白家族(Dias et al., 2003)、bHLH蛋白家族(Atchley et al., 1994)及大豆的DREB蛋白家族(Chen et al., 2009)的研究中都出现过相似的结果。
目前关于DREB基因在逆境胁迫下诱导表达的研究很多,DREB1A/CBF3(A-1)基因被低温诱导表达,而不能被干旱或者高盐诱导,DREB2A (A-2)基因能被干旱和高盐诱导表达,却不能低温诱导。A-1和A-2组的基因不依赖ABA信号转导途径(Liu et al., 1998),TINY2 (A-4), GhDBP1 (A-5), GmDREB2 (A-5)及ZmDBF1 (A-6)都是能被胁迫诱导表达的基因,同时它们是ABA依赖型的(Wei et al., 2005;Huang et al., 2006;Kizis et al., 2002;Chen et al., 2007)。但是关于植物特定组织诱导表达的研究却很少。曾有研究报道,盐处理后,榆钱菠菜(Atriplex hortensis)的AhDREB1基因(Shen et al., 2003b)在根部被诱导表达,在茎和叶中却不能被诱导表达。逆境胁迫下,大豆中的GmDREBa和GmDREBb基因在根、茎、叶中都能被诱导表达,而GmDREBc基因只能在根部被高度诱导表达(Li et al., 2005)。荧光定量PCR结果表明,LpDREB基因在成熟牛枝子的根、茎、叶中具有不同的表达水平,其中根部的表达量最高。干旱胁迫处理后,牛枝子根、茎和叶中LpDREB基因均被诱导,表达水平呈上升趋势,并且根部达到显著差异水平,这一结果说明牛枝子根部对逆境胁迫的应答响应优于茎和叶。这种结果是比较合理的,因为同茎和叶相比,根部能够较早地响应来自土壤中的胁迫,诱导大量基因表达启动抗逆调控网络,从而使植物适应不良环境继续生存下去。研究植物特定组织诱导表达机制对林木育种具有重要的意义。
在植物基因组中,SNP被认为是最丰富的且能够稳定遗传的新一代分子标记(Rafalski et al., 2002)。对LpDREB基因进行SNPs检测,该基因非编码区有SNPs位点6个,编码区有17个,非编码区SNPs位点的数量少于编码区,这一结果同前人对人类(Bensen et al., 2003)、大豆(Zhu et al., 2003)和玉米(Ching et al., 2002)的研究结果相反,可能是非编码区未被完全获得,样本量较少或者LpDREB基因同其他基因相比具有自身的特点,比如热点突变区域多分布在编码区等。23个SNPs位点中转换12个,颠换11个,比值为1.09 ( < 2),同大豆DREB基因中SNPs位点转换与颠换的比例1:1相似(Zhu et al., 2003),出现这一结果的原因可能是SNPs位点个数较少或者样本量较少。编码区的17个SNPs位点中,9个为同义突变,其中只有1个位于该基因的保守区域内,说明LpDREB基因的DREBP/AP2区域在生物进化过程中是高度保守的;8个为错义突变,错义突变的SNPs位点导致了氨基酸的改变,这些氨基酸的改变是否引起基因功能的改变,需要进行下一步的表型与基因型的关联分析。牛枝子LpDREB基因SNPs同义突变与非同义突变的比值为1.1,可能是由于LpDREB基因受到负向选择压力的作用。
综上所述,牛枝子LpDREB基因受干旱胁迫被诱导表达,该基因是牛枝子抗旱的重要候选基因。牛枝子LpDREB基因与大豆GmDREB3基因亲缘关系最近,同其他物种的DREB蛋白质氨基酸序列比较,除保守区域外其余序列片段相似性较低。牛枝子LpDREB基因中有丰富的SNPs位点,这些位点既存在于编码区内,也存在于非编码区内,同时有部分SNPs位点导致了氨基酸种类的改变。牛枝子LpDREB基因的诱导表达特性及大量存在的SNPs位点为阐释牛枝子个体间抗旱机制差异提供了重要的分子基础,也为今后林木抗旱基因筛选、抗旱基因的研究及以基因为标记的分子标记辅助选择育种提供了新的思路。
| [] | 高琼, 陈晓阳, 杜金友, 等. 2005. 不同种和种源胡枝子的耐旱性差异研究. 北华大学学报:自然科学版, 6(3): 257–260. |
| [] | 李伟, 李慧, 陈晓阳, 等. 2007. 利用染色体步移PCR检测辐射松的单核苷酸多态性. 西北植物学报, 27(8): 1571–1576. |
| [] | 李新荣. 1997. 毛乌素沙地灌木资源区系特征及其保护对策. 自然资源学报, 12(1): 146–152. |
| [] | 骈瑞琪, 陈晓阳, 赵杨. 2005. 胡枝子属植物的遗传育种研究进展. 西部林业科学, 34(4): 105–110. |
| [] | 杨帆, 丁菲, 杜天真. 2010. 盐胁迫下构树DREB转录因子基因表达的实时荧光定量PCR分析. 林业科学, 46(4): 146–150. DOI:10.11707/j.1001-7488.20100422 |
| [] | Agarwal P K, Agarwal P, Reddy M K, et al. 2006. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants. Plant Cell Reports, 25(12): 126–1274. |
| [] | Atchley W, Fitch W, Bronner-Fraser M. 1994. Molecular evolution of the MyoD family of transcription factors. Proceedings of the National Academy of Sciences USA, 91(24): 11522–11526. DOI:10.1073/pnas.91.24.11522 |
| [] | Aulchenko Y S, de Koning D J, Haley C. 2007. Genome wide rapid association using mixed model and regression: a fast and simple method for genome wide pedigree-based quantitative trait loci association analysis. Genetics, 177(1): 577–585. DOI:10.1534/genetics.107.075614 |
| [] | Bensen J T, Langefeld C D, Hawkins G A, et al. 2003. Nucleotide variation, haplotype structure, and association with end-stage renal disease of the human interleukin-1 gene cluster. Genomics, 82(2): 194–217. DOI:10.1016/S0888-7543(03)00123-X |
| [] | Bouchabke O, Chang F, Simon M, et al. 2008. Natural variation in Arabidopsis thaliana as a tool for highlighting differential drought responses. PLoS one, 3(2): e1705. DOI:10.1371/journal.pone.0001705 |
| [] | Chen Ming, Xu Zhaoshi, Xia Lanqin, et al. 2009. Cold-induced modulation and functional analyses of the DRE-binding transcription factor gene, GmDREB3, in soybean (Glycine max L.). Journal of Experimental Botany, 60(1): 121–135. DOI:10.1093/jxb/ern269 |
| [] | Chen Ming, Wang Qiaoyan, Cheng Xianguo, et al. 2007. GmDREB2, a soybean DRE-binding transcription factor, conferred drought and high-salt tolerance in transgenic plants. Biochemical Biophysical Research Communications, 353(2): 299–305. DOI:10.1016/j.bbrc.2006.12.027 |
| [] | Ching A, Caldwell K S, Jung M, et al. 2002. SNP frequency, haplotype structure and linkage disequilibrium in elite maize inbred lines. BMC Genetics, 3: 19. DOI:10.1186/1471-2156-3-19 |
| [] | Dias A P, Braun E L, McMullen M D, et al. 2003. Recently duplicated maize R2R3 Myb genes provide evidence for distinct mechanisms of evolutionary divergence after duplication. Plant Physiology, 131(2): 610–620. DOI:10.1104/pp.012047 |
| [] | Haake V, Cook D, Riechmann J L, et al. 2002. Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis. Plant Physiology, 130(2): 639–648. DOI:10.1104/pp.006478 |
| [] | Huang Bo, Liu Jinyuan. 2006. A cotton dehydration responsive element binding protein functions as a transcriptional repressor of DRE-mediated gene expression. Biochemical and Biophysical Research Communications, 343(4): 1023–1031. DOI:10.1016/j.bbrc.2006.03.016 |
| [] | Ingvarsson P K. 2005. Nucleotide polymorphism and linkage disequilibrium within and among natural populations of European aspen (Populus tremula L., Salicaceae). Genetics, 169(2): 945–953. DOI:10.1534/genetics.104.034959 |
| [] | Kizis D, Pages M. 2002. Maize DRE-binding proteins DBF1 and DBF2 are involved in rab17 regulation through the drought-responsive element in an ABA-dependent pathway. The Plant Journal, 30(6): 679–689. DOI:10.1046/j.1365-313X.2002.01325.x |
| [] | Li Xueping, Tian Aiguo, Luo Guangzuo, et al. 2005. Soybean DRE-binding transcription factors that are responsive to abiotic stresses. Theoretical and Applied Genetics, 110(8): 1355–1362. DOI:10.1007/s00122-004-1867-6 |
| [] | Liu Ning, Zhong Naiqin, Wang Guiling, et al. 2007. Cloning and functional characterization of PpDBF1 gene encoding a DRE-binding transcription factor from Physcomitrella patens. Planta, 226(4): 827–838. DOI:10.1007/s00425-007-0529-8 |
| [] | Liu Qiang, Kasuga M, Sakuma Y, et al. 1998. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought and low-temperature-responsive gene expression, respectively, in Arabidopsis. Plant Cell, 10(8): 1391–1406. DOI:10.1105/tpc.10.8.1391 |
| [] | Murray M G, Thompson W R. 1980. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Research, 8(19): 4321–4325. DOI:10.1093/nar/8.19.4321 |
| [] | Nakashima K, Yamaguchi-Shinozaki K. 2006. Regulons involved in osmotic stress-responsive and cold stress-responsive gene expression in plants. Plant Physiology, 126(1): 62–71. DOI:10.1111/ppl.2006.126.issue-1 |
| [] | Rafalski A. 2002. Applications of single nucleotide polymorphisms in crop genetics. Current Opinion in Plant Biology, 5(2): 94–100. DOI:10.1016/S1369-5266(02)00240-6 |
| [] | Riechmann J L, Meyerowitz E M. 1998. The AP2/EREBP family of plant transcription factors. Journal of Biological Chemistry, 379(6): 633–646. |
| [] | Saleh A, Lumbreras V, Lopez C, et al. 2006. Maize DBF1-interactor protein 1 containing an R3H domain is a potential regulator of DBF1 activity in stress responses. The Plant Journal, 46(5): 747–757. DOI:10.1111/tpj.2006.46.issue-5 |
| [] | Shen Y G, Zhang W K, He S J, et al. 2003a. An EREBP/AP2 type protein in Triticum aestivum was a DRE-binding transcription factor induced by cold, dehydration and ABA stress. Theoretical and Applied Genetics, 106(5): 923–930. DOI:10.1007/s00122-002-1131-x |
| [] | Shen Y G, Zhang W K, Yan D Q, et al. 2003b. Characterization of a DRE-binding transcription factor from a halophyte Atriplex hortensis. Theoretical and Applied Genetics, 107(1): 155–161. DOI:10.1007/s00122-003-1226-z |
| [] | Shinozaki K, Yamaguchi-Shinozaki K. 2007. Gene networks involved in drought stress response and tolerance. Journal of Experimental Botany, 58(2): 221–227. |
| [] | Wang Qiuyun, Guan Yucheng, Wu Yaorong, et al. 2008. Overexpression of a rice OsDREB1F gene increases salt, drought and low temperature tolerance in both Arabidopsis and rice. Plant Molecular Biology, 67(6): 589–602. DOI:10.1007/s11103-008-9340-6 |
| [] | Wei Gang, Pan Yi, Lei Juan, et al. 2005. Molecular cloning, phylogenetic analysis, expressional prowling and in vitro studies of TINY2 from Arabidopsis thaliana. Journal of Biochemistry and Molecular Biology, 38(4): 440–446. |
| [] | Zhu Y L, Song Q J, Hyten D L, et al. 2003. Single-nucleotide polymorphisms in soybean. Genetics, 163(3): 1123–1134. |
 2012, Vol. 48
2012, Vol. 48