文章信息
- 刘雪梅, 刘瀛, 宋福南, 邢磊, 戴超, 杨传平
- Liu Xuemei, Liu Ying, Song Funan, Xing Lei, Dai Chao, Yang Chuanping
- 白桦雄花突变体早期发育差异表达基因的cDNA-AFLP分析
- Differential expression analysis of transcripts at early developmental stage of male inflorescence mutant of White Birch (Betula platyphylla) by cDNA-AFLP
- 林业科学, 2012, 48(5): 20-28.
- Scientia Silvae Sinicae, 2012, 48(5): 20-28.
-
文章历史
- 收稿日期:2011-09-02
- 修回日期:2011-10-20
-
作者相关文章
白桦(Betula platyphylla)是中国北方重要的阔叶树种之一,是典型的雌雄同株单性花树种,因此是研究单性花发育机制的理想材料。目前,白桦花发育研究主要集中在细胞学和系统发育研究,而分子生物学研究则较少,进展较慢。近年来,对桦木属(Betula)花发育调控基因的研究取得了一些成果,例如,已从欧洲白桦(Betula pendula)中分离出7个花发育相关的基因,其中包括6个MADS-box基因(Lemmetyinen et al., 2004;Elo et al., 2001)和1个SBP-box基因(Lannenpaa et al., 2004),这些基因可能作用于早期营养阶段向生殖阶段的转变。作用于开花时间调控路径的结点基因SOC1,参与营养生长和生殖生长(刘菲菲等, 2011),参与花序及花分生组织特异性的形成,促进其早花(Elo et al., 2001)。王超等(2008)发现了多个花发育相关基因,其功能涉及到调控花序形成、花分化及花粉管发育(杨传平等, 2006)。白桦雄花序发育初期的一些表达差异蛋白,可能与花分生组织及花器官的形成有关(杨传平等, 2004)。另外,通过外施激素研究了调控白桦花发育中成花基因的表达情况(Wang et al., 2011;Zhang et al., 2011)。虽然白桦花发育研究已有一些成果,但是,高等植物的花发育是一个复杂而精细的动态过程,作为该领域研究的新树种,其花发育分子机制研究还处于初期阶段,现有的成果还远不能揭示其发育机制,需要更深入系统的研究。
花发育突变体是研究花器官发育及其功能基因组学的理想材料。前期发现的天然白桦雄花序发育异常突变体,与正常的雄花序相比,该突变体在花序的着生位置、形态结构、花药形态及生殖发育等方面都有明显不同(刘雪梅等, 2010)。正常植株的雄花序着生在枝条的顶端,而突变植株上有2种不同的雄花序:一种与正常雄花序形态结构和着生位置等很相似(NL),但后期发育异常,表现为败育特征,且越冬后散粉时雄花序枯死,或者只有少数花药开裂散粉;另一种雄花序则为典型的雄花序突变体(M),其花药的形态及发育、减数分裂及雄配子体形成均严重异常,越冬后枯死,花药不开裂,导致败育(刘雪梅等, 2010)。因此,该突变体可用于白桦雄花发育机制的研究。
cDNA-AFLP技术效率高,重复性好,灵敏度高,可用于基因表达差异分析(Zhang et al., 2008;Capomaccio et al., 2009;安泽伟等, 2010;Shi et al., 2011),可在一定程度上揭示植物发育机制,并在相应的调控机制和遗传网络基本信息方面提供基础信息(Cnundde et al., 2003)。本研究采用cDNA-AFLP技术,以白桦雄花突变植株早期发育的2种雄花序(NL和M)为材料,分析二者的差异基因表达谱,共分离了81个ESTs,选取其中5个与花发育相关的ESTs分析其转录表达规律。该研究为今后进一步揭示白桦雄花发育及雄性不育机制奠定了基础,也为将来对重要的单性花发育相关基因进行生物学功能研究提供有价值的资料。
1 材料与方法 1.1 植物材料取2009年7月5日—8月27日(每3天取材1次)的突变植株的正常雄花序(NL)和突变雄花序(M)用于cDNA-AFLP分析。2010年7月17日的正常植株雄花序和突变植株的突变雄花序(M)、雌花序、幼叶、幼枝,及不同发育阶段(7月17日—8月27日每3天取材1次)的雄花序(M和NL)进行Real-time RT-PCR分析。材料采集后以液氮速冻后保存于-80 ℃。
1.2 总RNA的提取和cDNA的合成利用改进的CTAB方法提取各时期正常雄花序(NL)和突变雄花序(M)的总RNA,并用RNase-free Dnase I(Promega,Mandson,USA)对RNA进行DNA消化,分别将纯化后突变雄花序(M)与正常雄花序(NL)的RNA进行等量混合(终浓度为2 μg·μL-1)。采用M-MLV Rtase cDNA合成试剂盒(TaKaRa,Japan)合成双链cDNA。
1.3 cDNA-AFLP分析参考Bachem等(1998)的方法,并利用已优化的程序进行cDNA-AFLP分析(邢磊等, 2010)。预扩增PCR反应体系:5 μL连接产物,2 μL 10×PCR Buffer,1.2 μL MgCl2(25 mmol·L-1),2 μL dNTP(2.0 mmol·L-1),0.6 μL EcoRⅠ引物(20 μmol·L-1),0.6 μL MseⅠ引物(20 μmol·L-1),0.2 μL Taq酶(5 U·μL-1),加ddH2O至20 μL。预扩增条件如下:94 ℃ 3 min;94 ℃ 30 s,56 ℃ 30 s,72 ℃1 min,30个循环;72 ℃ 5 min。将预扩增产物稀释40倍,用于选择性扩增模板。选择性扩增PCR反应体系:5 μL稀释的预扩增产物,2 μL 10×PCR Buffer,1.2 μL MgCl2(25 mmol·L-1),2 μL dNTP(2.0 mmol·L-1),0.6 μL EcoRⅠ引物(20 μmol·L-1),0.6 μL MseⅠ引物(20 μmol·L-1),0.2 μL Taq酶(5 U·μL-1),加ddH2O至20 μL。选择性扩增条件如下:94 ℃ 3 min;94 ℃ 30 s,65 ℃(每个循环-0.7 ℃)30 s,72 ℃ 1 min,13个循环;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,23个循环;72 ℃ 5 min。扩增产物在6%的聚丙烯酰胺变性凝胶上电泳,银染检测。
1.4 差异片段的克隆及序列分析将差异表达片段回收纯化后克隆到PMD-18T载体中(TaKaRa,Japan)并测序,利用BLASTX程序与NCBI数据库进行同源性分析(Altschul et al., 1997)。应用GO(Gene Ontology Consortium)(http:∥www.geneontology.org)进行生物学功能分析,并运用KEGG软件对其代谢途径进行分析(http: ∥www.genome.jp/kegg/)(Kim et al., 2010)。
1.5 差异片段的Real-time RT-PCR分析用Primer 5.0软件设计引物(表 1)。反应试剂盒为SYBR Green Realtime PCR Master Mix(TOYOBO,Osaka,Japan),反应体系:10 μL SYBR Premix Ex Taq,2 μL cDNA模板,上下游引物各1 μL,6 μL ddH2O。扩增程序为:95 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,79 ℃ 1 s,共40个循环。试验设置3次重复,数据利用Opticon Monitor 2软件保存和分析,整个反应过程在OPTIONⅡ荧光定量PCR仪上进行。
|
|
运用cDNA-AFLP方法筛选出白桦突变雄花序(M)与正常雄花序(NL)超过4 500个差异cDNA片段,每对引物组合平均扩增20~30条片段,片段长度为100~500 bp,从中挑选280条多态性片段(图 1),回收后得到81条差异片段,在NCBI中应用BLASTX数据库进行基因同源性比对,并提交GenBank EST数据库(JK546624—JK546704)(表 2)。其中,51个ESTs(63%)在数据库中显示相似性较低即无匹配(no match),30个ESTs(37%)显示与已知蛋白同源性较高。另外,在81个ESTs中,25个ESTs(31%)在M中下调表达,33个ESTs(41%)在M中上调表达,13个ESTs(16%)在NL中特异性表达,10个ESTs在M中特异性表达。
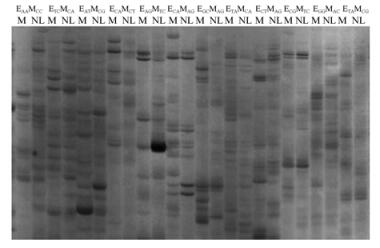
|
图 1 不同引物组合的cDNA-AFLP扩增图谱 Fig.1 The cDNA-AFLP map using different primers NL:突变株中类似正常雄花序;M:突变株中突变雄花序。下同。 NL: Normal-like male inflorescences in mutant; M: Mutant male inflorescences in mutant. The same below. |
|
|
对30个已知同源性蛋白的ESTs序列进行GO功能分类,30个ESTs均归类为生物学过程和分子功能,29个ESTs归于细胞组分。在生物学过程中(图 2),参与代谢过程和刺激反应过程均为13.33%,其他高表达ESTs参与细胞过程(10%)、发育过程(10%)、生物黏附(6.67%)等过程。另外,本研究结果显示碳水化合物的代谢和生物合成过程只存在于NL雄花序中,而生物调节和细胞成分的组织或起源过程则特异性地存在于突变雄花序M中。29个ESTs归于细胞组分,其中包含叶绿体(16.67%)、胞外区(20%)、内膜系统(10%)等细胞组分,与线粒体和液泡膜有关的ESTs特异出现于NL中。在分子功能分类中,参与结合活性的ESTs为30%,催化活性为13.33%,转移酶活性为13.33%,其中水解酶活性相关的ESTs只在NL中表达,而激酶活性和化学诱导活性则特异表达于M中(数据未列出)。这些差异暗示了突变雄花序发育的复杂性和多样性,其不同基因在雄花发育的过程中发挥着重要作用。
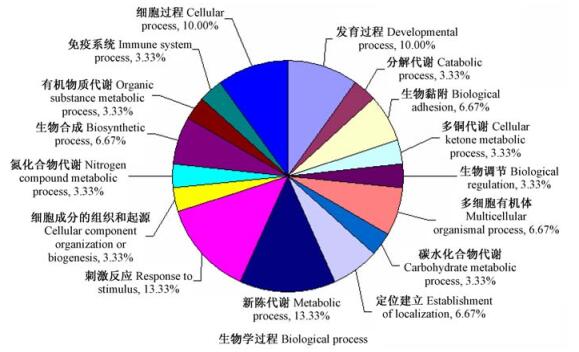
|
图 2 NL和M花序中差异序列的GO生物过程分析 Fig.2 GO biological process analysis of differential sequences expressed in NL and M inflorescences |
运用KEGG数据库对30个ESTs进行生物途径分析(表 3),6个ESTs(20%)参与新陈代谢途径,3个ESTs(10%)参与细胞周期,另外,分别有2个ESTs(6.67%)参与核糖体、ABC转运蛋白、吞噬体、次生代谢产物的合成和泛素介导的蛋白水解等途径。此外,有6个生化途径相关的7个ESTs在M中下调表达,另外5个生化途径相关的7个ESTs在M中上调表达(表 3)。这些突变雄花序的生化代谢途径差异说明了白桦雄花发育的复杂性,并提供了一些可能与雄性不育有关的代谢调控途径和基因。
|
|
选择5个ESTs进行qRT-PCR分析,其中3个ESTs在突变雄花序(M)中下调表达(图 3),包括Bplmme15(MAP kinase,MAP激酶),Bplmme29(Ubiquitin-conjugating enzyme,泛素结合酶),Bplmme160(Cellulose synthase,纤维素合成酶);2个ESTs在突变雄花中上调表达(图 3),包括Bplmme158(Heat shock protein,热激蛋白),Bplmme252(Cytochrome P450,细胞色素P450)。此结果对应于cDNA-AFLP的差异表达,证明这种方法对NL和M花序之间差异表达的研究是可信的。
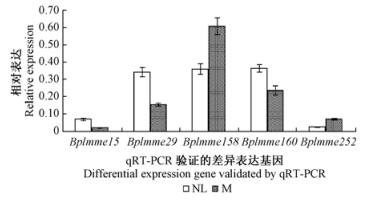
|
图 3 qRT-PCR验证NL和M花序中的差异表达基因 Fig.3 Differential expression gene validated by qRT-PCR in NL and M inflorescences M:突变植株中的突变雄花序;NL:突变植株中类似正常的雄花序。 M: Mutant male inflorescences in mutant plant; NL: Normal-like male inflorescences in mutant plant. |
为了探索这5个基因的转录表达特征和规律,首先进行了正常植株(WtP)和突变植株(MP)4种组织(雄花序、雌花序、幼叶和幼枝)的表达分析(图 4a, c, e, g, i)。结果表明,这些基因在不同组织表达情况有明显差异。与正常植株相比,这5个基因在突变植株的雌花、幼叶、幼枝中的表达情况有很大的不同,其中Bplmme15,Bplmme252 2个基因在突变植株的雌花中下调表达(图 4a, i),而其他3个基因则出现上调表达(图 4c, e, g)。突变植株的幼叶中Bplmme252出现下调表达(图 4i),而Bplmme15,Bplmme29,Bplmme160出现上调表达(图 4a, c, g)。此外,这种差异也出现在突变幼枝中,Bplmme15,Bplmme252下调表达(图 4a, i),Bplmme29,Bplmme158上调表达(图 4c, e)。5个基因中除Bplmme15和Bplmme158在突变植株的雄花序中表达无明显变化外(图 4a, e),其他3个基因(Bplmme29,Bplmme160,Bplmme252)均出现下调表达(图 4a, g, i)。另外,个别基因在2种植株中个别部位的表达有显著变化,例如Bplmme15在雌花中显著下调而在幼叶中显著上调表达(图 4a),Bplmme29在雄花中显著下调而在雌花中显著上调表达(图 4c)。可见,这些基因不仅参与生殖器官发育,也在营养器官中发挥一定的作用,同时,也暗示它们可能与突变植株的异常发育及雄性不育有关。
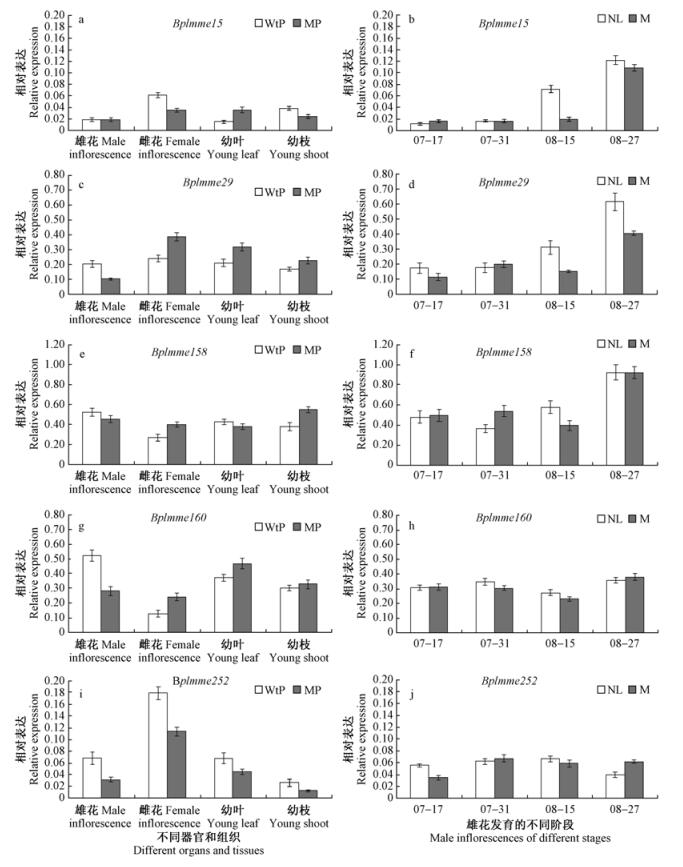
|
图 4 Real-time RT-PCR分析5个基因在白桦植株各组织和雄花序发育的不同阶段的表达情况 Fig.4 Real-time RT-PCR analyses of five genes transcription expressions in white birch plant(tissues) and male inflorescences mutant(stages) WtP:野生型植株;MP:突变植株;NL:突变株中类似正常雄花序;M:突变株中突变雄花序。 WtP: Wild-type plant; MP: Mutant plant; NL: Normal-like male inflorescences in mutant; M: Mutant male inflorescences in mutant. |
此外,本研究还对突变植株的2种雄花序(NL和M)减数分裂期间(7月中旬—8月末)的转录表达进行了分析。结果表明,5个基因在2种雄花序之间存在明显的差异表达(图 4b,d,f,h,j)。与NL相比,Bplmme15和Bplmme29在减数分裂晚期(8月15—27日)的表达量显著降低(图 4b,d),Bplmme160减数分裂中期(7月31日—8月15日)轻微下调表达(图 4h)。另外,Bplmme252在突变雄花减数分裂发育初期发生明显下调表达,而在减数分裂发育晚期出现明显的上调表达(图 4j)。推测这些基因在特定时期的表达变化与突变雄花序的减数分裂有关,它们可能是白桦雄花配子体发育中的重要基因。
3 讨论关于植物雄性不育性的机制已有很多报道,但对于生殖周期较长的白桦来说,其雄性不育的研究还未见报道,尤其是自然条件下形成的白桦花发育突变体更为少见。本研究应用cDNA-AFLP技术分析2种白桦早期发育雄花序(NL与M)的差异表达转录图谱,共获得81条差异表达ESTs,参与信号转导、能量代谢、胁迫反应、蛋白运输和转录等过程,这些基因差异表达的多样性,暗示白桦雄花序早期发育的复杂性。另外,有51个ESTs可视为新基因,说明白桦作为一个花发育基因组研究的新物种,还有很多方面值得更多的研究和探索。
本研究中,有58个(72%)ESTs序列在白桦突变体早期雄花中出现上调或下调表达,有23个(28%)ESTs序列特异性地表达于NL和M雄花中,这些差异片段共产生18个生物代谢途径(表 3),由此可以推测,这些差异表达的基因及其参与的代谢途径可能与白桦雄性不育有着某种联系。要特别指出的是,这些差异表达基因还可能与雄花早期发育有关,它们参与的生物学过程主要包括发育、代谢、应激反应等过程(图 2)。研究结果显示白桦的雄花早期发育是由多基因参与及精确调控的复杂生物过程。
通过对5个ESTs(Bplmme15,Bplmme29,Bplmme160,Bplmme158和Bplmme252)的Real-time RT-PCR分析,发现它们在白桦不同组织和雄花序发育不同阶段中的表达量存在差异(图 4),表明它们在雄花的结构、功能和发育上发挥着不同的作用。例如Bplmme29(泛素结合酶)、Bplmme160(纤维素合成酶)和Bplmme158(热激蛋白)的表达量均明显高于Bplmme15(MAP激酶)和Bplmme252(细胞色素P450),平均相差约8~10倍(图 3),推测前3个基因在白桦发育过程中具有重要作用。同时,这5个基因不仅仅在雄花序中表现出表达差异,在其他组织(雌花、叶、枝条等)中的表达也有差异,说明这些基因除了与雄花序异常发育有关之外,也参与其他组织的代谢和基因表达系统。
雄配子体形成与花药发育、小孢子发生、减数分裂等多个过程密切相关,其中,减数分裂是非常重要的环节,不正常的减数分裂是导致雄性不育的重要原因之一。本研究中突变植株中的突变雄花序M的减数分裂延迟且异常,直接影响了小孢子和花粉的形成(刘雪梅等, 2010)。研究中发现有的基因在M进行减数分裂期间的表达发生了很大变化,推测这些基因与M早期花药发育、减数分裂和小孢子发育异常有着一定的联系,在雄性生殖发育中起着重要作用。例如,Bplmme15与拟南芥的MAP激酶有很高的同源性,该基因在植物体受到机械压力、创伤、盐害、极端温度、干旱缺水等信号传递过程中发挥着重要作用(宋东辉等, 2004; 江静等, 2003)。也有证据表明,MAP激酶家族成员在花粉中表达,并可能对花粉发育和萌发具有调节功能(Heberle-Bors et al., 2001)。Bplmme15在雌花、幼枝及突变雄花序M减数分裂晚期呈现较明显的下调表达(图 4b),暗示该基因表达受到抑制与突变植株的异常发育有密切关系。Bplmme29同源于拟南芥的泛素结合酶,该基因在植物中发挥重要的作用,如参与DNA的修复、维管的分化调控、光周期反应,缺素(王金利等, 2010)、植物衰老(Picton et al., 1993)及抗逆胁迫响应等(Zhou et al., 2010)。Bplmme29在突变雄花序M中减数分裂后期表达受到明显抑制(图 4d),推测该基因参与减数分裂后期发育。Bplmme160与桉树的纤维素合成酶有91%的同源性,该基因在植物细胞壁发育(Kalluri et al., 2004)及茎伸长(Wise et al., 2011)中具有重要作用。Bplmme160在突变植株MP的雄花序中下调表达,在雌花和幼叶中上调表达(图 4g),但在正常雄花序NL和突变雄花序M减数分裂期间的表达无明显差异(图 4h),推测Bplmme160在减数分裂中作用不大,但可能参与雌花及初生细胞壁发育。Bplmme158与黄瓜(Cucumis sativus)的热激蛋白有98%的同源性,该基因对植物抵抗胁迫环境和生长发育起着重要作用(胡雨晴等, 2011)。在突变植株的雌花和幼枝中的上调表达说明Bplmme158可能参与二者的发育过程(图 4e)。另外Bplmme158在M减数分裂晚期的下调(8月15日)和大量(8月27日)表达(图 4f)预示该基因可能主要参与减数分裂晚期发育,并与由此导致的雄性不育有关。Bplmme252同源于脂麻(Sesamum schinzianum)的细胞色素P450,该基因参与多种生化反应、生物体内代谢途径及催化机制(杨致荣等, 2003),也参与花发育的调控(Su et al., 2009)。Bplmme252在雌花序中表达量明显高于其他组织(图 4i),在突变雄花序M减数分裂早期和晚期的表达有明显改变(图 4j),说明该基因参与雌花的发育,并且在突变雄花减数分裂早晚期发育中发挥着重要的作用。
植物的器官发育是一个复杂而循序渐进的形态建成过程,其中涉及到很多代谢和调控过程,需要很多基因的协调表达来维持系统平衡,一旦这种平衡被打破,就会造成发育异常。本研究表明,白桦雄花异常发育突变植株不仅仅表现为雄花序的异常,其他组织器官中多个基因的表达也发生了较大改变,因此,本研究筛选的这些基因不仅与雄性不育有关,还可能参与了其他组织的发育过程。今后将对这些基因进行系统深入的生物学功能研究,以便于了解这些基因在白桦花器官和其他组织发育过程中的调控机制和作用。
| [] | 安泽伟, 陈根辉, 程汉, 等. 2010. 橡胶树冷应答转录组cDNA-AFLP分析. 林业科学, 46(3): 62–67. DOI:10.11707/j.1001-7488.20100310 |
| [] | 胡雨晴, 孙文婷, 马婧, 等. 2011. 蜡梅热激蛋白基因CpHSP1. 林业科学, 47(5): 162–167. DOI:10.11707/j.1001-7488.20110527 |
| [] | 江静, 安国勇, 王鹏程, 等. 2003. MAP激酶调节蚕豆保卫细胞中ABA诱导的H2O2产生. 科学通报, 48(12): 1256–1263. DOI:10.3321/j.issn:0023-074X.2003.12.005 |
| [] | 刘菲菲, 李慧玉, 王姗, 等. 2011. 白桦BpSOC1. 东北林业大学学报, 39(4): 1–4. |
| [] | 刘雪梅, 周菲, 邢磊, 等. 2010. 白桦雄花突变体及其细胞学特征. 东北林业大学学报, 38(6): 1–3. |
| [] | 宋东辉, 宋凤鸣, 郑重. 2004. MAP激酶在植物信号传递网络中的功能. 浙江大学学报:农业与生命科学版, 30(2): 119–126. |
| [] | 王超, 杨传平, 魏继承, 等. 2008. 白桦雌花序抑制性消减文库构建及EST分析. 植物研究, 28(3): 293–298. |
| [] | 王金利, 史胜青, 贾利强, 等. 2010. 植物泛素结合酶E2功能研究进展. 生物技术通报, 12(4): 7–10. |
| [] | 邢磊, 刘雪梅, 宋福南. 2010. 白桦cDNA-AFLP体系的优化和建立. 东北林业大学学报, 38(10): 15–18. DOI:10.3969/j.issn.1000-5382.2010.10.006 |
| [] | 杨传平, 姜静, 梁艳, 等. 2004. 白桦雄花序发育初期蛋白质的双向电泳图谱分析. 东北林业大学学报, 32(1): 1–4. |
| [] | 杨传平, 魏继承, 李同华, 等. 2006. 实时定量PCR法分析白桦中一花发育相关基因BpHEN的时序表达. 东北林业大学学报, 34(1): 1–6. |
| [] | 杨致荣, 毛雪, 杨致芬, 等. 2003. 细胞色素P450基因及其在植物改良中的应用. 遗传, 25(2): 237–240. |
| [] | Altschul S F, Madden T L, Schäffer A A, et al. 1997. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs. Nucleic Acids Res, 25(17): 3389–3402. DOI:10.1093/nar/25.17.3389 |
| [] | Bachem C W, Oomen R J F, Visser R G. 1998. Transript imaging with cDNA-AFLP: a step-by-step protocol. Plant Molecular Biology Reporter, 16(2): 157–173. DOI:10.1023/A:1007468801806 |
| [] | Capomaccio S, Barone P, Veronesi L R, et al. 2009. Isolation of genes from female sterile flowers in Medicago sativa. Sex Plant Reprod, 22(2): 97–107. DOI:10.1007/s00497-009-0095-2 |
| [] | Cnundde F, Moretti C, Porceddu A, et al. 2003. Transcript profiling on developing Petunia hybrida floral organs. Sex Plant Reprod, 16(2): 77–85. DOI:10.1007/s00497-003-0178-4 |
| [] | Elo A, Lemmetyinen J, Turunen M, et al. 2001. Three MADS-box genes similar to APETALA1. Physiologia Plantarum, 112(1): 95–103. DOI:10.1034/j.1399-3054.2001.1120113.x |
| [] | Heberle-Bors E, Voronin V, Touraev A, et al. 2001. MAP kinase signaling during pollen development. Sex Plant Reprod, 14(1/2): 15–19. |
| [] | Kim J, Kim S, Park H, et al. 2010. Array 2KEGG: Web-based tool of KEGG pathway analysis for gene expression profile. BioChip J, 4(2): 134–140. DOI:10.1007/s13206-010-4208-7 |
| [] | Kalluri U C, Joshi C P. 2004. Differential expression patterns of two cellulose synthase genes are associated with primary and secondary cell wall development in aspen trees. Planta, 220(1): 47–55. DOI:10.1007/s00425-004-1329-z |
| [] | Lannenpaa M, Janonen I, Holtta-Vuori M, et al. 2004. A new SBP box gene BpSPL1. Physiologia Plantarum, 120(3): 491–500. DOI:10.1111/ppl.2004.120.issue-3 |
| [] | Lemmetyinen J, Hassinen M, Elo A, et al. 2004. Functional characterization of SEPALLATA3. Physiologia Plantarum, 121(1): 149–162. DOI:10.1111/ppl.2004.121.issue-1 |
| [] | Picton S, Gray J E, Lowe A, et al. 1993. Sequence of a cloned tomato ubiquitin conjugating enzyme. Plant Physiology, 103(4): 1471–1472. DOI:10.1104/pp.103.4.1471 |
| [] | Shi C, Chaudhary S, Yu K, et al. 2011. Identification of candidate genes associated with CBB resistance in common bean HR45(Phaseolus vulgaris L) using cDNA-AFLP. Mol Biol Rep, 38(1): 75–81. DOI:10.1007/s11033-010-0079-1 |
| [] | Su V, Hsu B D. 2009. Transient expression of the cytochrome p450 CYP78A2 enhances anthocyanin production in flowers. Plant Mol Biol Rep, 28(2): 302–308. |
| [] | Wang Shan, Jiang Jing, Li Tianfang, et al. 2011. Influence of nitrogen, phosphorus and potassium fertilization on flowering and expression of flowering-associated genes in white birch(Betula platyphylla Suk.). Plant Mol Biol Rep, 29(4): 794–801. DOI:10.1007/s11105-010-0281-2 |
| [] | Wise H Z, Saxena I M, Brown R M. 2011. Isolation and characterization of the cellulose synthase genes PpCesA6. Cellulose, 18(2): 371–384. DOI:10.1007/s10570-010-9479-6 |
| [] | Zhang Lanlan, Chen Kunsong, Xu Changjie, et al. 2008. Identification and characterization of transcripts differentially expressed in peel and juice vesicles of immature and ripe orange(Citrus sinensis) fruit. Plant Mol Biol Rep, 26(2): 121–132. DOI:10.1007/s11105-008-0030-y |
| [] | Zhang Rongshu, Wang Yucheng, Wang Chao, et al. 2011. Time-course analysis of levels of indole-3-acetic acid and expression of auxin-responsive GH3 genes in Betula platyphylla. Plant Mol Biol Rep, 29(4): 898–905. DOI:10.1007/s11105-011-0306-5 |
| [] | Zhou Guo'an, Chang Ruzhen, Qiu Lijuan. 2010. Overexpression of soybean ubiquitin-conjugating enzyme gene GmUBC2. Plant Mol Biol, 72(4/5): 357–367. |
 2012, Vol. 48
2012, Vol. 48




