文章信息
- 马春雷, 姚明哲, 王新超, 金基强, 陈亮
- Ma Chunlei, Yao Mingzhe, Wang Xinchao, Jin Jiqiang, Chen Liang
- 茶树2个MYB转录因子基因的克隆及表达分析
- Cloning and Expression of Two MYB Transcription Factors in Tea Plant (Camellia sinensis)
- 林业科学, 2012, 48(3): 31-37.
- Scientia Silvae Sinicae, 2012, 48(3): 31-37.
-
文章历史
- 收稿日期:2011-07-04
- 修回日期:2011-11-07
-
作者相关文章
转录因子是一类通过特异的结合靶基因启动子区的顺式作用元件来调节目标基因表达的DNA结合蛋白,一般由DNA结合区、转录调控区、核定位信号区和寡聚化位点这4个功能区域组成。MYB蛋白是植物转录因子中最大的家族之一,根据其含有Myb结构域的数量,可将其分为3类:含有1个Myb结构域的R1蛋白,含有2个Myb结构域的R2R3蛋白以及含有3个Myb结构域的R1R2R3蛋白。植物中绝大多数MYB蛋白是R2R3型,由数量众多的基因家族编码(许志茹等,2008)。如TT2属于拟南芥(Arabidopsis thaliana)R2R3-MYB超家族133个成员中的一个,有2个内含子,外显子编码258个氨基酸,蛋白N端具有R2R3-MYB DNA结合域,特异地在发育种子的内种皮中调控结构基因DFR,LDOX,ANR,TT12等的表达(Nesi et al., 2001)。Deluc等(2006;2008)从葡萄(Vitis vinifera)中分离得到了VvMYB5a和VvMYB5b基因,它们主要在果皮、果肉和种子的发育早期表达,而由Jeong等(2006)克隆的VvmybA1基因则在浆果皮中特异表达,它们都可以通过其转录激活作用,提高葡萄皮中花青素生物合成相关基因的表达水平,进而增加葡萄皮中的花青素含量。
已有的研究表明,很多茶树(Camellia sinensis)品种在夏秋季节,受光照、温度等因素的影响,其幼嫩芽叶会出现不同程度的紫化现象,进一步的理化分析表明紫色芽叶往往含有较高的花青素含量(季鹏章等,2010;李双伶等,2009;萧力争等,2009)。花青素是一类具有多个酚羟基的黄酮类化合物,广泛存在于植物的花、茎、叶和果实中,是植物体内最重要的水溶性色素之一。以花青素含量较高的紫色芽叶为原料加工而成的绿茶,色泽偏黑,苦涩味较重,严重影响茶叶的品质。而将其加工成红茶后,除了外形和叶底稍次外,其他感官品质因子的表现要比绿色芽叶加工的红茶好(萧力争等,2008b)。同大多数高等植物代谢途径一样,花青素的合成主要受2类基因的控制,一类是结构基因,编码花青素生物合成的催化酶;另一类则是调节基因,编码的转录因子应答外界刺激,调控基因的时空表达。在茶树中,目前绝大多数参与花青素合成与转化的功能基因已得到分离克隆(马春雷等,2006;夏涛等,2009;Chen et al., 2005),部分酶活性研究也已展开,但相关调节基因的研究还鲜有报道。作者所在课题组在前期进行‘龙井’群体紫芽资源和绿芽资源芯片杂交分析时,筛选到一批可能与茶树的芽叶紫化现象相关的差异表达基因,其中上调表达最高的几个基因中包含了2个转录调控基因MYB转录因子和WD40转录因子,分别在紫芽资源中上调2.25倍和2.54倍(马春雷等,2011)。本研究根据筛选到的MYB转录因子的部分序列,采用RT-PCR和RACE技术从茶树中克隆了2个茶树转录因子基因CsMYB1和CsMYB2,并对其进行了全面的生物信息学分析,随后采用荧光定量PCR技术检测了2个转录因子在茶树不同组织中的表达特性,及遮荫处理条件下的表达规律,为研究它们在茶树花青素代谢途径中的作用奠定基础。
1 材料与方法 1.1 材料以茶树品种‘龙井43’和紫芽资源‘龙井紫芽’为试验材料,分别于茶树的不同发育阶段采集‘龙井43’的叶片、花、种子和萌发芽,幼根采自‘龙井43’的种子苗。用遮阳网搭建遮阳棚,于2011年5月1日起,将4盆扦插2年的‘龙井紫芽’置于其中,另外4盆对照置于其外,放入遮阳棚前,均轻修剪处理,3周后分别采摘长势一致的茶树新梢,液氮冷冻处理,-80 ℃冰箱中保存备用。
1.2 总RNA的提取及检测采用植物总RNA快速提取试剂盒RNAiso-mate for Plant Tissue (Takara),按说明书操作提取‘龙井紫芽’新梢和‘龙井43’的花、叶片及萌发芽的总RNA;参照史成颖等(2007)建立的改良CTAB法提取茶树种子和根的总RNA。用微量紫外检测仪NanoDrop测定提取的RNA样品浓度,1.2%变性琼脂糖凝胶电泳检测RNA质量。
1.3 CsMYB1和CsMYB2全长基因克隆由于茶树MYB类基因的命名还缺乏一个统一的规则,故这里将芯片杂交筛选到的茶树MYB基因暂命名为CsMYB1,同时搜索实验室前期构建的新梢文库的测序数据库,得到另一个MYB型转录因子基因的部分序列,将其命名为CsMYB2。利用NCBI上的Blastx软件对得到的2条EST片段进行序列同源比对,结果表明CsMYB1和CsMYB2的开放阅读框都不完整,CsMYB1缺少3′端和5′端,而CsMYB2缺少3′端。因此根据2个基因的已知片段,设计了6条嵌套的基因特异引物(表 1),并参照Clontech公司的Smart RACE试剂盒说明书,将提取的‘龙井紫芽’叶片总RNA反转录为cDNA,以稀释10倍的cDNA为模板进行PCR反应。反应体系为20 μL,其中10×PCR Buffer (Mg2+ plus) 2 μL,dNTPs (10 mmol·L-1 each) 0.4 μL,模板cDNA 0.4 μL,PCR特异引物和通用引物(10 mmol·L-1)各0.2 μL,Taq DNA聚合酶(5 U·μL-1) 0.2 μL,加水至终体积20 μL,反应在MJ-PTC220型PCR仪(MJ Research,Inc.,Waltham,MA,USA)上进行。反应条件为:94 ℃预变性3 min;94 ℃变性45 s,54 ℃复性45 s,72 ℃延伸45 s,35个循环;最后72 ℃再延伸5 min。然后将得到的PCR产物分别用蒸馏水稀释10倍,取0.4 μL作为模板,分别以CsMYB1-3RACE-2,CsMYB1-5RACE-2,CsMYB2-3RACE-2和通用引物进行第2轮PCR扩增。反应条件除退火温度升至60 ℃以外,其他均相同。扩增产物在1.2%琼脂糖凝胶上电泳检测,随后利用多功能DNA纯化回收试剂盒回收扩增产物,并连接至pTA2载体,转化大肠杆菌(Escherichia coli)TG1,筛选阳性克隆测序。
|
|
利用DNAstar软件进行序列的拼接;利用DNAtool软件分析基因的最大阅读框,并结合Blastp结果,推测全长基因编码的氨基酸序列,计算蛋白质的分子量大小及其理论等电点;利用NCBI网站的Blastx程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源序列比对,分析其保守区;使用Clustal W软件进行氨基酸序列的多重比对,再使用Mega 3.0软件构建系统进化树;通过SignalP 3.0 Server (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽及核定位信号信息。
1.5 茶树不同组织中2个转录因子的表达特性分析以茶树18S rRNA为内标基因,设计基因特异引物18S rRNA-A和18S rRNA-S;同时根据克隆得到的CsMYB1和CsMYB2全长序列设计荧光定量引物CsMYB1-RTS,CsMYB1-RTA,CsMYB2-RTS和CsMYB2-RTA(表 1)。按照PrimeScriptTM RT reagent kit (Takara)操作手册将提取的‘龙井43’根、叶、种子、花蕊和花瓣反转录成cDNA,稀释5倍做模板。荧光定量反应体系为:SYBR Premix Ex Taq 25 μL,上、下游引物(10 mmol·L-1)各2 μL,ROX Dye Ⅱ 1 μL,cDNA 4 μL,加水至终体积50 μL。反应在ABI PRISM 7500实时定量PCR仪上进行,程序为95 ℃变性30 s,94 ℃变性15 s,60 ℃退火延伸34 s,40个循环,于60 ℃收集荧光,反应结束后分析荧光值变化曲线和融解曲线,每个反应重复3次。
1.6 遮荫处理下花青素代谢相关基因表达分析大量研究表明,植物黄酮类化合物的代谢及其相关基因的表达与外界环境因素密切相关。因此,选取了一个龙井群体种中的自然突变资源——‘龙井紫芽’作为研究材料,正常情况下其新梢芽叶一年四季均呈紫色,对其进行遮荫处理3周后,收集遮荫处理和对照茶树的新生一芽二叶,采用盐酸比色法分别测定其花青素相对含量(萧力争等,2008a);同时采用荧光定量PCR技术检测2个转录因子在遮荫处理下的表达情况,试验采用相对定量的方法,以18S rRNA为内标基因,所用引物见表 1,具体过程参照PrimeScriptTM RT reagent kit(Takara)操作手册,每个反应重复3次,采用2-ΔΔCt 算法分析结果(Livak et al., 2001)。
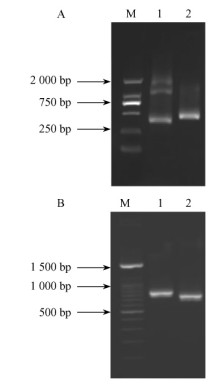
2 结果与分析 2.1 转录因子CsMYB1和CsMYB2全长序列的获得经过2轮PCR扩增后,CsMYB1的3′RACE和5′RACE电泳结果显示,分别在400 bp和500 bp处有2条较清晰的电泳带,同时在3′RACE的1 000 bp和2 000 bp处还存在2条弱带(图 1A);将所有4条片段分别纯化回收,并将其与pTA2载体连接,转入TG1感受态细胞中扩繁,然后利用菌液PCR的方法筛选阳性克隆测序,结果显示500 bp和400 bp的2条片段与原有的EST拼接在一起,组成了1条1 301 bp长的序列,而另外2条序列则为非特异扩增;用OFR软件对序列进行分析,最后确定其开放阅读框长879 bp,编码292个氨基酸,推测的蛋白分子量约为32.9 ku,理论等电点为8.13。CsMYB2进行3′RACE-PCR扩增后,电泳显示,其第1轮扩增和第2轮扩增结果分别为800 bp和700 bp的单一条带(图 1B);将700 bp片段纯化回收测序,并将测序结果与原EST拼接,得到了1条全长1 020 bp的cDNA序列,其中开放阅读框长675 bp,编码224个氨基酸,推测的蛋白分子量约为25.4 ku,理论等电点为9.05。2个基因的核苷酸序列及其推导的氨基酸序列如图 2所示,它们在GenBank的登录号分别为HQ660373和HQ660374。
|
图 1 RACE扩增产物的电泳结果 Fig.1 Electrophoresis results of RACE M:DNA标准分子量;A:1,2. CsMYB1的3′RACE和5′RACE电泳结果;B:1,2. CsMYB2的3′RACE电泳结果。 M: DNA marker; A: 1, 2: 3′RACE and 5′RACE amplification product of CsMYB1; B: 1, 2. 3′RACE amplification product of CsMYB2. |

|
图 2 茶树转录因子CsMYB1和CsMYB2的全长序列和推测氨基酸序列 Fig.2 Nucleotide and putative amino acid sequences of the CsMYB1 and CsMYB2 of tea plant 开放阅读框用下划线标出,myb结构域用阴影标出,保守氨基酸用斜体标出。 The open reading frame is underlined, myb domains are marked in shadow, and the conserved amino acids are denoted in italic type. |
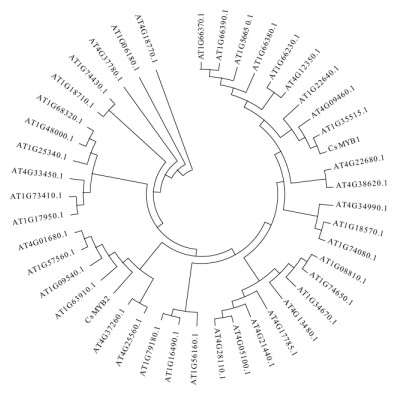
序列分析表明,CsMYB1和CsMYB2编码蛋白的N端都具有2个典型的MYB DNA结合域:R2(12aa—64aa)与R3 (65aa—115aa)。R2结构域自第17 aa开始,每间隔19个aa有1个保守的疏水氨基酸残基色氨酸(W,17aa,37aa,57aa);R3结构域中第1个W为苯丙氨酸(F,70aa)所取代,自苯丙氨酸起每间隔18个aa有1个W残基(89aa,108aa)。此外,CsMYB1和CsMYB2在R3结构域的下游都含有1个相对保守的C1(LIXXGIDPXTHR)基序,这也是转录因子激活结构域常见的特征,说明它们编码的蛋白是典型的R2R3-MYB转录因子家族成员。通过SignalP 3.0对2个转录因子进行信号肽预测,结果未发现推测蛋白含有信号肽,并预测它们都是非分泌型蛋白。利用Blastp程序对2条基因推导的氨基酸序列进行比对,结果显示2条序列间的氨基酸相似度为53%。利用Blastx对NCBI的蛋白数据库进行同源性搜索,发现与它们同源的序列全部为MYB蛋白,CsMYB1与陆地棉(Gossypium hirsutum)MYB1、蓖麻(Ricinus communis)MYB1和白花紫露草(Tradescantia fluminensis) MYB1的相似性分别为57%,55%,53%;CsMYB2与葡萄MYBC2、陆地棉MYB6和莽吉柿(Garcinia mangostana)MYB1的相似性分别为75%,64%,61%。应用Clustal W软件将2个茶树MYB转录因子与NCBI数据库下载的42个拟南芥R2R3-MYB蛋白进行多重比对(Thompson et al., 1994;Felsenstein,1985),再使用MEGA 3软件中的邻位相连法完成系统树的构建(Saitou et al., 1987;Kumar et al., 2004),分析结果(图 3)表明,CsMYB1与拟南芥的MYB8和AtMYB6关系最近,而CsMYB2与拟南芥中的AtMYB103关系最近,它们分别在拟南芥的逆境胁迫和细胞发育过程中起调控作用(Stracke et al., 2001)。
|
图 3 茶树转录因子CsMYB1和CsMYB2与拟南芥MYB基因家族系统树分析 Fig.3 Phylogenetic tree analysis of CsMYB1, CsMYB2 and 42 MYB proteins from Arabidopsis thaliana |
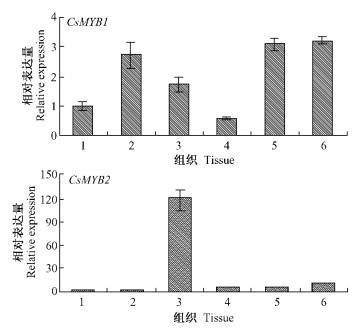
利用荧光定量PCR技术对CsMYB1和CsMYB2在不同组织中的表达模式进行分析。结果(图 4)显示:2个基因在茶树根、种子、叶、萌发芽、花瓣和花蕊中均有表达,应属组成型表达基因,但表达量各不相同;CsMYB1在花蕊、花瓣及种子中的表达相对较高,分别是根中表达量的3.2,3.0和2.7倍,而在萌发芽中表达最低;CsMYB2在不同组织中的表达差异较大,其在叶片中的相对表达量是根中的100多倍,即使同CsMYB2表达量相对较高的花蕊比较,其表达量也是它的十几倍,说明该基因可能主要在叶片中行使其表达调控功能。
|
图 4 CsMYB1和CsMYB2在茶树不同组织中的表达特性 Fig.4 Result of gene expression in different tissues of tea plant 1.幼根Young root; 2.种子Seed; 3.叶Leaf; 4.萌发芽Germination bud; 5.花瓣Petal; 6.花蕊Pistil. |
遮荫处理3周后,田间观测发现,遮荫处理茶树新生芽叶的紫化程度大大降低。花青素相对含量测定结果表明:遮荫处理的‘龙井紫芽’一芽二叶花青素含量为6.38花青素单位·g-1,而正常光照下的‘龙井紫芽’一芽二叶花青素含量为20.35花青素单位·g-1,遮荫处理使其相对花青素含量降低了3倍多。分别提取其总RNA进行荧光定量PCR分析,结果(图 5)显示,遮荫处理对CsMYB2的表达基本没有影响,但却明显增加了CsMYB1的表达量,说明茶树花青素含量的变化可能与CsMYB1的表达有一定的相关性。

|
图 5 遮荫处理下2个转录因子基因的表达变化 Fig.5 Expression analysis of CsMYB1 and CsMYB2 under treatments of shading |
高等植物的基因表达是一个精确调控的过程,转录水平的调节是很多基因表达调控的主要方式,转录因子作为植物中的调节基因,在这个阶段起重要的作用(房栋等,2008)。MYB蛋白是植物中最重要的转录因子之一,广泛参与植物的逆境胁迫、细胞分化、叶片形态建成以及次生代谢调控等生物过程(Martin et al., 1997)。其中R2R3-MYB型转录因子作为MYB蛋白最大的一类,主要参与植物花青苷的代谢调节(许志茹等,2008)。自Paz-Ares等(1987)从玉米(Zea mays)中发现第1个植物MYB基因C1以来,已有大量MYB蛋白基因在不同植物中得到分离克隆。但已有的研究表明,不同物种、不同个体,甚至同一个体的不同组织之间,MYB转录因子的表达特性和功能都存在较明显的差异,因此,克隆茶树MYB蛋白基因,并研究其功能,对于阐明茶树转录水平的代谢调控机制具有重要意义。Ban等(2007)从苹果(Malus×domestica)果皮中分离了MdMYBA基因,该基因表达具有组织和品种特性,将其转化苹果苗子叶的瞬时表达结果显示,子叶上出现微红色的点状物,同时检测到其转基因烟草(Nicotiana tabacum)中有花青素积累。Mano等(2007)从番薯(Ipomoea batatas)的地下器官中分离得到IbMYB1和IbMYB2s,其中IbMYB1特异地调节块根果肉CHS,CHI,F3H,DFR,ANS和3GT的表达和花青素的积累。本文通过对‘龙井紫芽’遮荫后新生芽叶中花青素含量及2个转录因子基因的表达变化进行分析,结果表明:遮荫处理能降低茶树新梢中的花青素含量,并改变芽叶颜色,同时CsMYB1的表达量也出现明显的增加,但它在此过程中是否起到了转录抑制作用则还有待进一步的验证。
目前已鉴定的绝大多数MYB蛋白都是在植物代谢调控中起转录激活作用,但除了具有正调控作用之外,花青素合成过程中还存在具有抑制作用的MYB蛋白。如Aharoni等(2001)从草莓(Fragaria)栽培变种中分离的FaMYB1基因在红色果实中特异表达,将其转入烟草后,FaMYB1的过量表达会抑制花和雄蕊中花青素的积累,这些组织中花青素合成途径后期的催化酶基因的表达量和催化酶含量也有所下降。另外基因表达调控方式也是多种多样的,近年来有关Myb类转录因子与其他转录因子家族成员尤其是bHLH类转录因子相互作用,通过组合调控的方式调节植物次生代谢,进而影响植物花器官、果实和叶片着色的研究,已成为育种工作者的研究热点。如在模式植物拟南芥中,TT8既可与MYB因子TT2结合,调控种皮色素合成,也可与MYB因子PAP1结合,调控多种器官中花青素苷的合成,靶基因为DFR和BAN (Nesi et al., 2000;Baudry et al., 2004;2006)。矮牵牛(Petunia hybrida)的AN1基因也编码bHLH蛋白,它与AN2和AN4编码的MYB蛋白具有复杂的作用方式,如叶中AN1的表达受AN2调控,花粉囊中AN1的表达受AN4调控,在AN1突变体花瓣中,花青素合成相关基因CHS,CHI和F3H正常表达,而DFR和ANS的表达降低(Quattrocchio et al., 2006)。作者所在课题组前期进行的芯片杂交结果中除了本研究分离克隆的CsMYB1基因外,还包含另一个转录因子WD40,它们是否通过某种作用方式共同调控茶树花青素的代谢还有待进一步的研究。另外序列同源性和基因表达模式分析虽被认为是推测基因功能的重要标准,但功能分析才能最终说明基因在植物发育过程中的作用,因此,将构建转录因子的表达载体,检测其转录调控活性及亚细胞定位情况,并进一步通过同源和异源转化对该基因的功能进行鉴定。
| [] | 房栋, 吕俊宏, 郭旺珍, 等. 2008. 一个新的棉花MYB类基因(GhTF1)的克隆及染色体定位分析. 作物学报, 34(2): 207–211. |
| [] | 季鹏章, 梁名志, 宋维希, 等. 2010. 茶树珍稀品种"紫娟"的叶片色素含量与叶色变化的关系研究. 西南农业学报, 23(6): 1860–1862. |
| [] | 李双伶, 郭俊凌, 杜晓. 2009. 茶树紫色芽叶中花青素的提取-层析分离及初步鉴定. 安徽农业科学, 37(28): 13799–13802. DOI:10.3969/j.issn.0517-6611.2009.28.138 |
| [] | 马春雷, 陈亮. 2006. 茶树功能基因分离克隆研究进展. 分子植物育种, 4(3S): 16–22. |
| [] | 马春雷, 姚明哲, 王新超, 等. 2011. 利用基因芯片筛选茶树芽叶紫化相关基因. 茶叶科学, 31(1): 59–65. |
| [] | 史成颖, 宛晓春, 江昌俊, 等. 2007. 提取高质量茶树总RNA的方法研究. 安徽农业大学学报, 34(3): 364–368. |
| [] | 夏涛, 高丽萍. 2009. 类黄酮及茶儿茶素生物合成途径及其调控研究进展. 中国农业科学, 42(8): 2899–2908. |
| [] | 萧力争, 李勤, 谭正初, 等. 2008a. 茶树紫色芽叶分级标准研究. 云南农业大学学报, 23(5): 668–672. |
| [] | 萧力争, 胡祥文, 龚志华, 等. 2008b. 紫色芽叶红茶适制性研究. 天然产物研究与开发, 20(3): 545–548. |
| [] | 萧力争, 苏晓倩, 李勤, 等. 2009. 紫芽品种茶树春梢芽叶生化成分分析. 福建农林大学学报:自然科学版, 38(1): 30–33. |
| [] | 许志茹, 李春雷, 崔国新, 等. 2008. 植物花青素合成中的MYB蛋白. 植物生理学通讯, 44(3): 597–604. |
| [] | Aharoni A, De Vos C H R, Wein M, et al. 2001. The strawberry FaMYB1 transcription factor suppresses anthocyanin and flavonol accumulation in transgenic tobacco. The Plant Journal, 28(3): 319–332. DOI:10.1046/j.1365-313X.2001.01154.x |
| [] | Ban Y, Honda C, Hatsuyama Y, et al. 2007. Isolation and functional analysis of a MYB transcription factor gene that is a key regulator for the development of red coloration in apple skin. Plant and Cell Physiology, 48(7): 958–970. DOI:10.1093/pcp/pcm066 |
| [] | Baudry A, Caboche M, Lepiniec L. 2006. TT8 controls its own expression in a feedback regulation involving TTG1 and homologous MYB and bHLH factors, allowing a strong and cell-specific accumulation of flavonoids in Arabidopsis thaliana. The Plant Journal, 46(5): 768–779. DOI:10.1111/tpj.2006.46.issue-5 |
| [] | Baudry A, Heim M A, Dubreucq B, et al. 2004. TT2, TT8, and TTG1 synergistically specify the expression of BANYULS and proanthocyanidin biosynthesis in Arabidopsis thaliana. The Plant Journal, 39(3): 366–380. DOI:10.1111/tpj.2004.39.issue-3 |
| [] | Chen L, Zhao L P, Gao Q K. 2005. Generation and analysis of expressed sequence tags from the tender shoot cDNA library of tea plant (Camellia sinensis). Plant Science, 168(2): 359–363. DOI:10.1016/j.plantsci.2004.08.009 |
| [] | Deluc L, Barrieu F, Marchive C, et al. 2006. Characterization of a grapevine R2R3-MYB transcription factor that regulates the phenylpropanoid pathway. Plant Physiology, 140(2): 499–511. DOI:10.1104/pp.105.067231 |
| [] | Deluc L, Bogs J, Walker A R, et al. 2008. The transcription factor VvMYB5b contributes to the regulation of anthocyanin and proanthocyanidin biosynthesis in developing grape berries. Plant Physiology, 147(4): 2041–2053. DOI:10.1104/pp.108.118919 |
| [] | Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution, 39(4): 783–791. DOI:10.1111/j.1558-5646.1985.tb00420.x |
| [] | Jeong S T, Goto-Yamamoto N, Hashizume K, et al. 2006. Expression of VvmybA1 gene and anthocyanin accumulation in various grape organs. American Journal of Enology and Viticulture, 57(4): 507–510. |
| [] | Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Briefings in Bioinformatics, 5(2): 150–163. DOI:10.1093/bib/5.2.150 |
| [] | Livak K J, Schmittgen T D. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method. Methods, 25(4): 402–408. DOI:10.1006/meth.2001.1262 |
| [] | Mano H, Ogasawara F, Sato K, et al. 2007. Isolation of a regulatory gene of anthocyanin biosynthesis in tuberous roots of purple-fleshed sweet potato. Plant Physiology, 143(3): 1252–1268. DOI:10.1104/pp.106.094425 |
| [] | Martin C, Paz-Ares J. 1997. MYB transcription factors in plants. Trends in Genetics, 13(2): 67–73. DOI:10.1016/S0168-9525(96)10049-4 |
| [] | Nesi N, Debeaujon I, Jond C, et al. 2000. The TT8 gene encodes a basic helix-loop-helix domain protein required for expression of DFR and BAN genes in Arabidopsis siliques. The Plant Cell, 12(10): 1863–1878. DOI:10.1105/tpc.12.10.1863 |
| [] | Nesi N, Jond C, Debeaujon I, et al. 2001. The Arabidopsis TT2 gene encodes an R2R3 MYB domain protein that acts as a key determinant for proanthocyanidin accumulation in developing seed. The Plant Cell, 13(9): 2099–2114. |
| [] | Paz-Ares J, Ghosal D, Wienand U, et al. 1987. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators. EMBO Journal, 6(12): 3553–3558. |
| [] | Quattrocchio F, Verweij W, Kroon A, et al. 2006. PH4 of Petunia is an R2R3 MYB protein that activates vacuolar acidification through interactions with basic-helix-loop-helix transcription factors of the anthocyanin pathway. The Plant Cell, 18(5): 1274–1291. DOI:10.1105/tpc.105.034041 |
| [] | Saitou N, Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular Biology and Evolution, 4(4): 406–425. |
| [] | Stracke R, Werber M, Weisshaar B. 2001. The R2R3-MYB gene family in Arabidopsis thaliana. Current Opinion in Plant Biology, 4(5): 447–456. DOI:10.1016/S1369-5266(00)00199-0 |
| [] | Thompson J D, Higgins D G, Gibson T J. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 22(22): 4673–4680. DOI:10.1093/nar/22.22.4673 |
 2012, Vol. 48
2012, Vol. 48


