文章信息
- 唐琴, 曾秀丽, 廖明安, 潘光堂, 扎西, 龚君华, 次仁卓嘎
- Tang Qin, Zeng Xiuli, Liao Ming'an, Pan Guangtang, Zha Xi, Gong Junhua, Ciren Zhuoga
- 大花黄牡丹遗传多样性的SRAP分析
- SRAP Analysis of Genetic Diversity of Paeonia ludlowii in Tibet
- 林业科学, 2012, 48(1): 70-76.
- Scientia Silvae Sinicae, 2012, 48(1): 70-76.
-
文章历史
- 收稿日期:2011-03-11
- 修回日期:2011-11-22
-
作者相关文章
2. 西藏自治区农牧科学院蔬菜研究所 拉萨 850032;
3. 四川农业大学玉米研究所 雅安 625014
2. Institute of Vegetables, Tibet Academy of Agricultural and Animal Husbandry Sciences Lahsa 850032;
3. Institute of Maize, Sichuan Agricultural University Ya'an 625014
大花黄牡丹(Paeonia ludlowii)是芍药科(Paeoniaceae)芍药属(Paeonia)牡丹组(Section Moutan)肉质花盘亚组(Subsection Delavayanae)植物(Hong, 1997), 为西藏特有的丛生灌木。大花黄牡丹天然分布于西藏林芝、米林等地, 生于高海拔(2 700 m以上)、半干旱半湿润环境, 对水分依赖性较大(邢震等, 2007), 常见于路边、半山腰、小溪边及河边等。大花黄牡丹染色体数目2n=10(李嘉珏等, 1998), 营专性种子繁殖(成仿云等, 1997), 虫媒传粉。野生大花黄牡丹株高可达3.5 m, 花大、色艳, 根含有丹皮酚, 是常用的藏药材, 因而具极高的观赏价值和经济价值; 野生牡丹中只有大花黄牡丹和黄牡丹(Paeonia delavayi var.lutea)拥有黄色花基因, 因此, 大花黄牡丹还是培育名、特、优黄色系、橙色系牡丹品种及高大切花品种的优良种质资源。更为重要的是, 大花黄牡丹属窄布种, 由于生境破坏和人为大量采掘, 分布区域日益缩小, 数量逐步减少, 处于濒危状态(汪松等, 2004)。
大花黄牡丹的遗传多样性及居群遗传结构研究对于保护其野生资源、选育新品种、丰富牡丹园艺品种的花色等有着重要意义。但大花黄牡丹的发现较晚, 且分布范围狭窄, 国内甘肃榆中和平牡丹园等少数单位成功进行了引种栽培(龚洵等, 2003)。受研究材料限制, 有关大花黄牡丹的分类地位(李嘉珏等, 1998; 洪德元等, 1999)、生态学(杨小林等, 2007; 杨翔等, 2010; 苏建荣等, 2010)、遗传学(林启冰等, 2004)及孢粉学(曾秀丽等, 2009)等已有一些报道, 但对其居群水平上遗传多样性的研究鲜有报道。
相关序列扩增多态性(sequence related amplified polymorphism, SRAP)是一种新的分子标记技术(Li et al., 2001), 分别由独特设计的17和18个碱基组成的上、下游引物优先对基因组DNA开放阅读框、内含子和启动子区域进行特异扩增, 因不同物种、不同个体启动子、内含子和间隔序列长度不同而产生多态性。该标记具有简便、高效、产率高、重复性好、易测序、便于克隆目标片段的特点, 目前已成功地应用于野牛草(Buchloe dactyloides)(Budak et al., 2004)、甜菜(Beta vulgaris)(Nagl et al., 2007)、牡丹(Paeonia suffruticosa)(Han et al., 2008a; 2008b)、芭蕉(Musa)(Youssef et al., 2011)等植物。本研究首次对西藏自治区林芝县和米林县的珍贵野生资源大花黄牡丹进行SRAP分析, 旨在探讨大花黄牡丹居群水平上DNA的遗传多样性, 为该种的新品种选育及保护策略等提供分子生物学基础。
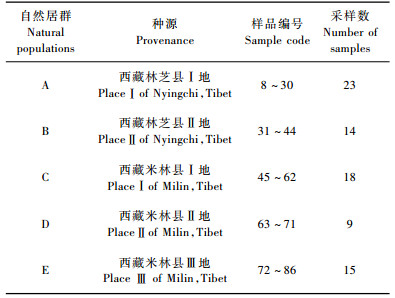
1 材料与方法 1.1 材料试验材料为野生大花黄牡丹未展开的幼叶, 于2009年5月采自西藏自治区林芝县和米林县。79个单株分别来自5个不同的自然居群(表 1), 居群间的距离均在50 km以上, 先对居群按生态和形态的多样性原则进行初步调查, 再根据居群大小选取9 ~ 23个单株, 分单株采集健康叶片(单株间距不少于5 m), 经硅胶快速干燥、密封后常温保存备用。为保护西藏濒危野生植物资源, 以字母A, B, C, D, E示各自然居群。
|
|
基因组DNA提取参照Doyle等(1987)的改良CTAB法, 用1.5%琼脂糖凝胶电泳检测其质量, 核酸蛋白分析仪检测纯度, DNA样品溶于100 μL的TE溶液中, - 20 ℃保存备用。
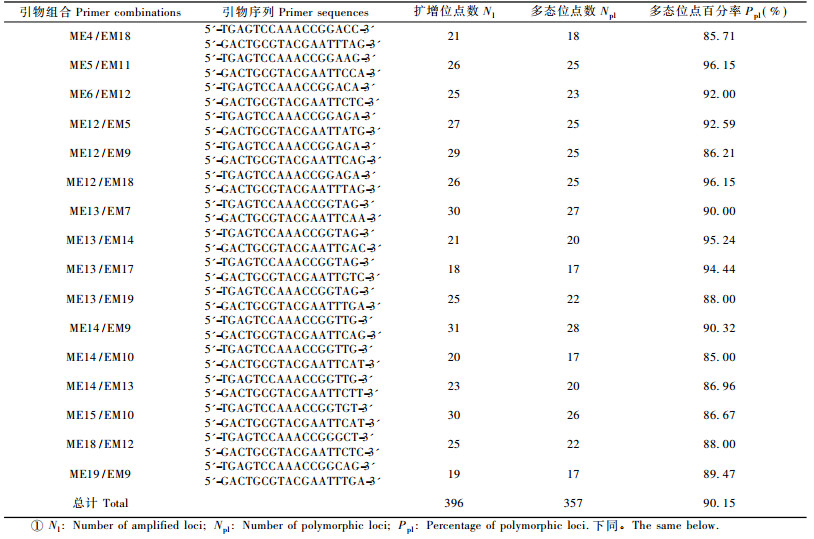
1.3 SRAP引物筛选由上海英俊公司合成的SRAP上、下游引物各20条组成400个引物组合, 从中筛选出条带清晰、稳定性和重复性好且条带较多的16对引物用于SRAP扩增(引物序列见表 2)。
|
|
用正交设计对PCR反应体系进行优化, 筛选出20 μL的反应体系为:模板DNA 30 ng, Mg2+ 3.0 mmol· L-1, dNTPs 0.4 mmol· L-1, 引物各0.5 μmol· L-1, Taq DNA聚合酶2 U, 并用15 μL石蜡油覆盖混合物。反应于PTC-200型PCR扩增仪(美国BIO-RAD公司)中进行, 扩增程序参照郝青等(2008):94 ℃预变性3 min; 94 ℃变性30 s, 37 ℃退火30 s, 72 ℃延伸l min 30 s, 退火温度每个循环增加1 ℃, 共8个循环; 94 ℃变性30 s, 50 ℃退火30 s, 72 ℃延伸l min 30 s, 32个循环; 72 ℃延伸7 min。扩增产物用6%变性聚丙烯酰胺凝胶电泳分离, 恒定功率75 W条件下预电泳30 min, 取4.5 μL产物上样, 电泳1.5 h, 采用银染法(王燕青等, 2009)进行显色反应, 统计、照相。每次扩增及电泳均设1个不加模板DNA的对照。
1.5 统计分析以100 bp DNA ladder(购自天根生化科技有限公司)为分子质量标准, 同一引物扩增的电泳迁移率一致的条带被认为有同源性, 属同一位点产物, 按有(1)、无(0)记录不同引物扩增结果, 构成SRAP “0,1”表型数据矩阵。用POPGENE1.32(Yeh et al., 1999)软件计算各居群多态位点百分率(Ppl)、Nei's遗传距离(D)、遗传分化系数(GST)。参照Goulart等(2005)的方法, 用Shannon表型多样性指数(Ηo)分析和计算居群内的平均遗传多样性(Ηpop)和总遗传多样性(Ηsp):Ηo =-ΣPilog2Pi/N, 式中, Pi为某位点的表型频率, N为位点数; Ηpop=1/nΣΗo, Ηsp=-ΣPlog2P/n, 式中, n为居群数, P为n个居群的综合表型频率; Ηpop/Ηsp是居群内多样性所占比例, (Ηsp-Ηpop)/Ηsp为居群间多样性所占比例。用DCFA1.1(张富民等, 2002)软件计算欧氏距离平方矩阵, 并将其作为输入文件用WINAMOVA (Excoffier et al., 1992)软件进行分子方差分析, 计算居群内和居群间的变异方差分布和Φ统计量距离(ΦST), 并用Mantel test对居群间的遗传距离和地理距离进行相关性检验。根据Nei's遗传距离, 对各居群进行UPGMA聚类分析; 用NTSYS-PC 2.1 (Rohlf, 2000)软件计算Jaccard相似系数, 对所有个体进行聚类分析。
2 结果与分析 2.1 SRAP-PCR扩增结果分析用筛选出的16个SRAP引物组合对79份大花黄牡丹样品进行DNA扩增, 共检测到396个清晰且可重复的有效位点, 其中多态性位点357个, 多态位点百分率为90.15%(表 2)。各对引物扩增的位点数为18 ~ 31个, 扩增片段大小在110 ~ 1 500 bp之间, 每个引物均扩增出多态片段, 多态百分率(85.00% ~ 96.15%)因引物而异。
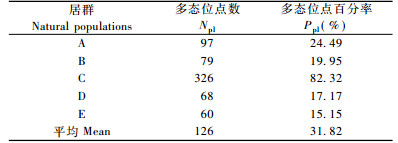
2.2 大花黄牡丹各居群的遗传多样性分析由表 3可知, 在居群水平上, 所有引物所扩增产物的多态性在不同居群中的分布数量不同。5个居群的多态位点数为60 ~ 326个, 居群C扩增出多态位点326个, 是其他居群多态位点数的3.4 ~ 5.4倍。不同居群的Ppl在15.15% ~ 82.32%之间, 其中居群C的Ppl最高, 居群D和E的较低, Ppl依次为自然居群C>A>B>D>E, 平均31.82%。以上结果表明, 5个居群中自然居群C的遗传多样性最高, 其余4个自然居群的Ppl较接近。
|
|
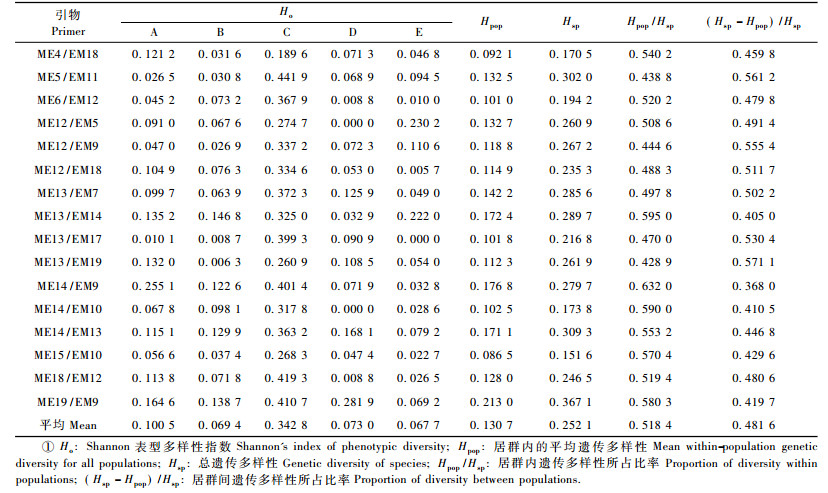
由Shannon表型多样性指数估测的大花黄牡丹各居群内的遗传多样性存在一定差异(表 4), 变幅为0.069 4 ~ 0.342 8, 平均值为0.130 7; 5个居群的遗传多样性顺序为居群C>A>D>B>E, 这与各居群的Ppl大小顺序基本一致。不同引物检测到的遗传多样性大小也有差异, 其中引物对ME19/EM9检测的遗传多样性最高, 平均0.213 0, 而引物对ME15/EM10检测的最低, 平均0.086 5。
|
|
从表 4还可看出, 大花黄牡丹居群内和居群间的遗传多样性在引物间变动较大, 说明各引物扩增出的不同多态位点对遗传多样性的贡献是不同的。各居群内的遗传多样性(Ηpop)平均为0.130 7, 总的遗传多样性(Ηsp)平均为0.252 1, 居群间的遗传变异占总的遗传变异的48.16%, 居群内的遗传变异仅占51.84%。可见, 大花黄牡丹居群间存在较高水平的遗传分化。
2.3 大花黄牡丹居群的遗传结构AMOVA分析显示总的遗传变异中有41.58%存在于居群间, 有58.42%存在于居群内(表 5), ΦST值为0.415 8(P<0.001), 表明大花黄牡丹居群间的遗传变异对总的遗传变异贡献较大, 居群分化较显著。对于相同的SRAP数据, 由POPGENE计算的居群间的基因分化系数GST值为0.430 9, 通过Shannon表型多样性指数求出的居群间遗传多样性所占比例[(Ηsp-Ηpop)/Ηsp]为48.16%, 也显示了居群间分化水平较高。
|
|
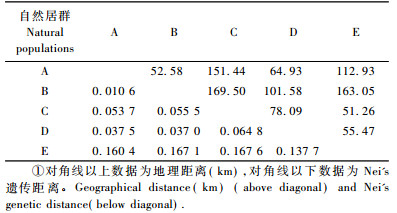
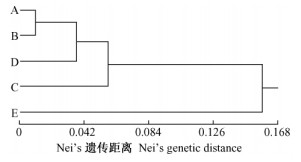
大花黄牡丹各居群间Nei's遗传距离的变化范围为0.010 6 ~ 0.167 6, 以居群C和E之间的遗传距离为最大, 居群A和B间的遗传距离最小(表 6)。Mantel检验结果表明大花黄牡丹居群间的遗传距离和地理距离之间相关不显著(r=0.597 5, P=0.905 5)。根据Nei's遗传距离对各居群进行UPGMA聚类可知(图 1), 同属西藏林芝县的居群A和B聚在一起, 而同属米林县的3个居群不能聚在一起, 尤其是居群E与其他4个居群之间的遗传距离显著高于其他4个居群间的遗传距离。
|
|
|
图 1 大花黄牡丹居群间Nei's遗传距离的UPGMA聚类 Figure 1 UPGMA dendrogram of Paeonia ludlowii based on Nei's genetic distance |
为了解大花黄牡丹居群内个体的变异幅度, 对所有的单株进行了UPGMA聚类(图 2)。聚类结果表明:所有个体的遗传相似系数变幅在0.47 ~ 0.99, 同一自然居群的个体并不能完全聚在一起。大部分来源于同一居群的个体表现出较为密切的亲缘关系, 如A, B, D, E 4个居群内的个体间相似系数均较高; 但也有一些居群的个体未能聚在一起, 如遗传相似系数为0.60时, C居群的5个单株(样品48, 51, 58, 59, 61)聚为一支, 该居群的其余个体与其他居群的个体聚为一支。
一般而言, 分布范围狭窄的特有植物种的多态性水平比广布种低(王艇等, 2006), 但近来不少研究发现某些特有和濒危物种仍保持较高的变异水平, 如中国鹅掌楸(Liriodendron chinense)(刘丹等, 2006)等。本研究中大花黄牡丹种水平上的多态位点百分率高达90%以上, 高于同一亚组的滇牡丹(Paeonia delavayi)(79.31%)(杨淑达等, 2005), 表明大花黄牡丹具有较高水平的遗传多样性。在所研究的5个居群中, 居群C的遗传多样性最高(Ppl = 82.32%, Shannon表型多样性指数估测的多样性为0.342 8), 野外调查发现该居群处于保存较完好的原有生境条件下, 可能因此维持了丰富的遗传多样性(邢震等, 2007); 其他居群因天然分布于林缘坡麓次生灌木林或河谷台地, 人为、牲畜活动频繁, 生境屡遭破坏, 加之对其过度挖掘, 使个体数锐减, 导致遗传多样性减少。
3.2 大花黄牡丹居群的遗传分化Meekins等(2001)曾报道过Shannon表型多样性指数对显性标记不能直接测度杂合体导致的偏差不敏感。在本研究中, 应用AMOVA分析得出的ΦST(0.415 8)值与GST值(0.430 9)接近, 二者略小于通过Shannon表型多样性指数计算得出的居群间遗传变异(0.481 6), 但它们均揭示了大花黄牡丹居群间存在较高水平的遗传分化, 这为居群间选优提供了可能。居群遗传结构往往受很多因素的影响, 包括生境片断化、基因流、繁育系统、种子散播机制等(Ge et al., 2005; Fischer et al., 1998)。本研究中, 导致大花黄牡丹居群间遗传分化较大的原因可能与种子、花粉的传播有关。大花黄牡丹营专性种子繁殖, 种子较大, 成熟时直径可达1.3 cm(Hong et al., 2001), 靠重力传播, 而且属虫媒传粉, 其花粉散布与传粉昆虫的活动范围和生活习性有关, 这些因素可能限制了居群间的基因流。一些居群个体较少, 因生境片段化严重, 易发生遗传漂变, 从而导致居群内遗传变异降低, 居群间遗传分化加剧, 如居群E可能发生了遗传漂变, 居群聚类图上它与其他4个居群之间的遗传距离显著高于其他4个居群间的遗传距离。
生境的不均一性往往是居群内个体差异或居群差异的直接原因(陈家宽, 1994)。西藏高原隆起后, 由于高原上升运动的差异性和第四纪以来的冰期与间冰期气候之交替影响, 形成了高原十分独特的复杂的自然环境(陈万勇, 1980)。个体聚类图上大多数居群内个体表现出较为密切的亲缘关系如居群B, D, E; 但也有一些居群的个体未能聚在一起, 如C居群, 且该居群的部分样品(编号48, 51, 58, 59, 61)在每对引物对其进行DNA扩增时均能产生多个特异性条带, 这从DNA水平上反映了这些个体与居群内其他个体间存在较大的差异。值得注意的是, 与其他居群相比, 居群C的分布地离冰川融水较近, 因此造成C居群内个体间差异及C居群与其他居群间差异的因素可能与水因子有关, 但这需要进一步的研究。
3.3 保护与利用策略作者通过2年的大规模野外调查, 仅发现了5个较大的大花黄牡丹居群[后来有文献报道山南地区亦分布有1个居群(倪圣武, 2009)], 这5个居群分布范围相当狭窄, 大部分居群分布于人类活动频繁的地方, 而且从植株叶形、叶色、花色、花瓣数目、雄蕊、雌蕊、种子等表型来看均变化不大, 本研究应用SRAP标记还揭示出了居群间存在较大的遗传分化, 这些说明其野生种群面临着濒危的巨大风险, 因此, 迫切需要采取综合有效的措施保护大花黄牡丹的遗传多样性。其一, 在分布地建立保护区, 对遗传多样性高的居群进行就地保护, 如本研究中遗传多样性较高的自然居群C; 其二, 要重视种质资源的收集, 建立种质资源库进行迁地保护, 针对大花黄牡丹的遗传变异特点, 尽量增加收集的居群数量和居群内的家系数, 保证迁地保护的有效性; 其三, 通过实生苗培育或组织培养途径将基因频率较小的个体进行扩繁后回归自然, 可能是保护大花黄牡丹种质资源的有效途径。以观赏为目标开展大花黄牡丹的定向培育, 探讨其在园林中的应用, 亦将对其保护起到重要的推动作用, 大花黄牡丹丰富的遗传多样性为品种选育提供了种质保证。
成仿云, 李嘉珏, 陈德忠. 1997. 中国野生牡丹自然繁殖特性研究[J]. 园艺学报, 24(2): 180-184. |
陈家宽. 1994. 居群概念和方法在植物分类学中的应用∥陈家宽, 杨继.植物进化生物学[M]. 武汉: 武汉大学出版社: 365-374.
|
陈万勇. 1980. 西藏林芝盆地新生代晚期的自然环境[J]. 古脊椎动物与古人类, 18(1): 52-58. |
龚洵, 潘跃芝, 杨志云. 2003. 滇牡丹的多样性和现状评估[J]. 西北植物学报, 23(2): 218-223. |
郝青, 刘政安, 舒庆艳, 等. 2008. 中国首例芍药牡丹远缘杂交种的发现及鉴定[J]. 园艺学报, 35(6): 853-858. |
洪德元, 潘开玉. 1999. 芍药属牡丹组的分类历史和分类处理[J]. 植物分类学报, 37(4): 351-368. |
李嘉珏, 陈德忠, 于玲, 等. 1998. 大花黄牡丹分类学地位的研究[J]. 植物研究, 18(4): 152-155. |
林启冰, 周志钦, 赵宣, 等. 2004. 基于Adh基因家族序列的牡丹组(Sect.Moutan DC.)种间关系[J]. 园艺学报, 31(5): 627-632. |
刘丹, 顾万春, 杨传平. 2006. 中国鹅掌楸遗传多样性研究[J]. 林业科学, 41(1): 57-64. |
倪圣武. 2009. 紫牡丹、黄牡丹、大花黄牡丹引种与迁地保护研究. 北京林业大学硕士学位论文. http://cdmd.cnki.com.cn/Article/CDMD-10022-2009161406.htm
|
苏建荣, 刘万德, 郎学东, 等. 2010. 濒危植物大花黄牡丹与生境地群落特征的关系[J]. 林业科学研究, 23(4): 487-492. |
汪松, 解炎. 2004. 中国物种红色名录[M]. 北京: 高等教育出版社.
|
王艇, 苏应娟, 欧阳蒲月, 等. 2006. 利用RAPD标记分析濒危植物白豆杉种群的遗传结构[J]. 生态学报, 26(7): 2313-2321. |
王燕青, 季孔庶. 2009. 利用正交设计优化牡丹SRAP-PCR反应体系[J]. 分子植物育种, 7(1): 199-203. |
邢震, 张启翔, 次仁. 2007. 西藏大花黄牡丹生境概况初步调查[J]. 江苏农业科学, (4): 250-253. |
杨小林, 王秋菊, 兰小中, 等. 2007. 濒危植物大花黄牡丹(Paeonia ludlowii)种群数量动态[J]. 生态学报, 27(3): 1242-1247. |
杨淑达, 施苏华, 龚洵, 等. 2005. 滇牡丹遗传多样性的ISSR分析[J]. 生物多样性, 13(2): 105-111. |
杨翔, 卢杰. 2010. 大花黄牡丹群落主要种群的生态位研究[J]. 江苏农业科学, (1): 314-318. |
张富民, 葛颂. 2002. 群体遗传学研究中的数据处理方法Ⅰ[J]. RAPD数据的AMOVA分析.生物多样性, 10(4): 438-444. |
曾秀丽, 代安国, 李青, 等. 2009. 部分牡丹花粉粒超微结构的研究初报. 四川农业大学学报, 27(4): 466-470. http://www.cqvip.com/qk/91109X/200904/32547064.html
|
Budak H, Shearman R C, Parmaksiz I. 2004. Molecular characterization of buffalograss germplasm using sequence-related amplified polymorphism markers[J]. Theoretical and Applied Genetics, 108(2): 328-334. DOI:10.1007/s00122-003-1428-4 |
Doyle J J, Doyle J L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochemical Bulletin, 19(1): 11-15. |
Excoffier L, Smouse P E, Quattro J M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J]. Genetics, 131(2): 479-491. |
Fischer M, Matthies D. 1998. RAPD variation in relation to population size and plant fitness in the rare Gentianella germanica(Gentianaceae)[J]. American Journal of Botany, 85(6): 811-819. DOI:10.2307/2446416 |
Ge Xuejun, Zhang Linbing, Yuan Yongmin, et al. 2005. Strong genetic differentiation of the East-Himalayan Megacodon stylophorus(Gentianaceae) detected by inter-simple sequence repeats(ISSR)[J]. Biodiversity and Conservation, 14(4): 849-861. DOI:10.1007/s10531-004-0655-6 |
Goulart M F, Ribeiro S P, Lovato M B. 2005. Genetic, morphological and spatial characterization of two populations of Mabea fistulifera Mart(Euphorbiaceae), in different successional stages[J]. Brazilian Archives of Biology and Technology, 48(2): 275-284. DOI:10.1590/S1516-89132005000200015 |
Han Xiaoyan, Wang Liangsheng, Liu Zheng'an, et al. 2008a. Characterization of sequence-related amplified polymorphism markers analysis of tree peony bud sports[J]. Scientia Horticulturae, 115(3): 261-267. DOI:10.1016/j.scienta.2007.09.003 |
Han Xiaoyan, Wang Liangsheng, Shu Qingyan, et al. 2008b. Molecular characterization of tree peony germplasm using Sequence-Related Amplified Polymorphism markers[J]. Biochemical Genetics, 46(3/4): 162-179. |
Hong Deyuan. 1997. Paeonia(Paeoniaceae) in Xizang(Tibet)[J]. Novon, 7(2): 156-161. DOI:10.2307/3392188 |
Hong Deyuan, Pan Kaiyu, Nicholas J T. 2001. Paeoniaceae∥Wu Z Y, Raven P H.Flora of China:Vol.6[M]. Beijing: Science Press and Missouri Botanic Garden Press.
|
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 103(2/3): 455-461. |
Meekins J F, Ballard H E, Mccarthy B C. 2001. Genetic variation and molecular biogeography of a North American invasive plant species(Alliaria petiolata, Brassicaceae)[J]. International Journal of PlantSciences, 162(1): 161-169. |
Nagl N, Weiland J, Lewellen R. 2007. Detection of DNA polymorphism in sugar beet bulks by SRAP and RAPD markers[J]. Journal of Biotechnology, 131(2). |
Rohlf F J. 2000. NTSYS-pc:Numerical taxonomy and multivariate analysis system, version 2.1[M]. Exeter Software: Setauket, New York.
|
Yeh F C, Yang R C, Boyle T.1999.POPGENE version 1.32, microsoft window-based freeware for population genetic analysis.Edmonton, Canada:University of Alberta. https://sites.ualberta.ca/~fyeh/popgene.pdf
|
Youssef M, James A C, Rivera-Madrid R, et al. 2011. Musa genetic diversity revealed by SRAP and AFLP[J]. Molecular Biotechnology, 47(3): 189-199. DOI:10.1007/s12033-010-9328-8 |
 2012, Vol. 48
2012, Vol. 48