文章信息
- 王旭, 杨少辉, 王洁华
- Wang Xu, Yang Shaohui, Wang Jiehua
- CLE多肽激素信号转导在植物维管系统发育分化中的作用
- The Function of CLE Peptide Hormone-Mediated Signaling Transduction in the Development and Differentiation of Plant Vascular System
- 林业科学, 2011, 47(11): 160-165.
- Scientia Silvae Sinicae, 2011, 47(11): 160-165.
-
文章历史
- 收稿日期:2011-03-12
- 修回日期:2011-04-28
-
作者相关文章
分生组织(meristem)是位于植物体的一定部位、具有持续或周期性分裂能力的细胞群,常见于植物体生长的幼嫩部位。在一个成熟的植物胚胎上,胚根和胚芽顶端的原分生组织是植株形成中各种组织和器官的主要来源。原分生组织通过细胞分裂和分化,形成由原表皮层、基本分生组织和原形成层组成的初生分生组织。原形成层分化初生维管组织后,分生组织细胞继续保留在初生木质部和初生韧皮部之间,与由基本薄壁组织恢复分生能力后形成的细胞层共同构成维管形成层,并在生长季节进行活跃的细胞分裂,向内和向外分别形成次生木质部和次生韧皮部,使根和茎不断加粗生长(Scheres, 2007)。维管形成层属侧生分生组织,与之对应,植物的顶端分生组织(apical meristem)位于维管植物茎和根的顶端,分别称为茎端分生组织(shoot apical meristem, SAM)和根端分生组织(root apical meristem, RAM)。
多肽激素是1991年首次在番茄(Lycopersicon esculentum)中被报道的激素性多肽蛋白质,是植物体内天然存在的一类生理活性物质(Pearce et al., 1991)。多肽激素不但作为信号分子,通过细胞与细胞之间的短距离信息交流,参与植物生长、发育及抗逆等许多生命过程,而且对于维持多种分生组织中干细胞分裂与分化之间的平衡起着关键的作用。对于维管组织而言,多肽激素介导的胞间通讯与交流不但决定了它特殊的放射状结构,也是维管组织行使复杂生理功能的分子基础(Hirakawa et al., 2010a;2010b)。本文主要介绍多肽激素尤其是CLE(CLAVATA3/Embryo surrounding region-related)多肽及其受体激酶如何通过细胞间的信号转导,对维管分生组织的发育及形成层的分化进行调节。
1 CLE多肽激素的结构及其分类CLE基因在拟南芥(Arabidopsis thaliana)、棉花(Gossypium spp.)、番茄、大豆(Glycine max)和紫花苜蓿(Medicago sativa)等双子叶植物以及水稻(Oryza sativa)、玉米(Zea mays)、小麦(Triticum aestivum)等单子叶植物中均有表达(Cock et al., 2001)。CLE基因具有不同的组织特异性表达方式,例如,CLV3在SAM中心区域的原套层表达;CLE19和CLE40在根部特异表达;而CLE2和CLE16则在多个组织中均有表达。另外,一些CLE基因(如CLE10, CLE26, CLE27)的转录水平在体外细胞进行木质化的过程中会有所提高(Kubo et al., 2005)。
CLE基因编码的蛋白具有3个共同特点: 1) 蛋白分子量小( < 10 ku);2) N末端带有分泌信号肽;3) C末端或其附近含有1个保守的CLE基序(CLE motif)。域交换(domain swap)实验及缺失分析发现,CLV3的CLE域可以不依赖于相邻的侧翼序列而发挥作用(Fiers et al., 2005)。在CaMV35S: :CLV3转基因愈伤组织中,利用飞行时间质谱仪(MALDI-TOF MS)所获得的活性最高的CLV3多肽是由含有2个羟脯氨酸的13个氨基酸组成的,其中1个羟脯氨酸含有3个阿拉伯糖残基修饰(Ohyama et al., 2009)。
拟南芥和水稻的基因组中分别含有大概83和47个CLE基因(Etchells et al., 2010;Jun et al., 2008;Kinoshita et al., 2007)。按照CLE多肽生理作用的不同,可将其分为4类: 1) CLV3等能够抑制SAM和RAM中干细胞增殖分裂的CLE多肽统称为A型(Kinoshita et al., 2007;Kondo et al., 2006);2) 与A型相比,CLE25和CLE26虽然也可以抑制SAM和RAM中干细胞的分裂,但对根初生木质部中导管细胞的分化缺乏抑制作用,统称为B型(Kondo et al., 2011);3) 编码导管分化抑制因子TDIF (tracheary element differentiation inhibitory factor)的CLE41/CLE44多肽,以及同样具有TDIF活性但氨基酸序列相差1个的CLE42多肽统称为C型(Ito et al., 2006);4) 尚未发现对SAM及维管发育具有明显作用的CLE多肽统称为D型(Hirakawa et al., 2011)。其中,A型CLE多肽有可能是通过特异性地降低ARR5和ARR6基因的表达来激活细胞分裂素的信号传导,从而导致根组织中的初生木质部导管无法形成(Kiba et al., 2003;To et al., 2004)。CLV2受体与CRN/SOL2激酶可能作为信号传导的下游组件共同参与这一途径(Kondo et al., 2011)。
2 导管分化抑制因子TDIF及其生理功能百日草(Zinnia elegans)细胞培养系统是诱导和分析细胞木质化的有效体系,目前已被用来鉴定与木质部分化相关的关键基因、蛋白以及信号分子等。通过在百日草细胞的培养基中添加生长素和细胞分裂素,能够使游离的叶肉细胞分化为导管分子TE(tracheary element)。2006年,日本研究人员发现,一种含有12个氨基酸的短肽可以抑制TE的分化,并将其命名为导管分子分化的抑制因子TDIF (tracheary element differentiation inhibitory factors)(Ito et al., 2006)。TDIF的氨基酸序列为HEVHypSGHypNPISN,与拟南芥CLE41和CLE44多肽的C-末端12个氨基酸序列相同,并与CLE42和CLE46的C-末端序列具有高度的同源性。
将TDIF添加到浸没培养的拟南芥幼苗中,可以造成拟南芥叶脉中木质部导管束的中断,但不会破坏韧皮部和原形成层束的结构(Hirakawa et al., 2008),这表明TDIF能够特异性地抑制原形成层细胞分化为导管分子。过量表达CLE41和CLE44的转基因拟南芥不但具有上述的不连续导管束,同时由于原形成层细胞的过度分裂,其维管区域明显增大,这种现象尤其在下胚轴更加显著(Hirakawa et al., 2008)。因此,在维管组织的发育分化过程中,TDIF具有2个重要的作用:抑制形成层细胞向木质部细胞的分化;促进原形成层细胞的分裂(Hirakawa et al., 2008)。
拟南芥基因组编码至少26种CLE多肽(Ito et al., 2006;Jun et al., 2008)。通过对人工化学合成的、由12个氨基酸组成的各种CLE多肽进行活性分析发现,除CLE42和CLE41/44多肽具有较强的TDIF活性外,其他的CLE十二肽均无此活性(Ito et al., 2006)。TDIF属于C型CLE多肽,它能够抑制形成层细胞的分化,而促进其分裂;而上文提及的CLV3,则属于A型CLE多肽,它的作用是促进形成层细胞向木质部细胞分化,这表明在维管组织发育过程中,CLE多肽具有生物学功能的多样性。
3 识别CLE肽的类受体激酶以及TDIF的受体TDR/PXY植物类受体蛋白激酶(receptor like protein kinases, RLKs)是一类定位在质膜上,具有胞外结构域、跨膜结构域和胞内激酶域的蛋白质。RLKs通过与配体的特异性结合来激活胞内激酶域的自磷酸化和互磷酸化活性,将胞外信号转换为胞质信号,连接动态的转录和激素调节网络,从而影响植物的生长和发育。富含亮氨酸重复序列(LRRs)的类受体蛋白激酶(LRR-RLKs),是植物RLKs中最大的一类。拟南芥的基因组中含有200多个、分为13个亚族的LRR-RLKs,占全部RLKs的一半以上(Shiu et al., 2004)。目前,已经陆续发现了一些LRR-RLK基因在分生组织发育、干细胞伸长、授粉作用、抵御疾病、共生、器官的形成和解离、胞囊胚的形成等方面具有功能,但大多数LRR-RLKs及其配体的功能仍是未知的(Morillo et al., 2006),也只有大概50个LRR-RLKs基因在数据库中有一定的功能注释(Shiu et al., 2004)。
目前在植物分生组织中研究最为深入的LRR-RLK是CLV1蛋白。在细胞内,CLV1和CLV2(一个与CLV1类似的富含亮氨酸重复顺序类受体蛋白)通过共价二硫键连接形成一个180 ku的、位于细胞膜表面受体的异二聚体(Trotochaud et al., 1999)。CLV1-CLV2受体复合物继而同CLV3多肽激素发生识别与作用,将信号传递到邻近的细胞,最终抑制WUS基因的表达。由于WUS可以增强CLV3的表达水平(Brand et al., 2000;Haecker et al., 2001),因此在CLV复合体与WUS之间形成了一个非常精确的反馈调节环,控制着茎端分生组织中干细胞的数目(Brand et al., 2000;Schoof et al., 2000)。WUS负责维持干细胞的未分化状态,增加茎生长点中干细胞的数目;而CLV1/CLV2/CLV3复合体则负责产生一个促进干细胞分化的信号,从而限制茎端生长点中干细胞的数目(Clark et al., 1997;Fletcher et al., 1999;Jeong et al., 1999;Schoof et al., 2000)。
利用AtGenExpress公共数据库及对百日草的分化细胞(Demura et al., 2002)和拟南芥的木质化细胞(Kubo et al., 2005)进行基因表达谱分析发现: LRR-RLK XI亚族的一些成员与维管系统的发育密切相关。利用TDIF可以抑制原形成层细胞分化为木质部导管这一特点,研究人员筛选到对TDIF作用不敏感的突变体,从而分离到TDIF受体基因TDR(TDIF receptor)。生化分析表明TDIF多肽可与TDR的胞外结构域直接进行特异性的结合(Hirakawa et al., 2008)。由于对内源TDIF敏感性的缺失,tdr突变体中的原形成层细胞分裂次数减少,胚轴中的某些木质部导管直接与韧皮部相连,失去了本应位于两者之间的原形成层细胞。TDR在另一正向遗传学研究中又被命名为PXY(phloem intercalated with xylem),同另外3个具有序列相似性的RLKs(PXL1,PXY-LIKE1和PXL2)共同维持着维管组织内细胞的极性排列,并对形成层细胞的定向分裂具有促进作用(Fisher et al., 2007)。
4 TDIF/TDR信号识别在维管系统发育中的作用机制TDIF与CLV3同属CLE多肽,而TDR/PXY与CLV1也都属于LRR-RLK XI亚族,这暗示着可能有一个类似于SAM中的CLV1/CLV3系统在维管组织中决定着形成层细胞的命运。WOX4基因属于WUS-相关的同源框(WOX)基因家族,位于TDIF-TDR信号途径的下游(Hirakawa et al., 2010b)。在拟南芥中通过RNAi方法降低WOX4的表达会对形成层细胞的分裂产生强烈的抑制作用,同时延缓和减少韧皮部标记基因APL1 (ALTERED PHLOEM1)以及形成层和初生木质部的标记基因ATHB8的表达,这表明WOX4具有促进形成层细胞分裂的作用(Ji et al., 2010)。但与此同时,WOX4却不能够对形成层细胞的分化产生抑制作用。在tdr突变体中,靠近韧皮部的原形成层细胞会成为木质部细胞(Hirakawa et al., 2008);而在wox4突变体中,虽然原形成层细胞的数目有所减少,但是木质部和韧皮部之间至少保留有1层原形成层细胞(Hirakawa et al., 2010b)。由此证明,WOX4只负责TDIF-TDR途径中的一条信号通路,即只调节原形成层细胞的分裂(图 1);TDIF-TDR途径中负责抑制维管干细胞向木质部细胞分化的信号通路其下游元件目前仍未知。

|
图 1 维管组织中TDIF-TDR信号通路介导形成层细胞的分化 Figure 1 TDIF-TDR signaling pathway in vascular system mediating the differentiation of meristem cells 参见Refer to: Hirakawa et al., 2010a;2011. |
TDIF-TDR-WOX4信号途径与CLV3-CLV1-WUS途径具有明显的不同之处: 1)CLV3通过抑制WUS的表达来限制干细胞数目,而TDIF是促进WOX4的表达,从而增强干细胞的分裂。clv1突变体中分生组织细胞加强分裂,生长点变大(Clark et al., 1993;De Smet et al., 2008),而tdr/pxy和cle41突变体中则是形成层细胞分裂减少(Hirakawa et al., 2010a;2010b;Hirakawa et al., 2008);2)WUS采用非细胞自主性作用机制(Mayer et al., 1998),而WOX4则是细胞自主性机制(Hirakawa et al., 2010b);3)WUS通过反馈调节环来影响CLV3的表达(Brand et al., 2000;Schoof et al., 2000),而WOX4并不对CLE41/CLE44的表达产生影响(Hirakawa et al., 2010b)。
由于TDR主要在原形成层/形成层位置表达(Fisher et al., 2007;Hirakawa et al., 2008),而CLE41和CLE44特定地在韧皮部及其附近区域表达(Hirakawa et al., 2008),因此Hirakawa等(2010a)提出CLE41/44/TDIF可能在从韧皮部到原形成层再到木质部的区域内形成一个浓度梯度,以此决定原形成层细胞的分裂和分化(图 1)。在韧皮部附近,原形成层细胞接受高浓度的TDIF,进行分裂却不分化;而在离韧皮部较远的位置,由于TDIF的浓度较低,原形成层细胞得以分化为木质部细胞。如果CLE41在木质部特异性IRX3启动子或者35S启动子的控制下进行异位表达,形成层细胞的分裂方向以及维管系统中木质部与韧皮部的极性分布都将被破坏;而如果CLE41是在韧皮部特异性启动子SUC2的控制下表达,上述结果则不会出现,由此验证了信号分子合成位置的重要性(Etchells et al., 2010),也说明TDIF可以通过非细胞自主性的方式在韧皮部和木质部之间实现信息交换与沟通,从而调控原形成层的命运。与拟南芥相比,在木本模式植物杨树中,有关CLE多肽激素如何影响形成层分生组织的发育与分化的研究甚少。目前从山杨(Populus davidiana)中分离到WUS的同源基因PttHB3和CLV1的同源基因PttRLK3,二者在维管形成层区域均有表达,且在木质部端的表达水平高于韧皮部端(Schrader et al., 2004),这说明山杨的形成层区域可能也存在着一个类似于茎端分生组织的CLV-WUS的信号途径,但这些基因在功能上是否类似于它们的拟南芥同源基因以及它们如何参与形成层的活动,则需要进一步的研究来决定。
目前拟南芥中大多数CLE基因的功能还没有得到确认。18个CLE基因的过量表达会产生功能获得性表型(Strabala et al., 2006)。CLV3及多个CLE家族成员过量表达的转基因植株具有与wus突变体类似的表型,说明这些CLE基因功能相似。CLE功能的冗余性也体现在clv3突变体的表型可被其他多个CLE基因所挽救(Hobe et al., 2003;Ni et al., 2006)。另外,尽管某些CLE基因,如CLE6,CLV3和CLE19基因单独过量表达时,并不影响维管组织的发育,但与TDIF一起使用时则可以增强形成层细胞的分裂(Whitford et al., 2008),这说明CLE6,CLV3和CLE19是与TDIF协同作用的,它们的受体可能不同于CLV1-CLV2受体复合物,但也不排除同一个CLE受体同时识别多个CLE多肽激素的可能。实验表明含有3个阿拉伯糖残基的CLE2多肽可与CLV1结合,其效率几乎同CLV3相同(Ohyama et al., 2009)。因此在维管系统中,不同的受体和CLE多肽之间可以发生多种的识别与作用,由此组成复杂的胞间交流信号,共同来影响维管组织的发育(图 2)。另外,CLE多肽激素介导的信号途径对维管系统发育的影响作用并不是独立的。目前已知在维管系统内,CLE多肽对原形成层细胞分裂的促进作用受到生长素正向调控,也受到生长素运输抑制剂NPA(1-naphtylphthalamic acid)的负向调控(Whitford et al., 2008)。在拟南芥和杨树中,原形成层/形成层的细胞分裂为细胞分裂素和其信号传导中的其他元件所控制(Hejatko et al., 2009;Mahonen et al., 2006;Matsumoto-Kitano et al., 2008)。因此,CLE多肽对于维管干细胞命运的控制是通过与其他植物生长激素,包括生长素和细胞分裂素的信号途径进行沟通交流来实现的(图 2)。
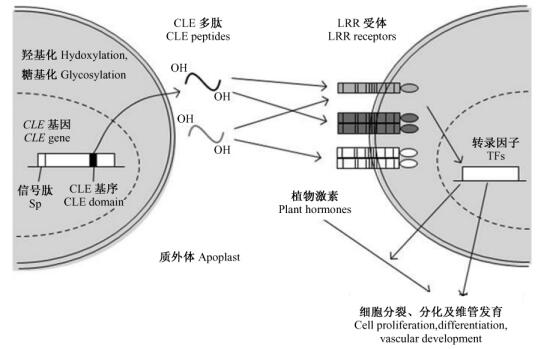
|
图 2 CLE多肽信号转导途径对维管发育的调控模型 Figure 2 A regulation model of the CLE peptide signaling transduction pathway on vasculature development 参见Refer to: Hirakawa et al., 2010a. TFs:Transcription factors; Sp: N-terminal signal peptide. |
综上所述,CLE基因家族代表着植物中一类新的多肽激素,它们通过细胞间的短距离通讯控制分生组织细胞在分裂和分化2种命运中进行选择。与此同时,负责识别CLE多肽激素的受体可能大多数都属于一个更大规模的基因家族——LRR-RLK基因家族。过去几年间,通过对CLV3, CLE19, CLE40和TDIF的研究,初步了解了多肽激素及其受体的生物学功能。但目前仍有一些重要的问题亟待解决。例如为不同的CLE多肽找到它们各自对应的受体;理解CLE多肽的剪切加工与修饰机制,找到那些负责对多肽前体进行剪切及对脯氨酸进行羟基化以及糖基化的酶类;鉴定激素配体与受体结合识别后激活的信号转导下游元件,阐明胞内磷酸化和去磷酸化的分子机制,以及找到那些受到信号途径调控的转录因子以及它们的目标基因;理解CLE多肽与其他植物激素之间的相互作用,研究不同的激素通路如何在单细胞水平上进行整合形成信号网络从而控制细胞行为;寻找木质部起源的、能够控制韧皮部发育的CLE信号等等。以上这些问题的答案,不但将填补目前对多肽激素调节维管系统发育的认识上的空白,也将有助于对多肽激素的分子作用机制有更深的了解。目前有关维管系统发育的分子基础研究仍比较肤浅,特别是对于木本植物维管形成层的起始、维持及分化的分子决定机制方面还需要大量的研究工作。由于林木形体大、世代长,杨树木质部的发育远比拟南芥复杂,只有结合功能基因组学和生物信息学等研究方法,并大力借鉴拟南芥的相应研究成果,才能更快、更准确地分析次生维管系统不同发育阶段所涉及的基因表达和调控网络,才可能实现人为地调控次生维管系统发育过程,进行材性改良的最终目标。
Brand U, Fletcher J C, Hobe M, et al. 2000. Dependence of stem cell fate in Arabidopsis on a feedback loop regulated by CLV3 activity[J]. Science, 289(5479): 617-619. DOI:10.1126/science.289.5479.617 |
Clark S E, Running M P, Meyerowitz E M. 1993. CLAVATA1, a regulator of meristem and flower development in Arabidopsis[J]. Development, 119(2): 397-418. |
Clark S E, Williams R W, Meyerowitz E M. 1997. The CLAVATA1 gene encodes a putative receptor kinase that controls shoot and floral meristem size in Arabidopsis[J]. Cell, 89(4): 575-585. DOI:10.1016/S0092-8674(00)80239-1 |
Cock J M, McCormick S. 2001. A large family of genes that share homology with CLAVATA3[J]. Plant Physiol, 126(3): 939-942. DOI:10.1104/pp.126.3.939 |
De Smet I, Vassileva V, De Rybel B, et al. 2008. Receptor-like kinase ACR4 restricts formative cell divisions in the Arabidopsis root[J]. Science, 322(5901): 594-597. DOI:10.1126/science.1160158 |
Demura T, Tashiro G, Horiguchi G, et al. 2002. Visualization by comprehensive microarray analysis of gene expression programs during transdifferentiation of mesophyll cells into xylem cells[J]. Proc Natl Acad Sci U S A, 99(24): 15794-15799. DOI:10.1073/pnas.232590499 |
Etchells J P, Turner S R. 2010. The PXY-CLE41 receptor ligand pair defines a multifunctional pathway that controls the rate and orientation of vascular cell division[J]. Development, 137(5): 767-774. DOI:10.1242/dev.044941 |
Fiers M, Golemiec E, Xu J, et al. 2005. The 14-amino acid CLV3, CLE19, and CLE40 peptides trigger consumption of the root meristem in Arabidopsis through a CLAVATA2-dependent pathway[J]. Plant Cell, 17(9): 2542-2553. DOI:10.1105/tpc.105.034009 |
Fisher K, Turner S. 2007. PXY, a receptor-like kinase essential for maintaining polarity during plant vascular-tissue development[J]. Curr Biol, 17(12): 1061-1066. DOI:10.1016/j.cub.2007.05.049 |
Fletcher L C, Brand U, Running M P, et al. 1999. Signaling of cell fate decisions by CLAVATA3 in Arabidopsis shoot meristems[J]. Science, 283(5409): 1911-1914. DOI:10.1126/science.283.5409.1911 |
Haecker A, Laux T. 2001. Cell-cell signaling in the shoot meristem[J]. Curr Opin Plant Biol, 4(5): 441-446. DOI:10.1016/S1369-5266(00)00198-9 |
Hejatko J, Ryu H, Kim G T, et al. 2009. The histidine kinases CYTOKININ-INDEPENDENT1 and ARABIDOPSIS HISTIDINE KINASE2 and 3 regulate vascular tissue development in Arabidopsis shoots[J]. Plant Cell, 21(7): 2008-2021. DOI:10.1105/tpc.109.066696 |
Hirakawa Y, Kondo Y, Fukuda H. 2010a. Regulation of vascular development by CLE peptide-receptor systems[J]. J Integr Plant Biol, 52(1): 8-16. DOI:10.1111/jipb.2010.52.issue-1 |
Hirakawa Y, Kondo Y, Fukuda H. 2010b. TDIF peptide signaling regulates vascular stem cell proliferation via the WOX4 homeobox gene in Arabidopsis[J]. Plant Cell, 22(8): 2618-2629. DOI:10.1105/tpc.110.076083 |
Hirakawa Y, Kondo Y, Fukuda H. 2011. Establishment and maintenance of vascular cell communities through local signaling[J]. Current Opinion in Plant Biology, 14(1): 17-23. DOI:10.1016/j.pbi.2010.09.011 |
Hirakawa Y, Shinohara H, Kondo Y, et al. 2008. Non-cell-autonomous control of vascular stem cell fate by a CLE peptide/receptor system[J]. Proc Natl Acad Sci USA, 105(39): 15208-15213. DOI:10.1073/pnas.0808444105 |
Hobe M, Muller R, Grunewald M, et al. 2003. Loss of CLE40, a protein functionally equivalent to the stem cell restricting signal CLV3, enhances root waving in Arabidopsis[J]. Dev Genes Evol, 213(8): 371-381. DOI:10.1007/s00427-003-0329-5 |
Ito Y, Nakanomyo I, Motose H, et al. 2006. Dodeca-CLE peptides as suppressors of plant stem cell differentiation[J]. Science, 313(5788): 842-845. DOI:10.1126/science.1128436 |
Jeong S, Trotochaud A E, Clark S E. 1999. The Arabidopsis CLAVATA2 gene encodes a receptor-like protein required for the stability of the CLAVATA1 receptor-like kinase[J]. Plant Cell, 11(10): 1925-1934. DOI:10.1105/tpc.11.10.1925 |
Ji J, Strable J, Shimizu R, et al. 2010. WOX4 promotes procambial development[J]. Plant Physiol, 152(3): 1346-1356. DOI:10.1104/pp.109.149641 |
Jun J H, Fiume E, Fletcher J C. 2008. The CLE family of plant polypeptide signaling molecules[J]. Cell Mol Life Sci, 65(5): 743-755. DOI:10.1007/s00018-007-7411-5 |
Kiba T, Yamada H, Sato S, et al. 2003. The type-A response regulator, ARR15, acts as a negative regulator in the cytokinin-mediated signal transduction in Arabidopsis thaliana[J]. Plant Cell Physiol, 44(8): 868-874. DOI:10.1093/pcp/pcg108 |
Kinoshita A, Nakamura Y, Sasaki E, et al. 2007. Gain-of-function phenotypes of chemically synthetic CLAVATA3/ESR-related CLE peptides in Arabidopsis thaliana and Oryza sativa[J]. Plant Cell Physiol, 48(12): 1821-1825. DOI:10.1093/pcp/pcm154 |
Kondo T, Sawa S, Kinoshita A, et al. 2006. A plant peptide encoded by CLV3 identified by in situ MALDI-TOF MS analysis[J]. Science, 313(5788): 845-848. DOI:10.1126/science.1128439 |
Kondo Y, Hirakawa Y, Kieber J J, et al. 2011. CLE peptides can negatively regulate protoxylem vessel formation via cytokinin signaling[J]. Plant Cell Physiol, 52(1): 37-48. DOI:10.1093/pcp/pcq129 |
Kubo M, Udagawa M, Nishikubo N, et al. 2005. Transcription switches for protoxylem and metaxylem vessel formation[J]. Genes Dev, 19(16): 1855-1860. DOI:10.1101/gad.1331305 |
Mahonen A P, Bishopp A, Higuchi M, et al. 2006. Cytokinin signaling and its inhibitor AHP6 regulate cell fate during vascular development[J]. Science, 311(5757): 94-98. DOI:10.1126/science.1118875 |
Matsumoto-Kitano M, Kusumoto T, Tarkowski P, et al. 2008. Cytokinins are central regulators of cambial activity[J]. Proc Natl Acad Sci U S A, 105(50): 20027-20031. DOI:10.1073/pnas.0805619105 |
Mayer K F X, Schoof H, Haecker A, et al. 1998. Role of WUSCHEL in regulating stem cell fate in the Arabidopsis shoot meristem[J]. Cell, 95(6): 805-815. DOI:10.1016/S0092-8674(00)81703-1 |
Morillo S A, Tax F E. 2006. Functional analysis of receptor-like kinases in monocots and dicots[J]. Curr Opin Plant Biol, 9(5): 460-469. DOI:10.1016/j.pbi.2006.07.009 |
Ni J, Clark S E. 2006. Evidence for functional conservation, sufficiency, and proteolytic processing of the CLAVATA3 CLE domain[J]. Plant Physiol, 140(2): 726-733. DOI:10.1104/pp.105.072678 |
Ohyama K, Shinohara H, Ogawa-Ohnishi M, et al. 2009. A glycopeptide regulating stem cell fate in Arabidopsis thaliana[J]. Nat Chem Biol, 5(8): 578-580. DOI:10.1038/nchembio.182 |
Pearce G, Strydom D, Johnson S, et al. 1991. A polypeptide from tomato leaves induces wound-inducible proteinase inhibitor proteins[J]. Science, 253(5022): 895-897. DOI:10.1126/science.253.5022.895 |
Scheres B. 2007. Stem-cell niches: nursery rhymes across kingdoms[J]. Nat Rev Mol Cell Biol, 8(5): 345-354. DOI:10.1038/nrm2164 |
Schoof H, Lenhard M, Haecker A, et al. 2000. The stem cell population of Arabidopsis shoot meristems is maintained by a regulatory loop between the CLAVATA and WUSCHEL genes[J]. Cell, 100(6): 635-644. DOI:10.1016/S0092-8674(00)80700-X |
Schrader J, Moyle R, Bhalerao R, et al. 2004. Cambial meristem dormancy in trees involves extensive remodelling of the transcriptome[J]. Plant Journal, 40(2): 173-187. DOI:10.1111/tpj.2004.40.issue-2 |
Shiu S H, Karlowski W M, Pan R, et al. 2004. Comparative analysis of the receptor-like kinase family in Arabidopsis and rice[J]. Plant Cell, 16(5): 1220-1234. DOI:10.1105/tpc.020834 |
Strabala T J, O′Donnell P J, Smit A M, et al. 2006. Gain-of-function phenotypes of many CLAVATA3/ESR genes, including four new family members, correlate with tandem variations in the conserved CLAVATA3/ESR domain[J]. Plant Physiol, 140(4): 1331-1344. DOI:10.1104/pp.105.075515 |
To J P, Haberer G, Ferreira F J, et al. 2004. Type-A Arabidopsis response regulators are partially redundant negative regulators of cytokinin signaling[J]. Plant Cell, 16(3): 658-671. DOI:10.1105/tpc.018978 |
Trotochaud A E, Hao T, Wu G, et al. 1999. The CLAVATA1 receptor-like kinase requires CLAVATA3 for its assembly into a signaling complex that includes KAPP and a Rho-related protein[J]. Plant Cell, 11(3): 393-406. DOI:10.1105/tpc.11.3.393 |
Whitford R, Fernandez A, De Groodt R, et al. 2008. Plant CLE peptides from two distinct functional classes synergistically induce division of vascular cells[J]. Proc Natl Acad Sci USA, 105(47): 18625-18630. DOI:10.1073/pnas.0809395105 |
 2011, Vol. 47
2011, Vol. 47

