文章信息
- 陈亚娟, 王宏芝, 李瑞芬, 张中保, 崔莉洁, 魏建华
- Chen Yajuan, Wang Hongzhi, Li Ruifen, Zhang Zhongbao, Cui Lijie, Wei Jianhua
- 毛白杨纤维素合酶基因家族部分成员的克隆及表达
- Isolation and Expression Profile of Some Members of Cellulose Synthase Gene Family in Populus tomentosa
- 林业科学, 2011, 47(10): 70-75.
- Scientia Silvae Sinicae, 2011, 47(10): 70-75.
-
文章历史
- 收稿日期:2011-01-06
- 修回日期:2011-04-25
-
作者相关文章
纤维素是木材的主要组分,是地球上最丰富的重要可再生资源,自然界每年可生产约1 800亿t的纤维素。在生物能源和制浆造纸工业中,纤维素的含量和存在形式直接影响发酵产物液体乙醇的产率和制浆造纸过程中纸浆得率(Chiang et al., 1988;Sassner et al., 2005)。因此,深入研究林木植物中纤维素的生物合成过程,挖掘控制木材纤维品质和产量的重要基因,进而通过遗传操作改良植物的纤维品质具有重要的理论和实践意义。
在高等植物中,纤维素合酶(cellulose synthase,CesA)催化纤维素的合成(Chandrashekhar et al., 2007)。纤维素合酶在细胞膜上组装成纤维素合酶复合体(cellulose synthase complexs,CSCs),它由6个大亚基组成,在细胞膜上呈六边玫瑰形,推测每个大亚基可能包含6个CesA蛋白(Doblin et al., 2002;Somerville,2006;Mutwil et al., 2008)。目前研究表明,植物初生、次生细胞壁的形成涉及不同的纤维素合酶(Saxena et al., 2000;Haigler et al., 2001;Desprez et al., 2007;Atanassov et al., 2009)。拟南芥(Arabidopsis thaliana)中,CesA1, CesA3和CesA6形成的复合体参与初生壁纤维素合成(Wang et al., 2008),而CesA4,CesA7和CesA8参与次生壁纤维素的合成(Atanassov et al., 2009)。
林木植物的次生细胞壁形成远比拟南芥复杂,已有研究对林木中一些参与细胞壁形成的纤维素合酶编码基因进行了表达分析和功能验证。杨树(Populus)全基因组测序的完成促进了杨树中参与细胞壁合成的纤维素合酶基因的研究。通过序列分析,在毛果杨(P. trichocarpa)基因组中已发现18个CesA基因(Soraya et al., 2005),其中PtCesA4, PtCesA5, PtCesA7, PtCesA8, PtCesA17和PtCesA18在木质部特异高表达(Suzuki et al., 2006)。最近,Song等(2010)发现,杨树木质部细胞膜上至少存在2种纤维素合酶复合体,复合体Ⅰ由CesA4, CesA7, CesA8, CesA17和CesA18组成,参与细胞次生壁的合成;复合体Ⅱ由CesA3, CesA10, CesA11, CesA13, CesA15和CesA16组成,参与细胞初生壁和次生壁的合成。然而,纤维素合酶在杨树次生细胞壁形成中的功能鉴定还需要深入研究。
杨树是林木分子生物学研究的模式植物,也是北半球重要的用材树种。揭示杨树中纤维素合酶基因及其功能,尤其是参与次生细胞壁形成的纤维素合酶基因,对于利用现代分子生物学手段改良毛白杨(P. tomentosa)木材品质具有重要的现实意义。毛白杨是我国特有乡土树种,在材性品质和生态适应性上具有明显优势,是优良的纸浆材树种和潜在的生物质能源树种。塔形毛白杨CV-BJHR01(P. tomentosa ‘BJHR01’)(审定编号:国S-SC-PT-001-2006)新品系是经过人工杂交和多年选择而培育出的杨树新品种。该品系杨树具有生长快、抗性强、树冠狭窄、雄株或弱雌性等优良特性,是城市绿化、农田防护和短周期工业用材林的优良品种。本研究从塔形毛白杨CV-BJHR01中克隆了7个纤维素合酶基因,结合生物信息学分析初步预测了其功能;运用GeXP系统研究了这些纤维素合酶基因的组织特异性表达模式,为揭示这些基因的生物学功能提供了实验数据。
1 材料与方法 1.1 植物材料塔形毛白杨CV-BJHR01,温室培养3个月,培养温度为(25±2)℃,光照时间为16 h·d-1,光照强度为40~45 μmol·m-2s-1。
1.2 试验方法 1.2.1 RNA的提取使用柱式植物RNAOUT2.0试剂盒(天恩泽)提取毛白杨叶片(6—8节间幼嫩叶片)和次生木质部(1—3节间)总RNA。由于RNA容易降解,取材过程要迅速,剥去树皮后,用灭菌刀片刮取树干和树皮内侧的材料,放入液氮速冻过的离心管,马上投入液氮中。具体总RNA提取过程按照试剂盒说明书进行。
1.2.2 基因克隆根据已发表的毛果杨纤维素合酶基因全序列,在基因3’UTR和5’UTR区设计引物,采用巢式PCR的方法扩增毛白杨纤维素合酶基因,引物序列见表 1。cDNA的合成使用Invitrogen公司的SuperScriptⅢ First-Strand Synthesis System试剂盒,具体操作按照试剂盒说明书进行。PCR扩增产物纯化后克隆到pGEM-T Easy(Promega)载体上,进行测序分析。
|
|
DNA序列利用DNAMAN软件进行组装和分析;数据库搜索利用NCBI BLAST搜索程序(Altshul et al., 1990);采用Clustal W(Thompson et al., 1994)进行多序列比对;使用Mega4.0软件(Tamura et al., 2007)对10个拟南芥CesA基因、11个水稻(Oryza sativa)CesA基因、18个毛果杨CesA基因和7个完整的毛白杨CesA基因构建NJ(Neighbor-Joining Method,邻接法)系统发育树。拟南芥基因的数据来自NCBI,水稻基因数据来自水稻基因组计划注释数据库(http://rice.plantbiology.msu.edu),毛果杨基因序列来自毛果杨基因组文库(http://genome.jgi-psf.org/Poptr1_1/)。
1.2.4 基于GeXP遗传分析系统的基因表达分析引物设计选择在基因序列的非保守区,引物序列见表 2。毛白杨叶片和次生木质部总RNA的提取方法同上。使用GenomeLab GeXP Start Kit(Beckman Coulter)试剂盒进行反转录(RT)及多重PCR(XP-PCR)反应。以Actin基因的表达水平作为对照,同时设无模板质控(NTC)和无反转录酶质控(NRTC)对照。
|
|
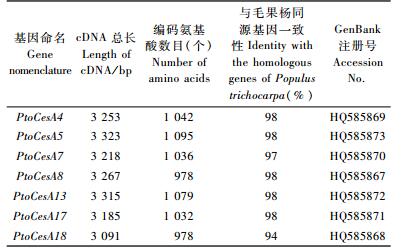
以毛白杨次生木质部总RNA为模板,通过RT-PCR扩增纤维素合酶基因,获得大小约为3 100 bp的DNA片段(图 1)。获得的序列经分析均具有完整的阅读框,包含部分UTR区序列。经NCBI BLAST数据库搜索,以及同毛果杨和拟南芥纤维素合酶基因家族比对,分别命名为PtoCesA4,PtoCesA5,PtoCesA7,PtoCesA8,PtoCesA13,PtoCesA17和PtoCesA18,基因命名(表 3)参考Kumar等(2009)。DNAMAN分析表明,克隆的毛白杨纤维素合酶基因与毛果杨基因组文库登记的同源基因在氨基酸水平上的一致性为94%~98%(表 3)。
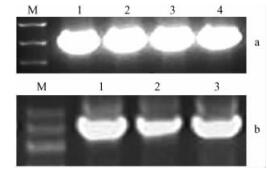
|
图 1 纤维素合酶基因扩增结果 Figure 1 Products of CesA amplification a.M:DNA分子量标准DNA marker Ⅲ;1-4:CesA5, CesA7, CesA13, CesA17基因巢式扩增结果Nested PCR products of CesA5, CesA7, CesA13, CesA17 respectively. b.M:DNA分子量标准DNA marker Ⅲ;1-3:CesA4, CesA8, CesA18基因巢式扩增结果Nested PCR products of CesA4, CesA8, CesA18 respectively. |
|
|
使用Mega4.0软件建立系统进化树(图 2)。从进化树上可以看出,毛白杨PtoCesA13与拟南芥AtCesA3属于同一亚族,毛白杨PtoCesA5与拟南芥AtCesA2, AtCesA5, AtCesA6及AtCesA9属于同一亚族。拟南芥研究表明,AtCesA1, AtCesA3和AtCesA6组成纤维素合酶复合体,参与初生细胞壁合成;同时AtCesA2, AtCesA5和AtCesA9被认为是AtCesA6的替代物,这些蛋白彼此之间存在功能冗余(Persson et al., 2005;Sonia et al., 2006;Desprez et al., 2007)。由此推测毛白杨PtoCesA5和PtoCesA13在初生壁形成中发挥作用。

|
图 2 拟南芥、水稻、毛果杨和毛白杨纤维素合酶构建的邻接树 Figure 2 The neighbor-joining tree of Arabidopsis thaliana, Oryza sativa, Populus trichocarpa and P. tomentosa CesAs proteins 拟南芥数据来自NCBI Data of Arabidopsis thaliana was from NCBI:AtCesA1(AF027172);AtCesA2 (AF027173);AtCesA3 (AF027174);AtCesA4(AF458083);AtCesA5 (NM-121024);AtCesA6 (NM-125870);AtCesA7(AF088917);AtCesA8(AF267742);AtCesA9(NM-127746);AtCesA10(NM-128111). 水稻数据来自RGAP Data of Oryza sativa was from Rice Genome Annotation Project:OsCesA1(LOC_Os05g08370);OsCesA2(LOC_Os03g59340);OsCesA3(LOC_Os07g24190);OsCesA4(LOC_Os01g54620);OsCesA5(LOC_Os03g62090);OsCesA6(LOC_Os07g14850);OsCesA7(LOC_Os10g32980);OsCesA8(LOC_Os07g10770);OsCesA9(LOC_Os09g25490);OsCesA10(LOC_Os12g29300);OsCesA11(LOC_Os06g39970). 毛果杨数据来自毛果杨基因组文库Data of populus trichocarpa was from Populus trichocarpa genome database:PtCesA1(582479);PtCesA2(551308);PtCesA3(691465);PtCesA4(691997);PtCesA5(714760);PtCesA6(71761);PtCesA7(717646);PtCesA8(568682);PtCesA9(695949);PtCesA10(702007);PtCesA11(344723);PtCesA12(560520);PtCesA13(697507);PtCesA14(576348);PtCesA15(56570);PtCesA16(97727);PtCesA17(578717);PtCesA18(555650). |
毛白杨PtoCesA4与PtCesA4,AtCesA4和OsCesA7属于同一个亚族;PtoCesA7和PtoCesA17与PtCesA7,PtCesA17,AtCesA7和OsCesA9属于同一亚族;PtoCesA8和PtoCesA18与PtCesA8,PtCesA18,AtCesA8和OsCesA4属于同一个亚族。研究表明AtCesA4,AtCesA7和AtCesA8共同参与细胞次生壁的合成(Turner et al., 1997;Taylor et al., 2003;Atanassov et al., 2009)。OsCesA4,OsCesA7和OsCesA9在功能上并不冗余,可能形成一个纤维素合酶复合体参与次生细胞壁合成(Tanaka et al., 2003)。由此推测毛白杨PtoCesA4,PtoCesA7/17和PtoCesA8/18参与细胞次生壁的合成。
2.2 毛白杨纤维素合酶基因的表达分析 2.2.1 用于GeXP多重分析的基因引物有效性验证采用PCR对设计的特定基因引物进行验证和优化,筛选出能够有效扩增的特异引物。PCR产物经毛细管电泳分离,以检验特定引物扩增的有效性和扩增片段的大小。结果表明,本研究中最终选定的引物,对目标基因能进行有效特异性扩增,为基因的表达分析奠定了基础。尝试了多对不同引物,PtoCesA13均没有得到有效扩增,可能是由于基因转录丰度没有达到系统可以检测的水平。
2.2.2 GeXP系统分析杨树纤维素合酶基因的表达特性为了鉴定在次生木质部特异表达的毛白杨纤维素合酶基因,利用GeXP系统分别分析了毛白杨叶片和次生木质部纤维素合酶基因的表达(图 3, 图 4)。结果表明:毛白杨次生木质部和叶片纤维素合酶基因家族表达谱存在明显差异,其中PtoCesA4, PtoCesA7, PtoCesA8, PtoCesA17和PtoCesA18在茎次生木质部中的转录丰度较高(图 4),说明这些基因可能主要参与毛白杨次生细胞壁的形成;而PtoCesA5在叶片中的转录丰度明显高于次生木质部(图 4)。由于叶片中维管系统较少,推测PtoCesA5可能在初生壁合成中发挥作用。以上这些结果与生物信息学分析预测的基因功能相吻合。
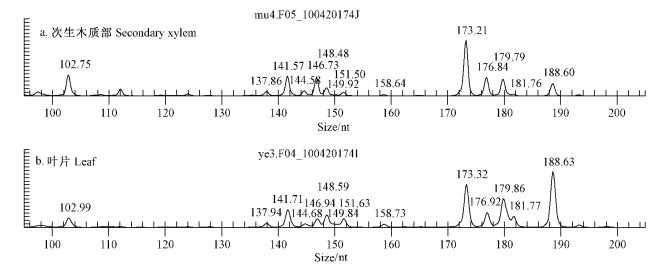
|
图 3 毛白杨次生木质部和叶片纤维素合酶基因家族表达谱的毛细管电泳 Figure 3 Representative capillary electropherograms corresponding to gene expression profiles of cellulose synthase from Populus tomentosa in secondary xylem and leaves |
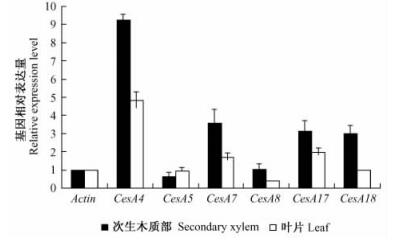
|
图 4 毛白杨次生木质部和叶片纤维素合酶转录丰度的比较 Figure 4 Expression levels of cellulose synthase genes from Populus tomentosa in secondary xylem and leaves 基因表达水平为相对值,对照Actin基因的表达水平定为1,试验设3次重复。The expression levels of genes are compared with that of Actin, which is set to 1. Error bars represent the SE of three biological replicates. |
本研究利用RT-PCR的方法,从毛白杨中分离了纤维素合酶基因PtoCesA4, PtoCesA5, PtoCesA7, PtoCesA8, PtoCesA13, PtoCes17和PtoCesA18。通过与拟南芥、水稻以及毛果杨基因序列比对,推测毛白杨PtoCesA5和PtoCesA13参与毛白杨初生壁的形成,PtoCesA4, PtoCesA7, PtoCesA8, PtoCesA17和PtoCesA18在毛白杨次生壁形成过程中发挥作用,这与Suzuki等(2006)和Song等(2010)在毛果杨中的研究结论相一致。然而,在Suzuki等(2006)的研究中,毛果杨PtCesA4转录表达水平最低,PtCesA18最高;而在本试验中PtoCesA4的转录表达水平最高。Suzuki等(2006)的研究显示木质部特异表达基因在其他组织中的表达量都非常低,而本研究通过GeXP系统分析其表达水平差异较小,如在木质部特异高水平表达的基因PtoCesA4,PtoCesA7,PtoCesA17等在叶片中的表达量也较高。以上差异可能是由于本试验取材树龄小、木质部还没有充分木质化所致。引起这些差异的具体原因还有待进一步的研究。
Suzuki等(2006)在毛果杨4种组织中均检测不到PtCesA3的表达,本试验在毛白杨叶片和次生木质部中均检测到了PtoCesA3基因的表达,但是表达量相对较低。PtoCesA13在本试验中未能得到有效扩增。根据系统进化树分析,PtoCesA13与AtCesA3,PtCesA13和PtCesA3进化上亲缘关系最近;Song等(2010)的研究表明毛果杨中,PtCesA3和PtCesA13既参与初生壁的合成也参与次生壁的合成,AtCesA3参与了拟南芥初生细胞壁的形成。根据以上结果毛白杨PtoCesA3和PtoCesA13在细胞壁形成过程中的作用还有待于进一步的试验验证。
本研究克隆了毛白杨7个CesA基因,利用GeXP遗传分析系统对毛白杨纤维素合酶家族基因在不同部位的表达进行了研究,为进一步鉴定毛白杨纤维素合酶基因的功能提供了实验依据。本实验室选取了部分次生木质部高表达基因进行了进一步的过表达和抑制表达研究,目前已获得了一部分转基因株系,基因功能的验证还在进行中。
Altshul F, Gish W, Miller W, et al. 1990. Basic local alignment search tool[J]. J Mol Biol, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
Atanassov H, Pittman J K, Turner S R. 2009. Elucidating the mechanisms of assembly and subunit interaction of the cellulose synthase complex of Arabidopsis secondary cell walls[J]. Journal of Biological Chemistry, 284(6): 3833-3841. DOI:10.1074/jbc.M807456200 |
Chandrashekhar P J, Shawn D Mansfield. 2007. The cellulose paradox-simple molecule, complex biosynthesis[J]. Current Opinion in Plant Biology, 10(3): 220-226. DOI:10.1016/j.pbi.2007.04.013 |
Chiang V L, Puumala R J, Takeuchi H. 1988. Comparison of softwood and hardwood kraft pulping[J]. Tappi J, 71(9): 173-176. |
Desprez T, Juraniec M, Crowell E F. 2007. Organization of cellulose synthease complexes involved in primary cell wall synthesis in Arabidopsis thaliana[J]. PNAS, 104(39): 15572-15577. DOI:10.1073/pnas.0706569104 |
Doblin M S, Kurek I, Jacob-Wilk D, et al. 2002. Cellulose biosynthesis in plants:from genes to rosettes[J]. Plant and Cell Physiology, 43(12): 1407-1420. DOI:10.1093/pcp/pcf164 |
Haigler C H, Datcheva I, Hogan P S, et al. 2001. Carbon partitioning to cellulose synthesis[J]. Plant Mol Biol, 47(1/2): 29-51. DOI:10.1023/A:1010615027986 |
Kumar M, Thammannagowda S, Bulone V, et al. 2009. An update on the nomenclature for the cellulose synthase genes in Populus[J]. Trends in Plant Science, 14(5): 248-254. |
Mutwil M, Debolt S, Persson S. 2008. Cellulose synthesis: a complex complex[J]. Current Opinion in Plant Biology, 11(3): 252-257. DOI:10.1016/j.pbi.2008.03.007 |
Persson S, Wei H, Milne J, et al. 2005. Identification of genes required for cellulose synthesis by regression analysis of public microarray data sets[J]. Proc Natl Acad Sci U S A, 102(24): 8633-8638. DOI:10.1073/pnas.0503392102 |
Sassner P, Galbe M, Zacchi G. 2005. Steam pretreatment of Salix with and without SO2 impregnation for production of bioethanol[J]. Appl Biochem Biotech, 124: 1101-1118. DOI:10.1385/ABAB:124:1-3 |
Saxena I M, Brown R M Jr. 2000. Cellulose synthase and related enzymes[J]. Current Opinion in Plant Biology, 3(6): 523-531. DOI:10.1016/S1369-5266(00)00125-4 |
Somerville C. 2006. Cellulose synthesis in higher plants[J]. Annual Review of Cell and Developmental Biology, 22(1): 53-78. DOI:10.1146/annurev.cellbio.22.022206.160206 |
Song D L, Shen J H, Li L G. 2010. Characterization of cellulose synthase complexes in Populus xylem differentiation[J]. New Phytologist, 187(3): 777-790. DOI:10.1111/j.1469-8137.2010.03315.x |
Sonia B, Christopher J B, Neil G, et al. 2006. Interactions between MUR10/Ces7-dependent secondary cellulose biosynthesis and primary cell wall structure[J]. Plant Physiology, 142(4): 1353-1363. DOI:10.1104/pp.106.087700 |
Soraya D, Mats L, Lars A, et al. 2005. The genome sequence of black cottonwood (Populus trichocarpa) reveals 18 conserved cellulose synthase (CesA) genes[J]. Planta, 221(5): 739-746. DOI:10.1007/s00425-005-1498-4 |
Suzuki S, Li L G, Sun Y H, et al. 2006. The cellulose synthase gene superfamily and biochemical functions of xylem-specific cellulose synthase-like genes in Populus trichocarpa[J]. Plant Physiology, 142(3): 1233-1245. DOI:10.1104/pp.106.086678 |
Tamura K, Dudley J, Nei M, et al. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
Tanaka K, Murata K, Yamazaki M, et al. 2003. Three distinct rice cellulose synthase catalytic subunit genes required for cellulose synthesis in the secondary wall[J]. Plant Physiology, 133(1): 73-83. DOI:10.1104/pp.103.022442 |
Taylor N G, Howells R M, Huttly A K, et al. 2003. Interactions among three distinct CesA proteins essential for cellulose synthesis[J]. PNAS, 100(3): 1450-1455. DOI:10.1073/pnas.0337628100 |
Thompson J D, Higgins D G, Gibson T J. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673 |
Turner S R, Somerville C R. 1997. Collapsed xylem phenotype of Arabidopsis identifies mutants deficient in cellulose deposition in the secondary cell wall[J]. Plant Cell, 9(5): 689-701. DOI:10.1105/tpc.9.5.689 |
Wang J, Elliott J E, Williamson R E. 2008. Features of the primary wall CesA complex in wild type and cellulose-deficient mutants of Arabidopsis thaliana[J]. Journal of Experimental Botany, 59(10): 2627-2637. DOI:10.1093/jxb/ern125 |
 2011, Vol. 47
2011, Vol. 47