文章信息
- 胥猛, 谢雯凡, 潘惠新, 苏晓华, 张守攻, 黄敏仁
- Xu Meng, Xie Wenfan, Pan Huixin, Su Xiaohua, Zhang Shougong, Huang Minren
- 杨树ARGONAUTE基因的克隆及序列分析
- Cloning and Characterization of ARGONAUTE Genes in Populus
- 林业科学, 2011, 47(3): 46-51.
- Scientia Silvae Sinicae, 2011, 47(3): 46-51.
-
文章历史
- 收稿日期:2010-10-21
- 修回日期:2010-12-28
-
作者相关文章
2. 中国林业科学研究院林业研究所 北京 100091
2. Research Institute of Forestry, Chinese Academy of Forestry Beijing 100091
RNA沉默(RNA silencing)是一种通过mRNA降解、翻译抑制或染色体修饰等方式调控基因表达和抵抗外源遗传因子入侵的生物现象。它存在2种既有联系又有区别的途径:siRNA(small interference RNA)和miRNA (microRNA)。尽管引发RNA沉默的来源不同,但siRNA和miRNA都参与构成结构相似的RNA诱导沉默复合体(RNA-Induced Silencing Complex,RISC),在作用方式上二者有很大的相似性。作为RISC的核心元件,Argonaute (AGO)蛋白在RNA沉默中发挥关键作用,与小RNA在生物体中共同参与维持基因组稳定、调控组织发育、响应逆境胁迫以及激发免疫防御(李超等,2009)。小RNA直接与AGO蛋白结合,赋予靶向作用专一性,而AGO蛋白决定RNA沉默功能。
AGO蛋白是一个高度保守而又庞大的家族,以含有PAZ和Piwi 2个主要结构域为共同特征。PAZ结构域是小RNA的结合位点。尽管所有类型的小RNA在某种程度上都可与任一AGO蛋白结合,但对于特定的小RNA,与AGO的结合有明显的偏好: AGO1蛋白主要与miRNA结合,AGO2偏好结合tasiRNA,AGO4结合rasiRNA,而AGO5蛋白与非翻译序列衍生的小RNA结合; 而且,AGO蛋白对小RNA的5’端核苷酸也有很强的偏好:与AGO1结合的小RNA 5’端86%是尿苷,与AGO2结合的小RNA 5’端93%是腺苷,与AGO4结合的小RNA 5’端79%是腺苷,与AGO5结合的小RNA 5’端83%是胞苷(Mi et al., 2008)。C端的Piwi结构域是RISC行使切割功能的活性中心,具有类似RNaseH催化中心的结构特征,3个高度保守的氨基酸残基(DDH或DDD)对应于RNaseH的DDE催化模体。近年来,AGO蛋白家族的基本结构、功能及其作用机制日益成为研究的焦点。拟南芥(Arabidopsis thaliana)有10个AGO蛋白家族成员(Vaucheret,2008),水稻(Oryza sativa)至少也有19个AGO蛋白(Kapoor et al., 2008)。这个庞大的蛋白家族不仅在各个物种中分布的数量和种类具有物种差异性,而且成员之间所发挥的生物学功能也具有特异性。迄今为止,有关AGO蛋白家族的功能和作用机制还远未阐述清楚,并且已有的研究主要集中在拟南芥和水稻等草本植物中,而林木中关于这类基因的研究远远滞后,鲜有报道(Tahir et al., 2006)。
根是植物长期适应陆地生境而形成的一个重要器官。深入研究植物根系的生长发育机制,有利于全面了解植物的整体生物学特性及其功能,从而为保护和开发利用植物资源、改良品质等提供理论依据。杨树(Populus)是世界中纬度平原地区栽培最为广泛的树种之一,也是我国主要的用材树种。在我国长江中下游及黄淮地区主要栽种黑杨派(Sect. Aigeiros)南方型无性系,包括美洲黑杨(P. deltoides)和欧美杨(P.× euramericana)。由于黑杨派无性系的繁殖方式是通过硬枝扦插进行,扦插生根难易程度直接影响造林成活率,因此,生根性状在其遗传改良研究中占有重要的地位。南林895杨(P.× euramericana ‘NL895’)和南林3244杨(P. deltoides ‘NL3244’)是南京林业大学在“九五”期间选育的速生、优质、高产的杨树单板用材新品种,它们的生根能力存在显著差异。长期以来,杨树遗传改良研究更多的是集中在地上部分,而对根系发育性状的认识和了解比较缺乏。为此,本研究从毛果杨(P.trichocarpa) POPTR_0001s22120.1基因预测模型入手,以南林895杨和南林3244杨为研究材料,从其不定根中分离得到编码AGO蛋白的同源基因PeAGO5和PdAGO5。在此基础上,进行了序列同源性比对和系统进化树分析,并利用实时定量PCR技术检测了PeAGO5基因在杨树扦插生根过程中的表达模式,从而为深入了解AGO蛋白家族在杨树发育中的调控机制提供新信息。
1 材料与方法 1.1 植物材料南林895杨和南林3244杨硬枝插穗采集于1年生扦插苗,水培生根。在水培生根过程中,根据肉眼可辨的发育形态变化确定5个取样点(Xu et al., 2011):DB (dormant bark tissues休眠皮层; 第1天),RC (root calli愈伤组织; 第32天),ER (emerging roots新生根; 第48天),PR (primary adventitious roots初生不定根; 第54天)和LR (lateral roots侧根; 第65天)。不同发育阶段的生根材料经液氮速冻后,-75 ℃超低温冰箱保存备用。
1.2 总RNA提取与cDNA第1链的合成采用改进的CTAB-LiCl方法抽提杨树不定根总RNA(Xu et al., 2009),总RNA的纯化及DNase Ⅰ消化参照RNeasy Plant Mini Kit(Qiagen,USA)说明书进行。用SuperScriptTM Ⅲ First-Strand Synthesis System for RT-PCR (Invitrogen)试剂盒进行反转录,合成cDNA第1链。
1.3 目的基因克隆及序列测定根据Affymetrix杨树基因组芯片上PtpAffx. 200557.1.S1_at探针信息,比对确定靶基因在毛果杨基因组上的基因预测模型(estExt_Genewise1 _v1. C_LG_I9749),在其预测的开放阅读框(Open Reading Frame,ORF)区域以外设计基因特异引物AGO-f:5’-GCATCCGGCTACCTTTC-3’和AGO-r:5’-CCACACAAAAGAAGGGCCAACTATA-3’,分别以南林895杨和南林3244杨初生不定根(ER) cDNA为模板,进行目的基因的PCR扩增。PCR反应扩增体系如下:反转录cDNA模板30~50 ng,10×LA PCR Buffer(Mg2+ Free) 5.0 μL,MgCl2 (25 mmol·L-1) 5.0 μL,正反引物(10 μmol·L-1)各2.0 μL,高保真TakaRa LA Taq(5 U·μL-1) 0.5 μL,加灭菌Milli-Q水补至50.0 μL。PCR反应程序: 94 ℃预变性4 min; 94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸3 min,30个循环; 72 ℃延伸7 min。目的扩增片段克隆参照TaKaRa公司的pMD19-T simple Vetor说明书进行,序列测定由上海英骏生物技术有限公司完成。
1.4 序列分析利用FGENESH (http://mendel.cs.rhul.ac.uk/mendel.php?topic=fgen)软件进行ORF确定及氨基酸序列推测。在线工具Expasy Protparma (http://www.expasy.ch/tools/protparam.html)用于分析蛋白质的分子量、等电点和氨基酸组成。通过Pfam数据库推测蛋白质结构域(http://pfam.sanger.ac.uk/)。蛋白质跨膜区预测、信号肽和二级结构分析分别采用TMHMM (http://www.cbs.dtu.dk/services/TMHMM/),SignalP (http://www.cbs.dtu.dk/services/SignalP/)和SOPMA (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)工具进行。利用MEGA4.0的邻接(Neighbor Joining,NJ)算法构建AGO蛋白的系统进化树(Tamura et al., 2007)。
1.5 qRT-PCR表达分析南林895杨硬枝插穗生根过程中,5个不同发育时间点获取的cDNA样品用于目的基因的实时定量PCR(quantitative real-time PCR,qRT-PCR)表达分析。采用Primer Express Software version 3.0 (Applied Biosystems)软件进行qRT-PCR引物设计,PeAGO-f: 5′-GGAAGACTCTAGCCCGTCGAT-3′,PeAGO-r: 5′-GGTTACCTCTGGCCAGTCCAT-3′。泛素延伸蛋白基因UBQ (ubiquitin extension protein)为qRT-PCR的内参照,扩增引物为UBQ-f: 5′-CCAAGCCCAAG AAGATCAAGC-3′和UBQ-r: 5′-GCACCGCACTCAGCATTAGG-3′。qRT-PCR采用SYBR Green Realtime PCR Master Mix (TOYOBO),在ABI 7500 Realtime PCR Systems (Applied Biosystems)上完成,每反转录样品3次重复。反应体系为:2×SYBR Green Realtime PCR Master Mix 10 μL,正反引物(10 mmol· L-1)各1 μL,cDNA 3 μL (10~50 ng),灭菌Milli-Q水5 μL。反应程序为:95 ℃ 1 min; 95 ℃ 15 s,60 ℃ 1 min,40个循环。
2 结果与分析 2.1 PeAGO5,PdAGO5基因的分离及序列分析在前期的研究中,Affymetrix杨树基因组芯片分析结果显示杨树POPTR _0001s22120.1基因(探针PtpAffx.200557.1.S1_at,基因预测模型estExt_Genewise1_v1.C _LG _I9749,Blastp注释为AGO5)参与杨树不定根发育调控。为了分离该基因,利用基因特异扩增引物AGO-f和AGO-r,分别以欧美杨和美洲黑杨不定根cDNA为模板进行PCR扩增,琼脂糖凝胶电泳分离获得大小约2 800 bp特异条带。测序结果表明,2个cDNA克隆均为2 809 bp,包含起始和终止密码子,61~2 784 bp为1个开放阅读框,分别命名为PeAGO5和PdAGO5,均编码907个氨基酸(图 1),预测蛋白质分子量分别为102.31 ku和102.34 ku,理论等电点(pI)分别为9.34和9.7。PeAGO5编码氨基酸中,含量最高的为丝氨酸(Ser,74,8.2%),其次是缬氨酸(Val,71,7.8%)、亮氨酸(Leu,68,7.5%)和精氨酸(Arg,63,6.9%); 酸性氨基酸(Asp,Glu)总数95个,碱性氨基酸(Arg,Lys)总数125个; GRAVY (grand average of hydropathicity)值为-0.405。与之相似,PdAGO5编码氨基酸中,丝氨酸(Ser,73,8.0%)含量最高,其次也是缬氨酸(Val,71,7.8%)、亮氨酸(Leu,69,7.6%)和精氨酸(Arg,63,6.9%); 酸性氨基酸(Asp,Glu)总数95个,碱性氨基酸(Arg,Lys)总数128个; GRAVY值为-0.412。蛋白质跨膜区和信号肽分析结果表明,PeAGO5和PdAGO5蛋白都不具有跨膜区域,不存在信号肽酶切位点,推测是一种非分泌、亲水性蛋白。

|
图 1 PeAGO5/PdAGO5氨基酸序列及结构域分析 Figure 1 Amino acid alignment and domain analysis of PeAGO5/PdAGO5 |
从欧美杨和美洲黑杨不定根中分离到的PeAGO5/PdAGO5基因,其编码蛋白具有AGO家族典型的结构域。PeAGO5和PdAGO5基因编码氨基酸序列具有较高的相似性,氨基酸序列之间仅有7个差异位点(图 1,粗体示差异位点,其中括号内为PdAGO5)。Pfam数据库检索显示,PeAGO5和PdAGO5均含有3个共同的结构域,DUF1785,PAZ和Piwi结构域(图 1)。N端的DUF1785 (208—260aa)结构域,具体功能尚不清楚。PAZ (265— 396aa)结构域含有132个氨基酸。C端的Piwi (553—871aa)结构域包含3个高度保守氨基酸残基(2个天冬氨酸和1个组氨酸,DDH)。它们与拟南芥AGO蛋白家族相似性分别为64.8%~32.2%和64.3%~31.9%,其中,与AtAGO5 (AT2G27880.1)相似性最高。多重序列比对表明,无论PeAGO5和PdAGO5之间及其与拟南芥10个AGO蛋白家族成员之间的相似性高低,它们都含有2个高度保守的结构域,PAZ和Piwi。图 2示Piwi结构域氨基酸序列比对结果,箭头标注Piwi盒中高度保守的催化三联体DDH/DDD元件(Kapoor et al., 2008)。SOPMA分析表明α螺旋(α helix)和无规则卷曲(random coil)是PeAGO5和PdAGO5蛋白二级结构的主要成分。PeAGO5蛋白二级结构中含有31.42%的α螺旋、17.86%的延伸链(extended strand)、3.31%的β转角(β turn)和47.41%的无规则卷曲; 与其相似,PdAGO5蛋白二级结构中含有32.30%的α螺旋、16.76%的延伸链、3.97%的β转角和46.97%的无规则卷曲。

|
图 2 PeAGO5/PdAGO5与拟南芥AGO蛋白家族Piwi结构域比较 Figure 2 Piwi domain alignment of PeAGO5/PdAGO5 and Arabidopsis thaliana AGO proteins 箭头所示为Piwi结构域中最重要的3个保守氨基酸残基DDH或DDD. The conserved DDH or DDD triad residues are marked with upward arrows. |
为了分析杨树PeAGO5/PdAGO5蛋白与拟南芥、水稻AGO蛋白家族成员的系统进化关系,采用NJ算法构建了上述AGO蛋白系统进化树(图 3)。拟南芥和水稻AGO蛋白成员并非单独聚类形成各自的进化枝,而是相互交叉进化,这说明AGO蛋白产生于单子叶和双子叶植物分化之前。拟南芥有10个AGO蛋白成员,分属3个进化枝:进化枝1包括AtAGO1,AtAGO5和AtAGO10;进化枝2包括AtAGO2,AtAGO3和AtAGO7;进化枝3包括AtAGO4,AtAGO6,AtAGO8和AtAGO9 (Vaucheret,2008)。杨树PeAGO5和PdAGO5蛋白归属进化枝1,且与AtAGO5归为同一个小的分支,这为推测PeAGO5/PdAGO5的功能提供一定的线索。AtAGO1是目前拟南芥中研究最透彻、参与通路最多、功能最为重要的AGO蛋白成员(李超等,2009)。AtAGO10/PNH/ZLL基因通过抑制mi165/166对靶mRNA的剪切,参与顶端分生组织和叶极性建立的调控(Liu et al., 2009)。AGO5蛋白与非翻译序列衍生的小RNA结合,生物信息分析表明,与AGO5结合的小RNA5’端83%是胞苷(Mi et al., 2008); 通过反向遗传学确定的ago5突变体没有表现出明显的发育缺陷(Vaucheret,2008)。
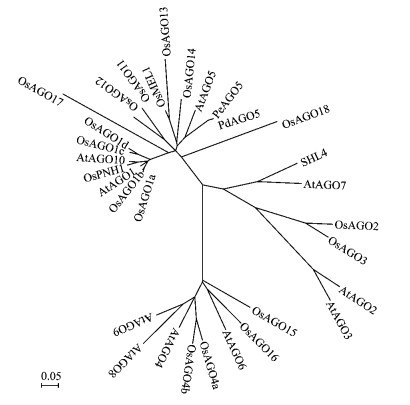
|
图 3 PeAGO5/PdAGO5与拟南芥、水稻AGO蛋白家族系统进化树分析 Figure 3 Phylogenetic tree of PeAGO5/PdAGO5 and Arabidopsis thaliana/rice AGO family proteins At:拟南芥 Arabidopsis thaliana; Os:水稻 Oryza sativa; Pe:欧美杨 Populus×euramericana; Pd:美洲黑杨 Populus deltoides. |
为了检测PeAGO5基因在杨树不定根发育中的表达模式,分别收集硬枝插穗水培生根过程中5个不同发育时间点的生根材料,以UBQ为内参基因,进行qRT-PCR分析。结果显示,PeAGO5在5个不同发育时间点存在显著差异表达(图 4)。该基因在休眠皮层中表达量最高,随着愈伤组织的产生和新生根的出现,该基因的表达量逐渐降低,并在初生不定根时最低,从这个结果看,该基因是抑制不定根的发生或负调控因子; 然而在形成侧根时,该基因表达量又升高很多,暗示它可能参与根系形态发育。在拟南芥中,AtAGO5蛋白是AtAGO1较为密切的旁系同源体(the second-closest paralogue of AGO1),虽然ago5突变体没有表现出明显的发育缺陷(Vaucheret,2008),但AtAGO1不仅能够调节激素介导的不定根发生(Sorin et al., 2005),而且独立于SHR/SCR途径调控根的辐射形态建成(Miyashima et al., 2009)。因此,推测PeAGO5基因在杨树扦插生根过程中,既参与不定根的形成,又调控根的辐射形态建成。由于南林3244杨属于难生根无性系,收集的水培生根材料有限,因此,未能进行PdAGO5基因的qRT-PCR表达分析。
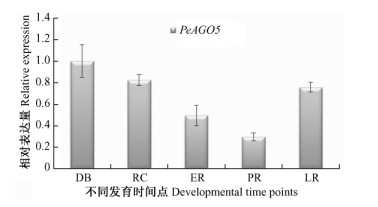
|
图 4 PeAGO5基因在杨树扦插生根中的表达模式 Figure 4 Expression pattern of PeAGO5 during Populus adventitious root development DB:休眠皮层 Dormant bark tissues; RC:愈伤组织 Root calli; ER:新生根 Emerging roots; PR:初生不定根 Primary adventitious roots; LR:侧根 Lateral roots. |
林木世代长、高度杂合、遗传负荷大。在林木中开展AGO蛋白家族成员的分离、鉴定和功能研究将会为全面深入阐明这类蛋白的调控机制奠定基础。杨树是首个完成基因组序列测定的模式树木,生长迅速,无性繁殖容易,种间可杂交,组织培养和遗传转化体系比较完善,遗传学研究有较好基础,其功能基因组学的研究将为其他林木的同类研究提供理论和方法上的直接借鉴(Tuskan et al., 2006; Jansson et al., 2007; 胥猛等,2009)。
前期研究结果表明:杨树POPTR_0001s22120.1基因在南林895杨和南林3244杨硬枝扦插生根过程中差异表达(Zhu et al., 2007)。本研究以此为基础,分别从南林895杨和南林3244杨不定根中分离其cDNA克隆,2个目的cDNA片段均含有完整的开放阅读框; 二者编码氨基酸序列间仅有7个差异位点,均含有高度保守的PAZ和Piwi结构域,具有切割活性所必需的催化三联体DDH (Tolia et al., 2007); 与拟南芥AtAGO1,AtAGO5和AtAGO10同属一个进化分支,与AtAGO5蛋白相似性分别为64.8%和64.3%。进一步分析表明,PeAGO5和PdAGO5蛋白具有相似的氨基酸组成和二级结构,这种序列上的高度同源性和结构上的高度相似性说明它们在杨树不定根发育中具有相似的功能。序列相似性比对和基因表达谱分析是推测基因功能的重要途径。qRT-PCR技术以其特异性强、灵敏度高、重复性好等优点成为基因表达模式分析的重要工具,已经被广泛应用于植物功能基因组学研究(Czechowski et al., 2004; Ruijter et al., 2009)。在此研究中,采用该技术分析了PeAGO5基因在杨树硬枝扦插生根过程中的表达情况,结果揭示,PeAGO5既参与不定根的发生,又调控侧根辐射形态建成。
对PeAGO5/PdAGO5等扦插生根相关基因的分离克隆及进一步的功能鉴定不仅有利于深入了解这些基因在林木生长发育中的作用机制,也是改良生根性状的基础。本研究从序列上的同源性和结构上的相似性以及基因表达模式,探讨了PeAGO5/PdAGO5基因可能的生物学功能,但尚需通过构建过量或RNAi表达载体进行同源或异源转化才能最 终鉴定它们在发育进程中的作用。
李超, 杜志游, 陈集双. 2009. 解读AGO蛋白结构及其功能[J]. 中国生物化学与分子生物学报, 25(11): 969-976. |
胥猛, 潘惠新, 张博, 等. 2009. 林木遗传改良种的分子生物学研究进展[J]. 林业科学, 45(1): 136-143. DOI:10.11707/j.1001-7488.20090124 |
Czechowski T, Bari R P, Stitt M, et al. 2004. Real-time RT-PCR profiling of over 1400 Arabidopsis transcription factors: unprecedented sensitivity reveal novel root-and shoot-specific genes[J]. Plant J, 38(2): 366-379. DOI:10.1111/tpj.2004.38.issue-2 |
Jansson S, Douglas C J. 2007. Populus: A model system for plant biology[J]. Annu Rev Plant Biol, 58: 435-458. DOI:10.1146/annurev.arplant.58.032806.103956 |
Kapoor M, Arora R, Lama T, et al. 2008. Genome-wide identification, organization and phylogenetic analysis of Dicer-like, Argonaute and RNA-dependent RNA polymerase gene families and their expression analysis during reproductive development and stress in rice[J]. BMC Genomics, 9: 451. DOI:10.1186/1471-2164-9-451 |
Liu Qili, Yao Xiaozhen, Pi Limin, et al. 2009. The ARGONAUTE10 gene modulates shoot apical meristem maintenance and establishment of leaf polarity by repressing miR165/166 in Arabidopsis[J]. Plant J, 58(1): 27-40. DOI:10.1111/j.1365-313X.2008.03757.x |
Miyashima S, Hashimoto T, Nakajima K. 2009. ARGONAUTE1 acts in Arabidopsis root radial pattern formation independently of the SHR/ SCR pathway[J]. Plant Cell Physiol, 50(3): 626-634. DOI:10.1093/pcp/pcp020 |
Mi Shijun, Cai Tao, Hu Yugang, et al. 2008. Sorting of small RNAs into Arabidopsis argonaute complexes is directed by the 5′ terminal nucleotide[J]. Cell, 133(1): 116-127. DOI:10.1016/j.cell.2008.02.034 |
Ruijter J M, Ramakers C, Hoogaars W, et al. 2009. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data[J]. Nucleic Acids Res, 37(6): e45. DOI:10.1093/nar/gkp045 |
Sorin C, Bussell J D, Camus I, et al. 2005. Auxin and light control of adventitious rooting in Arabidopsis require ARGONAUTE1[J]. Plant Cell, 17: 1343-1359. DOI:10.1105/tpc.105.031625 |
Tahir M, Law D A, Stasolla C. 2006. Molecular characterization of PgAGO, a novel conifer gene of the ARGONAUTE family expressed in apical cells and required for somatic embryo development in spruce[J]. Tree Physiology, 26(10): 1257-1270. DOI:10.1093/treephys/26.10.1257 |
Tamura K, Dudley J, Nei M, et al. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 |
Tolia N H, Joshua-Tor L. 2007. Slicer and the argonautes[J]. Nat Chem Bio, 3: 36-43. DOI:10.1038/nchembio848 |
Tuskan G A, DiFazio S, Jansson S, et al. 2006. The genome of black cottonwood, Populus trichocarpa (Torr & Gray)[J]. Science, 313(5793): 1596-1604. DOI:10.1126/science.1128691 |
Vaucheret H. 2008. Plant ARGONAUTES[J]. Trends Plant Sci, 13(7): 350-358. DOI:10.1016/j.tplants.2008.04.007 |
Xu Meng, Zhang Bo, Su Xiaohua, et al. 2011. Reference gene selection for quantitative real-time polymerase chain reaction in Populus[J]. Aanlytical Biochemistry, 408(2): 337-339. DOI:10.1016/j.ab.2010.08.044 |
Xu Meng, Zhang Bo, Yao H S, et al. 2009. Isolation of high quality RNA and molecular manipulations with various tissues of Populus[J]. Russ J Plant Physiol, 56(5): 716-719. DOI:10.1134/S1021443709050197 |
Zhu Qihui, Guo Anyuan, Gao Ge, et al. 2007. DPTF: a database of poplar transcription factors[J]. Bioinformatics, 23(10): 1307-1308. DOI:10.1093/bioinformatics/btm113 |
 2011, Vol. 47
2011, Vol. 47

