文章信息
- 王铁娟, 李伟琼, 张淑艳, 孟翔飞
- Wang Tiejuan, Li Weiqiong, Zhang Shuyan, Meng Xiangfei
- 差不嘎蒿自然种群的遗传多样性与遗传分化
- Genetic Diversity and Differentiation of Five Natural Populations of Artemisia halodendron
- 林业科学, 2010, 46(12): 171-175.
- Scientia Silvae Sinicae, 2010, 46(12): 171-175.
-
文章历史
- 收稿日期:2009-10-26
- 修回日期:2010-06-14
-
作者相关文章
差不嘎蒿(Artemisia halodendron)也称盐蒿,是菊科(Compositae)蒿属(Artemisia)的多年生半灌木,为中旱生沙生植物。可生长于流动沙丘的背风坡、半流动沙丘、半固定沙地、固定沙地和干旱坡地,在半流动、半固定沙地优势作用明显(李进,1991; 常学礼等,1996)。差不嘎蒿为四倍体,2n = 4x = 36 (乔有明等,1990; 王凌诗,2000),主要以风媒传粉。它是我国东部沙地植被的重要建群植物,主要分布于科尔沁沙地和呼伦贝尔沙地,国外也仅分布于与呼伦贝尔邻近的蒙古的东蒙古、中哈尔哈,俄罗斯的达乌里(王铁娟,2005a)。其主根粗长,根状茎发达,茎基部营养枝常贴于地面生长,并萌生不定根,扎入土壤,能够很好地抵御风蚀和沙埋等恶劣环境,其种子壁有胶质物,遇水吸湿膨胀能胶结沙粒及保水供种子萌发,是良好的固沙植物。差不嘎蒿的嫩枝及叶可入药,有止咳、镇喘、祛痰、消炎、解表之效,蒙医用于治疗慢性气管炎及支气管哮喘等(林有润,1991; 富象乾,1993)。差不嘎蒿作为重要的固沙植物,对其研究较多,但多集中在形态解剖、花粉、核型、分布格局、群落作用和生理生态学等方面(蒋林等,1993; 1996; 潮洛蒙等,1999; 周海燕等,2002; 王先之等,2004; 尹航等,2006),在分子水平上只有王铁娟等(2007)对该种及其近缘种的亲缘关系进行过研究报道。
ISSR(Inter Simple Sequence Repeat)分子标记是一种以微卫星序列为引物,进行多位点PCR扩增的技术。以其快速、稳定的特点广泛用于植物品种鉴定、遗传作图、基因定位、遗传多样性、进化及分子生态学研究中(王建波,2002; 李海生,2004; Feyissa et al., 2007; 张颖娟等,2008; Crema et al., 2009; Rossi et al., 2009)。本文采用ISSR技术对我国5个差不嘎蒿自然种群进行了分析,旨在揭示其遗传结构和遗传多样性的水平,为差不嘎蒿种质资源的利用与保护提供重要的基础资料。
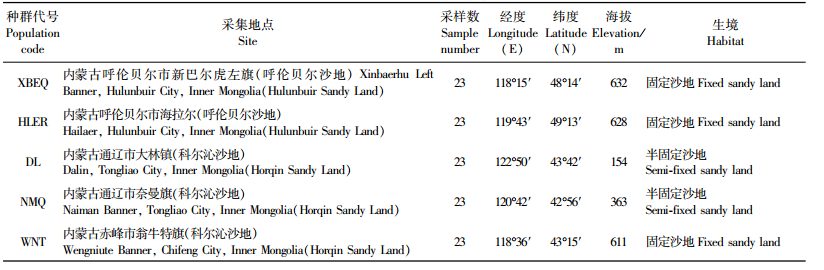
1 材料与方法 1.1 材料采集与处理根据我国差不嘎蒿的地理分布范围设置了5个采样地点,取样地点地理间隔大,有着较好的涵盖性,取样点和生境见表 1和图 1。在种群内随机选取植物,采集嫩叶,用硅胶快速干燥,带回实验室于-20 ℃下保存。
|
|

|
图 1 取样分布图 Figure 1 Map showing locations of the sample populations |
1) 基因组DNA的提取 采用CTAB法(邹喻苹等,2001; 王铁娟等,2004a)提取DNA,根据差不嘎蒿的特点略作改动。采用0.7%琼脂糖凝胶电泳检测DNA样品,并估计DNA的大致含量,用0.1 × TE将DNA浓度稀释至0.005 g·L-1,于4 ℃储存。
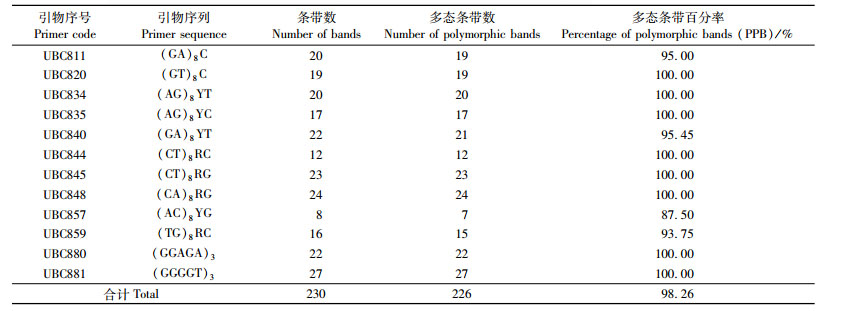
2) ISSR的反应体系 ISSR引物根据加拿大哥伦比亚大学公布的序列,由上海生工生物工程公司合成。从63条ISSR引物中筛选出12条能够扩增出清晰条带的引物(表 2),用于所有样品的扩增。经优化试验得出反应体系(20 μL)为: 0.005 g·L-1的模板DNA 6 μL,2 × Master Mix 10 μL(天为时代公司,包含Taq DNA聚合酶、dNTPs、MgCl2、反应缓冲液、PCR反应的增强剂和优化剂以及稳定剂),0.3 μmol·L-1引物1.6 μL,ddH2O 2.4 μL。每个引物设有空白对照,即以双蒸水代替模板。扩增反应在PTC-100TM型PCR仪上进行。扩增程序为: 94 ℃预变性5 min; 94 ℃变性30 s,53 ℃退火1.5 min,72 ℃延伸1 min,进行38个循环; 72 ℃延伸7 min,4 ℃保温。
|
|
3) 数据处理 用GeneRuler TM 100 bp DNA Ladder为分子量标记,同一引物的扩增产物在电泳中迁移率相同的条带被认为是同源性的,按清晰可见的强带或反复出现的弱带记为1,无带记为0,形成二元数据矩阵。计算各种群的多态条带百分率(PPB),Shannon表型多样性指数(Ho)按照Ho=-Σpi log2 pi(pi为种群中第i个条带的频率)计算(Shannon et al., 1949),并计算种群(Hpop)和物种(Hsp)水平的多样性以及种群内和种群间多样性组分[分别为Hpop/Hsp和(Hsp-Hpop)/Hsp]。应用WINAMOVA 1.55软件(Excoffier et al., 1992)计算种群内、种群间的遗传变异组分和ΦST,并计算种群间遗传距离。用Mantel软件(Mantel,1967)检测遗传距离和地理距离之间的相关性。利用NTSYS pc2.1软件(Rohlf,2000)对种群进行UPGMA聚类分析。
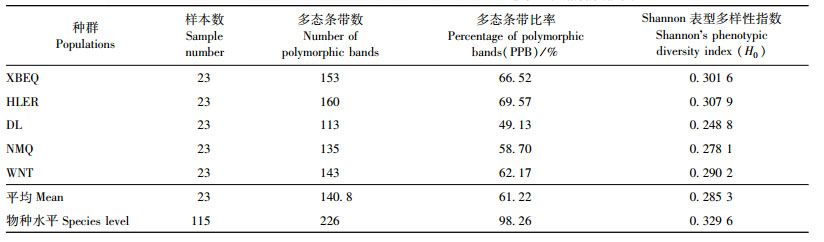
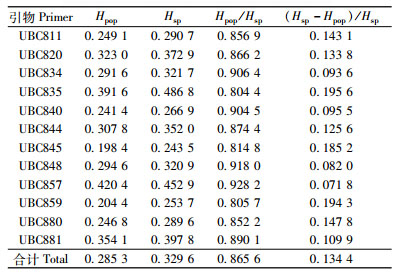
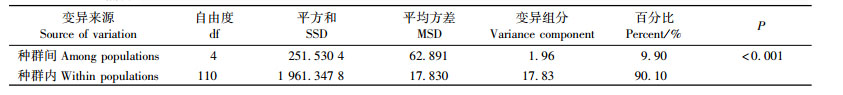
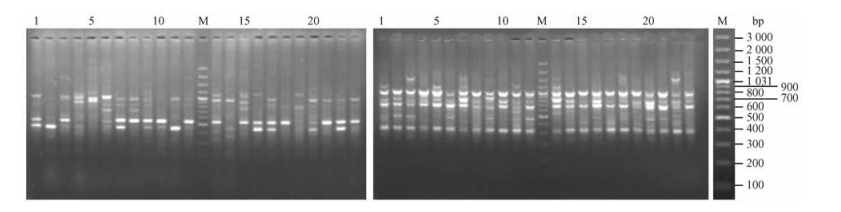
2 结果与分析 2.1 遗传多样性用筛选的12个引物对5个种群的115个样品进行ISSR-PCR分析,共得到230个条带,平均每个引物为19个,绝大多数条带集中在300 ~ 2 000 bp (图 2),其多态条带226个(表 2)。由表 3的统计结果显示,差不嘎蒿在物种水平上的多态条带百分率(PPB)为98.26%,Shannon表型多样性指数(Ho)为0.329 6;在种群水平上,多态条带百分率为61.22%,Shannon指数为0.285 3。
|
图 2 引物UBC 880对翁牛特旗种群(左)和引物UBC 811对海拉尔种群(右)的扩增结果 Figure 2 ISSR amplification results with primer UBC 880 for WNT population(left) and primer UBC 811 for HLER population (right) M:100 bp DNA分子量标准100 bp DNA ladder. |
|
|
用Shannon表型多样性指数对差不嘎蒿不同种群遗传变异的分析结果(表 4)表明,有13.44%的遗传变异存在于种群间。AMOVA分析(表 5)显示,遗传变异主要分布在种群内,占总变异的90.10%,而只有9.90%分布在种群间。2种方法均反映出差不嘎蒿种群间发生了较小的遗传分化。
|
|
|
|
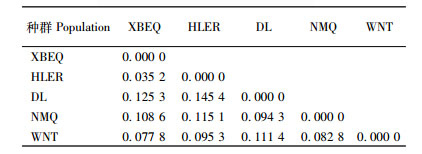
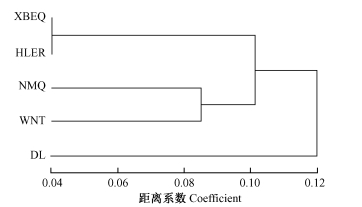
应用WIN-AMOVA 1.55软件计算的种群间ΦST遗传距离(Ci et al., 2008)见表 6,各种群的遗传距离均较小,其中遗传距离最大的是海拉尔种群(HLER)和通辽市大林种群(DL),最小的是新巴尔虎左旗(XBEQ)和海拉尔2个种群。用NTSYS-pc 2.1对5个种群的ΦST遗传距离进行UPGMA聚类分析的结果见图 3,同属呼伦贝尔沙地且遗传距离最小的海拉尔种群与新巴尔虎左旗种群首先聚到一起构成一支; 科尔沁沙地的奈曼旗(NMQ)种群和翁牛特旗(WNT)种群聚到一起构成一支,科尔沁沙地的通辽市大林种群(DL)单独构成1个分支。为研究地理距离对差不嘎蒿遗传结构的影响,对各种群间的地理距离和遗传距离进行了Mantel检验,结果表明,种群间的地理距离和遗传距离之间存在显著正相关(r = 0.65,P < 0.05)。
|
|
|
图 3 5个差不嘎蒿自然种群的UPGMA聚类图 Figure 3 Dendrogram resulting from UPGMA analysis of five natural populations of A. halodendron |
遗传多样性是物种长期进化的结果,是种群生存和发展的前提。从研究结果看,12个引物检测到230个条带,且多态条带百分率高达98.26%,与其他沙蒿类植物研究的结果接近(王铁娟等,2004a; 2004b; 2005b),各个种群在49.13% ~ 69.57%之间,大小顺序为HLER > XBEQ > WNT > NMQ > DL。Shannon表型多样性指数(Ho)为0.329 6,各种群在0.248 8 ~ 0.307 9之间,其大小顺序与多态条带百分率一致。2种方法均显示差不嘎蒿有着高的遗传多样性,以海拉尔种群(HLER)最高,通辽大林种群(DL)最低,并且呼伦贝尔沙地的2个种群高于科尔沁沙地的3个种群。这可能与科尔沁沙地受人类影响较多有关,尤其是通辽一带为农牧交错区,且是本种分布区的东南边缘,人类的干扰造成生境的片断化,种群较小从而遗传多样性较低。
根据Shannon指数和AMOVA分析种群的遗传分化,差不嘎蒿种群间发生了较小的遗传分化,2个指标的计算结果较接近,分别显示只有13.44%和9.90%的遗传变异存在于种群间。在物种水平上影响遗传变异大小的因素主要为分类地位和分布范围,而在种群水平上最为重要的为繁育系统(Hamrick et al., 1990)。差不嘎蒿虽然为狭域分布种,但作为蒿属这样进化类群的成员,高的遗传多样性使其对环境有着很好的生态适应。差不嘎蒿间断分布于呼伦贝尔沙地和科尔沁沙地,中间被大兴安岭所隔,但种群间遗传分化很小,这主要是由于本种为风媒传粉,花粉粒小,能够随风远距离传播,易于种群间的基因交流。经Mantel检验表明5个差不嘎蒿种群的地理距离和遗传距离间存在显著正相关,说明地理隔离对种群分化产生了一定的影响。
差不嘎蒿可以生长在不同固定化程度的沙地中,在半流动和半固定沙地中优势作用明显。根据常学礼等(1996)的研究,差不嘎蒿有3种繁殖方式:由被沙埋后枝条上的不定芽萌生出茎生苗,这种方式在半流动、半固定和固定沙丘上均是最主要的繁殖方式; 由地下根茎萌生出幼苗,这种方式在以上3种类型沙地中也均存在; 以种子萌发形成实生苗,流动沙丘以此种繁殖方式为主,而到了固定沙丘阶段则几乎不存在。因此在沙地群落演替的初期,种子的来源对种群的遗传多样性有着重要的影响,由于差不嘎蒿绝大部分遗传变异存在于种群内部,在治沙时,从种群内采集种子进行播种就可以达到高的遗传多样性。
常学礼, 赵学勇, 李胜功, 等. 1996. 差不嘎蒿在科尔沁沙地草场植被中的作用[J]. 中国草地, 16(1): 27-31. |
潮洛蒙, 朴顺姬, 智瑞年, 等. 1999. 不同类型沙地中差不嘎蒿种群分布格局的研究[J]. 中国沙漠, 19(1): 45-48. |
富象乾. 1993. 内蒙古植物志:第4卷[M]. 2版. 呼和浩特: 内蒙古人民出版社: 639-641.
|
蒋林, 林有润. 1993. 中国蒿属植物比较形态和解剖学研究(Ⅰ)叶表皮结构[J]. 植物研究, 13(4): 353-369. |
蒋林, 林有润. 1996. 中国蒿属植物比较形态和解剖学研究(Ⅱ)花粉形态[J]. 热带亚热带植物学报, 4(3): 1-14. |
李海生. 2004. ISSR分子标记及其在植物遗传多样性分析中的应用[J]. 生物学通报, 39(2): 19-20. |
李进. 1991. 差不嘎蒿的分布及其在天然植被演替中的地位[J]. 中国沙漠, 11(2): 55-60. |
林有润. 1991. 中国植物志:第76卷第2分册[M]. 北京: 科学出版社: 180-200.
|
乔有明, 富象乾, 阎贵兴, 等. 1990. 20种蒿属植物的染色体核型研究[J]. 中国草地, (6): 24-31. |
王建波. 2002. ISSR分子标记及其在植物遗传学研究中的应用[J]. 遗传, 24(5): 613-616. |
王凌诗. 2000. 中国东北蒿属龙蒿组植物核型研究[J]. 植物研究, 20(4): 402-410. |
王铁娟, 韩国栋. 2007. 中国蒿属沙生地理替代种亲缘关系的RAPD分析[J]. 植物研究, 27(4): 449-454. |
王铁娟, 王静, 吴恩岐. 2004a. 褐沙蒿遗传分化的研究[J]. 内蒙古师范大学学报, 33(4): 428-436. |
王铁娟, 杨持, 马静, 等. 2004b. 油蒿种群遗传分化的RAPD分析[J]. 内蒙古大学学报, 35(4): 399-403. |
王铁娟, 杨持, 吕桂芬, 等. 2005a. 中国北部六种沙蒿的地理替代规律及其主导生态因子[J]. 生态学报, 25(5): 1012-1018. |
王铁娟, 杨持, 马静, 等. 2005b. 籽蒿的地理分布与遗传分化[J]. 植物生态学报, 29(1): 122-127. |
王先之, 李锋瑞, 赵丽娅. 2004. 异龄差不嘎蒿地上生物量的分配格局[J]. 兰州大学学报, 40(6): 69-71. |
尹航, 朴顺姬, 王振杰, 等. 2006. 科尔沁沙地差巴嘎蒿群落及种群生态特征[J]. 应用生态学报, 17(7): 1169-1173. |
张颖娟, 王玉山. 2008. 濒危小灌木长叶红砂种群的遗传多样性[J]. 林业科学, 44(12): 43-47. DOI:10.3321/j.issn:1001-7488.2008.12.008 |
周海燕, 赵爱芬. 2002. 科尔沁草原主要牧草冷蒿和差不嘎蒿的生理生态学特性与竞争机制[J]. 生态学报, 22(6): 895-900. |
邹喻苹, 葛颂, 王晓东. 2001. 系统与进化植物学中的分子标记[M]. 北京: 科学出版社: 9-41.
|
Ci Xiuqin, Chen Junqiu, Li Qiaoming, et al. 2008. AFLP and ISSR analysis reveals high genetic variation and inter-population differentiation in fragmented populations of the endangered Litsea szemaois (Lauraceae) from Southwest China[J]. Plant Syst Evol, 273(3/4): 237-246. |
Crema S, Cristofolini G, Rossi M, et al. 2009. High genetic diversity detected in the endemic Primula apennina Widmer (Primulaceae) using ISSR fingerprinting[J]. Plant Syst Evol, 280(1/2): 29-36. |
Excoffier L, Smouse P E, Quattro J M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data[J]. Genetics, 131: 479-491. |
Feyissa T, Nybom H, Bartish I V, et al. 2007. Analysis of genetic diversity in the endangered tropical tree species Hagenia abyssinica using ISSR markers[J]. Genet Resour Crop Evol, 54(5): 947-958. DOI:10.1007/s10722-006-9155-8 |
Hamrick J L, Godt M J W. 1990. Allozyme diversity in plant species// Brown A H D, Clegg M T, Kahler A L, et al. Plant Population Genetics, Breeding, and Genetic Resources. Sunderland, MA: Sinauer, 43-63.
|
Mantel N. 1967. The detection of disease clustering and a generalized regression approach[J]. Cancer Res, 27: 209-220. |
Rohlf F J. 2000. NTSYS-pc: Numerical Taxonomy and Multivariate Analysis System: Version 2. 1. Exeter Software, Setauket, New York. http://www.scienceopen.com/review?vid=7890bbd2-8f00-4e7a-9a9d-dc6877647ced
|
Rossi A A B, de Oliveira L O, Venturini B A, et al. 2009. Genetic diversity and geographic differentiation of disjunct Atlantic and Amazonian populations of Psychotria ipecacuanha (Rubiaceae)[J]. Genetica, 136(1): 57-67. DOI:10.1007/s10709-008-9309-z |
Shannon C E, Weaver W. 1949. The mathematical theory of communication[M]. Urban: University of Illinois Press.
|
 2010, Vol. 46
2010, Vol. 46