文章信息
- 段续伟, 邓舒, 沈元月, 冯永庆, 秦岭
- Duan Xuwei, Deng Shu, Shen Yuanyue, Feng Yongqing, Qin Ling
- 板栗CmAPs基因的克隆及在芽变短雄花序中的表达分析
- Cloning and Expression Analysis of CmAPs in Mutant Short Catkin of Castanea mollissima
- 林业科学, 2010, 46(12): 49-55.
- Scientia Silvae Sinicae, 2010, 46(12): 49-55.
- DOI: 10.11707/j.1001-7488.20101208
-
文章历史
- 收稿日期:2009-09-17
- 修回日期:2010-01-18
-
作者相关文章
板栗(Castanea mollissima)为壳斗科(Fagaceae)栗属(Castanea)植物,是我国特有的优良坚果类果树,其果实营养丰富,含有人体所需的大量营养元素。板栗雌雄异花同株,同一植株雌雄花朵比例可达1: 2 400 ~ 1: 4 000,过多的雄花发育形成,消耗树体营养,影响雌花的发育,是限制板栗高产的重要原因之一,因此选育雌雄花比例大、产量高的品种是板栗育种的目标之一。项目组在北京密云发现的极短雄花序变异类型是一种珍贵的种质资源,其雄花序短小,短雄花序发育过程中,花序上部变黄,弯曲,最后枯死脱落,而基部花序正常发育仍具有授粉能力(冯永庆等,2005)。利用透射电镜观察花序上部发育中的细胞超微结构发现:细胞呈现有序的死亡变化,细胞核内发生染色质凝聚,继而核仁消失,核质降解,核膜破裂,核解体; 液泡内吞现象明显,形成很多小液泡; 叶绿体和线粒体也逐渐解体; 花序上部衰亡过程中所有细胞器在膜包裹下有序地降解,表现出植物细胞程序性死亡的典型形态学特征(张婧等,2007),由此证明了细胞的程序性死亡的发生诱导了芽变雄花序的短缩。
天冬氨酸蛋白酶(aspartic proteinases,APs)是一类重要的蛋白水解酶,参与机体的新陈代谢及生物调控作用。广泛分布于动物、植物、真菌、酵母,一些细菌和病毒,组成了4个不同的蛋白水解酶超家族(Davies,1990; Rawlings et al., 1995)。APs存在于单子叶植物(Doi et al., 1980; Belozersky et al., 1989; Sarkkinen et al., 1992)、双子叶植物(Polanowski et al., 1985; Rodrigo et al., 1989)和裸子植物(Bourgeois et al., 1991),这类酶在不同植物的不同部位被发现。它们具有一个共同的特征就是其活性位点都包含有2个催化性天冬氨酸残基,但其在不同植物不同组织中的确切功能并未被充分认知(Mutlu et al., 1999)。在种子发育和萌芽过程中该类酶被认为参与各种分解蛋白功能,参与了甘蓝(Brassica) (D'Hondt et al., 1993)和拟南芥(Arabidopsis thaliana) (Gruis et al., 2002)2S清蛋白的成熟和麦类种子贮藏蛋白水解启动(Belozersky et al., 1989)。在导管元件和筛胞(Rodrigo et al., 1991; Runeberg-Roos et al., 1998)发育过程中,APs在致病性蛋白降解过程中起了很重要的作用。众多数据表明在大麦(Hordeum vulgare)的生命周期的不同阶段,大麦天冬氨酸蛋白酶在蛋白质加工和代谢中起着不同的作用(Paris et al., 1996; Runeberg-Roos et al., 1991; Törmäkangas et al., 1994),而且,大麦天冬氨酸蛋白酶参与大麦根导管元件发育过程中的程序性死亡(Lindholm et al., 2000; Runeberg-Roos et al., 1998)。在拟南芥的繁殖和胚胎发育中证实1个拟南芥APs起到了抑制程序性死亡的作用(Ge et al., 2005)。
本试验以板栗正常雄花序和芽变短雄花序为试材,拟克隆获得天冬氨酸蛋白酶基因,并对其基因及其编码蛋白序列进行分析,同时,采用实时荧光定量PCR技术对不同时期的天冬氨酸蛋白酶基因表达量进行检测和分析,研究天冬氨酸蛋白酶在芽变雄花序短缩过程中的变化和作用,以揭示芽变板栗短雄花序形成的分子机制。
1 材料与方法 1.1 试验材料试材为板栗正常花序和芽变短雄花序,源于北京市密云板栗产区。从2008年5月3日花序萌发开始,到5月27日花序出现雌花簇为止,从芽变枝与正常枝同时采集雄花序,共采样7次,3个重复。
1.2 总RNA的提取及cDNA第1链的合成用EASYspin植物RNA快速提取试剂盒(北京博迈德科技发展有限公司)提取芽变花序和正常花序总RNA。以提取的总RNA为模板,利用Clontech SMARTTM Library试剂盒(购自Clontech)合成3 ’-RACE和5’-RACE cDNA第1链。
1.3 板栗天冬氨酸蛋白酶基因序列的克隆根据GenBank中公布的木薯(Manihot esculenta) (AF266465.1)、可可(Theobroma cacao) (AJ313384.1)、豇豆(Vigna unguiculata) (U61396.2)等植物的天冬氨酸蛋白酶的基因序列的保守区域设计兼并引物APs-s1: TGGDWCTCR AGGTGTAAGTA和APs-a1: AGSTWAWGCAGSYT CVGCAA,以正常花序cDNA为模板进行PCR扩增。PCR产物经回收、纯化,与PMD19-T Vector(购自宝生物工程公司)连接,转化DH5α感受态细胞,蓝白斑筛选后测序(上海生工生物工程技术服务有限公司)。
利用得到的主要片段分别设计3’和5’端引物,APsL: GCCCAACGACTATCATCACT,APsR: GACAAACTGCCACAATCAAC,UPM通用引物来自RACE试剂盒。根据试剂盒说明,转录产物的3’末端用APsL和UPM引物扩增,其PCR反应条件为: 94 ℃预变性5 min; 94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸2 min,32个循环; 72 ℃延伸10 min。转录产物的5’末端使用APsR和UPM引物扩增,其PCR反应条件为: 94 ℃预变性5 min; 94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸2 min,32个循环; 72 ℃延伸10 min。PCR产物经回收、纯化,与PMD19-T Vector连接,转化DH5α感受态细胞,蓝白斑筛选后测序。
1.4 核酸与氨基酸的序列分析及进化分析用DNAMAN5.2.2软件拼接以上序列得到板栗天冬氨酸蛋白酶基因全长cDNA序列。根据测得的核酸序列,由ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析天冬氨酸蛋白酶基因的开放读码框; 利用BLAST软件(http://www.ncbi.nlm.nih.gov/BLAST)进行序列相似性分析; 利用ProtScale (http://ca.expasy.org/tools/protscale.html)分析氨基酸序列的亲/疏水性; 使用TMHMM (http://genome.cbs.dtu.dk/services/TMHMM)对蛋白质进行跨膜分析; 蛋白质结构的综合预测采用CPHmodels 2.0 Server在线分析和SMART在线分析。采用MEGA4.0进行蛋白序列比对和进化树构建。
1.5 实时荧光定量PCR分析分7个时期从芽变枝与正常枝同时采集雄花序,分别提取RNA,使用M-MuLV First cDNA Synthesis Kit(购自上海生工生物工程技术服务有限公司)合成第1链cDNA,按照SYBR Premix Ex Taq试剂盒(TaKaRa)操作指南,采用实时荧光定量PCR(real-time quantitative PCR,RT-qPCR)的方法,检测基因的相对表达量。为避免DNA污染,设计了跨内含子引物,APs-s2: 5′-GTCCAATGGGAGAA TCAGCA-3′; APs-a2: 5′-CAGAGC GGTAAATCCA CTAA-3′,产物长度为161 bp。以actin(ev253704)为内参基因设计引物,A-s: 5′-TTGACTATGAG CAGGAACTT-3′; A-a: 5′-TTGTAGGTGGTCTCGTGA AT-3′,产物长度为178 bp。对反转录所得的cDNA按体积分别进行梯度稀释(1,1/10,1/100,1/1 000,1/10 000),实施荧光定量反应,然后绘制相对标准曲线。CmAPs基因RT-qPCR扩增的反应体系为20 μL,包括: 10 μL SYBR premix Ex Taq混合液,2 μL cDNA,0.4 μL APs-s (10 μmol·L-1),0.4 μL APs-a(10 μmol·L-1),7.2 μL ddH2O。反应程序为: 94 ℃预变性2 min; 94 ℃变性20 s,58 ℃退火30 s,72 ℃延伸30 s,40次循环,每次循环第3步进行荧光采集; 最后95 ℃变性1 min,退火至55 ℃ (每隔10 s上升0.15 ℃)后保温1 min,接着检测其荧光值,绘制熔点曲线。内参基因的actin PCR扩增的反应体系为20 μL,包括: 10 μL SYBR premix Ex Taq混合液,2 μL cDNA,0.4 μL A-s (10 μmol·L-1),0.4 μL A-a(10 μmol·L-1),7.2 μL ddH2O。其反应程序与CmAPs基因的反应程序相同,标准品cDNA和待测样品均设置3次重复。
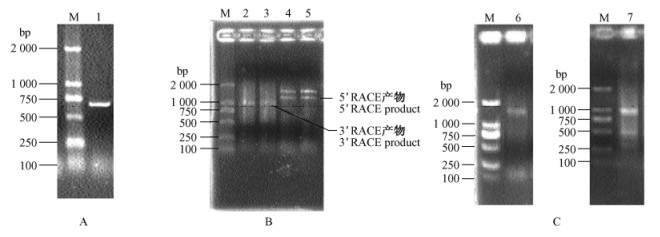
2 结果与分析 2.1 天冬氨酸蛋白酶基因的克隆与序列比较以正常花序RNA反转录的cDNA为模板,扩增出1条665 bp的单一特异性条带(图 1A)。回收纯化产物后经测序并进行序列同源性比较表明,该片段与蓖麻(Ricinus communis) (EEF32480.1)、可可(CAC86003.1)和葡萄(Vitis vinifera) (XP_002279049.1)的该家族基因分别具有86%,81%和81%的氨基酸序列相似性,证明该扩增产物是天冬氨酸蛋白酶基因片段,这是进一步通过RACE法克隆CmAPs基因全长序列的前提条件。
|
图 1 CmAPs基因的扩增结果 Figure 1 The PCR result based on CmAPs gene A. CmAPs基因片段的PCR扩增结果; B. CmAPs基因在正常花序中的3’RACE和5’RACE PCR扩增结果; C. CmAPs基因在芽变花序中的3’-RACE和5’-RACE PCR扩增结果。M: DNA marker; 1: RT-PCR扩增产物; 2,3,7: 3’-RACE扩增产物; 4,5,6: 5’-RACE扩增产物。A. The PCR result based on CmAPs gene fragment; B. The PCR results of 3’RACE and 5’RACE for CmAPs in the normal catkin; C. The PCR results of 3’RACE and 5’RACE for CmAPs in the mutant short catkin. M: DNA marker; 1: RT-PCR amplified fragment of CmAPs gene; 2,3,7: 3’-RACE amplified fragment of CmAPs gene; 4,5,6: 5’-RACE amplified fragment of CmAPs gene. |
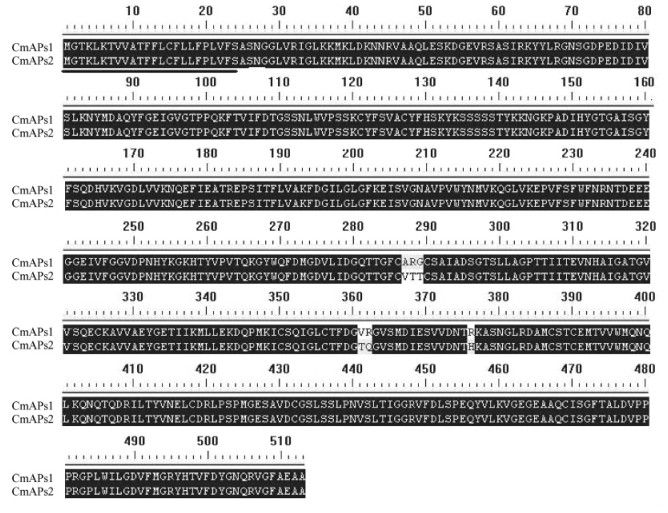
根据3’RACE和5’RACE测序结果(图 1B,C)拼读出正常花序和芽变花序中板栗天冬氨酸蛋白酶基因,全长序列分别为1 822 bp和1 909 bp。对该基因的序列进行分析表明,基因内部含有完整的开放阅读框架,大小均为1 539 bp,可编码513个氨基酸的完整阅读框架,包括起始密码子和终止密码子。利用DNAMAN5.2.2软件,对CmAPs基因编码的氨基酸序列分析,认为该基因具有完整开放阅读框,是板栗天冬氨酸蛋白酶基因的全长序列; 在对正常花序和芽变花序的cDNA为模板进行多次PCR扩增、测序,以排除测序误差后发现,在板栗正常花序和芽变花序中都存在着2个APs,暂命名为CmAPs1和CmAPs2(登陆号分别为GQ984143和GQ984144)。对二者序列比较得知CmAPs1和CmAPs2的cDNA序列上存在着少部分碱基差异,导致氨基酸序列的第287,288,289,361,362和376氨基酸发生差异(图 2)。
|
图 2 CmAPs1与CmAPs2氨基酸序列比对及差异位点 Figure 2 The Differences between predicted amino acid sequence of CmAPs1 and CmAPs2 划线部分24个氨基酸为转运肽信号。The putative signal of transit peptide is underlined. |
SignalP 3.0 Server在线分析表明CmAPs1和CmAPs2的前24个氨基酸疏水性较强,可能在第24 (Ser)和第25(Ala)氨基酸处存在信号肽切割位点。Expasy中ProtScale在线进行疏水性分析可见1 ~ 24位的区段具有典型信号肽特征,这与信号肽预测结果完全吻合。而且CmAPs1和CmAPs2亲疏水性在287 ~ 290位区段,还有361,362和372位存在差异,但是并未发生跨膜变化。
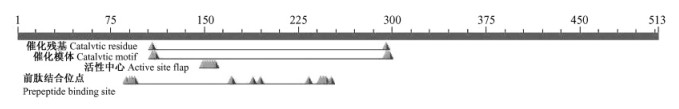
根据http://www.expasy.org/tools/pi_tool.html在线软件对编码氨基酸分子质量和等电点进行分析得到CmAPs1和CmAPs2的分子质量约为56 ku,CmAPs1预测pI值为5.77,CmAPs2的pI值为5.51。根据SMART和BLAST在线对编码蛋白的结构域和功能位点进行分析表明,Asp108,Asp295位2个催化性天冬氨酸残基组成它的活动中心,催化序为DTGS和DSGT,149 ~ 159位点DIHYGTGAISG为CmAPs的活性部位,Met86,Gln89,Tyr90,Phe91,Glu93,Asp170,Leu171,Ile187,Lys193,Phe232,Gly241,Gly242,Glu243,Ile244,Val245,Phe246,Asp250为CmAPs的肽结合位点(图 3)。
|
图 3 CmAPs氨基酸序列结构分析图 Figure 3 Amino acid sequence structure analysis of CmAPs |
利用CPHmodels 2.0 Server在线分析CmAPs1和CmAPs2的二级结构发现二者的二级结构存在着7处差异(图 4)。前2个差异处于活性中心区,初步推测它们的功能和活性可能也有所不同。

|
图 4 CmAPs1(左)和CmAPs2(右)的二级结构预测 Figure 4 The putative secondary structure of CmAPs1 (left) and CmAPs2 (right) 方框部分为CmAPs1和CmAPs2的二级结构差异点。The boxes are the differences between CmAPs1 and CmAPs2. |
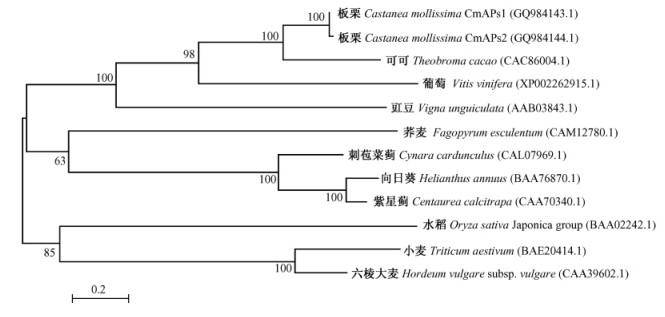
基于包括板栗在内的11种植物氨基酸序列,采用MEGA4.0软件以邻接法(Neighbor-Joining)构建进化树,建树模型选用Poisson correction距离法(图 5),并进行1 000次置信度检验。图 5显示进化树主要分为3大分支:板栗在遗传进化上与可可首先聚在一起,再与葡萄、豇豆形成一大类,蓼科(Polygonaceae)的荞麦(Fagopyrum esculentum)与刺苞菜蓟(Cynara cardunculus)、向日葵(Helianthus annuus)、紫星蓟(Centaurea calcitrapa)等菊科(Asteraceae)植物亲缘关系相对较近,形成一大类,禾本科(Poaceae)的六棱大麦(Hordeum vulgare subsp.vulgare)、小麦(Triticum aestivum)、水稻(Oryza sativa japonica group)为一分支。
|
图 5 基于APs氨基酸序列构建的系统进化树(NJ树) Figure 5 Phylogenetic tree based on APs amino acid sequence from eleven plant species (Neighbour-Joining) 节点旁数据为1 000次bootstrap检验后置信度值。Numbers at nodes represent bootstrap values(%) with 1 000 replicates. |
由iCycleriQTM自动测出的板栗属内参基因actin的回归方程、PCR扩增效率和相关系数分别为: Y =-3.447 8X + 34.269,105.3%和0.999*; 测出的CmAPs的回归方程、PCR扩增效率和相关系数分别为: Y =-2.551 5X + 41.755,168.3%和0.975*。说明该试验具有良好的重复性和较高扩增效率。熔点曲线分析出的actin和CmAPs均只有1个Tm值,分别为85 ℃和82.5 ℃,表明actin和CmAPs所用引物具有高度特异性,其PCR扩增产物均为目的片段,不含引物二聚体。
因为CmAPs1和CmAPs2序列差异位点很小,用荧光定量只能对二者总量进行检测,CmAPs基因在不同时期的正常花序和芽变花序中表达模式如图 6。以表达量最高的5月19日正常花序的表达量定为“1”,按照相对定量公式2-ΔΔCt作图(Kenneth et al., 2001)。由图 6可以看出,在5月19日前即板栗雄花序生长发育的雄花序原基形成期、花簇原基形成期和花朵原基形成期芽变花序中的CmAPs基因的表达量均高于正常花序,在5月19日CmAPs在芽变花序中表达量并没有和正常花序一样急剧升高,而是远远低于正常中表达量,5月19日之后基因在芽变花序和正常花序中表达量均降低,但芽变花序表达量仍低于正常花序。从同时期表达量差值的绝对值做线可以看出,在5月19日表达量差异最大,此时期芽变雄花序上部开始枯黄,处于花序发育的花被原基形成期。

|
图 6 CmAPs在不同时期正常花序和芽变花序中的表达量比较 Figure 6 Comparison of CmAPs expression between short catkins and normal catkins at different growth period |
植物APs广泛存在于不同植物的不同组织中,目前已经从种子(Doi et al., 1980; Belozersky et al., 1989; Runeberg-Roos et al., 1991; Sarkkinen et al., 1992; Stachowiak et al., 1994; Asakura et al., 1995; Hondt et al., 1993; Hiraiwa et al., 1997; Mutlu et al., 1999)、花(Heimgartner et al., 1990; Cordeiro et al., 1994; Verissimo et al., 1996)、花粉(Radlowski et al., 1996)和叶子(Rodrigo et al., 1989; 1991; Kuwabara et al., 1995; Schaller et al., 1996; Guevara et al., 2001)中被分离出来,但是APs的确切的功能还未被充分认知。本试验以板栗正常雄花序和芽变短雄花序为试材,成功克隆获得2个天冬氨酸蛋白酶基因全序列,为研究其功能提供了基础。
张婧(2007)认为板栗雄花序发育分为雄花序原基形成期、花簇原基形成期、花朵原基形成期、花被原基形成期、雄蕊原基形成期和花药形成期,通过电镜观察和TUNEL原位检测到芽变短雄花序内部细胞程序性死亡发生于花被原基形成期,从而导致雄花序的短缩。由于CmAPs1和CmAPs2序列上差异位点很小,所以本试验采用实时荧光定量PCR技术只能对不同发育时期的雄花序CmAPs1和CmAPs2基因的总表达量进行检测和分析,结果显示在发育的花被原基形成期即花序上部枯黄开始死亡时,CmAPs在芽变花序中表达量显著低于正常花序中表达量,这个时期与张婧(2007)得出的花被原基形成期发生程序性死亡的时期完全吻合。而Ge等(2005)利用反向遗传学方法证实1个拟南芥天冬氨酸蛋白酶在繁殖和胚胎发育中起到了抑制程序性死亡的作用,这与CmAPs在芽变花序表达量的变化趋势也相吻合。因此,初步推断板栗雄花序短缩与CmAPs在花被原基形成期的表达受阻存在紧密联系。
综合试验结果推测,CmAPs1和CmAPs2可能为1对等位基因,CmAPs1和CmAPs2差异位点为突变位点,但是该位点与芽变短雄花序的发育是否存在着密切关系,位点的差异导致蛋白结构的不同,功能是否也发生改变,从而导致该基因在花序发育过程中的表达发生差异,最终引起细胞程序性死亡,还需要小心去求证。CmAPs在芽变短雄花序程序性死亡过程中究竟具体起到何种作用还有待于进一步的研究。
冯永庆, 秦岭, 杨东生, 等. 2005. 板栗短雄花序芽变的主要特征特性研究[J]. 北京农学院学报, 20(3): 1-5. |
张婧, 董清华, 杨凯, 等. 2007. 板栗短雄花序异常死亡的超微结构观察[J]. 园艺学报, 34(3): 605-608. |
张婧. 2007. 板栗短雄花序发育期间细胞程序性死亡研究[J]. 北京农学院硕士学位论文. |
Asakura T, Abe K, Arai S. 1995. Evidence for the occurrence of multiple aspartic proteinases in rice seeds[J]. Biosci Biotechnol Biochem, 59(9): 1793-1794. DOI:10.1271/bbb.59.1793 |
Belozersky M A, Sarbakanova S T, Dunaevsky Y E. 1989. Aspartic proteinase from wheat seeds: isolation, properties and action on gliadin[J]. Planta, 177(3): 321-326. DOI:10.1007/BF00403589 |
Bourgeois J, Malek L. 1991. Purification and characterization of an aspartyl proteinase from dry jack pine seeds[J]. Seed Science Research, 1(3): 139-147. |
Cordeiro M, Pais M S, Brodelius P E. 1994. Tissue-specific expression of multiple forms of cyprosin (aspartic proteinase) in flowers of Cynara cardunculus[J]. Physiol Plant, 92(4): 645-653. DOI:10.1111/ppl.1994.92.issue-4 |
Davies D R. 1990. The structure and function of the aspartic proteinases[J]. Annual Review of Biophysics and Biophysical Chemistry, 19: 189-215. DOI:10.1146/annurev.bb.19.060190.001201 |
D'Hondt K, Bosch D, Van Damme J, et al. 1993. An aspartic proteinase present in seeds cleaves Arabidopsis 2S albumin precursors in vitro[J]. J Biol Chem, 268(28): 20884-20891. |
Doi E, Shibata D, Yonezewa D. 1980. Characterization of pepstatinsensitive acid protease in resting rice seeds[J]. Agric Biol Chem, 44(4): 741-747. |
Ge Xiaochun, Dietrich C, Matsuno, et al. 2005. An Arabidopsis aspartic protease functions as an anti-cell-death component in reproduction and embryogenesis[J]. EMBO reports, 6(3): 282-288. DOI:10.1038/sj.embor.7400357 |
Gruis D F, Selinger D A, Curran J M, et al. 2002. Redundant proteolytic mechanisms process seed storage proteins in the absence of seed-type members of the vacuolar processing enzyme family of cysteine proteases[J]. Plant Cell, 14(11): 2863-2882. DOI:10.1105/tpc.005009 |
Guevara M G, Daleo G R, Oliva C R. 2001. Purification and characterization of an aspartic proteinase from potato leaves[J]. Physiol Plant, 112(3): 321-326. DOI:10.1034/j.1399-3054.2001.1120304.x |
Heimgartner U, Pietrzak M, Geertsen R, et al. 1990. Purification and partial characterization of milk clotting proteases from flowers of Cynara cardunculus[J]. Phytochemistry, 29(5): 1405-1410. DOI:10.1016/0031-9422(90)80090-4 |
Hiraiwa N, Kondo M, Nishimura M, et al. 1997. An aspartic endopeptidase is involved in the breakdown of propeptides of storage proteins in protein-storage vacuoles of plants[J]. Eur J Biochem, 246(1): 133-141. DOI:10.1111/ejb.1997.246.issue-1 |
Kenneth J L, Thomas D S. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
Kuwabara T, Watanabe H, Tsuji S, et al. 1995. Lactate rise in the basal ganglia accompanying finger movements: a localized 1H-MRS study[J]. Brain Res, 670(2): 326-328. DOI:10.1016/0006-8993(94)01353-J |
Lindholm P, Kuittinen T, Sorry O, et al. 2000. Glycolysation of phytepsin and expression of dad1, dad2 and ost1 during onset of cell death in germinating barley scutella[J]. Mech Develop, 93(1/2): 169-173. |
Mutlu A, Gal S. 1999. Plant aspartic proteinases: enzymes on the way to a function[J]. Physiol Plant, 105(3): 569-576. DOI:10.1034/j.1399-3054.1999.105324.x |
Paris N, Stanley C M, Jones R L, et al. 1996. Plant cells contain two functionally distinguished vacuolar compartments[J]. Cell, 85(4): 563-572. DOI:10.1016/S0092-8674(00)81256-8 |
Polanowski A, Wilusz T, Kolaczkowska M K, et al. 1985. Purification and characterization of aspartic proteinases from Cucumis sativus and Cucurbita maxima seeds∥Kostka V. Aspartic proteinases and their inhibitors. Berlin: Walter de Gruyter Co., 49-52.
|
Radlowski M, Kalinowski A, Adamczyk J, et al. 1996. Proteolytic activity in the maize pollen wall[J]. Physiol Plantarum, 98(1): 172-178. DOI:10.1111/ppl.1996.98.issue-1 |
Rawlings N D, Barrett A J. 1995. Families of aspartic peptidases and those of unknown catalytic mechanism[J]. Methods Enzymol, 248(7): 105-120. |
Rodrigo I, Vera P, Conejero V. 1989. Degradation of tomato pathogenesis-related proteins by an endogenous 37-kDa aspartyl endoproteinase[J]. Eur J Biochem, 184(3): 663-669. DOI:10.1111/ejb.1989.184.issue-3 |
Runeberg-Roos P, Törmäkangas K, Ostman A. 1991. Primary structure of a barley-grain aspartic proteinase: A plant aspartic proteinase resembling mammalian cathepsin D[J]. Eur J Biochem, 202(3): 1021-1027. DOI:10.1111/ejb.1991.202.issue-3 |
Runeberg-Roos P, Saarma M. 1998. Phytepsin, a barley vacuolar aspartic proteinase is highly expressed during autolysis of developing tracheary elements and sieve cells[J]. Plant Journal, 15(1): 139-145. DOI:10.1046/j.1365-313X.1998.00187.x |
Sarkkinen P, Kalkkinen N, Tilgmann C, et al. 1992. Aspartic proteinase from barley grains is related to mammalian lysosomal cathepsin D[J]. Planta, 186(3): 317-323. |
Schaller A, Ryan C A. 1996. Molecular cloning of a tomato leaf cDNA encoding an aspartic protease, a systemic wound response protein[J]. Plant Mol Biol, 31(5): 1073-1077. DOI:10.1007/BF00040725 |
Stachowiak D, Wilimowska-Pelc A, Kolaczowska M, et al. 1994. Aspartic proteinase from the seeds of fig-leaf gourd (Cucurbita ficifolia)[J]. Acta Biochem Polonica, 41(2): 181-182. |
Törmäkangas K, Kervinen J, Östman A, et al. 1994. Tissue-specific localization of aspartic proteinase in developing and germinating barley grains[J]. Planta, 195(1): 116-125. |
Verissimo P, Faro C, Moir A J G, et al. 1996. Purification, characterization and partial amino acid sequencing of two new aspartic proteinases from fresh flowers of Cynara cardunculus L[J]. Eur J Biochem, 235(3): 762-768. DOI:10.1111/ejb.1996.235.issue-3 |
 2010, Vol. 46
2010, Vol. 46

