文章信息
- 李伟, StefanJansson, 陈晓阳
- Li Wei, Stefan Jansson, Chen Xiaoyang
- 欧洲山杨叶片衰老与光周期候选基因SNPs的关联分析
- Association Analysis Between Autumn Senescence and SNPs in the Candidate Genes of Populus tremula
- 林业科学, 2010, 46(12): 42-48.
- Scientia Silvae Sinicae, 2010, 46(12): 42-48.
- DOI: 10.11707/j.1001-7488.20101207
-
文章历史
- 收稿日期:2009-09-29
- 修回日期:2009-12-03
-
作者相关文章
2. 北京林业大学林木育种国家工程实验室 北京 100083;
3. 瑞典于默奥大学植物科学中心 于默奥 SE-90187;
4. 华南农业大学林学院 广州 510642
2. National Engineering Laboratory of Forest Tree Breeding, Beijing Forestry University Beijing 100083;
3. Umea Plant Science Center, Umea University Umea SE-90187;
4. College of Forestry, South China Agricultural University Guangzhou 510642
植物叶片衰老是一个高度调控的过程,它涉及到一系列生物大分子的降解和营养元素的重新利用(Lim et al., 2003)。这个过程中,1年生植物叶绿素中的氮元素随着叶片的脱落而丢失(Hörstensteiner,2006)。但是,对于落叶树种来说,如欧洲山杨(Populus tremula),随着秋季叶片的衰老,叶片蛋白质中的氮元素被转运到树皮储藏蛋白(bark storage proteins,BSP)中,这些蛋白在下一年春天树木生长时被重新活化和利用(Cooke et al., 2005),每年这种转移的氮元素占到叶片总氮元素的90%以上(Keskitalo et al., 2005)。对于高纬度地区的落叶树种来说,叶片的这种衰老机制是其长期生长适应的结果。如果叶片过早衰老,树木个体生长期变短,光合碳吸收减少,影响生长; 而衰老过晚,叶片会遭霜冻破坏,其中的氮元素流失,对来年树木的生长产生不利影响。由于氮元素的获取是北半球高纬度地区树木生长的主要限制因素,因此合理的秋季叶片衰老启动时间,代表了叶片中氮元素的最佳利用和碳元素合成代谢之间的一种平衡,并显示了对当地环境的一种适应(Nasholm et al., 1998; Fracheboud et al., 2009)。尽管叶片衰老研究对于落叶树种来说非常重要,但是目前关于引发和调控叶片衰老因素的了解还不是很多。最近,瑞典于默奥大学植物中心(Umea Plant Science Center)以欧洲山杨为材料,开展了有关叶片衰老的影响因素、叶片衰老的生理和生化变化(Keskitalo et al., 2005)及叶片衰老的基因差异表达(Bhalerao et al., 2003; Andersson et al., 2004)等方面的研究。最新的研究结果表明,欧洲山杨秋季叶片衰老的主要引发因素是光周期的缩短(Keskitalo et al., 2005; Fracheboud et al., 2009),树皮储藏蛋白基因的表达通过光敏色素受光周期的调控(Zhu et al., 2001)。这些发现有利地支持了欧洲山杨秋季叶片衰老受光周期调控假说。
近年来,鉴于人类遗传学领域研究中一些主要疾病基因位点的发现,利用连锁不平衡作图(linkage disequilibrium mapping,LD)定位植物复杂的数量性状核苷酸序列(quantitative trait nucleotide,QTN)成为了世界范围内的研究热点(Carlson et al., 2004)。2008年《Nature》发表的一篇论文,将LD作图称之为植物学领域的下一代遗传学,并预测在植物学领域将比在人类和动物学领域具有更广阔的应用前景(Nordborg et al., 2008)。大多数树木是异交繁殖,而且存在大量的天然群体,在进化过程中同源染色体之间发生了无数次的历史重组事件,使得在群体水平上中性的分子标记与数量性状位点之间的连锁不平衡消失,除非二者之间在群体水平上存在某一种或多种强烈的结构关系(Wilcox et al., 2007)。另外,林木具有无性繁殖、生长周期长、连年开花结实等特点,使得林木LD作图群体可多点进行、目标性状可连年观测、个体基因型长期稳定等,因此LD作图在林木复杂性状的基因定位方面更具优势。目前,国际上有关火炬松(Pinus taeda)木材密度、晚材发育(Gonzalez-Martinez et al., 2006)、纤维素含量、微纤丝角(microfibril angle) (Gonzalez-Martinez et al., 2007),桉树(Eucalyptus spp.)纤维素微纤丝角(Thumma et al., 2005),欧洲山杨花芽形成时间(Ingvarsson et al., 2008)的LD作图已有成功的报道。
单核苷酸多态性(single nucleotide polymorphism,SNP)是继限制性片段长度多态性(RFLP)和微卫星多态性(SSR)之后发展起来的第3代分子标记,它具有数量多、分布广泛、遗传稳定性高、重现性好且准确性高等特点,可以为任何一个待研究的基因内部或附近提供一系列标记,所以SNP技术是目前分析复杂性状连锁不平衡最好的工具(Choi et al.,2007)。利用SNP进行连锁不平衡作图的策略主要有2种,全基因组扫描和候选基因筛选法(Weiss et al., 2002; Hinds et al., 2005)。由于多数植物的全基因组信息有限,因此普遍采用候选基因筛选法进行关联遗传学研究。
光周期是植物感知周围环境的一种重要和稳定的外界信号,结合光周期信号的诱导,植物能够实现在最有利的时刻生长发育和繁殖。连续10年欧洲山杨秋季叶片衰老的观测,发现杨树个体叶片衰老起始的时间总是发生在一年当中相对固定的时刻,处于温度和湿度恒定的温室中的欧洲山杨个体,当光周期减少到一定程度叶片才开始衰老(Fracheboud et al., 2009)。这些研究结果揭示了光周期是决定欧洲山杨个体秋季叶片衰老的主要因素。光周期途径中涉及到很多的基因,具体与欧洲山杨秋季叶片衰老直接相关的基因或QTN目前还未见报道。本研究以光周期途径中涉及到的基因为候选基因,发掘其中的SNP位点,以无亲缘关系的自然生长的欧洲山杨为作图群体,进行候选基因SNP位点与秋季叶片衰老的关联分析。研究结果为揭示落叶树木秋季叶片衰老的分子机制及以候选基因为标记的杨树辅助选择育种(gene-assisted selection,GAS)奠定基础。
1 材料与方法 1.1 关联群体有关UmAsp (Umea Aspen)群体在Fracheboud文献中进行了详细描述。欧洲山杨在瑞典不作为用材和绿化树种,没有经过任何遗传改良和人为选择,现存的欧洲山杨群体为自然起源,反映了其自然变异。UmAsp群体建立于2006年,最初是由180个无关联的个体组成,2007年个体数扩大到了315个。为了便于表型测定和取样,沿路边选择个体并做标记,个体之间至少间隔50 m并确认属于不同的克隆。为了便于确认,每个个体拍照留存、GPS (Garmin 60 CSx)定位(Fracheboud et al., 2009)。
1.2 叶片衰老调查与统计按Fracheboud等(2009)方法建立叶片衰老对照卡(图 1)。将欧洲山杨秋季叶片衰老的整个过程分为8个阶段,分别给予0 ~ 7的分值,并配以照片,制成叶片衰老对照卡。每年自8月23日开始,以叶片衰老对照卡为标准,每周2次记录UmAsp群体中每个个体的衰老指数,直到最后一个体的叶片完全脱落为止。

|
图 1 欧洲山杨秋季叶片衰老对照卡 Figure 1 Autumn senescence score card of Populus tremula 0:全部深绿色叶片Uniformly green; 1:深绿色叶片多于浅绿色叶片More darker green leaves than paler green leaves; 2:浅绿色叶片多于深绿色叶片More paler green leaves than darker green leaves; 3:绿色叶片多于黄色叶片More green leaves than yellow leaves; 4:黄色叶片多于绿色叶片More yellow leaves than green leaves; 5:全部黄色叶片Only yellow leaves; 6:主要是金黄色或棕色叶片Mainly gold or brown leaves; 7: 90%叶片脱落More than 90% abscission. |
UmAsp群体的叶片衰老测定为2006-2008年,连续测量了3年。每年当中,每次调查分值的总和为个体叶片衰老的指数,达到分值3的时间表示叶片衰老的开始,达到分值7的时间表示叶片衰老结束,叶片从分值3 ~ 7经历的天数被认为是叶片衰老持续的时间或衰老的速率。
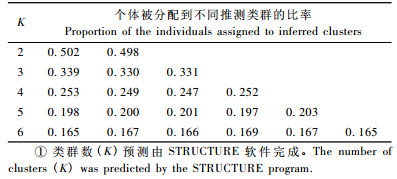
1.3 群体结构分析从毛果杨(Populus trichocarpa)基因组序列已被验证的SSR名单中(http://www.ornl.gov/sci/ipgc/Links.htm),选取6个中性的SSR位点(GCPM1831,ORPM312,ORPM327,PTR6,PTR8,FLC1B)。这些SSR位点已被应用于欧洲山杨的遗传进化研究(Hall et al., 2007)。利用荧光标记引物法对SSR片段进行扩增,采用3条引物进行PCR反应,即M13通用引物(18 bp,5 ’-TGTAAAAC GACGGCCAGT-3’)加尾的基因特异前向引物、基因特异反向引物和1条荧光标记的M13通用引物。PCR反应液在Becman-Coulter CEQ8000毛细管电泳测序仪(Becman-Coulter,Fullerton,CA)上运行,利用CEQ8000片段分析软件进行色谱显示和结果分析。将6个SSR位点在UmAsp群体中分布的数据在Structure软件上运行,分析群体结构。
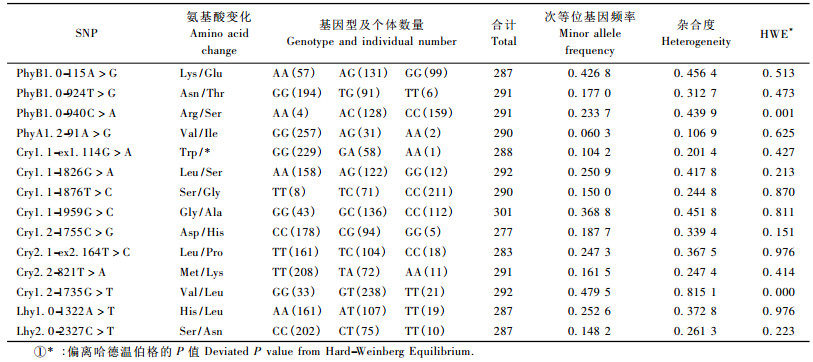
1.4 候选基因选择、SNP检测及基因型分型以光周期途径涉及的基因为候选基因(表 1),并从120个欧洲山杨个体中克隆其DNA序列,利用BioEdit软件选择引起氨基酸改变的SNP位点。
|
|
参照Latorra等(2003)的方法,针对选择的SNP位点,设计3 ’端锁定核苷酸(locked nucleic acid,LAN)的SNP特异引物。每个SNP位点设计3条引物,即同一位点上针对SNP位点的LAN特异引物各1条和另一方向的普通引物。SNP基因型分型由LAN引物与普通引物进行2次PCR反应,根据反应条带的有无确定SNP位点的分布。
1.5 统计分析欧洲山杨基因组中,染色体LD片段很短,在整个基因组平均500 bp以外的范围,LD值会迅速衰减(Ingvarsson,2005),所以很少的SNP位点可产生很多可能的单倍型; 随着单倍型数量的增多,基于单倍型的关联分析也就失去了有效性; 另外由二倍体估计可能存在的单倍型也存在很多不确定的因素。因此本研究采用了单一标记的关联分析,而未采用基于单倍型的关联分析。
根据固定效应模型,调用SAS8.0软件包GLM过程分析基因型效应对性状测定值的影响,分别采用LSMEAN进行最小二乘分析。单个位点的统计分析模型为: Y = u + G + e,式中,Y为性状测定值,u为群体均值,G为基因型效应,e为随机残差。
2 结果与分析 2.1 UmAsp群体的结构对LD作图来说,如果群体内部存在亲缘关系,可能导致虚假关联(spurious association)分析结果,使本来不存在联系的位点产生关联,或者使本来存在关联的位点导致统计学上的不关联,因此群体结构对于关联分析十分重要。在群体建立的时候已经考虑到这个问题,除了分布距离方面有明确规定外,对选择的相邻个体之间要确认属于不同的克隆。另外在分子水平上,选择了已应用于欧洲山杨遗传分化的6个中性的SSR位点,对UmAsp群体的遗传结构进行分析。数据分析采用STRUCTURE软件,此软件根据多位点基因型信息按照位点频率将群体划分为不同的类群(K),并且统计群体中个体属于不同类群的比率。结果显示,群体UmAsp中个体被分配到不同类群间的比率几乎是对称的(表 2)。如果群体内部存在亚群体或个体间存在亲缘关系,其个体被分配到不同亚群体的比率将是不对称的。因此,可以推断UmAsp群体中的个体不存在亚群体或亲缘关系。另外,对整个瑞典欧洲山杨群体(Swedish Aspen,SwAsp)的遗传分化研究结果显示,在分子水平上SwAsp采样群体中不存在亚群体(Hall et al., 2007),进一步证明了UmAsp是由无内部结构且无亲缘关系个体组成的群体,适合做SNPs与性状间的关联分析。
|
|
杨树是异交繁殖,作图群体的个体又是自然起源,因此候选基因内部存在很多的SNPs。树木个体间可遗传差异的最根本原因是控制该性状的DNA序列的改变,从而引起基因功能发生变化。因此本研究中的关联分析选择了候选基因中能够引起氨基酸改变的SNP位点。针对选择的SNP位点,设计SNP位点的特异引物,对UmAsp群体中所有个体进行基因型分型及分析(表 3)。结果显示,基因内部由于SNP引起的编码蛋白中氨基酸的改变比较普遍,本研究中此类SNPs占总SNP的30%左右,并随不同基因而有所差别,这类SNP与基因功能改变最为密切。UmAsp群体中各SNP基因型的比例差别较大,分布范围为1 ~ 257,反映了基因内部的SNP分布受基因流动或外界选择压力影响。异交繁殖的树木杂合体比例较高,本研究中欧洲山杨的杂合体比率最高为0.815,所选择的SNP位点都有很高的杂合体,这也是树木基因型分型比较困难的原因之一。在UmAsp群体中,各SNP位点的HWE (HardWeinberg Equilibrium)程度差别很大,有完全处于平衡状态的,但大部分偏离HWE平衡。其原因可能是UmAsp群体中的个体分布于整个瑞典于默奥(Umea)地区,个体间距离相差很远,不可能做到自由交配,因此也构不成孟德尔群体。这也反映了UmAsp群体是由无关联个体组成,不存在特殊结构,适合LD作图。
|
|
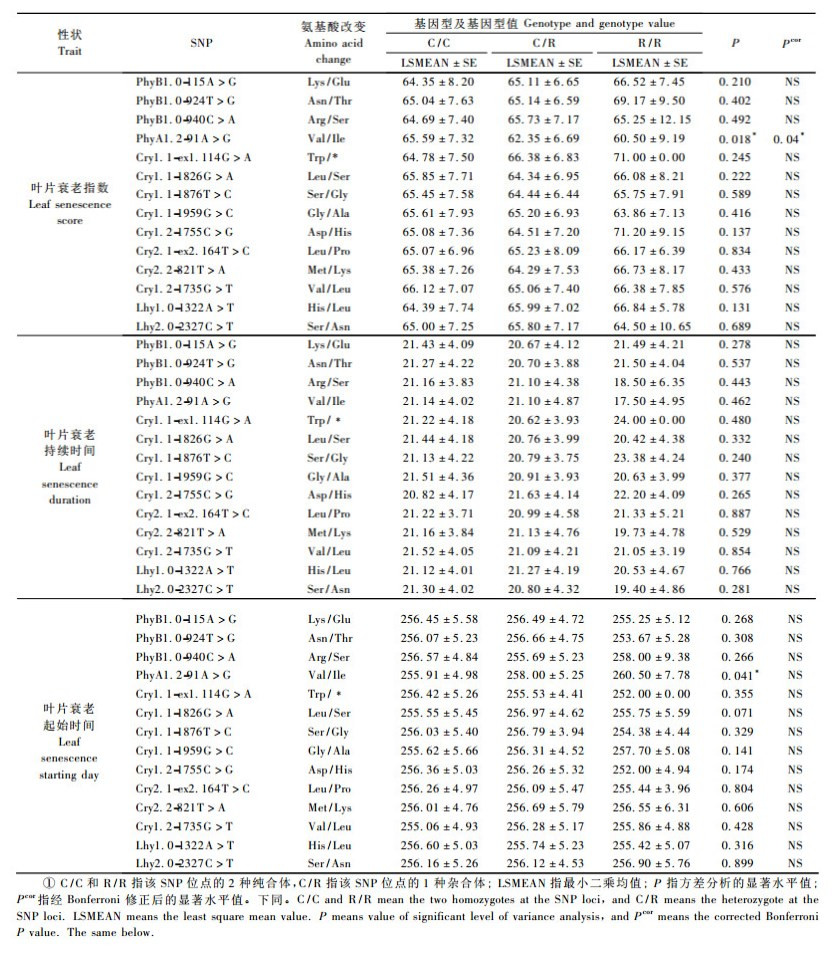
花芽形成(bud set)是叶片衰老之前另一个比较重要的发育过程,杨树花芽形成同样受光周期调控。虽然有关叶片衰老与花芽形成之间是相互独立的还是存在一定的联系,目前还没有报导,但是花芽形成是叶片衰老所必须的(Fracheboud et al., 2009)。因此,本研究叶片衰老指标中包括了叶片的衰老指数、叶片衰老持续时间和叶片衰老的起始时间。其中叶片衰老指数代表了欧洲山杨秋季衰老从花芽形成后,衰老前期准备、接受信号刺激、衰老开始、叶片脱落的整个过程。叶片衰老持续时间代表了叶片从衰老开始到叶片脱落的过程,显示了叶片衰老的速率。叶片衰老的起始时间是指在花芽形成后、叶片衰老前,叶片所经历的时间,在一定程度上代表了树木个体每年生长的时间。
结果(表 4)显示:在选择的SNP位点当中,光敏色素A中第291个核苷酸A和G的转换,导致了3种基因型AA、AG和GG产生,其基因型叶片衰老指数方差分析广义P值为0.018,经Bonferroni修正后的P值为0.04,均达到了显著水平。光周期候选基因中的SNP、叶片衰老持续时间基因型值之间没有显著差异,说明二者之间不存在线性相关。在与叶片衰老起始时间的方差分析结果当中,同样是光敏色素A中第291位点的3种基因型,方差分析广义P值为0.041,说明3种基因型值间存在显著差异,但是经Bonferroni修正后的显著水平大于0.05,又说明3种基因型值之间不存在显著差异。从结果来看,光敏色素A对于叶片的衰老很重要,除了与叶片衰老整个过程密切相关外,还与叶片衰老的起始时间可能存在某些关系。已有的研究结果表明,叶片衰老速率与外界环境有很大关系,叶片衰老一旦开始,温度能够影响衰老的速度(Fracheboud et al., 2009),这可能是所选择的SNP位点当中叶片衰老持续时间或叶片衰老速率基因型值之间存在差异但差异不显著的原因。
|
|
LD作图首先应用于人类疾病的遗传分析,随着高等植物全基因组测序的完成以及高通量基因型分型技术、测序技术发展带来的分子标记成本的降低,植物学领域的LD作图在世界范围内迅速展开(Zhu et al., 2008)。树木中LD作图可以利用无内部结构的天然群体,由于这种群体当中存在广泛的染色体历史重组事件,因此分子标记与连锁的QTN之间的遗传距离很短,使树木中数量性状位座(QTL)的图位克隆成为可能。另外,一般树木的LD程度较低,SNP更多的是位于表达基因内部或者与表达基因紧密连锁,为与QTL作图的分子标记辅助育种(marker-assisted selection,MAS)区别,基于LD作图的辅助选择育种称之为基因辅助选择育种(geneassisted selection,GAS) (Wilcox et al., 2007)。GAS可以应用于优树选择、家系间和家系内正向选择以及不同树种间的共线性选择。
光照是影响植物生长和发育的关键因素之一。植物体内感受光的受体有叶绿素、叶黄素和光敏色素,其中光敏色素(phytochrome,PHY)参与调节植物种子萌发、幼苗生长、茎的伸长、子叶伸展直至开花控制等许多生理过程(Zhu et al., 2001)。本研究结果表明,欧洲山杨个体叶片衰老差异与光敏色素A中第291个碱基的改变密切相关,在基因辅助选择育种时可以根据PHYA中第291个碱基的情况选择需要的基因型。叶片衰老持续时间与光周期候选SNPs不存在线性相关,这与前面的研究结果相一致,因为欧洲山杨一旦衰老开始,衰老持续的时间或衰老的速率与外界温度有关。在本研究的叶片衰老调查过程中也普遍存在一个现象,有些个体一直保持到很晚的时间才出现叶片颜色的变化,叶片衰老开始的时间很晚,但是一旦衰老开始,叶片很快全部脱落,这是欧洲山杨叶片衰老速率受温度控制的有利证据。叶片衰老起始时间与光周期候选基因有一定相关性,但强度不够。其原因可能是,叶片衰老只是叶片周期的一个特定阶段,在叶片衰老之前叶片要完成花芽的形成,在花芽形成之前任何的光周期都不能使叶片衰老,只有花芽形成之后,在短光照处理后叶片才出现衰老。
树木叶片的衰老是一个复杂的生理过程,欧洲山杨叶片衰老严格受光周期的调控。但是有些树种,如糖槭(Acer saccharum)、山毛榉(Fagus grandifolia)、黄桦(Betula alleghaniensis),研究结果显示其叶片衰老主要受温度控制,而且随着全球温度的升高,衰老的时间有所推迟,预示了光周期不是这些树种最主要的叶片衰老调控方式(Richardson et al., 2006)。叶片衰老机制差异的原因是高纬度地区树种的生长发育可能更容易受光周期调控。因为在高纬度地区,日照时间的变化十分明显,与季节变化相比光周期变化更为准确,这是乡土树种自然进化的结果。另外,树木叶片衰老受多种因素影响,光周期诱导的欧洲山杨叶片衰老可能与温度诱导的叶片衰老有区别。这需要进一步深入研究,因为这2种衰老机制的差异影响今后以基因为标记的落叶树种长生长周期的辅助选择育种研究。
致谢:
瑞典于默奥大学植物科学中心的 Lars Bjorken 参加了叶片衰老野外调查工作,同时还得到了 Yvan Fracheboud 的试验指导和帮助。
Andersson A, Keskitalo J, Sjodin A, et al. 2004. A transcriptional timetable of autumn senescence[J/ OL]. Genome Biology, 5 (4) : R24.1-R24.13. http://genomebiology.com/2004/5/4/R24.
|
Bhalerao R, Keskitalo J, Sterky F, et al. 2003. Gene expression in autumn leaves[J]. Plant Physiology, 131(2): 430-442. DOI:10.1104/pp.012732 |
Carlson C S, Eberle M A, Kruglyak L, et al. 2004. Mapping complex disease loci in whole genome association studies[J]. Nature, 429: 446-452. DOI:10.1038/nature02623 |
Choi I Y, Hyten D L, Matukumalli L K, et al. 2007. A soybean transcript map: gene distribution, haplotype and single nucleotide polymorphism analysis[J]. Genetics, 176: 685-696. DOI:10.1534/genetics.107.070821 |
Cooke J E K, Weih M. 2005. Nitrogen storage and seasonal nitrogen cycling in Populus: bridging molecular physiology and ecophysiology[J]. New Phytologist, 167(1): 19-30. DOI:10.1111/j.1469-8137.2005.01451.x |
Fracheboud Y, Luquez V, Sjödin A, et al. 2009. The control of autumn senescence in European Aspens (Populus tremula)[J]. Plant Physiol, 149(4): 1982-1991. DOI:10.1104/pp.108.133249 |
Gonzalez-Martinez S C, Ersoz E, Brown G R, et al. 2006. DNA sequence variation and selection of Tag single-nucleotide polymorphisms at candidate genes for drought-stress response in Pinus taeda L[J]. Genetics, 172: 1915-1926. DOI:10.1534/genetics.105.047126 |
Gonzalez-Martinez S C, Wheeler N C, Ersoz E, et al. 2007. Association genetics in Pinus taeda L.I. wood property traits[J]. Genetics, 175: 399-409. |
Hall D, Luquez V, Garcia M V, et al. 2007. Adaptive population differentiation in phenology across alatitudinal gradient in European aspen (Populus tremula L.) : a comparison of neutral markers, candidate genes and phenotypic traits[J]. Evolution, 61(12): 2849-2860. |
Hinds D A, Stuve L L, Nilsen G B, et al. 2005. Whole-genome patterns of common DNA variation in three human populations[J]. Science, 307(5172): 1072-1079. |
Hörstensteiner S. 2006. Chlorophyll degradation during senescence[J]. Annual Review of Plant Biology, 57: 55-77. DOI:10.1146/annurev.arplant.57.032905.105212 |
Ingvarsson P K, Garcia M V, Luquez V, et al. 2008. Nucleotide polymorphism and phenotypic associations within and around the phytochrome B2 locus in European aspen (Populus tremula, Salicaceae)[J]. Genetics, 178: 2217-2226. DOI:10.1534/genetics.107.082354 |
Ingvarsson P K. 2005. Nucleotide polymorphisms and linkage disequilibrium within and among natural populations of europen aspen (Populus tremula L., Salicaceae)[J]. Genetics, 169: 945-953. DOI:10.1534/genetics.104.034959 |
Keskitalo J, Bergquist G, Gardström P, et al. 2005. A cellular time table of autumn senescence[J]. Plant Physiology, 139(4): 1635-1648. DOI:10.1104/pp.105.066845 |
Latorra D, Campbell K, Wolter A, et al. 2003. Enhanced allele-specific PCR discrimination in SNP genotype using 3'locked nucleic acid (LAN) primers[J]. Human Mutation, 22: 79-85. DOI:10.1002/(ISSN)1098-1004 |
Lim P O, Woo H R, Nam H G. 2003. Molecular genetics of leaf senescence in Arabidopsis[J]. Trends in Plant Sciences, 8(6): 272-278. DOI:10.1016/S1360-1385(03)00103-1 |
Nasholm T, Ekblad A, Nordin A, et al. 1998. Boreal forest plants take up organic nitrogen[J]. Nature, 392: 914-916. DOI:10.1038/31921 |
Nordborg M, Weigel D. 2008. Next-generation genetics in plants[J]. Nature, 456: 720-723. DOI:10.1038/nature07629 |
Richardson A D, Bailey A S, Denny E G, et al. 2006. Phenology of a northern hardwood forest canopy[J]. Global Change Biology, 12(7): 1174-1188. DOI:10.1111/gcb.2006.12.issue-7 |
Thumma B R, Nolan M F. 2005. Polymorphisms in cinnamoyl CoA reductase (CCR) are associated with variation in microfibril angle in Eucalyptus spp[J]. Genetics, 173: 1257-1265. |
Weiss K M, Clark A G. 2002. Linkage disequilibrium and the mapping of complex human traits[J]. Trends in Genetics, 18(1): 19-24. DOI:10.1016/S0168-9525(01)02550-1 |
Wilcox P L, Echt C E, Burdon R D. 2007. Gene-assisted selection: application of association genetics for forest tree breeding∥Oraguzie N C, Rikkerink E H A, Gardiner S E, et al. Association mapping in plants. New York: Springer, 213-217.
|
Zhu B, Coleman G D. 2001. Phytochrome-mediated photoperiod perception, shoot growth, glutamine, calcium, and protein phosphorylation influence the activity of the poplar bark storage protein gene promoter (bspA)[J]. Plant Physiolgy, 126(1): 342-351. DOI:10.1104/pp.126.1.342 |
Zhu C S, Gore M, Buckler E S, et al. 2008. Status and prospects of association mapping in plants[J]. The Plant Genome, 1(1): 5-19. DOI:10.3835/plantgenome2008.02.0089 |
 2010, Vol. 46
2010, Vol. 46