文章信息
- 张飞, 陈发棣, 房伟民, 陈素梅
- Zhang Fei, Chen Fadi, Fang Weimin, Chen Sumei
- 菊花花器性状在F1代变异及相关联的SRAP分子标记
- Variation of Inflorescence Traits in F1 Progeny of Chrysanthemum and the Association with SRAP Markers
- 林业科学, 2010, 46(11): 162-167.
- Scientia Silvae Sinicae, 2010, 46(11): 162-167.
-
文章历史
- 收稿日期:2009-09-21
- 修回日期:2009-12-03
-
作者相关文章
2. 浙江省农业科学院花卉研究开发中心 杭州 311202
2. Flower Research and Development Centre, Zhejiang Academy of Agricultural Sciences Hangzhou 311202
花器是菊花(Dendranthema morifolium)观赏特性的重要构成因素,也是菊花品种改良的主要目标性状之一。花器性状比较复杂,主要由花序直径、舌状花数、瓣性和花色等组成。由于栽培菊花的遗传背景复杂,存在高度杂合、自交不亲和、近交衰退等现象,给菊花目标性状改良带来了很大困难。张飞等(2008)研究了花器性状在切花菊品种间的遗传变异情况; 菊花部分性状在杂种一代的变异情况也有相关报道(徐文辉等,2000; 陈发棣等,2003; 蒋甲福等,2003),但是研究的样本群体都比较小。赵静媛等(2009)成功筛选出与地被菊株型匍匐性状紧密连锁的RAPD标记并成功进行SCAR转化; 另外,种质资源评价(韩洁等,2007; 缪恒彬等,2007; 2008)、转基因(洪波等,2006; 孙磊等,2008; Yang et al., 2008; Jiang et al., 2010)、基因克隆(Miao et al., 2009)、表达序列标签(Chen et al., 2009)等在菊花研究中也有相关报道,这些研究为菊花遗传育种工作提供了一定的理论依据。
近年来,随着分子生物学的发展,数量性状基因定位(QTLs)已经成为植物数量性状遗传研究的热点之一,相关研究工作已经在杜鹃(Rhododendron) (Dunemann et al., 1999)、百合(Lilium) (Abe et al., 2002)、月季(Rosa) (Dugo et al., 2005; Oyant et al., 2008)等观赏植物中陆续报道。目前,数量性状基因定位的方法主要有方差分析法、区间作图法、复合区间作图法和混合线性模型方法等,这4种方法具有同一性,但也有各自的特点。方差分析法虽然难以确定QTLs的准确位置,但其简便易行,发现能力较高; 区间作图法可以较准确估计QTLs的效应及确定在某一特定区间的位置; 复合区间作图法可以消除在染色体上至多存在1个QTLs的假设并发现前2种方法发现不了的QTLs; 混合线性模型方法将效应估计和定位分析结合起来,进行多环境下的联合QTLs定位分析,提高了作图的精度和效率(Yang et al., 2005; 尹伟伦等,2005; 张坤谱,2008)。然而,由于方差分析法不需要以连锁遗传图谱为基础,对于像菊花等一些没有建立连锁遗传图谱的植物来说,方差分析法是检测与这类植物数量性状相关联或紧密连锁的分子标记或QTLs的首选方法,该方法已经成功应用于杨树(Populus) (李金花等,1999; 苏晓华等,2000; 黄烈健等,2004)、云南红豆杉(Taxus yunnanensis) (苏建荣等,2009)等林木或观赏植物中,但在菊花研究中尚无相关报道。相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)具有简便、快捷、稳定、高效、高共显性及在基因组中分布均匀等特点,主要是对开放阅读框进行扩增,提高了扩增结果与表现型的相关性,可以将遗传标记和基因克隆直接结合起来,其在分子生物学、遗传学等研究领域有着广泛的应用前景(Li et al., 2003; Pan et al., 2003)。鉴于上述现状,本研究在对菊花F1群体进行花器性状表型研究基础上,结合SRAP标记和单因素方差分析方法,寻找与菊花花器性状相关联的遗传标记,为进一步理解菊花花器性状的遗传基础,深入开展菊花花器性状相关基因克隆和分子标记辅助育种奠定基础。
1 材料与方法 1.1 试验材料供试材料为保存于南京农业大学中国菊花品种保存中心的夏菊品种‘奥运含笑’(父本)、秋菊品种‘雨花落英’(母本)及于2006年秋季通过两亲本之间杂交所获得的142个F1杂交后代单株。
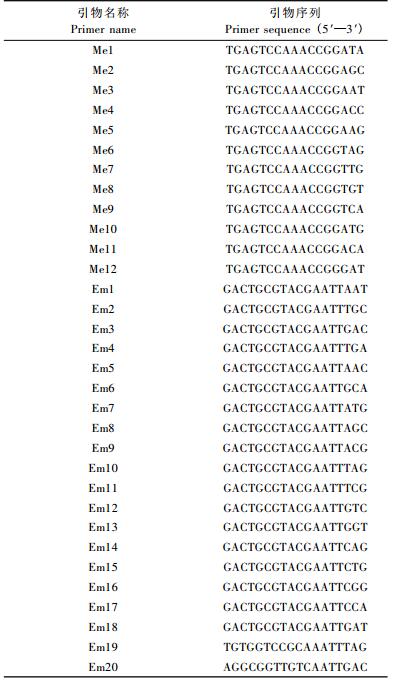
试验用的Lambda DNA、Taq DNA聚合酶、dNTPs以及100 bp DNA marker(100,300,500,700,1 000,1 500,2 000 bp)购自宝生物工程(大连)有限公司,SRAP引物(表 1)由上海英俊生物技术有限公司合成。
|
|
1) 基因组DNA的提取与检测 参照Murray等(1980)方法,菊花嫩叶采用CTAB微量法提取基因组DNA,Lambda DNA 1.0%琼脂糖凝胶电泳检测DNA质量和浓度,并用ddH2O稀释至25 ng·μL-1,-20 ℃保存备用。
2) SRAP-PCR反应与产物检测 SRAP-PCR反应体系参照张飞等(2009)方法,包括1×PCR buffer,Mg2+ 3.125 mmol·L-1、dNTPs 187.5 μmol· L-1、引物10 μmol·L-1、模板DNA 50 ng、Taq DNA聚合酶0.5 U,总体积20 μL。PCR扩增程序参照SRAP-PCR反应通用程序(Li et al., 2001) : 94 ℃预变性5 min; 94 ℃变性1 min,35 ℃复性1 min,72 ℃延伸1 min,5个循环; 94 ℃变性1 min,50 ℃复性1 min,72 ℃延伸1 min,35个循环; 72 ℃延伸10 min,10 ℃保存。扩增反应结束后,每管各加入6 ×溴酚蓝0.5 μL,离心混匀后,取2~3 μL扩增产物用8%非变性聚丙烯酰胺凝胶电泳,电极缓冲液为0.5×TBE。电泳结束后,采用陈宣等(2009)方法进行银染,最后在胶片观察灯上拍照记录、保存。
3) 花器性状的观测 于2007年盛花期,统计F1群体142个后代单株花器相关性状,包括花径、舌状花数、管状花数、花序外侧1~2轮的舌状花长和舌状花宽5个性状,单株重复测量3次。性状观测和统计方法参照李鸿渐(1993)在《中国菊花》中介绍的方法。
4) 与花器性状相关联的SRAP标记检测 采用Marker-based法(Singh et al., 1991),即根据标记基因型的种类将分离群体划分2组:有带位点记为1,无带记为0,对每一个位点按0、1将其分成2个组,对各位点中的2组进行性状差异的单因素方差分析,确定与不同数量性状相关的标记位点。用组间方差与总方差的比值求得该遗传标记位点产生的性状变异占性状总变异的百分率,即标记对该性状变异的贡献率。
1.3 数据统计分析利用Excel 2003软件进行性状和标记等基本数据的录入和整理; 性状描述型数据、柱形图及方差分析在SPSS 11.5软件中分析处理。
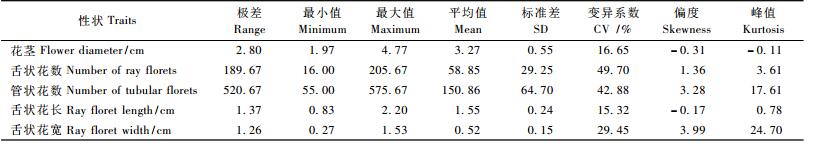
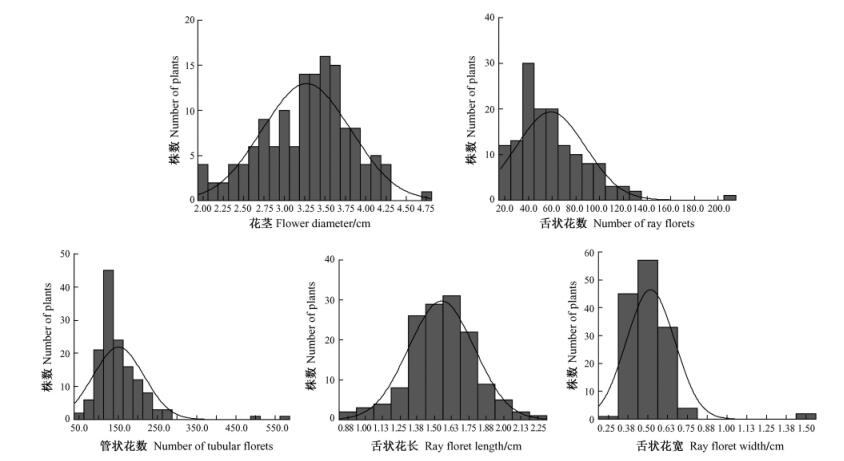
2 结果与分析 2.1 菊花花器相关性状在F1群体中的分离情况菊花花器性状表型值在F1代群体中分布的描述性统计结果见表 2。由表 2可以看出,5个花器性状表现出不同程度的变异,变异系数在15.32%~49.70%之间,其中舌状花数和管状花数的变异系数较大(40.0%以上)。说明5个花器性状在该F1代群体中个体间差异较大,表现出显著的分离情况,适合进行性状相关分子标记的筛选。从偏度和峰度结果(表 2)以及次数分布图(图 1)可知,5个性状在F1代群体中的分离情况基本符合正态分布,适合于进一步进行遗传分析。
|
|
|
图 1 菊花5个花器性状在F1代群体的次数分布图 Figure 1 Frequent distribution for the 5 inflorescence traits in the F1 population |
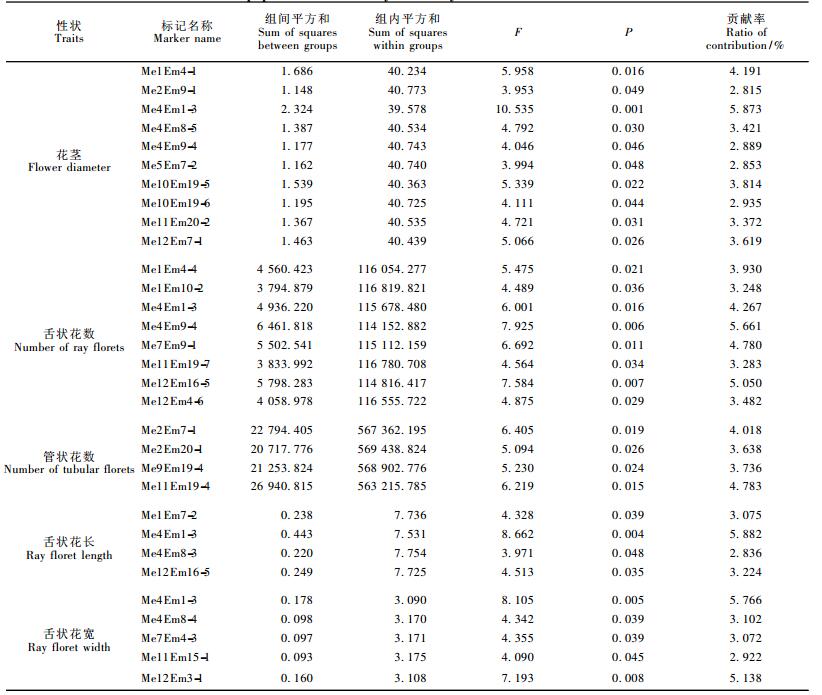
SRAP标记在菊花‘奥运含笑’、‘雨花落英’及其部分F1后代中分离的电泳图谱见图 2。通过单因素方差分析,检测到与菊花F1分离群体中5个花器性状显著关联的SRAP标记位点(表 3)。由表 3可知,与花径显著关联的SRAP标记有10个,其中极显著标记有1个,这些标记位点对花径变异的贡献率在2.815%~5.871%,累积贡献率为35.781%。与舌状花数显著关联的SRAP标记有8个,其中极显著标记有2个,这些标记位点对花径变异的贡献率在3.248%~5.661%,累积贡献率为33.702%。在所有的多态性SRAP标记中,与管状花数显著关联的遗传标记有4个,分别为Me2Em7-1,Me2Em20-1,Me9Em19-4和Me11Em19-4,对管状花数变异的贡献率分别为4.018%,3.638%,3.736%和4.783%,累积贡献率为16.175%。与舌状花长显著关联的SRAP标记位点分别为Me1Em7-2,Me4Em1-3,Me4Em8-3和Me12Em16-5,对舌状花长变异的贡献率分别为3.075%,5.882%,2.836%和3.224%,累积贡献率为15.018%,其中Me4Em1-3表现为极显著关联。在与舌状花宽关联的5个SRAP标记位点中,除Me4Em1-3和Me12Em3-1两个位点极显著关联外,其余3个表现为显著关联,各自可以解释舌状花宽变异的贡献率在2.922%~5.766%之间,累积贡献率为20%。
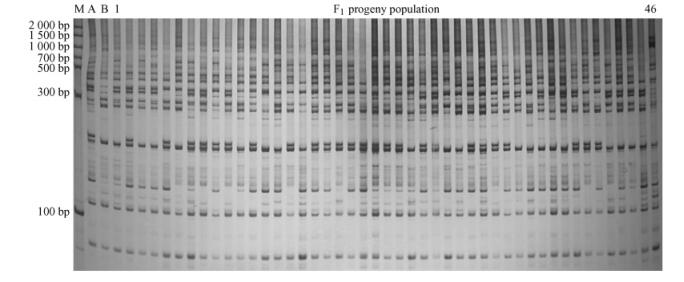
|
图 2 SRAP引物组合Me9Em19在菊花‘奥运含笑’、‘雨花落英’及其部分F1代中的扩增图谱 Figure 2 Amplification profiles of chrysanthemum 'Aoyunhanxiao', 'Yuhualuoying' and their F1 progeny with SRAP primer combination Me9Em19 M: DNA分子质量梯度DNA Marker; A: ‘奥运含笑’‘Aoyunhanxiao’; B: ‘雨花落英’‘Yuhualuoying’; 1-46: F1杂交后代F1 progeny. |
|
|
菊花是我国传统的名优花卉之一,在家庭和园林美化中具有重要的应用价值。花器性状作为菊花观赏特性的最直观表现,始终是菊花育种研究中最重要的目标性状之一。尽管有较多关于菊花常规杂交育种相关方面的研究(陈发棣等,2003; 蒋甲福等,2003),但是目前国内外均未见与菊花花器性状关联的分子标记或紧密连锁的QTLs定位研究相关报道。本研究结合菊花花器性状在F1代的表型变异及多态性SRAP标记数据,首次报道了与菊花花器性状相关联的SRAP分子标记,并获得了一些有意义的结果。通过单因素方差分析,共获得了与5个菊花花器性状显著关联的26个SRAP标记位点,筛选出与菊花花径、舌状花数、管状花数、舌状花长和舌状花宽显著相关联的SRAP标记位点,其分别为10,8,4,4和5个,其累计贡献率依次为35.781%,33.702%,16.175%,15.018%和20.0%。但是单个标记位点对目标性状变异的贡献率普遍较低,在2.815%~5.882%之间,表明其为微效多基因,且数量性状的连续变异是由于微效多基因的累加作用加上环境条件的影响而产生的。另外,对这些显著关联的标记位点进行双因素方差分析发现几乎都没有达显著水平,说明标记之间对花器性状的变异不存在交互作用(数据未列出)。
经典的数量遗传学研究表明,数量性状之间存在明显的相关性(李金花等,1999; 张飞等,2008),主要原因之一是存在一因多效现象。本研究结果为解释这类性状复杂的遗传背景及性状之间的相互关系提供了依据,即与某一数量性状关联的标记位点同时也与其他性状相关联。例如,在检测到的与菊花花器性状显著关联的SRAP标记位点中,Me4Em1-3与花径性状极显著关联,同时与舌状花数、舌状花长和舌状花宽也达到显著或极显著关联水平。
与其他作物相比较(尹伟伦等,2005; 苏建荣等,2009),本研究中检测到的单个遗传标记对目标性状的贡献率普遍较低,尽管如此,仍获得了一些比较有意义的SRAP分子标记位点,如Me4Em1-3对菊花花径、舌状花长和舌状花宽变异的贡献率分别为5.873%,5.882%和5.766%,Me4Em9-4和Me12Em16-5对舌状花数变异的贡献率分别高达5.661%和5.050%,Me12Em3-1对舌状花宽的贡献率为5.138%。借助这些与性状紧密连锁的分子标记,可以在育种中对有关QTLs的遗传行为进行动态跟踪,从而加强对数量性状的遗传操纵能力,提高育种中对数量性状优良基因型选择的准确性和预见性(黄秦军等,2004)。鉴于SRAP标记主要是对开放阅读框进行扩增,提高了扩增结果与性状的表现型的相关性(Li et al., 2003),因此,进一步应用这些与花器性状紧密关联的遗传标记或连锁的QTLs于基因克隆研究中,将会显著改善菊花分子标记辅助育种工作的效率。
当然,通过方差分析方法检测与植物数量性状相关联或紧密连锁的遗传标记或QTLs,有其客观局限性:不能确定标记是与1个还是几个QTLs连锁及其确切位置; 由于遗传效应与重组率混合在一起,容易低估QTLs的遗传效应; 容易出现假阳性等(张坤普,2008)。在本研究中,通过扩大研究群体在一定程度上减少了系统误差。许多研究表明,方差分析法和以图谱为基础的QTLs分析结果具有一致性(苏晓华等,2000; 尹伟伦等,2005),尽管如此,为了进一步了解菊花数量观赏性状的遗传基础,理想的研究手段应该是以遗传图谱为基础的分子标记或QTLs分析,相关研究工作正在进行中。
陈发棣, 蒋甲福, 郭维明. 2003. 小菊花器若干性状在F-1代的表现[J]. 园艺学报, 30(2): 175-182. |
陈宣, 郭海林, 薛丹丹, 等. 2009. 结缕草属植物耐盐性SRAP分子标记研究[J]. 草业学报, 18(2): 66-75. DOI:10.11686/cyxb20090211 |
韩洁, 胡楠, 李玉阁, 等. 2007. 菊花品种资源遗传多样性的AFLP分析[J]. 园艺学报, 34(4): 1041-1046. |
洪波, 仝证, 马男, 等. 2006. AtDREB1A基因在菊花中的异源表达提高了植株对干旱和盐渍胁迫的耐性[J]. 中国科学C辑, 36(3): 223-231. |
黄烈健, 苏晓华, 张香华, 等. 2004. 与杨树木材密度、纤维性状相关的SSR分子标记[J]. 遗传学报, 31(3): 299-304. |
黄秦军, 苏晓华, 黄烈健, 等. 2004. 美洲黑杨×青杨木材性状QTLs定位研究[J]. 林业科学, 40(2): 55-60. DOI:10.11707/j.1001-7488.20040210 |
蒋甲福, 陈发棣, 郭维明. 2003. 小菊杂种一代部分性状的遗传与变异[J]. 南京农业大学学报, 26(2): 11-15. |
李鸿渐. 1993. 中国菊花[M]. 南京: 江苏科学技术出版社.
|
李金花, 苏晓华, 张绮纹, 等. 1999. 用RAPD标记检测与杨树生长和物候期有关的QTLs[J]. 林业科学研究, 12(2): 111-117. |
缪恒彬, 陈发棣, 赵宏波. 2007. 85个大菊品种遗传多样性的ISSR分析[J]. 园艺学报, 34(5): 1243-1248. |
缪恒彬, 陈发棣, 赵宏波, 等. 2008. 应用ISSR对25个小菊品种进行遗传多样性分析及指纹图谱构建[J]. 中国农业科学, 41(11): 3735-3740. DOI:10.3864/j.issn.0578-1752.2008.11.039 |
苏建荣, 缪迎春, 张志钧. 2009. 云南红豆杉紫杉醇含量变异及其相关的RAPD分子标记[J]. 林业科学, 45(7): 16-20. DOI:10.11707/j.1001-7488.20090703 |
苏晓华, 李金花, 陈伯望, 等. 2000. 杨树叶片数量性状相关联标记及其图谱定位研究[J]. 林业科学, 36(1): 33-36. DOI:10.11707/j.1001-7488.20000106 |
孙磊, 张启翔, 周琳. 2008. 利用双T-DNA载体系统获得无选择标记转基因菊花[J]. 园艺学报, 35(5): 727-734. |
徐文辉, 高海卿, 陈华进. 2000. 菊花某些性状遗传规律的初步探讨[J]. 浙江林学院学报, 17(1): 37-14. |
尹伟伦, 胡建军. 2005. 杨树遗传图谱构建与数量性状基因定位[M]. 北京: 中国环境科学出版社.
|
张飞, 房伟民, 陈发棣, 等. 2008. 切花菊花器性状的遗传变异和相关性研究[J]. 浙江林学院学报, 25(3): 293-297. |
张飞, 陈发棣, 房伟民, 等. 2009. 菊花SRAP-PCR反应体系的优化与确立[J]. 植物资源与环境学报, 18(3): 44-49. |
张坤谱. 2008. 小麦分子遗传图谱的构建与数量性状基因定位. 山东农业大学博士学位论文. http: //cdmd. cnki. com. cn/Article/CDMD-10434-2008206854. htm
|
赵静媛, 陈素梅, 陈发棣. 2009. 与地被菊株型匍匐性连锁RAPD标记的SCAR转化[J]. 林业科学, 45(9): 147-150. DOI:10.11707/j.1001-7488.20090925 |
Abe H, Nakano M, Nakatsuka A, et al. 2002. Genetic analysis of floral anthocyanin pigmentation traits in Asiatic hybrid lily using molecular linkage maps[J]. Theoretical and Applied Genetics, 105(8): 1175-1182. DOI:10.1007/s00122-002-1053-7 |
Chen Sumei, Miao Hengbin, Chen Fadi, et al. 2009. Analysis of expressed sequence tags (ESTs) collected from the inflorescence of chrysanthemum[J]. Plant Molecular Biology Reporter, 27(40): 503-510. |
Dugo M L, Satovic Z, Millan T, et al. 2005. Genetic mapping of QTLs controlling horticultural traits in diploid roses[J]. Theoretical and Applied Genetics, 111(3): 511-520. DOI:10.1007/s00122-005-2042-4 |
Dunemann F, Kahnau R, Stange I. 1999. Analysis of complex leaf and flower characters in Rhododendron using a molecular linkage map[J]. Theoretical and Applied Genetics, 98(6/7): 1146-1155. |
Jiang Beibei, Miao Hengbin, Chen Sumei, et al. 2010. The Lateral suppressor-like gene, DgLsL, alternated the axillary branching in transgenic chrysanthemum (Chrysanthemum×morifolium) by modulating IAA and GA content[J]. Plant Molecular Biology Reporter, 28(1): 144-151. DOI:10.1007/s11105-009-0130-3 |
Li G, Gao M, Yang B, et al. 2003. Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J]. Theoretical and Applied Genetics, 107(1): 168-180. DOI:10.1007/s00122-003-1236-x |
Li G, Quiros C F. 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 103(2): 455-461. |
Miao Hengbin, Jiang Beibei, Chen Sumei, et al. 2009. Isolation of a gibberellin 20-oxidase cDNA from and characterization of its expression in chrysanthemum[J]. Plant Breeding, 128(1): 1-8. DOI:10.1111/pbr.2009.128.issue-1 |
Murray M, Thompson W F. 1980. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Res, 8(19): 4321-4325. DOI:10.1093/nar/8.19.4321 |
Oyant L H S, Crespel L, Rajapakse S, et al. 2008. Genetic linkage maps of rose constructed with new microsatellite markers and locating QTL controlling flowering traits[J]. Tree Genetic & Genomes, 4(1): 11-23. |
Pan Qinghua, Hu Zhendi, Tanisaka T, et al. 2003. Fine mapping of the blast resistance gene Pil5, linked to Pii on rice chromosome 9[J]. Acta Botanica Sinica, 45(7): 871-877. |
Singh P S, Gutierrez J A, Molina A, et al. 1991. Genetic diversity in cultivated common bean: Ⅱ Marker-base analysis of morphological and agronomic traits[J]. Crop Science, 31(1): 23-39. DOI:10.2135/cropsci1991.0011183X003100010005x |
Yang Jian, Zhu Jun. 2005. Methods for predicting superior genotypes in multiple environments based on QTL effects[J]. Theoretical and Applied Genetics, 110(7): 1268-1274. DOI:10.1007/s00122-005-1963-2 |
Yang Yanfang, Wu Jian, Zhu Kai, et al. 2008. Identifcation and characterization of two chrysanthemum (Dendranthema×morifolium) DREB genes, belonging to the AP2/EREBP family[J]. Molecular Biology Reports, 36(1): 71-81. |
 2010, Vol. 46
2010, Vol. 46