文章信息
- 闫女, 王丹, 高亚卉, 郝晓杰, 王祎玲
- Yan Nü, Wang Dan, Gao Yahui, Hao Xiaojie, Wang Yiling
- 七里峪不同海拔茶条槭种群的遗传多样性
- Genetic Diversity of Acer ginnala Populations at Different Elevation in Qiliyu Based on ISSR Markers
- 林业科学, 2010, 46(10): 50-56.
- Scientia Silvae Sinicae, 2010, 46(10): 50-56.
-
文章历史
- 收稿日期:2009-08-31
- 修回日期:2010-02-06
-
作者相关文章
七里峪国家级森林公园地处山西省霍州市境内(36°21′—36°45′N, 111°40′—112°21′E)。公园内由于山体高差悬殊, 地形变化复杂, 形成了不同的气候类型和不同的水热条件及其组合。土壤垂直分异明显, 随海拔升高呈现梯度变化。地带性植被是落叶阔叶林, 垂直分布明显, 沿海拔上升依次为灌草丛及农田带、低中山针叶林带、针叶阔叶混交林带、落叶阔叶林带、山地矮林和草甸带(郝晓杰, 2009)。
茶条槭(Acer ginnala)在七里峪森林公园主要生长在海拔1 200 ~ 2 000 m的杂木林中, 是槭树科(Aceraceae)槭属槭亚属茶条槭组的模式植物, 为多年生落叶灌木或小乔木, 喜温凉湿润气候, 抗寒、抗风雪。据分析, 槭属起源于侏罗纪, 是一种古老的物种, 在形态特征上, 槭亚属是槭属中最原始的一个亚属, 而茶条槭组是槭亚属中较为进化的类群(徐廷志, 1998; 田欣等, 2001; 2004)。茶条槭为二倍体, 其染色体基数为13(田欣等, 2004), 花杂性, 花盘外露, 雌雄蕊同株(Hall, 1951; Bock et al., 1980)。其果实为翅果, 自然状态下, 茶条槭结实率较高, 但种子普遍成熟度较低, 胚有生理后熟现象(梁鸣等, 2007)。茶条槭树形优美, 叶形清秀, 尤其是秋后叶色鲜红艳丽, 极具观赏价值, 可用于园林绿化; 嫩叶可加工成茶叶, 具有生津止渴、退热明目的功效; 其叶没食子酸含量高, 具有重要的经济价值(赵晶明等, 2003)。
一个物种的稳定存在与其种群的遗传多样性和遗传结构密切相关, 遗传多样性水平的高低可以反映一个物种适应环境的能力和对环境变迁持续进化的潜力, 遗传结构的空间分布与物种的繁育机制密切相关, 同时反映生态适应进化、环境变迁与自然选择效应(Sokal et al., 1989; Ge et al., 1998; 邹喻苹等, 2001)。目前用于遗传多样性研究的分子标记主要有SSR, ISSR和RFLP等, 其中ISSR(intersimple sequence repeat)分子标记由Zietkiewicz等(1994)创建, 是类似于RAPD的一种PCR技术。ISSR引物包含重复序列并在3’或5’端锚定单寡聚核苷酸, 比RAPD引物长, 退火温度比RAPD高, 结合了SSR和RAPD的优点, 因此ISSR具有比RAPD更高的可重复性和稳定性, 其产物多态性远比RFLP, SSR更加丰富, 可以提供更多的关于基因组的信息(Esselman et al., 1999)。该技术已广泛应用于指纹图谱(Dirlewanger et al., 1998)、遗传作图、种质资源鉴定(Arcade et al., 2000; Cekic et al., 2001)、遗传多样性(Kantety et al., 1995; 张杰等, 2007; 金则新等, 2007a; 2007b;杨明博等, 2008)以及种群遗传结构等方面(Mealor et al., 2006; Xu et al., 2003; Mehmet, 2004; 王金凤等, 2007; 李秀玲等, 2008; 江香梅等, 2009)。本研究拟采用ISSR技术探讨不同海拔茶条槭种群的遗传结构、遗传多样性水平、遗传变异的空间分布, 分析其与生境因子的相互关系, 目的在于初步揭示茶条槭种群的遗传变异, 了解种群变异的大小及空间分布, 为合理开发利用茶条槭资源提供一定的理论依据。
1 材料与方法 1.1 材料所用材料于2009年4月底采自山西七里峪森林公园, 从海拔1 200 m开始采集, 海拔每相隔100 m作为1个种群处理, 共采集8个种群, 分别记为A(1 900 m)、B(1 800 m)、C(1 700 m)、D (1 600 m)、E(1 500 m)、F(1 400 m)、G(1 300 m)和H(1 200 m)。为避免由于营养繁殖造成的同一株系基因型的重复采样, 2个植株之间的间距在30 m以上。每个种群随机抽取15个以上个体, 共142个个体采集嫩叶, 用硅胶迅速干燥, 带回实验室于-80 ℃超低温冰箱内保存备用。
1.2 试验方法 1.2.1 DNA提取采用2 × CTAB法(王关林等, 1998)提取茶条槭基因组DNA, 并作适当改进, 在植物细胞匀浆中用不含CTAB的缓冲液洗去多糖、多酚等次生代谢物, 沉淀稀释后的DNA重新抽提1次, 然后加入NaAc、2倍体积的预冷的无水乙醇, 沉淀DNA。最后用0.8%琼脂糖电泳检测样品DNA的质量, 用Eppendorf Biophtometer蛋白核酸分析仪测定DNA的浓度, 并将DNA的浓度稀释为50 ng·μL-1。
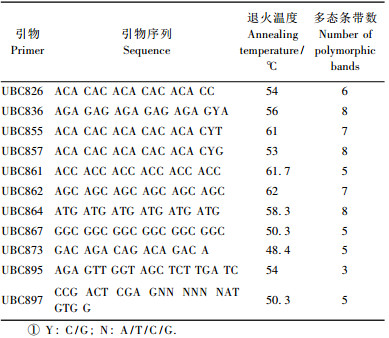
1.2.2 引物筛选ISSR引物根据加拿大哥伦比亚大学(University of British Columbia, Set No.9, No.801 ~ 900)公布的序列, 由上海生工生物工程公司合成。从茶条槭8个种群中, 每个种群任意选取1个个体, 利用其总DNA对100条引物进行筛选, 从中筛选出扩增条带清晰、重复性好的11条引物, 随后用这些引物对8个种群的所有个体进行PCR扩增(表 1)。
|
|
PCR反应体系10 μL: 3.0 mmol·L-1 Mg2+, 0.15 mmol·L-1 dNTPs, 0.5 U Taq DNA聚合酶, 0.2 μmol·L-1引物, 20 ~ 40 ng DNA模板。扩增反应程序为:94 ℃预变性4 min; 94 ℃变性1 min, 48 ~ 62 ℃(表 1)复性3 min, 72 ℃延伸2 min, 30个循环; 最后72 ℃延伸5 min, 4 ℃保存。
扩增产物用0.5% EB显色, 2%的琼脂糖凝胶进行电泳检测, 电压100 V, 电流50 mA, 电泳70 min。以100 bp DNA ladder作为Marker, 凝胶成像系统(LabWorks Software Version 310;UVP, Upland, CA 91786, USA)成像、观察、记录。
1.2.4 土壤因子的测定在每个样地内, 采用梅花取样法, 取0 ~ 30 cm土层的土壤混合样品, 分析土壤含水量、pH、有机质、氮、磷、钾等指标(中国土壤学会农业化学专业委员会, 1983)(表 2)。
|
|
ISSR为显性标记, 同一引物扩增产物中电泳迁移率一致的条带被认为具有同源性, 属于同一位点, 按照扩增条带的有无, 进行1, 0标记, 形成ISSR表型数据矩阵。用POPGEN软件(Yeh et al., 1997)估算种群的多态位点百分率(PPB, percentage of polymorphic band)和Shannon多样性指数(I)。
由于ISSR为显性遗传标记, 对于种群遗传多样性和种群间遗传分化的估算会有偏差, 因此基于贝叶斯统计, 利用HICKORY v1.0(Holsinger et al., 2002)进行种群遗传多样性(HB)和遗传分化(θB和GST-B)校正估算。同时应用ARLEQUIN v3.1软件(Excoffier et al., 2005)进行分子方差分析(AMOVA), 计算遗传变异在种群间和种群内的分布特征。
利用GenAlEx v6.0(Peakall et al., 2006)进行种群间相互关系的PCoA分析。应用STRUCTURE v2.0(Pritchard et al., 2004:http://pritch.bsd. uchicago.edu/structure.html.)软件中的连锁模型和等位基因非相关模型, 设定模拟参数burn-in 1 000 000次, MCMC 500 000次, 假定七里峪茶条槭种群的最大丛数为8(K), 进行贝叶斯种群结构分析, 分别从K = 1 ~ 8在非混合(no admixture)模式下进行估测(Li et al., 2009)。
采用Origin7.5(http://originlab.com软件对各项遗传多样性指数与环境因子进行拟合线性回归分析。
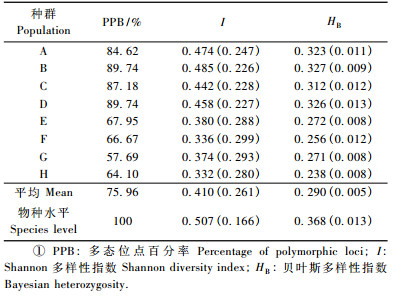
2 结果与分析 2.1 茶条槭种群的遗传多样性从100条ISSR引物中共筛选出11条能扩散出清晰条带、多态性高、稳定性好的引物。各引物检测出的多态条带见表 1。每条引物可扩增3 ~ 11条谱带, 扩增谱带长约200 ~ 2 500 bp, 共检测到78个位点。在物种水平上, 多态位点78个, 总多态位点百分率为100%(表 3)。Shannon多样性指数I, Bayesian指数HB分别为0.507和0.368。8个种群多态位点百分率变动范围为57.69% ~ 89.74%, 平均多态位点百分率75.96%。不同海拔8个种群的PPB以种群B最高(89.74%), 种群G最低(57.69%); Shannon多样性指数I中, 种群B最高, 种群H最低, 变幅在0.332 ~ 0.485, 平均为0.410。不同种群的HB指数为0.238 ~ 0.327, 种群B最高, 种群H最低, 平均为0.290(表 3)。在研究的所有种群中, 种群H的遗传多样性最低(PPB:64.10%; I:0.332;HB:0.238)。
|
|
利用ISSR数据对茶条槭8个种群进行AMOVA分析, 结果显示(表 4):种群内的遗传变异占总遗传变异的74.32%, 而种群间的遗传变异占总遗传变异的25.68%。由Bayesian方法估算的种群间的遗传分化程度分别为θB = 0.263, GST-B = 0.210, 同样表明遗传变异主要存在于种群内而不是种群间。
|
|
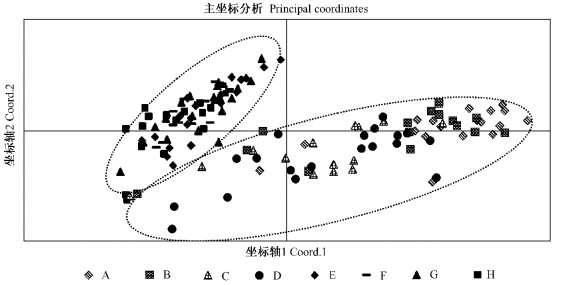
PCoA分析中, 第1坐标轴可以解释总遗传变异的43.64%, 第2坐标轴解释总遗传变异的18.56%。PCoA分析将所分析的8个茶条槭种群大体划分为2组:种群A, B, C, D为第1组, 种群E, F, G, H为第2组(图 1)。
|
图 1 茶条槭种群个体的主坐标分析 Figure 1 The Principal Coordinates analysis ( PCoA) for 142 A. ginnala individuals |
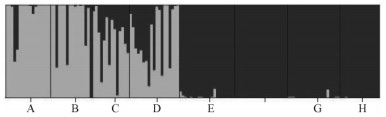
利用STRUCTURE软件中的no admixture模式, 在假定种群间的基因频率互不关联的情况下, 当lnP(D)(log-likelihood values)达到最大值时(Evanno et al., 2005), 取K = 2, 所分析的ISSR数据以最大的可能性聚集为2组(图 2)。这一结果与PCoA分析相一致。
|
图 2 利用STRUCTURE程序估测的茶条槭种群的遗传结构特征 Figure 2 Estimated genetic structure for K = 2 obtained with the STRUCTURE program for 8 populations of A.ginnala based on ISSR variation A-H:不同种群Different populations. |
为了进一步了解茶条槭种群的生态适应性, 对茶条槭种群的遗传多样性指数与生境因子进行了拟合线性回归分析。结果表明, 拟合较好的函数为二项式。种群的PPB(Y =-1 576.32 + 68.14x-0.35x2, R2 = 0.755*), I(Y = 1 305.07-2 597.21x + 7 636.77x2, R2 = 0.854**), HB(Y =-827.78 +10 219.61x-6 922.59x2, R2 = 0.816*)均与海拔(x)显著相关, 说明8个种群的遗传多样性存在随海拔升高而增大的趋势。另外, 种群的PPB, I, HB分别与土壤的含水量(PPB:Y =-1.72 + 0.05x-2.61x2, R2 = 0.894*; I:Y = 1.80-9.05x + 12.81x2, R2 = 0.725*; HB:Y = 4.87-34.95x + 65.02 x2, R2 = 0.827*)、氮含量(PPB:Y =-4.32 +0.13x-3.27x2, R2 = 0.253*; I:Y = 104.58-522.01x + 657.76x2, R2 = 0.709*; HB:Y = 189.84-1 337.46x + 2 363.03x2, R2 = 0.874*)也呈显著的相关关系。茶条槭种群的遗传多样性与其他生境因子没有显著相关关系。总体而言, 土壤含水量、全氮含量越高, 茶条槭种群的遗传多样性越高。
3 讨论 3.1 茶条槭种群的遗传多样性应用ISSR技术对筛选出的11个引物进行PCR扩增, 结果表明, 在物种水平上, 8个茶条槭种群存在着高的遗传多样性, 其多态位点百分率为100%。与槭树科其他植物相比, 茶条槭与金钱槭(Dipteronia sinensis)种群的遗传多样性(92.97%) (李珊, 2004)相近, 高于云南金钱槭(Dipteronia dyerana)(81.55%)、庙台槭(Acer miaotaiense) (53.13%)的遗传多样性(李珊, 2001; 2004)。
一个物种的遗传多样性是该物种长期进化的产物, 是各种因素综合作用的结果(王金凤等, 2007; 江香梅等, 2009), 与其生活型、繁育系统、地理分布、种子散布机制等生物学生态学特性有关(谢国文等, 2007; 何俊等, 2007)。茶条槭发展到现在至少有几百万年的历史, 世代重叠, 且结实量大(徐廷志, 1998; 梁鸣等, 2007)。其次, 茶条槭的花粉、种子散布以风媒为主, 有利于避免因小种群遗传漂变而致遗传多样性丧失。另外, 茶条槭种群表现出高的遗传多样性与其多样化的生境有关。它不但生长于山谷湿地、林缘、林下, 亦生长于向阳的山坡、灌丛, 干旱及湿润立地均可见茶条槭生长。由于长期适应多种多样化生境导致茶条槭的高遗传多样性。
在以往的相关研究中, 紫椴(Tilia amurensis)种群的遗传多样性随海拔的升高呈现逐渐降低的趋势(穆立蔷等, 2007), 生存环境恶劣、基因流动困难使处于高海拔地区的紫椴种群遗传多样性降低。不同海拔木荷(Schima superba)种群的多样性也呈现降低的模式, 金则新等(2007a)认为海拔升高导致温度的下降可引起植物生活期的缩短和开花、传粉物候期的延迟, 从而影响基因交流, 导致木荷高海拔种群遗传多样性水平的降低。而沙棘(Hippophae rhamnoides)、柴胡(Bupleurum chinense)种群的遗传多样性随海拔升高呈低—高—低的变异趋势, 这样的变异模式与其各种群所处生境的选择压力有关(赵春芳等, 2007; 黄玮等, 2008)。粉报春(Primula farinosa)种群的遗传差异则随海拔的升高而增大(Reisch et al., 2005), 群落类型的改变使得高海拔种群基因流加强引起高水平的遗传多样性。在本研究中, 茶条槭种群的遗传多样性指数I、HB与海拔之间均具有显著的正相关关系, 表明随着海拔的升高, 茶条槭种群的遗传多样性也随之升高。一般来讲, 海拔每升高100 m温度会下降0.56 ℃(杨玉坡等, 1992)。随着海拔的升高, 空气湿度也会增加。相关回归分析表明, 茶条槭种群各项遗传多样性指数与土壤含水量表现出显著的相关性。其他生境因子也不同于低海拔区域, 如紫外线UV-B的辐射剂量, 高海拔区域高于低海拔区域。高海拔区域特异的环境可以诱发各种各样的突变, 且大多数突变是可以遗传的, 这样高海拔种群比低海拔种群更易发生基因突变(Li et al., 1998)。野外调查发现, 从海拔1 300 m左右, 茶条槭开始成片出现, 1 300 ~ 1 800 m为其集中分布的地段, 通风透光, 相对其他生境更适合茶条槭的生长。海拔1 800 m以上, 茶条槭的数量逐渐减少, 零星出现于华北落叶松林缘。高海拔地区, 茶条槭所在生境类型多样, 群落结构及种类组成复杂, 选择压力较大, 因而表现出较高的遗传变异水平。
用I, HB估算的8个种群的遗传多样性大小顺序虽略有差异, 但都以种群H最低。种群H位于茶条槭生长的最低海拔, 茶条槭个体开始零星出现于鹅耳枥(Carpinus turczaninowii)林、辽东栎(Quercus wutaishanica)林、油松(Pinus tabulaeformis)林边缘和路边, 且邻近村庄, 受人为干扰较为严重。在野外调查中发现, 低海拔种群常常受到村民的樵砍和牲畜的啃食。人为干扰可能会造成种群变小, 易发生遗传漂变, 使得其遗传多样性丧失, 从而形成种群H遗传多样性较低的格局。
3.2 茶条槭种群的遗传结构AMOVA分子变异分析显示, 在茶条槭种群总遗传变异中, 有25.7%存在于种群间, 74.3%存在于种群内, 种群内和种群间变异均极显著(ΦST = 0.257, P < 0.001)。由Bayesian方法分析的种群遗传分化θB(0.263)和GST-B(0.210)进一步表明茶条槭种群的遗传变异主要存在于种群内(表 4)。
植物种群的遗传结构不仅决定于自身遗传基础、繁育系统及其起源进化历史进程, 而且还受到自然选择、遗传漂变、定居(recolonization)和基因流等因素的影响(Ishihama et al., 2005; 彭艳秋等, 2007)。茶条槭是多年生木本植物, 为风媒传粉植物, 花粉小而轻, 果实为翅果, 花粉和果实均可随风传送, 这就为种群间的基因交流提供了可能。茶条槭种群的遗传分化更多地存在于种群内, 还可能与茶条槭种子的结实和萌发特性有关(梁鸣等, 2007)。这些特性决定了茶条槭种子定居萌发成新个体能力弱、个体萌发整齐性差、个体差异显著, 致使种群内茶条槭个体发育不一、遗传差异显著。
虽然茶条槭种群的遗传变异主要存在于种群内, 但种群间仍然产生了一定的遗传分化(25.7%) (表 4, 图 1、2)。大量证据表明在一个连续的种群内遗传分化将很少发生, 但在较小地理范围内异质生境确实可以造成某些植物种群的遗传分化(李钧敏等, 2004; 张志红等, 2005; 赵利峰等, 2001)。七里峪山地垂直地带性明显, 不同海拔植被差异显著, 茶条槭种群群落生境类型多样, 土壤垂直分异明显(郝晓杰, 2009)。由此造成不同海拔种群所处生境的温度、水分、光照、土壤等生态因子发生变化, 不同种群承受的生境选择压力产生差异, 最终导致种群间发生了遗传分化。茶条槭种群各遗传多样性指数与土壤含水量和氮含量呈显著相关关系, 证实了茶条槭不同种群所处生境的异质性是引起种群分化的一个原因。相似的结果也见于以往相关研究中, 李进等(1998)在对不同海拔川滇高山栎(Quercus semicarpifolia)群体遗传结构进行研究时, 认为遗传多样性的分布规律与随着海拔而表现出的降雨量、温度等自然环境的变化是密切相关的。赵春芳等(2007)发现卧龙自然保护区不同海拔沙棘种群间存在的遗传变异是由于海拔不同引起生境的异质性所致。木荷不同海拔种群间的遗传分化同样是由于不同种群所面临的生境选择压力差异所致(金则新等, 2007a)。另外, 不同海拔显著的水热差异, 致使不同海拔种群物候存在差异, 野外调查中发现, 低海拔种群已开始展叶的时候, 高海拔种群还没开始萌芽, 生长周期的不同使得不同海拔种群每代基因的突变率发生差异(Ohsawa et al., 2008), 生育期的不重叠限制了花粉(基因)的迁入, 进而引起了种群间的分化。
郝晓杰. 2009. 霍山七里峪茶条槭种群生态学研究. 山西师范大学硕士学位论文. http://cdmd.cnki.com.cn/Article/CDMD-10118-1015606663.htm
|
何俊, 杨柏云, 陈少风, 等. 2007. 多叶重楼遗传多样性的ISSR分析[J]. 云南植物研究, 29(4): 388-392. |
黄玮, 孙平, 张文生, 等. 2008. 北京东灵山地区不同海拔柴胡居群的遗传多样性[J]. 植物遗传资源学报, 9(4): 453-457. |
江香梅, 温强, 叶金山, 等. 2009. 闽楠天然种群遗传多样性的RAPD分析[J]. 生态学报, 29(1): 438-443. |
金则新, 李钧敏, 蔡琰琳. 2007a. 不同海拔高度木荷种群遗传多样性的ISSR分析[J]. 生态学杂志, 26(8): 1143-1147. |
金则新, 李钧敏. 2007b. 珍稀濒危植物夏腊梅遗传多样性的分析[J]. 应用生态学报, 18(2): 247-253. |
李进, 陈可咏, 李渤生. 1998. 不同海拔高度川滇高山栎群体遗传多样性的变化[J]. 植物学报, 40(8): 761-767. |
李钧敏, 金则新, 钟章成. 2004. 不同海拔高度大血藤群体遗传多样性的RAPD分析及其与环境因子的相关性[J]. 生态学报, 24(3): 567-573. |
李秀玲, 陈健, 王刚. 2008. 西北地区红砂种群ISSR遗传变异的空间自相关分析[J]. 中国沙漠, 28(3): 468-472. |
李珊. 2001. 用RAPD标记检测庙台槭自然居群的遗传结构和遗传分化. 西北大学硕士学位论文. http://cdmd.cnki.com.cn/Article/CDMD-10697-2001011130.htm
|
李珊. 2004. 金钱槭属植物保护遗传学与分子亲缘地理学研究. 西北大学博士学位论文. http://cdmd.cnki.com.cn/Article/CDMD-10697-2010118502.htm
|
梁鸣, 张悦, 杨轶华, 等. 2007. 槭属植物种子解剖生物学的研究[J]. 林业科技, 32(3): 9-12. |
穆立蔷, 刘赢男. 2007. 不同地理分布区紫椴种群的遗传多样性变化[J]. 植物生态学报, 31(6): 1190-1198. |
彭艳秋, 邵剑文, 张小平, 等. 2007. 珍稀濒危植物安徽羽叶报春遗传多样性的RAPD分析[J]. 云南植物研究, 29(5): 549-553. |
田欣, 金巧军, 李德铢, 等. 2001. 槭树科花粉形态及其系统学意义[J]. 云南植物研究, 23(4): 457-465. |
田欣, 李德铢. 2004. 槭树科植物广义形态学性状分支分析[J]. 云南植物研究, 26(4): 387-397. |
王关林, 方洪筠. 1998. 植物基因工程原理与技术[M]. 北京: 科学出版社.
|
王金凤, ChiakiKuroda, 龚洵. 2007. 东俄洛橐吾遗传变异与分化的ISSR分析[J]. 云南植物研究, 29(5): 537-542. |
谢国文, 彭晓瑜, 郑燕玲, 等. 2007. 濒危植物永瓣藤遗传多样性的ISSR分析[J]. 林业科学, 43(8): 48-53. |
徐廷志. 1998. 槭属的系统演化与地理分布[J]. 云南植物研究, 20(4): 383-393. |
杨明博, 熊友才, 王红芳, 等. 2008. 黄土高原不同气象因子对中国沙棘亚居群遗传多样性的影响[J]. 应用生态学报, 19(1): 1-7. |
杨玉坡, 李承彪. 1992. 四川森林[M]. 北京: 中国林业出版社.
|
张杰, 吴迪, 汪春蕾, 等. 2007. 应用ISSR-PCR分析蒙古栎种群的遗传多样性[J]. 生物多样性, 15(3): 292-299. |
张志红, 唐恬, 周仁超, 等. 2005. 异质性生境对半红树植物海漆(Excoecaria agallocha)居群遗传结构的影响[J]. 遗传学报, 32(1): 1286-1292. |
赵春芳, 陈国娟, 王芋华, 等. 2007. 利用RAPD标记分析卧龙自然保护区不同海拔沙棘种群的遗传变异[J]. 应用与环境生物学报, 13(6): 753-758. |
赵晶明, 张玉兰, 于立明, 等. 2003. 茶条槭良种选择及其专用林培育[J]. 特种经济动植物, (5): 18. |
赵利峰, 李珊, 潘莹, 等. 2001. 华山新麦草自然居群沿海拔梯度的遗传分化[J]. 西北植物学报, 21(3): 391-400. |
中国土壤学会农业化学专业委员会. 1983. 土壤农业化学常规分析方法[M]. 北京: 科学出版社: 195-221.
|
邹喻苹, 葛颂, 王晓东. 2001. 系统与进化植物学中的分子标记[M]. 北京: 科学出版社: 1-266.
|
Arcade A, Anselin F, Faivre R P, et al. 2000. Application of AFLP, RAPD and ISSR markers to genetic mapping of European and Japanese larch[J]. Theoretical and Applied Genetics, 100(2): 299-307. DOI:10.1007/s001220050039 |
Bock K, Cour L F N, Jensen S R, et al. 1980. The structure of Acertannin[J]. Phytochemistry, 19(9): 2033. DOI:10.1016/0031-9422(80)83034-2 |
Cekic C, Battey N H, Wilkinson M J. 2001. The potential of ISSR-PCR primer pair combinations for genetic linkage analysis using the seasonal flowering locus in Fragaria as a mode[J]. Theoretical and Applied Genetics, 103(4): 540-546. DOI:10.1007/PL00002907 |
Dirlewanger E, Pronier V, Parvery C, et al. 1998. Genetic linkage map of peach [Prunus persica(L.) Batsch ] using morphological and molecular markers[J]. Theoretical and Applied Genetics, 97(5/6): 888-895. |
Esselman E J, Li J Q, Crawford D J, et al. 1999. Clonal diversity in the rare Calamagrostis porteri ssp.insperata(Poaceae):Comparative results for allozymes and random amplified polymorphic DNA(RAPD) and intersimple sequence repeat(ISSR) markers[J]. Molecular Ecology, 8(3): 443-451. DOI:10.1046/j.1365-294X.1999.00585.x |
Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Molecular Ecology, 14(8): 2611-2620. DOI:10.1111/mec.2005.14.issue-8 |
Excoffier L, Laval G, Schneider S. 2005. Arlequin(version 3.0):An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 1: 47-50. |
Ge Song, Hong Deyuan, Wang Haiqun, et al. 1998. Population genetic structure and conversation of an endangered conifer, Cathaya argyrophylla(Pinaceae)[J]. Int J Plant Sci, 159(2): 351-357. DOI:10.1086/297556 |
Hall B A. 1951. The floral anatomy of the genus Acer[J]. American Journal of Botany, 38(10): 793-799. DOI:10.2307/2438204 |
Holsinger K E, Lewis P O, Dey D K. 2002. A Bayesian approach to inferring population structure from dominant markers[J]. Molecular Ecology, 11(7): 1157-1164. DOI:10.1046/j.1365-294X.2002.01512.x |
Ishihama F, Ueno S, Tsumura Y, et al. 2005. Gene flow and inbreeding depression inferred from fine-scale genetic structure in an endangered heterostylous perennial, Primula sieboldii[J]. Molecular Ecology, 14(4): 983-990. DOI:10.1111/j.1365-294X.2005.02483.x |
Kantety R V, Zeng X P, Bennetzen J L, et al. 1995. Assessment of genetic diversity in tent and popcorn(Zea mays L.) inbred lines using inter-simple sequence repeat(ISSR) amplification[J]. Molecular Breeding, 1(4): 365-373. DOI:10.1007/BF01248414 |
Li Enxiang, Sun Yi, Qiu Yingxiong, et al. 2009. Phylogeography of two East Asian species in Croomia(Stemonaceae) inferred from chloroplast DNA and ISSR fingerprinting variation[J]. Molecular Phylogenetics and Evolution, 49(3): 702-714. |
Li Jin, Chen Keyong, Li Bosheng. 1998. The variation of genetic diversity of Quercus aquifolioides in different elevations[J]. Acta Bot Sin, 40(8): 761-767. |
Mealor B A, Hild A L. 2006. Potential selection in native grass populations by exotic invasion[J]. Molecular Ecology, 15(8): 2291-2300. DOI:10.1111/j.1365-294X.2006.02931.x |
Mehmet A S. 2004. Inter and intra-species inter simple sequence repeat(ISSR) variations in the genus Cicer[J]. Euphytica, 135(2): 229-238. DOI:10.1023/B:EUPH.0000014938.02019.f3 |
Ohsawa T, Saito Y, Sawada H, et al. 2008. Impact of altitude and topography on the genetic diversity of Quercus serrata populations in the Chichibu Mountains, central Japan[J]. Flora-Morphology, Distribution, Functional Ecology of Plants, 203(3): 187-196. DOI:10.1016/j.flora.2007.02.007 |
Peakall R, Smouse P E. 2006. GenAlEx 6:genetic analysis in Excel Population genetic software for teaching and research[J]. Molecular Ecology Notes, 6(1): 288-295. DOI:10.1111/men.2006.6.issue-1 |
Reisch C, Anke A, Röhl M. 2005. Molecular variation within and between ten populations of Primula farinose(Primulaceae) along an altitudinal gradient in the northern Alps[J]. Basic and Applied Ecology, 6(1): 35-45. DOI:10.1016/j.baae.2004.09.004 |
Sokal R R, Jacquez G M, Wooten M. 1989. Spatial autocorrelation analysis of migration and selection[J]. Genetics, 121(4): 845-855. |
Xu Li, Wang Yiling, Wang Xumei, et al. 2003. Genetic structure of Reaumuria soongorica population in Fukang Desert, Xinjiang and its relationship with ecological factors[J]. Acta Botanica Sinica, 45(7): 787-794. |
Yeh F C, Yang R C, Boyle T, et al.1997.POPGENE, the user friendly shareware for population genetic analysis.Molecular Biology and Biotechnology Center, University of Alberta, Edmonton, Canada. http://pub.chinasciencejournal.com/PlantScienceJournal/13462.jhtml
|
Zietkiewicz E, Rafalski A, Labuda D. 1994. Genome fingerprinting by simple sequence repeat(ISSR) -anchored polymerase chain reaction amplification[J]. Genomics, 20: 176-783. DOI:10.1006/geno.1994.1151 |
 2010, Vol. 46
2010, Vol. 46