文章信息
- 高志民, 郑波, 彭镇华
- Gao Zhimin, Zheng Bo, Peng Zhenhua
- 毛竹 PeMADS1 基因的克隆及转化拟南芥初步研究
- Isolation of PeMADS1 Gene from Phyllostachys edulis and Its Transformation in Arabidopsis thaliana
- 林业科学, 2010, 46(10): 37-41.
- Scientia Silvae Sinicae, 2010, 46(10): 37-41.
-
文章历史
- 收稿日期:2010-04-20
- 修回日期:2010-07-28
-
作者相关文章
2. 中国林业科学研究院林业研究所 北京 100091
2. Research Institute of Forestry, Chinese Academy of Forestry Beijing 100091
竹子开花生物学和生殖生物学一直是竹类植物研究中的难点。随着对竹类植物资源利用工业化程度的不断提高, 对竹子专用新品种的需求日趋凸显, 育种者在试图通过各种手段来培育竹子新品系以适应不同工业化利用的需求, 竹子开花生物学和生殖生物学研究又成为人们关注的焦点。不同竹种的开花时间、周期各异, 有的竹种每年开花一次, 有的需要几十年甚至上百年才开花, 没有稳定的规律性, 而且花粉的活力维持时间较短, 结实率较低(袁金玲等, 2005), 因此竹类植物杂交育种工作的开展举步维艰。
分子生物学技术的飞速发展为从分子水平来研究竹类植物的花发育提供了良好的借鉴, 尤其是模式植物拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)、金鱼草(Antirrhinum majus)、矮牵牛(Petunia hybrida)等开花调控基因及其功能的研究, 为开展竹子研究带来了新的思路。研究表明, 花发育过程中各类花器官是受器官控制基因调控的, 按照其功能分为不同的类型, 并先后有学者提出了花发育ABC模型、ABCD模型和ABCDE模型(Coen et al., 1991; Angenent et al., 1995; Ganesh et al., 2005), 使得花发育调控网络日趋完善。在花发育调控中有很大一类属于MADS-box基因家族, 该家族基因拥有MADS-box保守结构域, 是一类重要的调节基因, 参与器官发育、开花时间的控制, 对胚胎发育(Perry et al., 1999)、组织的分化(Weigel, 1995)和根的形成(Alvarez-Buylla et al., 2000)、种子和果实的发育(Gu et al., 1998; Liljegren et al., 1998)等起调控作用。
目前, 已经从丛生竹麻竹(Dendrocalamus latiflorus)、绿竹(Dendrocalamopsis oldhami)中分离了2个与竹子花发育密切相关的MADS-box基因, 并对其功能进行了分析(田波等, 2005; 高志民等, 2007), 然而对于散生竹MADS-box基因的相关研究尚未见报道。本文以散生竹—毛竹(Phyllostachys edulis)为材料, 采用RT-PCR和RACE技术分离到1个MADS-box基因, 并通过转化模式植物拟南芥对其功能进行了初步研究, 以期为通过基因工程进行竹子定向育种提供理论依据。
1 材料与方法 1.1 植物材料和菌株购买毛竹种子, 在本实验室播种于直径为10 cm的塑料盆中, 以蛭石为培养介质, 用1/3 B5培养液培养, 4个月的实生苗用于试验。大肠杆菌(Escherichia coli)菌株DH5α和根癌农杆菌(Agrobacterium tumefaciens)菌株EHA105由本实验室保存。
1.2 总RNA提取与cDNA合成以毛竹幼嫩叶片为材料, 采用Invitrogen公司的Trizol试剂提取总RNA(Gao et al., 2006), cDNA合成使用Promega公司的反转录试剂盒, 5′ cDNA合成采用Clontech公司的SMARTTM RACE试剂盒。
1.3 基因克隆与测序分析根据已知竹类植物的MADS-box基因(田波等, 2005; 高志民等, 2007)设计PCR引物, PeMADS-F: 5′-AGTTGAACTGAAGAGGATTGAG-3′; PeMADS-R: 5′-CTAAAGAACCCAACCAAGCATG-3′, 由上海生工生物工程技术服务有限公司合成。以毛竹cDNA为模板, 进行梯度PCR。反应体系20 μL:2 μL 10 × LA PCR Buffer(Mg2+ Plus), 3.2 μL dNTPs (2.5 mmol·L-1 each), 2 μL PeMADS-F(5 mmol·L-1), 2 μL PeMADS-R(5 mmol·L-1), 1 μL模板, 9.6 μL超纯水和0.2 μL LA DNA聚合酶。反应条件为:94 ℃预变性5 min; 94 ℃ 1 min, 55 ~ 65 ℃ 1 min, 72 ℃ 1.5 min, 35个循环; 72 ℃延伸10 min。PCR产物经电泳分析后, 用上海申能博彩生物科技有限公司的DNA试剂盒回收目的片段。按Promega公司的快速连接试剂盒操作流程, 将DNA片段连到pGEM-T easy载体上, 点击转化DH5α, 提取阳性克隆质粒并酶切图谱分析后, 送北京三博远志科技有限公司测序。
根据获得基因序列设计5′ RACE引物: PeMADS-Race1:5′-GCCAAGGTCTTCTCCAAGCAA ATGCCTC-3′; PeMADS-Race2:5′-ACTCGTGCC AGCACTCCCAAACTCATAG-3′。5′-RACE反应条件1为:94 ℃ 30 s, 72 ℃ 3 min, 5个循环; 94 ℃ 30 s, 70 ℃ 30 s, 72 ℃ 3 min, 5个循环; 94 ℃ 30 s, 68 ℃ 30 s, 72 ℃ 3 min, 25个循环。5′-RACE反应条件2为:94 ℃ 30 s, 68 ℃ 30 s, 72 ℃ 3 min, 39个循环。
1.4 基因植物表达载体的构建根据获得序列的编码区分别设计包含酶切位点的引物, PeMADS-F1:5′-tctagaATGGGGAGAGGGA GAGTTGAAC-3′; PeMADS-R1:5′-ggatccCTAAAGAA CCCAACCAAGCATG-3′。应用宝日医生物技术(北京)有限公司的高保真pyrobest DNA聚合酶进行扩增, 反应体系20 μL:2 μL 10 × pyrobest Buffer(Mg2+ Plus), 1.6 μL dNTP(2.5 mmol·L-1), 2 μL PeMADSF1(5 mmol·L-1), 2 μL PeMADS-R1(5 mmol·L-1), 1 μL模板, 11.3 μL超纯水和0.1 μL pyrobest DNA聚合酶。反应条件为:94 ℃预变性5 min; 94 ℃ 1 min, 55 ~ 65 ℃ 1 min, 72 ℃ 1.5 min, 35个循环; 72 ℃延伸10 min。PCR产物回收并加A后连接到到pGEM-T easy载体上测序。加A反应体系10 μL: 1 μL 10 × PCR Buffer(Mg2+ Plus), 1 μL DNA聚合酶, 1 μL dATP和7 μL回收DNA片段。反应条件为:72 ℃ 30 min。
测序后将正确的序列构建到植物表达载体pCAMBIA1300的多克隆位点。
1.5 拟南芥转化与检测用电击法将含有目的基因的植物表达载体质粒转入农杆菌(EHA105), 参照徐芳等(2005)的方法转化拟南芥。直接观察转基因植株的表型, 并提取转基因植株的基因组DNA, 进行PCR检测。提取转基因阳性植株和野生型拟南芥的RNA, 反转录成cDNA, 用拟南芥的ACT7(U27811)作内标(ACT7-f: 5′-ATGGCCGATGGTGAGGATATTC-3′; ACT7-r:5′-GAAGCATTTCCTGTGAACAATCG-3′), 来检查目的基因在转基因植株中的表达情况。反应条件为: 94 ℃预变性5 min; 94 ℃ 1 min, 56 ℃ 1 min, 72 ℃ 1 min 20 s, 28个循环; 72 ℃延伸10 min。
2 结果与分析 2.1 基因克隆与结构分析PeMADS-F和PeMADS-R的PCR产物电泳结果显示, 在0.7 kb有1条明显的亮带, 且随着退火温度的提高目的条带有增加的趋势, 而非特异扩增产物减弱(图 1)。以59.3 ℃为退火温度进行大量扩增, 将目的片段回收并测序。测序结果表明, 插入片段为709 bp, 通过比较分析表明, 该片段为MADSbox基因的部分序列。用PeMADS-Race1与试剂盒中的通用引物UPM配对, 在5′-RACE反应条件1下扩增, 产物电泳没有明显的目的条带; 将产物稀释50倍做模板, 用PeMADS-Race2与通用引物NUP配对, 在5′-RACE反应条件2下扩增, 产物电泳显示在0.4 kb处有一明显条带。目的条带经测序后与先前获得的片段进行拼接, 获得长776 bp的cDNA序列, 包含了5′UTR(53 bp)和1个完整的开放阅读框(723 bp), 命名为 PeMADS1 (GenBank登记号: EU327784)。
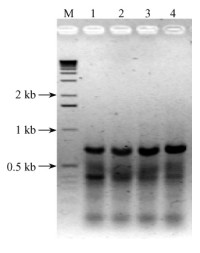
|
图 1 梯度PCR产物电泳结果 Figure 1 DNA fragments amplified by gradient PCR 1.55.9 ℃产物; 2.56.7 ℃产物; 3.57.8 ℃产物; 4.59.3 ℃产物; M:1 kb分子质量标记。 l:55.9 ℃ fragments; 2:56.7 ℃ fragments; 3:57.8 ℃ fragments; 4:59.3 ℃ fragments; M:1 kb molecular marker. |
序列分析表明, PeMADS1 具有典型的植物MADS-box基因结构, 与已知竹类植物的MADS-box基因相比, 在核酸水平 PeMADS1 与绿竹BoMADS1 (高志民等, 2007)、麻竹的DlMADS18(田波等, 2005)编码区的一致性分别为99.7%, 67.3%, 其中仅第6位、第22位的碱基与BoMADS1不同, 在 PeMADS1 中为G和C, 在BoMADS1中为A和T; 而在蛋白水平则分别为100%, 69.9%, 其等电点和分子量与BoMADS1的完全一致。同时, PeMADS1 编码的蛋白质与其他物种的MADS-box蛋白有着较高的一致性, 其中与水仙(Narcissus tazetta var. chinensis)的NtMADS3、石刁柏(Asparagus officinalis)的pAOM3(AAQ83835)、藏红花(Crocus sativus)的AGL6a(ABK35281)、百子莲(Agapanthus praecox)的SEP1(BAC66964)、风信子(Hyacinthus orientalis)的SEP1(AAT88088)等的一致性都在80%以上, 其中与水仙的NtMADS3(高志民等, 2009)一致性最高, 达88.0%, 与模式植物拟南芥的E类功能基因AGL6的一致性达57.2%。因此, 推测 PeMADS1 基因应属于E类功能基因。
2.3 植物表达载体的构建与检测以PeMADS-F1和PeMADS-R1为引物进行扩增, 将目的条带连接到pGEM-T easy载体上测序。结果显示, 扩增片段为735 bp, 包含了 PeMADS1 基因的完整编码区(723 bp)和酶切位点(12 bp)。因为 PeMADS1 基因内部包含SacⅠ酶切位点, 所以先分别用SacⅠ/EcoRⅠ和HindⅠ/XbaⅠ双酶切将pBI121的NOS终止区域和35S引入到载体pCAMBIA1300的多克隆位点; 然后用XbaⅠ/ BamHⅠ双酶切将 PeMADS1 基因片段插入到XbaⅠ和BamHⅠ酶切位点之间, 即获得了由35S启动子调控 PeMADS1 基因编码区的植物表达载体, 构建过程如图 2所示。
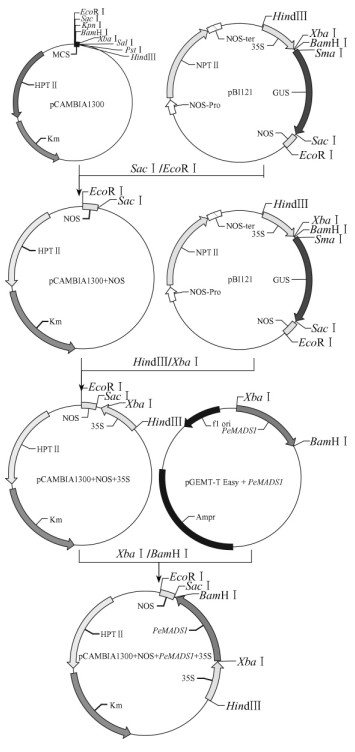
|
图 2 PeMADS1 基因表达载体的构建 Figure 2 Construction of PeMADS1 expression vector |
根据载体多克隆位点和 PeMADS1 基因所包含的酶切位点, 分别选用HindⅠ/EcoRⅠ、XbaⅠ/ BamHⅠ双酶切, HindⅢ、SacⅠ单酶切载体质粒, 酶切指纹与预期的完全相符(图 3), 表明所构建的 PeMADS1 基因植物表达载体是正确的。

|
图 3 PeMADS1 植物表达载体酶切检测 Figure 3 The plant expression vector of PeMADS1 digested by different restriction enzymes 1:HindⅠ; 2:HindⅠ/EcoRⅠ; 3:XbaⅠ/BamH Ⅰ; 4: SacⅠ; M:1 kb ladder. |
将构建的 PeMADS1 基因植物表达载体转入EHA105, 应用蘸花法转化拟南芥(Col-0), 收取转化后拟南芥种子, 经卡那霉素抗性筛选, 得到抗性植株, 培养并收获种子再播种。对转 PeMADS1 基因拟南芥T1代30个株系的表型观察发现, 与正常的拟南芥野生型相比, 转基因株系在苗期就有6株莲座叶卷曲的表型, 且随着生长发育, 开花明显提前, 比野生型平均提早7天(图 4A); 有15株只开花, 不结实, 且长势较弱(图 4B); 有3株营养生长延长, 直至4个月尚未抽薹(图 4C)。转 PeMADS1 基因拟南芥的表型说明 PeMADS1 可能参与毛竹发育过程中由营养生长向生殖生长转化的调控。
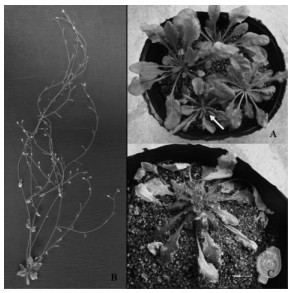
|
图 4 转 PeMADS1 基因拟南芥植株的表型 Figure 4 Phenotype of PeMADS1 gene transgenic plants of A.thaliana A.卷叶; B.早花; C.发育迟缓。 A.Curled rosette; B.Early flowering; C.Stunt. |
提取转 PeMADS1 基因拟南芥T2代的基因组DNA, 用引物PeMADS-F1和PeMADS-R1进行PCR检测。结果表明:阴性对照野生型拟南芥和水均无特异性条带, 而转 PeMADS1 基因的植株约在0.7 kb都有一明显的特征条带(图 5), 证明 PeMADS1 基因已经转入拟南芥。

|
图 5 转 PeMADS1 基因拟南芥植株PCR检测 Figure 5 PCR analysis of transformed plants of A.thaliana for PeMADS1 gene 1.野生型拟南芥; 2-7.转 PeMADS1 基因植株; 8.水对照; M. 1 kb分子量标记。 1.Wild type; 2-7.Transformed plants; 8.H2O control; M.1 kb molecular marker. |
分别以野生型拟南芥和表型明显的转基因阳性植株(卷叶、早花、延迟开花)的cDNA为模板, 用拟南芥的ACT7基因作内标, 调整cDNA的用量使之丰度一致后, 用引物PeMADS-F1和PeMADS-R1进行 PeMADS1 基因表达情况检测。结果表明: PeMADS1 基因在转基因植株(2, 3, 4)中得到了表达, 而野生型拟南芥没有检测到(图 6)。

|
图 6 转 PeMADS1 基因拟南芥植株RT-PCR检测 Figure 6 RT-PCR analysis of transformed plants of A.thaliana for PeMADS1 gene 1.野生型拟南芥; 2-4.转 PeMADS1 基因植株。 1.Wild type; 2-4.Transformed plants. |
毛竹是我国重要的经济竹种, 也是最受竹农欢迎的竹种之一, 然而造林主要以无性繁殖为主, 其品种来源仅限于对自然变异的选种, 杂交育种一直没有实质性进展。应用现代分子生物学技术来研究竹子开花生物学特性, 对于突破常规育种的局限性, 加速育种进程具有重要意义。本研究从毛竹(散生竹)中分离了 PeMADS1 基因, 其编码的蛋白质与其他属种的MADS-box有着较高的一致性, 证明MADS-box在进化上是高度保守的, 尤其是与来自绿竹(丛生竹)的BoMADS1完全一致, 意味着毛竹与绿竹在系统进化中可能处于极为相近的位置。在对 PeMADS1 基因及其编码蛋白序列分析的基础上, 推测该基因为E类功能基因。
研究表明, E类功能基因在植物由营养生长转向生殖生长的过程中起着重要作用(Immink et al., 1999)。本研究构建 PeMADS1 基因过量表达载体并转化模式植物拟南芥, T1代表型表明, 35S∷ PeMADS1 转基因拟南芥与35S ∷ DlMADS18, 35S∷OMADS1, 35S∷ NtMADS3及35S∷ DAL1的转基因后代有很多相似之处(田波等, 2005; Hsu et al., 2003; 高志民等, 2009; Ganesh et al., 2005), 如开花提早、莲座叶卷曲等。同时, 分子检测证实了目的基因在转基因植株内得到表达。因此, 可以初步推测 PeMADS1 基因可能参与毛竹的花发育, 而在毛竹花发育过程中 PeMADS1 基因是如何发挥作用的, 尚需通过竹子转基因来进一步证实。
高志民, 刘颖丽, 李雪平, 等. 2007. 一个绿竹MADS-box基因的克隆与序列分析[J]. 分子植物育种, 5(6): 866-870. |
高志民, 彭镇华, 陈段芬. 2009. 中国水仙NtMADS3基因全长的克隆与表达分析[J]. 北京林业大学学报, 31(3): 96-101. |
田波, 陈永燕, 严远鑫, 等. 2005. 一个竹类植物MADS盒基因的克隆及其在拟南芥中的表达[J]. 科学通报, 50(2): 145-151. |
徐芳, 熊爱生, 彭日荷, 等. 2005. 植物遗传转化的新方法:Floral Dip[J]. 中国蔬菜, (3): 29-31. |
袁金玲, 傅懋毅, 庄金坤, 等. 2005. 几个丛生竹开花授粉特性及麻竹苗期初步选择[J]. 竹子研究汇刊, 24(3): 9-13. |
Alvarez-Buylla E R, Liljegren S J, Palaz S, et al. 2000. MADS-box gene evolution beyond flowers:expression in pollen, endosperm, guard cells, roots and trichomes[J]. The Plant Journal, 24(4): 457-466. DOI:10.1046/j.1365-313x.2000.00891.x |
Angenent G C, Franken J, Busscher M, et al. 1995. A novel class of MADS-box genes is involved in ovule development in petunia[J]. Plant Cell, 7(10): 1569-1582. DOI:10.1105/tpc.7.10.1569 |
Coen E S, Meyerowitz E M. 1991. The war of the whorls:genetic interactions controlling flower development[J]. Nature, 353(6339): 31-37. DOI:10.1038/353031a0 |
Gao Zhimin, Li Xueping, Li Lubin, et al. 2006. An effective method for total RNA isolation from bamboo[J]. Chinese Forestry Science and Technology, 5(3): 52-54. |
Ganesh K A, Kiyomi A, Muneo Y, et al. 2005. Conservation of the Efunction for floral organ identity in rice revealed by the analysis of tissue culture-induced loss-of-function mutants of the OsMADS1 gene[J]. Plant Molecular Biology, 59(1): 125-135. DOI:10.1007/s11103-005-2161-y |
Gu Q, Ferrandiz C, Yanofsky M F, et al. 1998. The FRUITFULL MADSbox gene mediates cell differentiation during Arabidopsis fruit development[J]. Development, 125(8): 1509-1517. |
Hsu H F, Huang C H, Chou L T, et al. 2003. Ectopic expression of an orchid(Oncidium Gower Ramsey) AGL6-like gene promotes flowering by activating flowering time genes in Arabidopsis thaliana[J]. Plant Cell Physiology, 44(8): 783-794. DOI:10.1093/pcp/pcg099 |
Immink R G H, Hannapel D J, Ferrario S F, et al. 1999. A petunia MADS-box gene involved in the transition from vegetative to reproductive development[J]. Development, 126(22): 5117-5126. |
Liljegren S J, Ferrandiz C, Alvarez-Buylla R, et al. 1998. Arabidopsis MADS-box genes involved in fruit dehiscence[J]. Flowering Newsletter, 25: 9-19. |
Perry S E, Lehti M D, Fernandeze D E, et al. 1999. The MADS-domain protein AGAMOUS-like 15 accumulates in embryonic tissues with diverse origins[J]. Plant Physiology, 120(1): 121-129. DOI:10.1104/pp.120.1.121 |
Weigel D. 1995. The genetics of flower development:from floral induction to ovule morphogenesis[J]. Annual Review of Genetics, 29: 19-39. DOI:10.1146/annurev.ge.29.120195.000315 |
 2010, Vol. 46
2010, Vol. 46

