文章信息
- 王猛, 曹福祥, 龙绛雪
- Wang Meng, Cao Fuxiang, Long Jiangxue
- 马尾松捕光叶绿素a/b结合蛋白基因cab-Pm1的克隆与原核表达
- Cloning and Prokaryotic Expression of the Light Harvesting Chlorophyll a/b Binding Protein Gene from Pinus massoniana
- 林业科学, 2010, 46(9): 172-177.
- Scientia Silvae Sinicae, 2010, 46(9): 172-177.
-
文章历史
- 收稿日期:2009-11-20
- 修回日期:2010-01-09
-
作者相关文章
光合作用是光合生物将光能转化为化学能,并同时将无机物转化成有机物贮存在生物体的过程。现已证明光合生物对光能的吸收、传递和转化都是在光合膜上具有一定的分子排列和空间构象的色素蛋白复合体上进行的(郁飞等,2001)。在高等植物体内,捕光叶绿素a/b结合蛋白基因(cab)属于光合系统基因(Raghvendra,1998),其编码的蛋白质与色素形成捕光色素蛋白复合体(LHC),是一类把能量迅速传至反应中心引起光化学反应的色素蛋白复合体。大部分的叶绿素分子都结合在光系统Ⅰ(PSⅠ)和光系统Ⅱ(PSⅡ)的天然色素蛋白复合物上。真核生物的PSⅠ复合物包括了一系列捕光色素蛋白即LHCⅠ,现已知道LHCⅠ蛋白由核基因编码,在细胞质中合成后在引导肽的作用下被运输进叶绿体并整合到类囊体膜上(郁飞等,2001)。LHCⅡ是一个重要的膜蛋白,是绿色植物叶绿体类囊体中含量最丰富的一种天线蛋白,行使光能传递、转化和分配等功能,在植物光合作用中起到关键的作用(孙钦秒等,2000)。
马尾松(Pinus massoniana)在我国分布广,数量最多,在南方各省森林蓄积量中,马尾松占一半以上。由于松材线虫的入侵导致大量马尾松感病死亡,给我国的林业造成重大损失。在感病后的马尾松生理变化中,有比较明显的光合作用下降,针叶变色及叶绿素含量下降的现象(王猛,2006)。关于感病后松树的生理变化研究的比较多,但是关于其分子机制的研究还比较少。
本研究根据植物cab家族基因保守序列,设计引物,利用RT-PCR和RACE技术从马尾松中克隆到全长cab基因,并对该基因构建原核表达载体,在大肠杆菌中诱导表达。旨在为研究感病后松树生理变化的分子机制以及病虫害对其影响奠定基础。
1 材料与方法 1.1 材料1) 植物材料 马尾松2年生实生苗购于湖南省桃江县,移栽苗圃后,取其新鲜叶片用于试验。
2) 菌株与质粒 感受态细胞DH5α和BL21(DE3)购自Tiangen,克隆载体pGEM-T easy和pET-24a(+)分别购自Promega公司和Novagen公司。
3) 主要试剂 核酸分子质量标准、蛋白分子量标准、Taq DNA聚合酶、EcoRⅠ、NdeⅠ购自TaKaRa公司;Concert Plant RNA Reagent购自Invitrogen;SMART RACE cDNA Amplification kit购自Clontech;DNA凝胶纯化回收试剂盒购自北京鼎国生物科技公司;质粒小量提取试剂盒购自OMEGA;反转录试剂盒购自Promega。其他试剂均为国产分析纯。
1.2 方法1) 马尾松总 RNA提取与cDNA合成取马尾松实生苗幼嫩新鲜叶片,加入液氮碾磨,转入1.5 mL的离心管,具体步骤按照Concert Plant RNA Reagent说明书操作,得到的总RNA样品储存-76 ℃超低温冰箱中。cDNA第1链的合成按照Promega公司的反转录试剂盒说明书进行操作, 得到的DNA第1链-20 ℃保存备用。
2) 引物设计与合成 根据松科植物捕光叶绿素a/b结合蛋白基因家族的同源序列设计PCR引物,引物序列由上海生工生物工程技术服务公司合成。
HcpF:5’-ATCGGGTCGTGCGCTTTC-3’;HcpR:5’-TCCTTCACCTTCAGCTCCGC-3’;3GSP:5’-AGAAATGGAGTAAAGTTCGGGGAGGC-3’;5GSP:5’-CTGACACTTTTTTGGTGGTCGCCTTCC-3’。
3) 叶绿素捕光蛋白基因克隆与测序 以合成的cDNA为模板,进行核心序列PCR扩增。引物HcpF和HcpR反应条件为:94 ℃预变性3 min;94 ℃ 30 s,50 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃延伸10 min,扩增完毕后4 ℃终止反应。PCR产物经1%琼脂糖凝胶电泳后回收、纯化。将纯化的PCR产物与pGEM-T easy载体连接,重组子转化DH5α大肠杆菌后37 ℃培养;挑取单个白色菌落于37 ℃过夜培养后抽提质粒,PCR鉴定,送上海生工公司测序。为了获得基因的全长,根据测序结果设计引物3GSP和5GSP分别与SMART RACE试剂盒通用引物(UPM)配对进行RACE扩增。反应条件为94 ℃预变性3 min;94 ℃ 30 s,58 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃延伸10 min。扩增产物回收连接及转化同上,送样测序。
4) 序列的拼接与生物信息学分析 根据获得的核心序列、3’RACE末端和5’ RACE末端进行拼接以获得基因的全长,DNA序列相似性分析在NCBI站点上的BLAST进行。推导的氨基酸序列分析用Genedoc软件,多重序列比对和进化树分析用Vector NTI 10.0软件和DNAStar软件。推测蛋白的相关生物学信息在http://www.expasy.ch/tools/中进行,使用SignalP3.0 Server在线工具预测蛋白质信号肽。
5) 表达载体的构建及目的蛋白的诱导表达 根据所获得的基因全长,采用直接添加酶切位点方法,设计1对引物扩增叶绿素捕光蛋白基因的编码区,

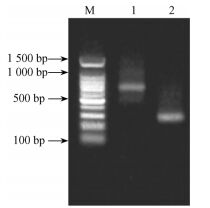
以马尾松总RNA反转录合成的cDNA为模板,扩增出来产物通过1%的琼脂糖凝胶中电泳,得到1条约650 bp片段(图 1)。经过凝胶纯化回收后,按照Promega的pGEM-T easy载体操作流程,连接到载体中,转化DH5α后在含有氨苄青霉素培养基上培养,经过蓝白筛选后挑取阳性克隆后送到上海生工进行测序。测序结果表明,插入片段为648 bp。应用Blast在线软件,将序列与已经登录的基因进行同源性比较分析,发现所获得片段与cab基因家族有较高的同源性。使用3’和5’末端特异性引物,做RACE扩增,引物3GSP和UPM扩增产物在700 bp左右有1个亮带(图 2中1);引物5GSP和UPM扩增产物在300 bp处有1个亮带(图 2中2)。将PCR产物回收连接后转化,挑取阳性克隆进行测序。

|
图 1 RT-PCR产物电泳图 Figure 1 DNA fragment amplified by RT-PCR M:100 bp分子质量标记100 bp ladder marker;1:RT-PCR产物The product of RT-PCR. |
|
图 2 3’RACE和5’RACE PCR产物电泳图 Figure 2 DNA fragment amplified by 3'RACE and 5'RACE 1:3’RACE产物Product of 3’RACE;2:5’RACE产物Product of 5’RACE;M:100 bp分子质量标记100 bp ladder marker. |
通过已经获得的核心序列与3’RACE和5’RACE序列的拼接得到基因全长cDNA序列,全长为1 062 bp,含有1个完整编码区(825 bp),该基因编码274个氨基酸、3’非翻译区(160 bp),包含25个poly(A)、5’非翻译区(67 bp)。GenBanK登录号为GU073386。对该基因的推导的氨基酸序列进行Protparam程序预测分析,该基因编码274个氨基酸,分子质量为28.98 ku,理论等电点为5.24,亲水指数为0.052。使用MOTIFSCAN对GU073386序列的编码区分析,结果显示序列的编码区第73~241位包含典型的捕光叶绿素a/b结合蛋白功能域(chlorophyll a/b binding domain),第18~20,40~42,45~47,243~245位为蛋白激酶C的磷酸化位点。5~10,13~18,28~33,117~122,131~136,178~183,187~192位为N-豆蔻酰化位点,42~45,47~50位为cAMP和cGMP依赖性的蛋白激酶磷酸化位点。该基因命名为cab-Pm1(cab gene from P.massoniana)。
2.2 与其他物种的cab基因比对分析通过Blast软件分析,在NCBI核酸数据库中,基因序列比较的得分为287~1 762,其中与S73603(黑松Pinus thunbergii)序列相似性最高,得分为1 762,一致性为98%,其次与欧洲赤松(Pinus sylvestris)和小干松(Pinus contorta)的得分分别为1 746和1 659,一致性分别为98%和95%。在蛋白质数据库中,分值为451~550,与cab-Pm1同源性最高的也是欧洲赤松,得分550。使用Genedoc软件,对9种松科植物的氨基酸同源性进行比对分析(图 3)。图中黑色区域为高度保守的相同氨基酸序列,灰色区域为部分相同氨基酸序列,白色区域为差异的氨基酸序列。从分析结果可以看出,cab-Pm1基因编码的蛋白与其他松科植物的cab基因有很高的相似性,与南亚松(Pinus merkusii)一致性达到99.2%,与北美云杉(Picea sitchensis)一致性最低, 为90.5%,充分说明cab家族基因在进化过程中的保守性。对上述9种同源性较高的物种的进化树分析表明,种间遗传距离更近,属间遗传距离较远,与分类学一致(图 4)。

|
图 3 cab-Pm1基因编码的氨基酸与其他cab基因的编码氨基酸多序列比较 Figure 3 Multiple sequence alignments of amino acids encoded by cab-Pm1 with amino acids encoded by the different cab genes CAA57408:欧洲云杉Picea abies;ABK22510:北美云杉Picea sitchensis;CAC38830:小干松Pinus contorta;AAU89251:西黄松Pinus ponderosa;AAU89252:辐射松Pinus radiata;ACY66805:马尾松Pinus massoniana;AAU89254:南亚松Pinus merkusii;CAA32658:欧洲赤松Pinus sylvestris;AAC78690:黑松Pinus thunbergii. |

|
图 4 不同物种cab基因编码蛋白系统进化树分析 Figure 4 Phylogenetic tree analysis of proteins encoded by cab gene |
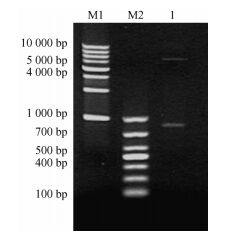
根据设计的引物PmF和PmR进行PCR,扩增产物凝胶电泳显示在800 bp左右有1条清晰条带(图 5),与预测基因的开放读码框大小相符。对阳性克隆菌提取质粒进行双酶切检验,电泳检测在800 bp和5 000 bp处各有1条带,符合目的基因和载体片段大小(图 6),进一步表明cab-Pm1基因已经成功插入pET-24a的多克隆位点。

|
图 5 cab-Pm1 ORF的扩增图 Figure 5 cab-Pm1 ORF DNA fragment amplified by PCR M:100 bp分子质量标记100 bp ladder marker;1:The product of cab-Pm1 ORF. |
|
图 6 重组表达载体的酶切检测 Figure 6 The recombinant expression vector checked by restriction enzymes 1:NdeⅠ/EcoRⅠ;M1:1 kb分子质量标记1 kb ladder marker;M2:DL1000分子质量标记DL1000 DNA marker. |
将所克隆的重组表达载体转入到BL21(DE3)菌株,在37 ℃条件下培养至A600为0.8时,加入IPTG至终浓度为1 mmol·L-1进行诱导表达蛋白,取1,2,3,4 h时间段的菌液进行15% SDS-PAGE电泳,在空载体质粒菌液里没有表达目的蛋白条带,而其他诱导菌在29 ku左右有明显的特异条带产生(图 7)。对不同浓度的IPTG对目的蛋白诱导4 h表达分析,结果显示当IPTG浓度逐渐增加时,目的蛋白(约29.0 ku)的表达量逐渐增大,当浓度增加到0.1 mmol·L-1,目的蛋白表达量最大(图 8)。由此也解释在1 mmol·L-1浓度下IPTG在不同诱导时间表达蛋白无明显差异的原因。

|
图 7 诱导时间对pET-cab-Pm1表达的影响 Figure 7 The expression of pET-cab-Pm1 in different time M:蛋白分子质量标准Protein molecular standard marker;1:空载体pET-24a(+);2-5:分别为加入1 mmol·L-1 IPTG诱导下1,2,3,4 h的表达产物Induced pET-cab-Pm1 at 1,2,3 and 4 h respectively. |

|
图 8 IPTG浓度对pET-cab-Pm1表达的影响 Figure 8 The expression of pET-cab-Pm1 in different concentration of IPTG M:蛋白分子质量标准Protein molecular standard marker;1:未加IPTG的pET-cab-Pm1 Non-induced pET-cab-Pm1;2-5:分别为0.01,0.05,0.1,0.5 mmol·L-1IPTG终浓度诱导的pET-cab-Pm1 Induced with 0.01,0.05,0.1,0.5 mmol·L-1 of IPTG. |
与国际核酸和蛋白质数据库联网进行Blast比对分析,cab-Pm1相似性最高是松属(Pinus)植物,其次为云杉属(Picea)。在松属、云杉属、沙梨(Pyrus pyrifolia var. culta)、野蕉(Musa balbisiana)、Lemna gibba、高粱(Sorghum bicolor)、地中海柏木(Cupressus sempervirens)、稀脉浮萍(Lemna paucicostata)、蓖麻(Ricinus communis)、毛竹(Phyllostachys edulis)、风信子(Hyacinthus orientalis)、大麦(Hordeum vulgare)、Nicotiana sylvestris等十几个不同的物种,Blast比较结果中的Score分值为287~1 762,其中属内cab-Pm1与黑松S73603序列相似性最高,Score分值为1 762,一致性(identity)为98%(988/1003)。在不同属之间,则与北美云杉相似性最高,相似性达到95%,说明属内同源性高于属间同源性。对cab-Pm1编码的氨基酸序列进行比对,得出了和核酸序列比对相一致的结论。在NCBI蛋白质数据库中共搜索到与cab-Pm1编码氨基酸有一定相似性的100条序列中,Score分值最低为451,最高为550,其中相似性最高是黑松,为268/274 (97%)。cab-Pm1编码区第73~241位包含典型的捕光叶绿素a/b结合蛋白功能域,其功能可能是与色素分子结合;蛋白激酶C的磷酸化位点均存在毛竹、水稻(Oryza sativa)、豌豆(Pisum sativum)、胡杨(Populus euphratica) cab编码的蛋白上,但是位置和数量不同。cAMP和cGMP依赖性的蛋白激酶磷酸化位点,在已报道的禾本科(Gramineae)植物中没有发现,N-豆蔻酰化位点的数目和位置与毛竹差异较大。这些位点可能与该基因转录后的调节有关(Allen et al., 1997)。
有研究表明在裸子植物,特别是松柏类植物cab基因的表达不受光的调控(Mukai et al., 1991;1992),并认为这是由于松柏类植物cab基因的上游缺乏光响应顺式因子(light-responsive cis-element)所致(Mukai et al., 1992)。对于马尾松cab基因的调控还有待进一步的研究。感染松树枯萎病的马尾松体内水分减少,造成叶绿素含量降低,影响了马尾松正常的生理功能,是造成马尾松大量死亡的原因之一。研究表明在水分胁迫下叶绿素的丧失,叶绿素a/b比值的增加及光合单位的减小都是LHC蛋白含量减少所致,故认为LHC蛋白是水分胁迫易攻击的一个靶子(Alberte et al., 1977;Vapaavuori et al., 1982)。cab在许多生物和非生物环境胁迫下表达都会受到影响,从而影响植物的光合作用(Chur et al., 2003;Gomber et al., 2006;Kufryk et al., 2008)。
cab-Pm1基因在大肠杆菌BL21(DE3)中的诱导发现,在1 mmol·L-1的IPTG诱导条件下,1 h的蛋白表达量和4 h的蛋白表达量几乎一样,说明推荐的浓度比较大,在进行IPTG不同浓度的诱导试验中发现,当浓度达到0.1 mmol·L-1时已经可以诱导大量的目的蛋白表达。本研究获得的马尾松捕光叶绿素a/b基因是否与抗性生理机制相关,尚需进一步深入研究,对马尾松感病后其生理功能变化的分子机制研究将是一个复杂而漫长的过程,下一步的研究工作是Cab蛋白的纯化及生理功能的研究。
孙钦秒, 冷静, 李良璧, 等. 2000. 高等植物光系统Ⅱ捕光色素蛋白复合体结构与功能研究的新进展[J]. 植物学通报, 17(4): 289-301. |
王猛. 2006. 湿地松对松材线虫抗病性的主要理化指标影响的研究. 中南林业科技大学硕士学位论文.
|
郁飞, 唐崇钦, 辛越勇, 等. 2001. 光系统Ⅰ(PSⅠ)的结构与功能研究进展[J]. 植物学通报, 18(3): 266-275. |
Alberte R S, Thornber J P, Fiscus E L. 1977. Water stress effects on the content and organization of chlorophyll in mesphyll and bundle sheath chloroplasts of maize[J]. Plant Physiol, 59: 351-353. DOI:10.1104/pp.59.3.351 |
Allen J F, Nilsson A. 1997. Redox signaling and the structural basis of regulation of photosynthesis by protein phosphorylation[J]. Physiol Plant, 100(4): 863-868. DOI:10.1111/ppl.1997.100.issue-4 |
Churin Y, Adam E, Kozma-Bognar L, et al. 2003. Characterization of two Myb-like transcription factors binding to CAB promoters in wheat and barley[J]. Plant Mol Biol, 52(2): 447-462. DOI:10.1023/A:1023934232662 |
Gomber T J, Etienne P, Ourr Y A, et al. 2006. The expression patterns of SAG12/Cab genes reveal the spatial and temporal progression of leaf senescence in Brassica napus L. with sensitivity to the environment[J]. J Exp Bot, 57(9): 1949-1956. |
Kufryk G, Hernandez-Prieto M A, Kieselbach T, et al. 2008. Association of small CAB-like proteins (SCPs) of Synechocystis sp.PCC 6803 with photosystem Ⅱ[J]. Photosynth Res, 95(2/3): 135-145. |
Mukai Y, Yamamoto N, Koshiba T. 1991. Light-independent and tissue-specific accumulation of light-harvesting chlorophyll a/b binding protein and ribulose bisphosphace carboxylase in dark-grow pine seedlings[J]. Plant Cell Physiol, 32: 1303. |
Mukai Y, Tazaki K, Fujii T, et al. 1992. Light-independent expression of three photosynthetic genes, cab, rbsS and rbcL in coniferous plants[J]. Plant Cell Physiol, 33(7): 859-866. |
Raghvendra A S. 1998. Photosythesis: a comprehensive treatise[M]. Cambridge: Cambridge Univ Press: 72-86.
|
Vapaavuori E, Nurmi A. 1982. Chlorophyll-protein complexes in Salix sp. Aquatica Gigantea under strong and weak light Ⅱ.effect of water stress on the chlorophyll protein complexes and chloroplast ultrastructure[J]. Plant Cell Physiol, 23(5): 791-801. |
 2010, Vol. 46
2010, Vol. 46

