文章信息
- 王源秀, 徐立安, 黄敏仁, 王明庥, 张美荣
- Wang Yuanxiu, Xu Li'an, Huang Minren, Wang Mingxiu, Zhang Meirong
- 利用不同作图软件构建响叶杨×银白杨遗传图谱
- Comparison of Genetic Linkage Maps of Populus adenopoda × P. alba Hybrid Constructed by Different Mapping Softwares
- 林业科学, 2010, 46(7): 55-63.
- Scientia Silvae Sinicae, 2010, 46(7): 55-63.
-
文章历史
- 收稿日期:2009-06-19
- 修回日期:2009-09-07
-
作者相关文章
2. 南京林业大学林木遗传与生物技术省部共建教育部重点实验室 南京 210037
2. Key Laboratory of Forest Genetics & Biotechnology of Ministry of Education Nanjing Forestry University Nanjing 210037
杨树(Populus)具有良好的生物学特性,如生长快、易繁殖、基因组较小(~480 Mbp)、易转化等,是目前公认的模式树种。随着杨树全基因组序列测定的完成,杨树在多年生植物的基因组研究中将起到重要作用(Tuskan et al., 2006; Jansson et al., 2007)。
目前为止,共有27张杨树遗传连锁图谱发表,涉及白杨派(Sect. Leuce)、黑杨派(Sect. Aigerio)和青杨派(Sect. Tacamahaca)的13个树种(Cervera et al., 2004; Gaudet et al., 2008; Woolbright et al., 2008; Pakull et al., 2009; Zhang et al., 2009)。其中Yin等(2001)曾利用RAPD分子标记技术和“拟测交”策略获得了中等密度的银白杨(P. alba)连锁图谱和响叶杨(P. adenopoda)图谱的1个连锁框架; 由于该研究使用的是RAPD显性随机标记,不具可比性; 同时该研究的作图群体也较小(只包含80个单株),随着群体的不断缩小,后续研究受到极大局限。因此本研究重建2树种的作图群体,重建的F1作图群体包含1 000多个单株。响叶杨和银白杨是我国优良乡土树种,属于杨属中比较原始的白杨派,白杨派树种在杨属的遗传进化和系统发育研究中占重要地位,具有较高的研究价值。Pakull等(2009)利用AFLP和SSR标记及JoinMap作图软件构建了白杨派2树种欧洲山杨(P. tremula)和美洲山杨(P. tremuloides)的遗传连锁图谱,并利用连锁到2个图谱上的SSR标记与杨树一致性图谱(consensus map)(http://popgenome.ag.utk.edu/cgi-bin/cmap/map_set_info; Yin et al., 2004)进行了比较,发现图谱间存在较好的标记共线性(collinearity)。这是首次发表包含SSR标记的白杨派树种的遗传图谱。
MapMaker和JoinMap作图软件是林木遗传图谱构建研究的常用软件。Lander等(1987)编写的MapMaker软件作图大多使用“拟测交”作图策略(Grattapaglia et al., 1994),默认位点间的连锁相为相引相,分别构建父母本的连锁图谱。但林木为异交物种,遗传组成高度杂合,对于特定的1个杂交组合所构建的作图群体,其相邻标记的分离类型达7种之多,可能的分离比有1:1:1:1, 1:2:1, 3:1和1:1,有些共显性标记具有完全信息,如ab×cd(4个等位基因),如果使用“拟测交”策略作图,将损失大量的遗传信息(张博等,2003)。Van Ooijen等(2001)编写的JoinMap作图软件考虑到了不同分离类型的标记位点间的连锁分析,可用于异交的全同胞群体的遗传作图; 但对于一列有序的标记位点,MapMaker使用隐马尔可夫链模型(HMM)、多位点连锁分析估计遗传距离,而JoinMap使用最小二乘法(LS)估计相邻位点间的遗传距离,没有使用多位点连锁分析。施季森等(2006)编写的作图软件FsLinkageMap,使用是隐马尔科夫链模型的方法,并考虑到了位点间的连锁相信息,进行了多位点连锁分析,可以利用林木F1作图群体的遗传信息构建遗传连锁图谱。
本研究利用SSR和SRAP(sequence related amplified polymorphism)标记技术采集响叶杨×银白杨F1作图群体的遗传信息,采用FsLinkageMap 2.0(施季森等,2006)和JoinMap 4.0作图软件(Van Ooijen, 2006)构建响叶杨×银白杨的整合图谱(integrated map),并进行二者的比较分析; 同时利用FsLinkageMap 2.0和MapMaker 3.0软件对母本响叶杨的遗传信息进行连锁分析,构建响叶杨的遗传图谱,并进行对比分析。希望本研究能为杨属内基因组图谱比较研究提供详尽的证据。
1 材料与方法 1.1 作图群体从1 100个响叶杨×银白杨成活F1代中随机挑选189个构成作图群体。作图群体的具体信息和DNA的提取方法见Wang等(2009)。
1.2 SSR和SRAP分子标记共对1 142对SSR引物和163个SRAP引物组合进行了筛选。通过筛选,1 142对SSR引物中检测到264对多态引物,共扩增出286个分离位点,其中47个位点为1:1:1:1分离。163个SRAP引物组合中40个组合产生多态DNA片段,共获得131个多态位点。引物的具体信息和检测方法见Wang等(2009)。
1.3 图谱构建利用χ2(P=0.001, P=0.005)对标记进行孟德尔分离分析,偏分离标记保留在数据中用于计算。利用Excel软件准备数据。利用FsLinkageMap2.0(施季森等,2006; http://202.119.214.18/software/FsLinkageMap/)构建响叶杨×银白杨的整合图谱和响叶杨的遗传图谱,都是在Lod>3.0、重组率R<0.3条件下估计连锁群。采用JoinMap4.0构建响叶杨×银白杨的整合图谱,在重组率R≤0.25情况下对标记进行分群,设置连锁分析参数为: Rec=0.45,Lod=1.0,Jump=5。
利用MapMaker3.0构建的母本响叶杨的遗传图谱是根据“拟测交”策略,参照其中“F2 backcross”数据形式进行计算分析的。命令“TRIPLE ERROR DETECTION”和“ERROR DETECTION”用于检测错误。先进行两点分析,再分群,Lod值和最大重组率取默认值(3.0和0.5)。连锁群中标记的顺序是通过多点分析决定的。大于8个标记的连锁群先用“THREE POINT”命令分析,然后用“ORDER”命令选择标记的第1顺序,第1顺序由“Ripple”命令决定标记的先后顺序,最后用“TRY”命令将其他标记加到最优顺序之中。小于或等于8个标记的连锁群,用“COMPARE”命令决定标记的顺序。估算标记间遗传距离均利用Kosambi作图函数。绘制图谱利用软件Mapchart2.2(Voorrips, 2002)。
1.4 连锁图谱的参数计算遗传图谱实际长度为框架图长度,即连锁群(大于4个标记)长度之和; 连锁群总长度包括连锁群、三联体和连锁对在内所有连锁长度的总和。
根据Hulbert等(1988)提出的方法估算基因组长度。利用Lange等(1982)基于标记随机分布的假设提出的函数估计图谱的基因组覆盖度。
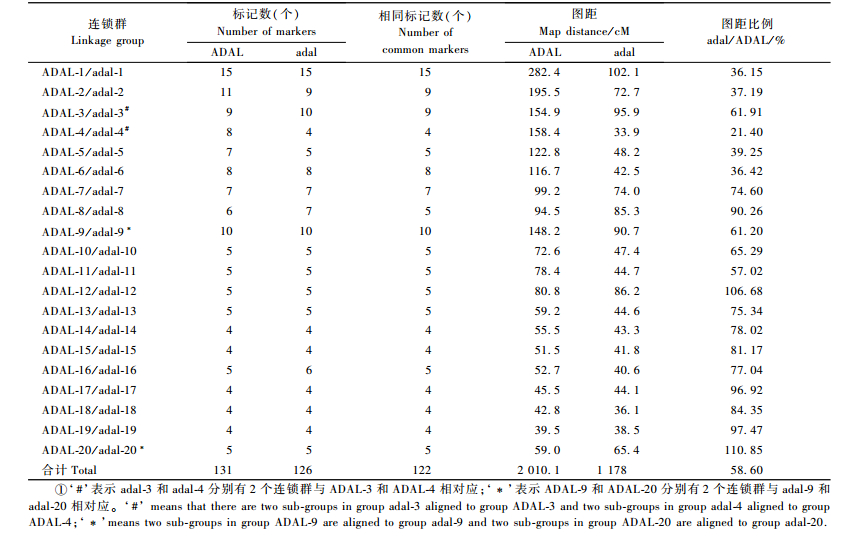
2 结果与分析 2.1 标记分离分析利用χ2检测标记分离: ab×aa和aa×ab期望分离比1:1(d=1), ab×cd期望分离比1:1:1:1(d=3),ab×ab期望分离比1:2:1(d=2)。通过计算发现有70个(16.79%)标记偏离于期望分离比(P=0.005);其中连锁到FsLinkageMap2.0构建的整合图上的偏分离标记有16个(18.57%),连锁到JoinMap4.0构建的整合图上的偏分离标记有9个(12.86%)。研究发现偏分离标记有聚集现象,其中集中在ADAL-4连锁群的偏分离标记有6个(图 1)。

|
图 1 FsLinkageMap2.0与JoinMap4.0图谱的比较 Figure 1 Comparison of the maps separately based on FsLinkageMap2.0 and JoinMap4.0 ADAL:FsLinkageMap2.0作图; adal:JoinMap4.0作图。连锁群左为图距,右为标记。灰色部分代表偏分离区域。标记后缀‘d’代表偏分离(P=0.005),‘dd’代表严重偏分离(P=0.001)。下同。 'ADAL'is the map based on FsLinkageMap2.0 and'adal'is on JoinMap4.0. Numbers on the left of each group show absolute marker position. Marker names are on the right. Shaded regions indicate blocks of distortion. distorted markers note with the suffix "d" and "dd" which deviating at 0.001 < P≤0.005 and seriously deviating at P≤0.001 respectively. The same below. |
根据响叶杨×银白杨的整合图谱,当Lod=5.0时(Yin et al., 2004),重组率Rec=0.3,基于234个构图标记,检测到的连锁对数k=402,估计杨树基因组大小为2 695.56 cM,95%的置信区间为2 455.52~2 987.62 cM。图谱的基因组覆盖度估计为93.34%。
2.3 FsLinkageMap2.0作图与JoinMap4.0作图的比较FsLinkageMap2.0和JoinMap4.0软件都可以分析全同胞作图群体的全部遗传信息,可获得响叶杨×银白杨F1的整合图谱。本研究共获得417个分离位点参与构建图谱。利用FsLinkageMap2.0构建整合图,在重组率(Rec)<0.245,Lod>3.0时,图谱共分为62个连锁群[包含19个含有4个或以上标记的群,16个三联体(triplet),27个二联体(doublet)]和183个未检测到连锁的标记。对每一个群进行作图分析,放弃图距超过35 cM的标记,共有20个主要连锁群(包含4个或以上标记), 可成功构建框架图(图 1,连锁群命名为‘ADAL’+阿拉伯数字),包括了126个标记(102个SSR和24个SRAP)。连锁群图距39.5~282.4 cM,共1 966 cM; 平均每个连锁群长度98.3 cM; 标记间距平均15.6 cM(表 1, 图 1)。另外有16个三联体和28个二联体,共包含104个标记,图距分别为554.0 cM和473.5 cM(未列出),因此所获得的图谱总图距为2 993.5 cM。
|
|
利用JoinMap4.0构建整合图,在重组率仍为0.245的情况下,图谱共有63个连锁群[包含20个含有4个或以上标记的群,16个三联体(triplet),27个二联体(doublet)]和182个未检测到连锁的标记。对每一个群进行作图分析,也得到20个主要连锁群(包含4个或以上标记), 可成功构建框架图(图 1,连锁群命名为‘adal’+阿拉伯数字),其中adal-9连锁群是在重组率为0.25的情况下构建的,因为在重组率为0.245时,该连锁群被分为2部分,为了对比的方便,本研究使用了重组率为0.25时的连锁群。框架图包括了122个标记(100个SSR和22个SRAP),连锁群图距36.1~102.1 cM,共1 144.1 cM; 平均每个连锁群长度57.2 cM; 标记间距平均9.38 cM(表 1和图 1)。另外有16个三联体和29个二联体,共包含106个标记,图距分别为499.4, 515.0 cM(未列出); 因此所获得的图谱总图距为2 155.5 cM。
对二者获得的整合图谱的框架图进行比较(图 1),结果发现: 2张图谱的图距差异显著,adal图谱(即利用JoinMap4.0软件构建的整合图,下同)中大部分连锁群的图距小于ADAL图谱(即利用FsLinkageMap2.0软件构建的整合图,下同)的对应连锁群的图距(表 1,只有第12和20是略大于ADAL图谱的对应连锁群的图距),其中连锁群adal-1, adal-2, adal-4, adal-5和adal-6图距达不到ADAL图谱的对应连锁群的40%,总的来看,adal图谱的总图距只有ADAL的58.6%。2张图谱上共发现了122个相同的标记,根据连锁群上的相同标记对比发现,除第3, 4, 9, 20连锁群外,其余连锁群都发现唯一对应连锁群; ADAL-3连锁群对应adal图谱中的2个连锁群; ADAL-4连锁群只对应了adal图谱中的2个二联体,这是由于该连锁群中的标记大部分是偏分离标记,不同软件进行统计分析时产生了不同的结果; 而adal-9连锁群对应了ADAL图谱中2个连锁群; adal-20连锁群在ADAL图谱中的分成了1个二联体和1个三联体。在ADAL图谱上有10个标记(即G_93B, G_3466, G_901, P_2879C, ME12EM13_250, ME2SA4_380, G_2167, G_2894, ME12EM9_470, P_2681C)没有出现在adal图谱的对应连锁群上,在adal图谱上也有5个标记(即G_1894, G _1753A, G _880A, G_880C, G_3467, ME1OD3_300)没有出现在ADAL图谱的对应连锁群上。另外在第1, 2和9对应连锁群上的相同标记在连锁群上的排列顺序存在显著差异,在第5, 6, 10, 12, 16和20对应连锁群上的相同标记出现倒位现象。将第1对应连锁群上的相同SSR标记与杨树一致性图谱相比较(图 2),发现该对应连锁群与杨树一致性图谱第Ⅲ连锁群是同源连锁群,共有9个同源标记,但adal-1连锁群上同源标记的排列顺序与一致性图谱第Ⅲ连锁群的标记排列顺序更加符合。在其他对应连锁群与一致性图谱的比较中也发现类似现象。

|
图 2 FsLinkageMap2.0和JoinMap4.0图谱与杨树一致性图谱的对比 Figure 2 Alignment of the maps separately based on FsLinkage Map2.0 and JoinMap4.0 to poplar consensus map 左为FsLinkageMap2.0作图的第1连锁群;右为JoinMap4.0作图的第1连锁群;中间为杨树一致性图谱的第3连锁群。 The first group by FsLinkageMap 2.0 is on the left and one by JoinMap 4.0 on the right. The third group of the poplar consensus map is in the middle. |
通过响叶杨×银白杨F1作图群体获得的母本响叶杨的遗传信息,共209个标记(151个SSR和58个SRAP),利用FsLinkageMap2.0(Rec<0.3,Lod>3.0)和MapMaker3.0(Lod=3.0)进行连锁分析,获得其遗传图谱(分别命名为ad和AD图谱)。通过对2张图谱进行对比发现: 2图谱共有32个对应连锁群, 其中28个对应连锁群为1对1的关系,3个(ad-5/AD-5, ad-15/AD-15和ad-21/AD-21)为2对2的关系,1个(ad-12/AD-12)为3对1的关系,另外ad图谱中有3个(ad-A, ad-B和ad-C)而AD图谱中有2个(AD-A和AD-B)连锁群没对应连锁群; 2图谱的对应连锁群总图距差异不大(相差52.2 cM即2.47%),但有6个(ad-5/AD-5, ad-13/AD-13, ad-14/AD-14, ad-16/AD-16, ad-19/AD-19和ad-32/AD-32)对应连锁群的图距差异超过50%;对应连锁群上的113个相同标记(105个SSR和8个SRAP)中有5个(G_3869, G_3269, G_1065, O_207和G_2768)出现倒位现象, 2图谱上有9个相同标记(G_20, G_2020, G _40, O_30B, G_114, G_1146, ME13EM8_140, ME7EM16_290和ME6EM14_450)位于不同的对应连锁群上; 另外ad图谱123个SSR和22个SRAP标记中分别有10和11个没有出现在对应连锁群上, 各占8.13%和50.00%,AD图谱116个SSR和24个SRAP标记中分别有3和14个没有出现在对应连锁群上, 各占2.59%和58.33%(表 2和图 3),可见共显性标记比显性标记有更好的对应关系。
|
|

|
图 3 FsLinkageMap2.0与MapMaker3.0图谱的比较 Figure 3 Comparison of the maps separately based on FsLinkageMap2.0 and MapMaker3.0 ad:FsLinkageMap2.0作图; AD:MapMaker3.0作图。 'ad' is the map based on FsLinkageMap2.0 and 'AD' is on MapMaker3.0. |
本研究利用FsLinkageMap2.0和JoinMap4.0构建响叶杨×银白杨F1的整合图谱,在重组率为0.245的情况下,都有62个连锁群,含4个以上标记的连锁群有19个,与杨树单倍体染色体数(n=19)一致。对每个连锁群进行作图分析,最终都获得了包含20个主要连锁群的遗传框架图(图 1)。
根据F1的遗传图谱估计杨树基因组大小为2 695.56 cM,图谱的基因组覆盖度估计为93.34%。图谱标记数量和Lod值的选取将共同影响基因组大小的估计。一般情况下杨属树种的基因组大小估计需要标记数至少为100个,并且随着标记数的增加Lod值也增加,如100个标记Lod为3,当标记数为150个时Lod值取5(Yin et al., 2004)。本研究获得的标记数为234或235,当Lod值取5时估算的杨树基因组大于期望值2 410 cM(Yin et al., 2004)。当图谱上的标记覆盖全基因组后, 基因组随标记数的增加是收敛的,不会随标记数的增加而产生巨大变动。但在已经构建的杨属树种中,多数图谱揭示的是基因组长度随标记的增加呈非收敛性增加(Yin et al., 2001; Zhang et al., 2004; Gaudet et al., 2008; Woolbright et al., 2008; Zhang et al., 2009; Pakull et al., 2009),而且随着标记数的增加往往非连锁标记的数量也在增加,这可能与标记基因型的错误有关。
通常种间杂交都会产生偏分离现象,标记的偏分离现象普遍存在。偏分离标记会影响图谱的构建,但本研究为了对图谱进行比较,将所有偏分离标记都用于了连锁分析。利用χ2检测发现连锁到FsLinkageMap2.0构建的框架图上的偏分离标记有16个,占总标记数的12.2%,这与Gaudet等(2008)发表的欧洲黑杨(P. nigra)的父母本遗传图谱上的偏分离标记的比例(分别为10%和14%)相差不大,小于Zhang等(2009)公布的美洲黑杨(P. deltoides)和欧美杨(P. euramericana)遗传图谱上的偏分离标记的比例(分别20.4%和21.7%)。关于造成偏分离的原因很多,在本研究中可能是遗传隔离(genetic isolation)造成的,母本响叶杨是生长在温暖湿润的华东地区(南京),而父本银白杨是生长在干旱的新疆玛纳斯,二者的地理分布差异较大,存在杂交不亲和现象,杂交后得到的子代种子即使通过组培也只有部分能萌发; 培养出的校虹由温室移入大田时,由于少部分苗不适应条件的变化被损失掉,这些因素都会导致偏分离现象; 同时取样和基因型统计误差也会使标记产生偏分离(Kuang et al., 1999)。不过,由于本研究分析的标记较少,对于偏分离产生的原因还需要更多的证据。与发表的其他杨树图谱一样,本研究也发现偏分离标记有聚集现象(图 1),特别是偏分离标记P_2879(位于连锁群ADAL-4或adal-4上),在美洲山杨和欧洲黑杨的图谱上同样处于偏分离标记聚集区,说明这个偏分离标记聚集区的基因受到某种选择压力的影响(Yin et al., 2004; Gaudet et al., 2008; Woolbright et al., 2008; Zhang et al., 2009; Pakull et al., 2009)。
3.2 FsLinkageMap2.0与JoinMap4.0, MapMaker 3.0的比较软件FsLinkageMap2.0和JoinMap4.0都可以对杨树F1全同胞作图群体的全部遗传信息进行分析,在作图分析参数一致的情况下,二者构建的遗传框架图的图距大小差异显著,利用JoinMap4.0获得的框架图总图距达不到利用FsLinkageMap2.0获得的框架图总图距的60%;比较中发现122个相同标记,双方图谱上各有部分标记没有出现在对应的连锁群上,而是组成了三联体或二联体,122个相同标记都出现在对应连锁群上,但部分对应连锁群上标记的排列顺序差异较大; 这应与2软件使用的统计分析方法有关,2软件都考虑到了位点间的连锁相信息,因此大部分标记都出现在对应连锁群上; JoinMap是利用最小二乘法估计一列有序位点的相邻位点间的距离,先确定2个连锁关系最紧密的位点作为开始,每次只有1个信息含量最丰富的位点被添加分析,只有在拟合度测试中通过的才被接受,每一步都进行最优化洗牌(Van Ooijen et al., 2001; Van Ooijen,2006); FsLinkageMap软件对于每个连锁群,采用启发式搜索法进行多位点排序,对于最优的排序,利用隐马尔科夫链模型进行多位点连锁分析,得到相邻位点间的重组率,再根据作图函数将位点间的重组率转换成遗传距离(施季森等,2006); 由于分析方法的不同导致2个框架图的图距差异显著,且出现少数标记由于间距过大,连锁分析时没有连锁到相应的连锁群上。在与杨树一致性图谱进行比对发现,利用JoinMap4.0构建的响叶杨×银白杨遗传图谱的同源连锁群上的标记排列顺序更加符合一致性图谱的标记排列顺序,这可能与一致性图谱也是由JoinMap软件构建的有关。
FsLinkageMap和JoinMap软件可以直接分析F1作图群体中ab×ab(1:2:1)和ab×cd(1:1:1:1)类型的遗传标记,不需要采用“拟测交”的分析方法,这是MapMaker软件不能做到的,MapMaker采用的“拟测交”作图策略不能分析ab×ab类型的标记,ab×cd类型标记只利用了部分作为1:1分离位点的信息; 因此利用FsLinkageMap2.0所做的母本响叶杨图谱(即ad图谱)包含了更多的遗传信息,其中分离比为1:2:1的4个SSR标记(G_3058, G_3359, O_108和O_218)分别被连锁到ad-5, ad-6和ad-7连锁群上。另外ad图谱比利用MapMaker3.0构建的AD图谱增加了3个SSR标记(G_1686, G_1809和G_3751);同时FsLinkageMap2.0把3个SSR标记(O_84, O_48和P_2606)连锁到ad图谱上,而MapMaker3.0没有将其连锁到AD图谱上,相反,MapMaker3.0将3个SSR标记(O_23, O_156B和P_2818)连锁到AD图谱上,而FsLinkageMap2.0没有将其连锁到ad图谱上; 还分别有5和6个标记在ad和AD 2个图谱中出现倒位现象和位于不同的对应连锁群上。总的来看,共有10个分离比为1:1:1:1的SSR标记在2图谱中出现倒位现象或位于不同的对应连锁群或没有连锁到相应的图谱上(图 3)。出现这些现象,除了作图策略的原因外,也可能是由于抽样误差造成的或与排序的算法不同有关。
童春发等(2004)利用FsLinkageMap软件对1:1分离位点遗传信息进行连锁分析,构建的杉木遗传连锁图谱比原有的图谱(胡小华,2002)分别增加了26和28(共54)个AFLP标记,图距分别增加了583.6和963.2 cM,而本研究利用全部分离位点信息获得的图谱只比原图谱增加了5个标记,图距只增加了109 cM。这可能与构图使用的标记有关,大多数AFLP和SRAP标记都属于显性标记,而SSR属于共显性标记,本研究构图所使用的标记,72.25%是SSR共显性标记,27.75%是SRAP显性标记。利用显性和共显性标记混合作图时,相同性质的标记易于连锁在一起,这除了与标记的遗传特性有关外,还可能与基因型统计误差的倾向性密切相关; 同时连锁分析条件改变后,显性标记和共显性标记的连锁关系可能会随之发生改变。
利用FsLinkageMap2.0构建的ad图谱包含22个SRAP显性标记,比利用MapMaker3.0构建的AD图谱少2个,2个图谱分别有50%以上的显性标记没有出现在对应连锁群中,而没有出现在对应连锁群中的共显性标记SSR却不到10%,因此共显性标记比显性标记在2图谱中有更准确的对应关系; 而且本研究中有5个位于不同的对应连锁群上的SSR标记(G_20, G_2020, G _40, O_30B和G_114),利用其引物序列在杨树全基因组网站(http://genome.jgi-psf.org/Poptr1_1/Poptr1_1.home.html)上进行比对(blast),发现这5个标记在ad图谱的相应连锁群(ad-1, ad-3, ad-13, ad-15和ad-24)上的位置与毛果杨(P. trichocarpa)的同源连锁群上的区域相近(王源秀,2008),这说明利用FsLinkageMap构建的响叶杨的遗传图谱上共显性标记的排列顺序与标记在杨树全基因组中的相对位置更符合。
综上所述,不同的作图软件构建的遗传图谱具有不同的优缺点; 在基因组图谱比较研究中,利用相同作图软件构建的近缘物种的遗传图谱, 其比较研究所发现的标记同线性(synteny)或共线性(collinearity), 应具有更高的可靠性,可为后续的深入研究奠定基础。另外,鉴于林木高度杂合的遗传特点,可以通过选择适合的统计分析方法,充分利用林木作图群体的遗传信息,获得相对较高密度的遗传图谱,以提高遗传图谱在QTL作图或图位克隆或标记辅助选择以及基因组图谱精细比较研究中的利用价值。
胡小华. 2002. 杉木AFLP遗传图谱的构建及生长和形态相关性状QTL定位研究. 南京林业大学硕士学位论文. http://d.wanfangdata.com.cn/Thesis/Y439177
|
施季森, 童春发. 2006. 林木遗传图谱构建和QTL定位统计分析[M]. 北京: 科学出版社.
|
童春发, 施季森. 2004. 利用杉木的F1代群体构建遗传连锁图谱[J]. 遗传学报, 31(10): 1149-1156. |
王源秀. 2008. 响叶杨×银白杨遗传图谱构建及杨属图谱比较研究. 南京林业大学博士学位论文. http://d.wanfangdata.com.cn/Thesis/Y1296047
|
张博, 杜生明, 黄敏仁. 2003. 林木遗传图谱研究现状及发展趋势[J]. 中国生物工程杂志, 23(4): 14-18. |
Cervera M, Sewell M, Faivre-Rampant P, et al. 2004. Genome mapping in Populus//Kumar S, Fladung M. Molecular Genetics and Breeding of Forest Trees. New York: Food Product Press, 387-410. https://biblio.ugent.be/publication/303082
|
Gaudet M, Jorge V, Paolucci I, et al. 2008. Genetic linkage maps of Populus nigra L. including AFLPs SSRs SNPs and sex trait[J]. Tree Genetics & Genomes, 4: 25-36. |
Grattapaglia D, Sederoff R. 1994. Genetic linkage maps of Eucalyptus grandis and Eucalyptus urophylla using a pseudo-testcross: mapping strategy and RAPD markers[J]. Genetics, 137(4): 1121-1137. |
Hulbert S, Ilott T, Legg E, et al. 1988. Genetic analysis of the fungus, Bremia lactucae, using restriction fragment length polymorphisms[J]. Genetics, 120: 947-958. |
Jansson S, Douglas C. 2007. Populus: A model system for plant biology[J]. Annu Rev Plant Biol, 58: 435-458. DOI:10.1146/annurev.arplant.58.032806.103956 |
Kuang H, Bongarten B, Richardson T, et al. 1999. Genetic analysis of inbreeding depression in plus tree 850. 55 of Pinus radiata D. Don. Ⅰ. Genetic map with distorted markers[J]. Theor Appl Genet, 98: 697-703. |
Lander E S, Green P, Abrahamson J, et al. 1987. MAPMAKER: an interactive computer package for constructing primary genetic linkage maps of experimental and natural populations[J]. Genomics, 1: 174-181. DOI:10.1016/0888-7543(87)90010-3 |
Lange K, Boehnke M. 1982. How many polymorphic genes will it take to span the human genome?[J]. Am J HumGenet, 24: 842-845. |
Pakull B, Groppe K, Meyer M, et al. 2009. Genetic linkage mapping in aspen (Populus tremula L. and Populus tremuloides Michx.)[J]. Tree Genetics & Genomes, 5: 505-515. |
Tuskan G A, Jansson S, Bohlmann J, et al. 2006. The genome of black cottonwood, Populus trichocarpa (Tort. & Gray)[J]. Science, 313: 1596-1604. DOI:10.1126/science.1128691 |
Van Ooijen J W, Voorrips R E.2001. JoinMap 3.0, Software for the calculation of genetic linkage maps.Plant Research International, Wageningen, the Netherlands. https://www.researchgate.net/publication/241336396_Software_for_the_calculation_of_genetic_linkage_maps
|
Van Ooijen J W. 2006. Joinmap 4.0, Software for the calculation of genetic linkage maps in experimental populations. Kyazma BV, Wageningen, Netherlands. http://www.scienceopen.com/document?vid=b159fadd-708e-4c3e-9c91-bd395f88b847
|
Voorrips R E. 2002. MapChart: Software for the Graphical Presentation of Linkage Maps and QTLs[J]. J Hered, 93: 77-78. DOI:10.1093/jhered/93.1.77 |
Wang Yuanxiu, Sun Xiaoyan, Tan Biyue, et al. 2010. A genetic linkage map of Populus adenopoda Maxim. × P. alba L. hybrid based on SSR and SRAP markers[J]. Euphytica, 173: 193-205. |
Woolbright S A, DiFazio S P, Yin T, et al. 2008. A dense linkage map of hybrid cottonwood (Populus fremontii × P. angustifolia) contributes to long-term ecological research and comparison mapping in a model forest tree[J]. Heredity, 100: 59-70. DOI:10.1038/sj.hdy.6801063 |
Yin Tongming, Huang Minren, Wang Mingxiu, et al. 2001. Preliminary interspecific genetic maps of the Populus genome constructed from RAPD markers[J]. Genome, 4: 602-609. |
Yin Tongming, Difazio S P, Gunter L E, et al. 2004. Large-scale heterospecific segregation distortion in Populus revealed by a dense genetic map[J]. Theor Appl Genet, 109: 451-463. |
Zhang Bo, Tong Chunfa, Yin Tongming, et al. 2009. Detection of quantitative trait loci influencing growth trajectories of adventitious roots in Populus using functional mapping[J]. Tree Genet Genom, 5: 539-552. DOI:10.1007/s11295-009-0207-z |
Zhang Deqiang, Zhang Zhiyi, Yang Kai, et al. 2004. Genetic mapping in (Populus tomentosa × Populus bolleana) and Ptomentosa Carr. using AFLP markers[J]. Theor Appl Genet, 108: 657-662. DOI:10.1007/s00122-003-1478-7 |
 2010, Vol. 46
2010, Vol. 46