文章信息
- 蒋瑶, 陈其兵
- Jiang Yao, Chen Qibing
- 植物CBF1转录因子的生物信息学分析
- Bioinformatic Analysis of CBF1 Transcription Factors from the Plants
- 林业科学, 2010, 46(6): 43-50.
- Scientia Silvae Sinicae, 2010, 46(6): 43-50.
-
文章历史
- 收稿日期:2009-05-18
-
作者相关文章
CBF (CRT-binding factor)转录激活因子是一类受低温特异诱导的反式作用因子,可以调控多个抗寒基因启动子区上的顺式作用元件[CRT/DRE(C-repeat /dehydration responsive element)],促进启动子中含有这一调控元件的多个冷诱导和脱水诱导基因的表达,从而激活植物体内的多种耐逆机制。CBF能感受上游传递的低温信号并将信号向下游传递,对于增强植物适应和抵御低温的能力具有重要作用(曹云飞等,2007)。ICE1 (inducer of CBF expression 1)需要经过冷诱导的构型变化(可能是磷酸化和去磷酸化作用)调节活性,从而激活下游基因的表达(Chinnusamy et al., 2003)。近来有研究表明,1个R2R3类型的拟南芥(Arabidopsis thalina,At)AtMYB15转录因子与ICE1相互作用,在冷胁迫条件下,负调控CBF基因的表达(Agarwal et al., 2005)。Chinnusamy等(2007)认为在冷驯化状态下,通过泛素相关的蛋白水解对CBF1的诱导物ICE1负调控,而小泛素相关修饰物E3酶通过催化正调控ICE1。林茂等(2008)综述了植物CBF转录因子及其在基因工程中的应用,认为CBF转录因子的典型特征是:N端有核定位信号(nuclear location signal,NLS)区,C端有酸性激活区,中间有与DNA结合的AP2(APETALA2)结构域。钟克亚等(2006)认为CBF1转录因子作用途径为CBF转录因子→CRT/DRE基序→COR基因(cold-regulated gene)表达→植物抗寒性增加,并研究其抗寒的作用机制,为提高植物的抗寒性、培育抗寒作物品种提供新方向。张丽丽等(2008)认为COR基因编码的多肽具有两亲性α-螺旋结构域,在低温诱导下使得磷脂双分子层向内弯曲,来抵抗低温引起细胞脱水造成的膜损伤。水稻(Oryza sativa,Os)OsDREB1B(dehydration responsive element binding factor)在烟草(Nicotiana tabacum,Nt)中过表达能够提高转基因植株的抗氧化性和抗冻性,组成型OsDREB1B可诱导病原体相关的转基因植株表达(Gutha et al., 2008)。
已经从拟南芥(Novillo et al., 2007)、玉米(Zea may,Zm)(Nguyen et al., 2009)、油菜(Brassica napus,Bn)(庄静等,2008)、小麦(Triticum aestivum,Ta) (Badawi et al., 2008)和水稻(Ito et al., 2006)等多种植物中分离得到相关的CBF1,其中Shahrokhabadi等(2008)利用CODEHOP软件设计相关引物,已鉴定出低温胁迫相关的小麦同源DREB基因,发现DRE调控基因表达,能增强小麦的抗冻性。Zhu等(2007)综述了在植物低温驯化下,冷应答基因调控、代谢及RNA剪切和二级结构等方面的相互作用。但目前对不同植物CBF1的生物信息学分析未见报道,因此,利用一系列的生物信息学软件分析了不同植物CBF1蛋白的性质,并对其蛋白作对比,以期为进一步开展CBF1的基因功能和蛋白质的特性研究提供一定的理论基础,也为后期深入研究基因的结构与生物学功能提供参考,为分析其在不同信号途径相互作用中的功能和调控机制提供有力证据。
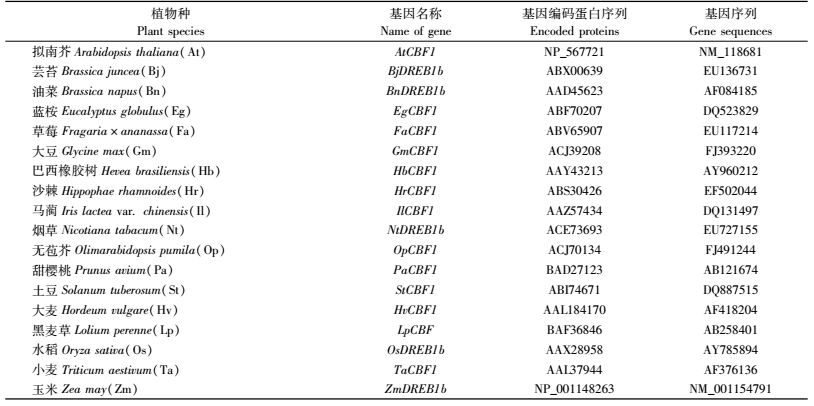
1 材料与方法 1.1 材料通过美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI:http://www.ncbi.nlm.nih.gov/)的核酸和蛋白质数据库寻找相应的数据资料,对已经克隆得到的CBF基因完整的cDNA(complemental DNA)序列和编码氨基酸序列进行数据搜索。收集得到的CBF基因完整cDNA和编码氨基酸序列各18条,其中双子叶植物13条,单子叶植物5条,单子叶植物全部为禾本科植物(表 1)。
|
|
采用ExPaSy的Prosite数据库(http://www.expasy.org/prosite/)、NCBI的Conserved Domains数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)(Marchler-Bauer et al., 2004)和Mega3.1 (Kumar et al., 2004)软件,对CBF1基因编码氨基酸序列进行氨基酸保守位点、可变位点、简约信息位点和单一多态位点分析。利用欧洲生物信息学研究所(European Bioinformatics Institute,EBI)在线的ClustalW2 (Larkin et al., 2007)程序,进行氨基酸序列的多重比对,寻找氨基酸保守区,并用Boxshade对其保守区域进行标记。
1.2.2 CBF1基因系统发生树的构建采用Mega3.1软件内置的ClustalW程序进行蛋白序列多重比对(比对采用程序默认的IUB记分矩阵),采用Mega3.1对ClustalW产生的多重比对结果构建系统发生树。参数设置:采取最大简约法(maximum parsimony,MP)构建系统树,采用随机逐步比较的方式搜索最佳系统树,对生成的系统发生树进行Bootstrap校正,最终生成系统发生树。
1.2.3 CBF1蛋白一级结构理化特性分析利用ExPaSy提供的Prot-Param软件和ProtScale软件分别进行氨基酸残基组成、蛋白质分子质量和亲/疏水性的在线分析;最后利用antheprot软件(http://antheprot-pbil.ibcp.fr/)(Barabasi et al., 2003)分析理论等电点。
1.2.4 CBF1蛋白二级结构分析利用ExPaSy的工具中的SOPMA软件在线预测分析α-螺旋(α-helix,H)、β-转角(β-turn,T)、无规则卷曲(random coil,C)以及延伸链(extended strand,E),然后利用网站ExPaSy工具中的PSORT软件预测蛋白亚细胞定位。
1.2.5 CBF1蛋白三级结构的分析三级结构的预测是蛋白质结构预测的重点,主要有以下几种方法:同源模建、折叠识别和从头预测法(Biocarta,2003)。利用ExPaSy工具中的Swiss-Model程序进行同源模建(Grill et al., 1998;Upinder et al., 1999)。它运用结构仿真模拟(ProModⅡ程序)和能量最小化分析(GROMOS96程序)构建目标序列的结构。
2 结果与分析 2.1 CBF1编码氨基酸的序列比对利用EBI在线的ClustalW2进行氨基酸序列多重比对,结果如图 1所示。根据比对结果发现,这18个CBF1转录因子在蛋白质氨基酸序列N端保守性较强,具有典型的CBF1转录因子结构特征,包含有PKK/RPAGRXKFXETRHP和FADSAW特征序列,可能起激活作用,而且推测这2个序列可能参与CBF蛋白的转运,前一个特征序列位于蛋白质N端,富含精氨酸(arginine,R)和赖氨酸(lysine,K)的碱性结构,而后一个特征序列位于AP2核心结合域,属于RAYD基元的保守结构域。因此,这2个特征序列可能参与对CRT/DRE调控元件的识别,调控下游基因表达。

|
图 1 18种植物CBF1类转录因子氨基酸序列比对的结果 Figure 1 Multiple alignment result of CBF1 transcription factors in 18 different plants 相同和保守的氨基酸残基用黑色和灰色分别表示,AP2和NLS用上划线表示,AP2区中YRG和RAYD 2个保守区分别用方框表示。C端保守域用最后3个方框表示。 Identical and conserved amino acid residues are indicated in black and gray, respectively; the AP2 domain and NLS are also indicated. Two boxes show the location of the YRG and RAYD elements in the AP2 domain, respectively.The last three boxes are the conserved domain in C terminal. |
保守结构域是蛋白质中能够折叠成特定三维结构的一段区域,其结构亚单位称为基序(motif),组成这些基序的氨基酸区段行使特异的功能,并蕴含各自的遗传进化信息。CBF1转录因子N端含有高度保守的AP2结构域,从而构成了植物特异性转录因子大家族。在ExPaSy的ScanProsite软件获得不同植物保守结构域,但其AP2核心结构域的位置存在差异,以AtCBF1保守结构域(图 2)为例。

|
图 2 AtCBF1保守域 Figure 2 AtCBF1 conserved domain in Arabidopsis thaliana |
利用Mega3.1软件构建CBF1转录因子的系统发生树(图 3)。由图 3可知,系统发生树中的CBF1转录因子主要聚为2类:第1类包括TaCBF1,LpCBF,OsCBF1,ZmDREB1b和HvCBF1单子叶植物的CBF1转录因子,用○标记;第2类AtCBF1, NtDREB1b, StCBF1和GmCBF1等双子叶植物的CBF1转录因子,用△标记。由此可以看出,这与植物中单子叶、双子叶植物的分类大体一致,表明基因和植物的进化过程密切相关。
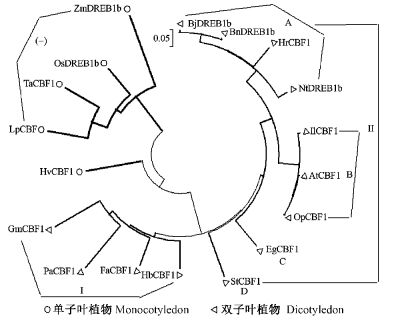
|
图 3 18种植物CBF1类转录因子的系统发生树 Figure 3 Phylogenetic tree of CBF1 transcription factors in 18 different plants |
第2类又可分为2大类:第Ⅰ大类由GmCBF1,FaCBF1,PaCBF1和HbCBF1组成;第Ⅱ大类分为4个小类:BnDREB1b,BjDREB1b,HrCBF1和NtDREB1b为A类,AtCBF1,OpCBF1和IlCBF1为B类,EgCBF1为C类,StCBF1为D类。
CBF1在植物中的进化优先于单、双子叶的分化时间,这种分类上的冲突是在植物中普遍存在的。有研究表明,基因的倍增和重组、水平的基因转移等都是这种差异存在的原因。
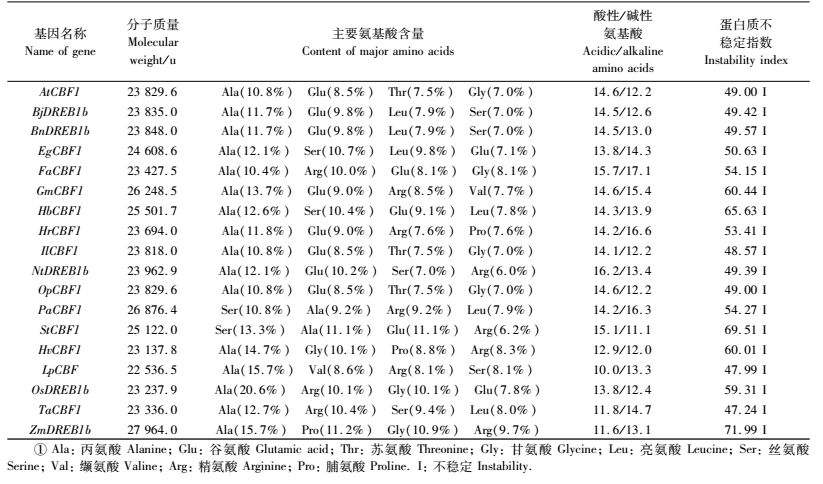
2.3 CBF1基因编码氨基酸组成特性通过Clustal W2在线软件对CBF1基因编码氨基酸序列多重比较,然后利用Mega3.1软件得到18个CBF1基因编码氨基酸的保守位点参数,其中保守位点47个,可变位点235个,简约信息位点154个,单一多态位点78个。所含氨基酸的种类主要是脂肪族类,中性氨基酸占大部分,而可变区与保守区的中性氨基酸的变化不具有方向性。
用Protparam预测CBF1编码蛋白的理化性质,图 2为不同植物CBF1氨基酸的组成及理化特性的比较,由此可以看出,不同植物CBF1的氨基酸分子质量较为一致,其氨基酸序列中主要氨基酸均含有非极性氨基酸——丙氨酸(alanine,Ala),且含量较高。酸性/碱性氨基酸比例也有所不同,双子叶植物中Eg,Fa和Gm酸性/碱性氨基酸比例小于1,说明这3种植物的碱性氨基酸占主导地位,其余双子叶植物则是酸性氨基酸占主导地位,其比值大于1;单子叶植物唯有大麦碱性氨基酸比例较高,比值小于1,其他单子叶植物氨基酸则是酸性氨基酸较高。通过Protparam软件分析不同植物蛋白,发现其蛋白均属于不稳定类蛋白。
2.4 CBF1蛋白等电点的预测及对比利用分析软件anthepro,由氨基酸序列预测不同植物CBF1蛋白的等电点。大部分植物CBF1蛋白都属于酸性等电点,其中酸性最强的是St,最弱的是Zm。唯有Lp是中性等电点,而Ta和Fa是碱性等电点。
2.5 CBF1蛋白的亲水/疏水性分析氨基酸序列决定蛋白质的功能,组成蛋白质的20种氨基酸各具特殊的侧链,当它们按照不同的序列关系组合时,就可形成多种多样的空间结构,使其具有不同生物学活性。疏水性是20种氨基酸都固有的特性,是影响蛋白质构象的重要因素,因此蛋白质疏水性/亲水性的预测和分析,对预测蛋白质生物学功能具有重要意义。用ProtScale的Kyte and Doolittle算法对不同植物CBF1氨基酸序列的疏水性/亲水性进行预测(图 4为AtCBF1的预测结果),并对特征基序采用anthepro软件表示亲水/疏水性(图 5为AtCBF1特征基序的预测结果),正值越大表示越疏水,负值越大表示越亲水,介于+0.5~-0.5之间的主要为两性氨基酸。结果表明,不同植物CBF1蛋白的氨基酸序列中亲水性、疏水性氨基酸都均匀分布在整个肽链中,亲水性氨基酸多于疏水性氨基酸,并且没有明显的疏水区域,因此可认为这些蛋白是亲水性蛋白。结果提示,大约在At中67-68区域具有很强的亲水性,91区域具有较强的疏水性(图 4),并预测其他植物可知,不同植物的CBF蛋白亲水/疏水性有一定差异,是由于植物行使生理功能存在差异。
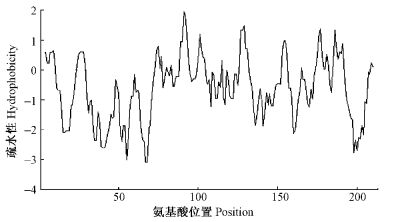
|
图 4 AtCBF1全序列亲水/疏水性图谱 Figure 4 Predicted hydrophilicity/hydrophobicity graph for CBF1 in Arabidopsis thaliana |
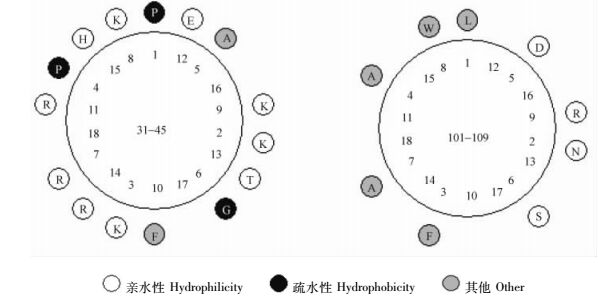
|
图 5 AtCBF1特征基序的亲水/疏水性图谱 Figure 5 Predicted hydrophilicity/hydrophobicity graph for specific motifs of CBF1 in Arabidopsis thaliana |
|
|
蛋白质的二级结构是多肽链中相邻多个氨基酸残基形成的局部肽链空间结构,由H(α-helix)、T(β-turn)、E(extended strand)和无规则C(random coil)等几种二级结构元件组成,具有特定的生物学活性。当H>45%,E < 5%时为all-alpha型;当H < 5%,E>45%时为all-beta型;当H>30%,E>20%时为alpha-beta型。用SOPMA预测不同植物CBF1氨基酸序列的二级结构,其中显示α-螺旋和无规则卷曲是不同植物CBF1最大量的结构元件,而β-转角和延伸链则散布于整个蛋白质中。统计表明,AtCBF1由23.00%的α-螺旋、15.49%的延伸链、2.35%的β-转角和59.15%的无规则卷曲组成,而OsDREB1b由35.78%的α-螺旋、12.84%的延伸链、2.75%的β-转角和48.62%的无规则卷曲组成。由此可以看出,不同植物间的二级结构在空间上发生了一定的变异。
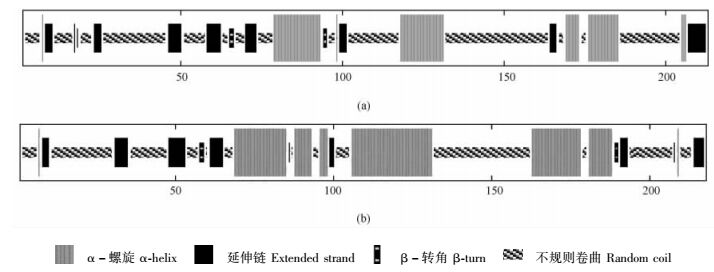
|
图 6 AtCBF1(a)和OsDREB1b(b)二级结构的预测结果 Figure 6 Predicted secondary structure for CBF1 in Arabidopsis thaliana (a) and OsDREB1b in Oryza sativa (b) |
用PSORT分析不同植物CBF1编码蛋白的亚细胞定位。由图 7可知,AtCBF1蛋白最终定位于细胞质的可能性为45.0%,定位于微体(过氧化物酶体)的可能性为35.3%,从而推断该蛋白可能定位于细胞质内。通过PSORT预测可知,大多数植物的蛋白亚细胞定位于细胞核中,少数植物的蛋白亚细胞定位于细胞质中,唯有少数蛋白定位于微体中,如OsDREB1b蛋白。由此可以看出,不同植物蛋白的亚细胞定位存在差异,是由于植物合成部位及行使生理功能所需的反应环境存在差异,也是由于其蛋白的结构、理化性质和功能不同所造成的差异,为此,探讨这些植物蛋白的亚细胞定位具有重大的意义。

|
图 7 拟南芥CBF1的亚细胞定位结果 Figure 7 Subcellular localization prediction of CBF1 protein in Arabidopsis thaliana |
蛋白质的三维结构,除了共价的肽键和二硫键,还靠大量的极其复杂的弱次级键共同作用,实际上折叠过程是一个正确途径和错误途径相互竞争的过程。利用Swiss-model建模服务器进行CBF1结构的三维建模,预测不同植物CBF1蛋白的三维结构。图 8为AtCBF1的三级结构预测结果,可看到α-螺旋、β-转角和β-折叠片,由此可看到其蛋白折叠空间结构构象的变化。

|
图 8 拟南芥CBF1的三维结构 Figure 8 Tertiary structure of CBF1 in Arabidopsis thaliana |
利用生物信息学方法指导试验工作的设计,可避免试验的盲目性和不必要的重复。在基因研究的相关文献报道中,利用生物信息学进行前期或后续分析越来越普遍。
CBF1转录因子是属于AP2转录因子家族,是植物中广泛存在的一类转录因子,这类转录因子主要参与植物的细胞周期、生长发育以及生物和非生物胁迫相关基因的表达调控,它们都含有非常保守的DNA结合区。CBF转录因子能够识别CRT/DRE元件,调控多个与同类性状有关的基因表达,在提高植物对环境胁迫耐性的分子育种中,改良或增强一个关键转录因子的调控能力,可使植物的耐逆性得到较为综合的改良(张晗等, 2006)。本研究对18种不同植物CBF1的生物信息学分析预测可得,CBF1转录因子主要由DNA结合域、NLS、转录调控结构域3个功能域组成,在植物体内起着激活或抑制基因表达的功能,符合CBF1的基本结构特征。CBF1结构域有YRG和RAYD 2个保守的结构域。在C端存在1个可能起激活作用的酸性结构域,在N端有1个可能作为NLS的富含R和K的碱性结构域,提高植物对非生物胁迫的抵抗能力。
唐宁等(2008)利用一系列生物信息学软件分析盐芥(Thellungiella salsuginea,Th)ThHKT1的一级和二级结构,并与拟南芥ThHKT1基因序列进行比对,从而对该基因的理化性质、结构特征和功能等进行预测和分析,研究结果表明,其基因编码蛋白分子式为:C2631H4064N636O707S21,属于稳定蛋白;二级结构主要是以无规则卷曲为主。本研究结果表明,18种不同植物CBF1蛋白为不稳定蛋白,其中α-螺旋和无规则卷曲成为最大量的结构元件,而β-转角和延伸链则散布于整个蛋白质中。
结构决定功能,仅通过对基因和氨基酸序列的了解并不能充分反映蛋白质的功能,故蛋白质空间结构的研究越来越得到重视(Sergiy et al., 2004)。本研究分析得知,AtCBF1三维空间结构的构象具有一定的特点,其中该区有1个α-螺旋、3个反向平行的β-折叠和2个β-转角。CBF1的氨基酸主要是脂肪族类,都含有非极性氨基酸Ala,其蛋白等电点多数为酸性。因此,应用生物信息学方法对已知序列进行比对、分析,推断和预测其结构和功能,为选择合适的试验方法提供理论参考,为这些基因的功能研究提供更全面的理论依据。
曹云飞, 张海娜, 肖凯. 2007. CBF转录因子介导的植物低温信号转导研究进展[J]. 棉花学报, 19(4): 304-311. |
林茂, 闫海霞, 眭顺照, 等. 2008. 植物CBF转录因子及其在基因工程中的应用[J]. 广西农业科学, 39(1): 21-25. |
唐宁, 杨平. 2008. 盐芥ThHKT1基因的生物信息学分析[J]. 药物生物技术, 15(6): 449-452. |
张晗, 信月芝, 郭惠明, 等. 2006. CBF转录因子及其在植物抗冷反应中的作用[J]. 核农学报, 20(5): 406-409. |
张丽丽, 李景富, 王傲雪. 2008. 转录激活因子CBF基因在植物抗冷分子机制中的作用[J]. 园艺学报, 35(5): 765-771. |
钟克亚, 叶妙水, 胡新文, 等. 2006. 转录因子CBF在植物抗寒中的重要作用[J]. 遗传, 28(2): 249-256. |
庄静, 周熙荣, 孙超才, 等. 2008. 油菜沪油15中AP2/ERF-B3亚族转录因子的克隆和生物信息学分析[J]. 分子细胞生物学报, 41(3): 192-206. |
Agarwal M, Hao Y, Kapoor A, et al. 2005. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF gene and in acquired freezing tolerance[J]. Current Opinion in Plant Biology, 10: 290-295. |
Badawi D, Reddy Y V, Agharbbaoui Z, et al. 2008. Structure and functional analysis of wheat ICE (inducer of CBF expression) genes[J]. Plant Cell Physiology, 49(8): 1237-1249. DOI:10.1093/pcp/pcn100 |
Barabasi A L, Bonabeau E. 2003. Scale-free network[J]. Sci Am, 5: 50. |
Biocarta. 2003. Charting pathways of life[EB/OL]. [2003-12-12]. http://www.biocarta.com/gene/CellSignaling.asp.
|
Chinnusamy V, Ohta M, Kanrar S, et al. 2003. ICE1: a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis[J]. Genes Dev, 17(8): 1043-1054. DOI:10.1101/gad.1077503 |
Chinnusamy V, Zhu J H, Zhu J K. 2007. Cold stress regulation of gene experession in plants[J]. Trends in Plant Science, 12(10): 1360-1385. |
Grill E, Himmelbach A. 1998. ABA signal transduction[J]. Current Opinion in Plant Biology, 412(1): 418. |
Gutha L R, Reddy A R. 2008. Rice DREB1B promoter shows distinct stress-specific responses, and the overexpression of cDNA in tobacco confers improved abiotic and biotic stress tolerance[J]. Plant Molecular Biology, 68: 533-555. DOI:10.1007/s11103-008-9391-8 |
Ito Y, Katsura K, Maruyama K, et al. 2006. Functional analysis of rice DREB1/CBF-type transcription factors involved in cold-responsive gene expression in transgenic rice[J]. Plant and Cell Physiology, 47(1): 141-153. DOI:10.1093/pcp/pci230 |
Kumar S, Tamura K, Nei M. 2004. MEGA3: Integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Briefings in Bioinformatics, 5: 150-163. DOI:10.1093/bib/5.2.150 |
Larkin M A, Blackshields G, Brown N P, et al. 2007. Clustal W and Clustal X version 2[J]. Bioinformatics, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
Marchler-Bauer A, Bryant S H. 2004. CD-Search: protein domain annotations on the fly. Nucleic Acids Res, 32(Web Server Issue): 327-331.
|
Nguyen H T, Leipner J, Stamp P, et al. 2009. Low temperature stress in maize (Zea mays L.) induces genes involved in photosynthesis and signal transduction as studied by suppression subtractive hybridization[J]. Plant Physiology and Biochemistry, 47(1): 116-122. |
Novillo F, Medina J, Salinas J. 2007. Arabidopsis CBF1 and CBF3 have a different function than CBF2 in cold acclimation and define different gene classes in the CBF regulon[J]. PNAS, 104(52): 21002-21007. DOI:10.1073/pnas.0705639105 |
Sergiy O G, Michail Y L, Oxana V G. 2004. To be folded or to be unfolded?[J]. Protein Sci, 13: 2871-2877. |
Shahrokhabadi K, Afshari R T, Alizade H, et al. 2008. Identification of DREB homologous genes in bread wheat via CODEHOP PCR primer design[J]. Pakistan Journal of Biological Science, 11(16): 1979-1986. DOI:10.3923/pjbs.2008.1979.1986 |
Upinder S, Bballa U S, Iyengar R. 1999. Emergent properties of networks of biological signaling pathways[J]. Science, 283: 381-387. DOI:10.1126/science.283.5400.381 |
Zhu J H, Dong C H, Zhu J K. 2007. Interplay between cold-responsive gene regulation, metabolism and RNA processing during plant cold acclimation[J]. Current Opinion in Plant Biology, 10: 290-295. DOI:10.1016/j.pbi.2007.04.010 |
 2010, Vol. 46
2010, Vol. 46