文章信息
- 唐征, 杨凯, 冯永庆, 曹庆芹, 沈元月, 秦岭
- Tang Zheng, Yang Kai, Feng Yongqing, Cao Qingqin, Shen Yuanyue, Qin Ling
- 板栗脂质转运蛋白基因的克隆及表达
- Cloning and Expression of Chestnut Lipid Transfer Protein
- 林业科学, 2010, 46(4): 43-48.
- Scientia Silvae Sinicae, 2010, 46(4): 43-48.
-
文章历史
- 收稿日期:2009-03-09
-
作者相关文章
2. 北京农学院生物技术系 北京 102206
2. Department of Biological Technology, Beijing University of Agriculture Beijing 102206
板栗(Castanea mollissima)是重要的生态与经济林树种,全国26个省市均有分布,中国板栗资源遗传多样性丰富,是抗性强的基础。本课题组近年来在北京市密云县板栗产区1株正常生长的栗树上发现雄花序显著短缩的自然芽变(冯永庆,2005),田间观察发现芽变株对栗树病害的抗性有所增强。AFLP分析表明短雄花序和对照之间的变异与遗传物质的改变相关(徐月等,2006),利用抑制性消减杂交技术构建板栗短雄花序对正常雄花序的差减文库,通过对文库克隆测序获得脂质转运蛋白基因片段(朱晓琴,2008)。植物脂质转运蛋白(lipid transfer proteins,LTPs)是一类定位于细胞外的碱性小分子蛋白,于高等植物中广泛存在。在菠菜(Spinacia oleracea var. samos)叶片中首次纯化得到脂质转运蛋白,其能在体外生物膜之间转运多种脂类物质(Kader et al., 1984),随后大量研究表明脂质转运蛋白对病原真菌和细菌具有抑制作用(葛晓春等,2002;Jayaraj et al., 2007),并且参与多种生物学过程(Suh et al., 2005;Cameron et al., 2006;Park et al., 2003)以及对各种环境胁迫具有适应性应答(Agnieszka et al., 2008;Wu et al., 2005)。
本研究运用RACE技术对板栗脂质转运蛋白基因进行克隆,荧光定量分析正常和芽变雄花序的LTP相对表达量,并通过原核表达初步探索其抑菌功能,为深入研究板栗芽变株的抗性强于正常株的分子机制奠定基础。
1 材料与方法 1.1 材料板栗雄花序采自北京密云县栗产区。
1.2 方法 1.2.1 cDNA全长的获得及序列分析板栗短雄花序与正常雄花序构建差减文库,根据随机挑取克隆测序得到的序列信息设计RACE引物。5′ RACE引物为: 5′-GGGCAGGTACATGAAGAAATACAAC-3′;3′ RACE引物为: 5′-GGAGTCTTCTTGTCGTT GTAG-3′。
利用CTAB法(Ying et al., 2002)提取板栗短雄花序总RNA,1%琼脂糖电泳检测,使用SMARTTM cDNA Library Construction Kit(Clontech公司)进行反转录,以合成的cDNA第1链为模板进行PCR扩增,电泳检测后回收,与pMD18-T载体连接,转化至大肠杆菌(Escherichia coli) DH5α,通过酶切或者菌体PCR检测筛选阳性克隆并测序。
测序获得的3′序列和5′序列用DNAStar软件进行拼接。通过BLAST在线进行序列相似性比对。亲/疏水性分析采用Expasy中ProtScale在线进行,蛋白质结构的综合预测采用CPHmodels 2.0 Server在线分析。采用MEGA 4.0进行蛋白序列比对和进化树构建,进行进化分析。
1.2.2 芽变与正常雄花序mRNA转录水平上荧光定量分析从芽变枝与正常枝同时采集雄花序,分别提取RNA,使用M-MLV First cDNA Synthesis Kit(购自上海生工)合成cDNA第1链,以actin(ev253704)为内参基因,采用SYBR Green I荧光染料法检测脂质转运蛋白基因的相对表达量。为避免DNA污染设计了LTP基因的跨内含子引物。LTP引物Ls: 5′-TGGCAAGTGTGGAGTCAA-3′;La: 5′-AGAGGAGCCCCATGTATG-3′,产物长度为162 bp。内参引物As: 5′-TTGACTATGAGCAGGAACTT-3′;Aa: 5′-TTGTAGGTGGTCTCGTGAAT-3′,产物长度为178 bp。
反应体系为: 10 μL Real Master Mix(TaKaRa),2 μL cDNA,0.5 μL primer(10 μmol·L-1),7 μL H2O;反应程序为: 95 ℃ 3 min,1个循环;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s(荧光采集),40个循环。PCR扩增结束后立即进行熔解曲线分析,以验证扩增特异性,其程序为:从60 ℃开始每升高0.5 ℃保持30 s,到95 ℃为止,连续升高71次。为了确保结果的可重复性,每个反应设置3次重复。
1.2.3 LTP的原核表达根据编码LTP成熟肽N端和C端的氨基酸顺序引入适当酶切位点设计表达引物,由上海生工公司合成。NL: (NcoⅠ)5′-GTACCATGGCTATATCATGTGGCCAAGTCC-3′;CL: (EcoRⅠ)5′-TCAGAATTCATCACTTCACGTTTTT GCAGTT-3′。
以反转录cDNA第1链为模板,NL与CL为引物,用Pfu酶进行PCR扩增,产物经NcoⅠ与EcoRⅠ酶切后与同样2种酶进行酶切后的表达载体pET32a(+)进行定向连接,构建原核表达质粒pET-LTP,转入DH5α,菌体PCR检测后送测序验证插入的序列及其读码框准确无误。
将质粒pET-LTP与载体pET32a(+)分别转入表达菌株Rosetta-gamiTM2(DE3),37 ℃生长约12 h后从平板上挑取单菌落接种于1 mL LB液体培养基(含Cam, Str, Tet和Amp 4种抗生素)中,30 ℃培养过夜,取1 mL过夜培养物接入100 mL四抗LB培养基中,30 ℃振荡培养至A600为0.6~1.0之间,加IPTG至终浓度为1 mmol·L-1,30 ℃诱导5 h后收集菌体。
根据葛晓春等(2002)方法进行渗透处理。使用Ni2+-chelating Sepharose fast flow柱进行融合蛋白的纯化,SDS-PAGE电泳鉴定采用4%浓缩胶和15%分离胶,100 V电泳至溴酚蓝离胶下沿约1 cm,拆胶,考马斯亮蓝R250染色。
从显症板栗种仁分离出的镰刀菌(Fusarium sp.)菌株接种至PDA培养基上,25 ℃培养至长满整皿,加5 mL无菌水于皿中,用推棒刮2圈,吸出菌悬液,在不同浓度LTP蛋白液中加入相同体积的菌悬液振荡摇匀,滴加在载玻片上保湿培养(25 ℃、黑暗),每浓度3个重复。每隔4 h在显微镜下观察分生孢子萌发数(以芽管长度超过孢子直径一半作为萌发标准),以pET32a(+)表达的担体蛋白为对照。当对照萌发率达到85%之后,检查所有处理的萌发率,按下式计算抑制孢子萌发率:抑制孢子萌发率(%)=(对照孢子萌发率-处理孢子萌发率)×100 /对照孢子萌发率。
2 结果与分析 2.1 板栗LTP结构特征通过RACE扩增和序列拼接后获得长度为660 bp的cDNA序列(图 1),经开放阅读框预测发现其包含1个完整的ORF(open reading frame),其中5′侧翼区长84 bp,3′侧翼区长为189 bp,编码118个氨基酸。进行BLASTX分析,发现其与陆地棉(Gossypium hirsutum)、草莓(Fragaria × ananassa)脂质转运蛋白基因氨基酸序列相似性为63%。经和陆地棉、草莓、拟南芥(Arabidopsis thaliana)等植物脂质转运蛋白多序列比对(图 2)显示此蛋白具有8个保守的半胱氨酸残基(C...C...CC...C XC...C...C),这是脂质转运蛋白的结构特征。

|
图 1 板栗LTP基因序列及由此推导的氨基酸序列 Figure 1 Nucleotide and deduced amino acid sequences of LTP from C. mollissima 下划线部分的26个氨基酸为信号肽。The signal peptide is underlined. |

|
图 2 板栗LTP与其他物种LTP蛋白序列比较 Figure 2 Comparison of the primary structure of LTP sequence from different plants 1.板栗Castanea mollissima; 2.拟南芥Arabidopsis thaliana; 3.草莓Fragaria × ananassa; 4.桃Prunus persica; 5.陆地棉Gossypium hirsutum. |
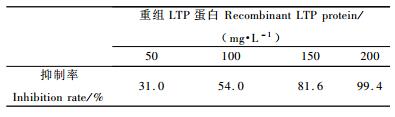
选用Expasy中ProtScale在线进行疏水性分析(窗口大小选择9,以Hphob. / Kyte & Doolittle标度计算信号),从图 3中可见该蛋白具有4个高疏水性区,其中1-26位的区段具有信号肽特征,与SignaIP 3.0 Server信号肽预测结果完全吻合。去除信号肽后板栗成熟脂质转运蛋白为92个氨基酸残基,分子质量约为9 ku,预测pI值为8.99,并且与脂质转运蛋白家族Ⅰ中的半保守氨基酸残基Tyr16, Tyr79(Lullien-Pellerin et al., 1999), Pro10, Asp43和Arg44(Yeats et al., 2008)保持一致。
|
图 3 板栗脂质转运蛋白(LTP)亲/疏水性分布曲线 Figure 3 The distribution curve of hydropathy of chestnut lipid transfer protein (LTP) |
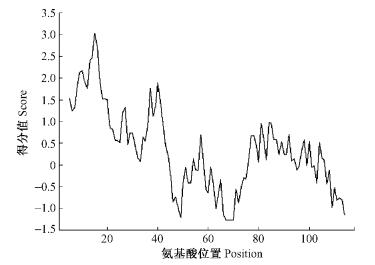
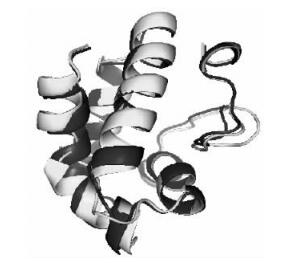
在PredictProtein网站上进行板栗成熟脂质转运蛋白二级结构分析(图 4)可见板栗脂质转运蛋白具有4个α螺旋(α-helice)和1个无规则卷曲的C末端。利用CPHmodels 2.0 Server分析板栗与拟南芥成熟脂质转运蛋白的空间结构(图 5)可见两者之间三极结构类似,并且与桃(Prunus persica)、绿豆(Vigna radiata)等通过核磁共振和X射线结晶法了解到的脂质转运蛋白三维结构也类似(Pasquato et al., 2006;Lin et al., 2005)。通过以上序列分析,可以断定克隆得到的cDNA片段为典型的植物脂质转运蛋白,GenBank登录号为FJ490676(基因)和ACL01093(蛋白)。

|
图 4 板栗脂质转运蛋白二级结构预测 Figure 4 Second structure of chestnut lipid transfer protein H. α-螺旋α-helix; C.无规则卷曲Random coil. |
|
图 5 板栗和拟南芥LTP空间结构比较 Figure 5 Overlay of structure of chestnut LTP with those of Arabidopsis thaliana 黑色为板栗,灰色为拟南芥。 The black is chestnut, the grey is Arabidopsis thaliana. |
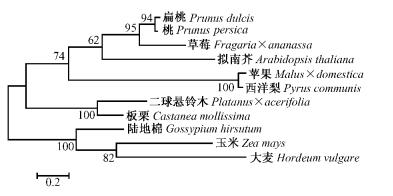
板栗与其他10种植物LTP蛋白质序列用MEGA 4.0软件采用邻接法(NJ)和Bootstrap的1 000次重复检测方法进行亲缘关系分析,结果表明在遗传进化上主要分为2类,板栗与二球悬铃木(Platanus × acerifolia)首先聚在一起,再与扁桃(Prunus dulcis)、桃、拟南芥、草莓、苹果(Malus × domestica)、西洋梨(Pyrus communis)形成一大类,而作物类玉米(Zea mays)、大麦(Hordeum vulgare)和陆地棉亲缘关系较近,单独为一类(图 6)。
|
图 6 板栗LTP与其他物种LTP的聚类分析 Figure 6 Phylogenetic analysis of chestnut LTP and other species LTP |
2008年5月10日为板栗雄花序发育初期,5月23日芽变雄花序顶部变黄。荧光定量分析发现板栗LTP在正常雄花序与短雄花序的mRNA表达水平上具有差异性(图 7),短雄花序mRNA表达量约是正常花序的3.2倍和5.0倍(设正常雄花序mRNA表达量为1)。
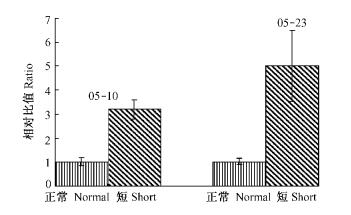
|
图 7 短雄花序与正常雄花序LTP表达量比较 Figure 7 Comparison of LTP expression levels of short male inflorescence with normal male inflorescence |
以反转录的板栗雄花序cDNA第1链为模板,NL与CL为引物,用Pfu酶进行PCR扩增,得到约300 bp的单一条带(图 8),与预期大小相符。PCR产物与pET32a(+)分别经NcoⅠ与EcoRⅠ双酶切后连接,构建原核表达质粒pET-LTP,转入DH5α,NcoⅠ与EcoRⅠ双酶切检测重组子,有1条300 bp左右的条带(图 9),测序结果表明插入的序列及其读码框准确无误。
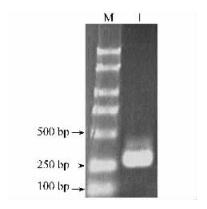
|
图 8 RT-PCR产物电泳 Figure 8 Analysis of RT-PCR product by agarose gel electrophoresis |
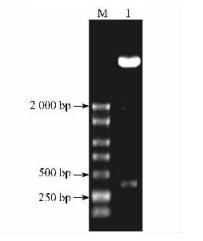
|
图 9 重组质粒酶切鉴定 Figure 9 Identification of recombinant plasmid pET-LTP by digestion |
融合蛋白诱导表达及纯化处理后的电泳结果见图 10,诱导5 h蛋白表达量明显大于诱导2 h的量,融合蛋白约为30 ku,无插入的pET32a(+)载体表达1个大小约为21 ku的担体蛋白,与预期相符。经过Ni2+-chelating Sepharose fast flow柱纯化后得到电泳纯的融合蛋白。
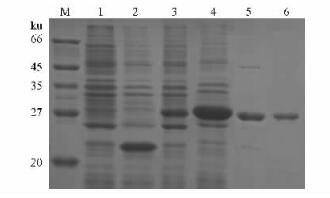
|
图 10 LTP重组蛋白的表达与纯化 Figure 10 Expression and purification of recombinant LTP protein M.蛋白marker.1.pET-LTP未诱导;2. pET32(+)诱导5 h;3. pET-LTP诱导2 h;4. pET-LTP诱导5 h;5.渗透处理的上清;6.纯化的融合蛋白。 M. Protein marker. 1.Untreated pET-LTP; 2.pET32a(+) induced for 5 h; 3.pET-LTP induced for 2 h; 4.pET-LTP induced for 5 h; 5.Supernatant after osmotic shock; 6.Purified thio-LTP fusion protein. |
将纯化的重组LTP和担体蛋白加入到板栗镰刀菌属病原真菌孢子悬液中暗培养,显微镜下观察孢子萌发情况。结果表明:浓度达到100 mg·L-1时,LTP抑制孢子萌发率为54%(表 1),可以明显延缓病原真菌孢子的萌发;浓度越大,影响越显著,浓度达到200 mg·L-1时,孢子几无明显的萌发表现。
|
|
植物脂质转运蛋白家族在种子植物中普遍存在,而在球蒴藓(Physcomitrella patens)及江南卷柏(Selaginella moellendorffii)的基因组草图中搜索不到该家族序列,可能在藓类和蕨类这些低等植物中不存在脂质转运蛋白。从多个物种的研究表明:纯化的植物LTPs蛋白在体外可抑制真菌和细菌的生长, 病原菌能诱导LTPs的表达,LTPs过表达的转基因植物对病原菌的抗性增强,可见脂质转运蛋白基因是植物体内的一种重要抗逆基因。目前植物LTPs抗菌的机制还不甚明确,但至少部分原因是增加病原菌而不是宿主细胞的通透性(Regente et al., 2005)。植物LTPs既能抑制革兰氏阴性菌,又能抑制革兰氏阳性菌和多种真菌的生长,但依个体来源不同而有一定的差异。绿豆种子LTPs对真菌茄腐镰刀菌(Fusarium solani)、根腐病菌(Fusarium oxysporum)等有较强的抗性,但对革兰氏阴性菌鼠伤寒沙门菌(Salmonella typhimurium)没有作用(Wang et al., 2004)。在向日葵(Helianthus annuus)种子中发现LTPs抑制茄腐镰刀菌孢子萌发的浓度为40 mg·L-1,50%抑制浓度为6.5 mg·L-1(Gonorazky et al., 2005;Regente et al., 2000)。板栗是原产我国的特色果树,我国板栗品质优良, 具有较高的抗逆性和抗病虫害能力,板栗脂质转运蛋白基因的克隆与抑菌功能的研究,对进一步揭示其结构和生物学功能以及在抗逆基因工程中的应用有一定的参考意义。
本课题组发现的板栗芽变枝雄花序变短,通过研究表明短雄花序发育中上部自然衰落的异常死亡为细胞程序性死亡(张婧等,2007),并且短花序营养状况等农艺性状优于对照,荧光定量分析短雄花序脂质转运蛋白mRNA表达量较正常花序高。大戟属植物Euphorbia lagascae苗中发现有类似现象,其胚乳在程序性死亡时LTP的表达量增加,Eklund等(2003)推测LTP与胚乳脂质再利用或者是充当蛋白酶抑制剂来保护生长的子叶有关。关于LTP作为蛋白酶抑制剂的说法目前还存在矛盾: Jose-Estanyol等(2004)提出一些蛋白酶抑制因子只是分子结构上与LTPs类似,均为具有8个位置保守半胱氨酸残基的小分子蛋白质,但它并不是属于真正的LTP家族;而在银杏(Ginkgo biloba)种子中分离的蛋白酶抑制因子具有脂质转运活性,但不具有抗真菌和细菌活性;Yoriko等(2008)认为该基因为脂质转运蛋白家族Ⅰ成员。板栗LTP在短雄花序中表达量更高,它是否有蛋白酶抑制作用以及在体内的具体功能还有待于进一步的研究。
冯永庆. 2005. 板栗短雄花序芽变形态、生化及细胞学研究. 乌鲁木齐: 新疆农业大学硕士学位论文.
|
葛晓春, 陈继超, 林奕, 等. 2002. 水稻非特异性脂质转移蛋白的原核表达、纯化及抑菌功能[J]. 生物化学与生物物理学报, 34(1): 83-87. |
徐月, 曹庆芹, 冯永庆, 等. 2006. 短雄花序板栗芽变的AFLP分析[J]. 园艺学报, 33(6): 1321-1324. |
张婧, 董清华, 杨凯, 等. 2007. 板栗短雄花序异常死亡的超微结构观察[J]. 园艺学报, 34(3): 605-608. |
朱晓琴. 2008. 应用抑制性消减杂交分离板栗短雄花序芽变相关基因. 北京农学院硕士学位论文. http://www.cqvip.com/QK/94996A/200903/30371242.html
|
Agnieszka K M, Pascal R, Tadeusz R, et al. 2008. The organ-dependent abundance of a Solanum lipid transfer protein is up-regulated upon osmotic constraints and associated with cold acclimation ability[J]. J Exp Bot, 59: 2191-2203. DOI:10.1093/jxb/ern088 |
Cameron K D, Teece M A, Smart L B. 2006. Increased accumulation of cuticular wax and expression of lipid transfer protein in response to periodic drying events in leaves of tree tobacco[J]. Plant Physiol, 140: 176-183. |
Eklund D M, Edqvist J. 2003. Localization of nonspecific lipid transfer proteins correlate with programmed cell death responses during endosperm degradation in Euphorbia lagascae seedlings[J]. Plant Physiol, 132: 1249-1259. DOI:10.1104/pp.103.020875 |
Gonorazky A G, Regente M C, de la Canal L. 2005. Stress induction and antimicrobial properties of a lipid transfer protein in germinating sunflower seeds[J]. J Plant Physiol, 162(6): 618-624. DOI:10.1016/j.jplph.2004.10.006 |
Jayaraj J, Punja Z K. 2007. Combined expression of chitinase and lipid transfer protein genes in transgenic carrot plants enhances resistance to foliar fungal pathogens[J]. Plant Cell Rep, 26: 1539-1546. DOI:10.1007/s00299-007-0368-x |
Jose-Estanyol M, Gomis-Ruth F X, Puigdomenech P. 2004. The eight cysteine motif, a versatile structure in plant proteins[J]. Plant Physiol Biochem, 42: 355-365. DOI:10.1016/j.plaphy.2004.03.009 |
Kader J C, Julienne M, Vergnolle C. 1984. Purification and characterization of a spinach-leaf protein capable of transferring phospholipids from liposomes to mitochondria or chloroplasts[J]. Eur J Biochem, 139: 411-416. DOI:10.1111/ejb.1984.139.issue-2 |
Lin K F, Liu Y N, Hsu S T, et al. 2005. Characterization and structural analyses of nonspecific lipid transfer protein 1 from mung bean[J]. Biochemistry, 44: 5703-5712. DOI:10.1021/bi047608v |
Lullien-Pellerin V, Devaux C, Ihorai T, et al. 1999. Production in Escherichia coli and site-directed mutagenesis of a 9-kDa nonspecific lipid transfer protein from wheat[J]. Eur J Biochem, 260: 861-868. DOI:10.1046/j.1432-1327.1999.00229.x |
Park S Y, Lord E M. 2003. Expression studies of SCA in lily and confirmation of its role in pollen tube adhesion[J]. Plant Mol Biol, 51: 183-189. DOI:10.1023/A:1021139502947 |
Pasquato N, Berni R, Folli C, et al. 2006. Crystal structure of peach Pru p 3, the prototypic member of the family of plant non-specific lipid transfer protein pan-allergens[J]. J Mol Biol, 356: 684-694. DOI:10.1016/j.jmb.2005.11.063 |
Regente M C, de la Canal L. 2000. Purification, characterization and antifungal properties of a lipid-transfer protein from sunflower (Helianthus annuus) seeds[J]. Physiol Plant, 110(2): 158-163. DOI:10.1034/j.1399-3054.2000.110203.x |
Regente M C, Giudici A M, Villalain J, et al. 2005. The cytotoxic properties of a plant lipid transfer protein involve membrane permeabilization of target cells[J]. Lett Appl Microbiol, 40: 183-189. DOI:10.1111/lam.2005.40.issue-3 |
Suh M C, Samuels A L, Jetter R, et al. 2005. Cuticular lipid composition, surface structure, and gene expression in Arabidopsis stem epidermis[J]. Plant Physiol, 139: 1649-1665. DOI:10.1104/pp.105.070805 |
Wang S Y, Wu J H, Ng T B, et al. 2004. A non-specific lipid transfer protein with antifungal and antibacterial activities from the mung bean[J]. Peptides, 25(8): 1235-1242. DOI:10.1016/j.peptides.2004.06.004 |
Wu Y R, Wang Q Y, Ma Y M, et al. 2005. Isolation and expression analysis of salt up-regulated ESTs in upland rice using PCR-based subtractive suppression hybridization method[J]. Plant Sci, 168(3): 847-853. DOI:10.1016/j.plantsci.2004.10.020 |
Yeats H T, Rose K C J. 2008. The biochemistry and biology of extracellular plant lipid-transfer proteins (LTPs)[J]. Protein Sci, 17: 191-198. DOI:10.1110/ps.073300108 |
Ying Z, Tao Y. 2002. RNA isolation from highly viscous samples rich in polyphenols and polysaccharides[J]. Plant Molecular Biology Reporter, 20(12): 417a-417e. |
Yoriko S, Ken-ichi H, Takuya M, et al. 2008. Proteinase inhibitor from Ginkgo seeds is a member of the plant nonspecific lipid transfer protein gene family[J]. Plant Physiol, 146: 1909-1919. DOI:10.1104/pp.107.111500 |
Yubero-Serrano E M, Moyano E, Medina-Escobar N, et al. 2003. Identification of a strawberry gene encoding a non-specific lipid transfer protein that responds to ABA, wounding and cold stress[J]. Exp Bot, 54(389): 1865-1877. DOI:10.1093/jxb/erg211 |
 2010, Vol. 46
2010, Vol. 46